利用Ⅲ型聚酮合酶CsyB起始单元的底物多样性体内合成csypyrone类化合物
2018-07-30潘丽霞朱婧王青艳申乃坤李亿杨登峰
潘丽霞,朱婧,王青艳,申乃坤,李亿,杨登峰,2
利用Ⅲ型聚酮合酶CsyB起始单元的底物多样性体内合成csypyrone类化合物
潘丽霞1,朱婧1,王青艳1,申乃坤1,李亿1,杨登峰1,2
1 广西科学院 非粮生物质酶解国家重点实验室 国家非粮生物质能源工程技术研究中心 广西生物质产业化工程院 广西生物炼制重点实验室,广西 南宁 530007 2 广西科学院 广西北部湾海洋研究中心 广西海洋天然产物与组合合成生物化学重点实验室,广西 南宁 530007

金城. 2018酶工程专刊序言. 生物工程学报, 2018, 34(7): 1021−1023.Jin C. Preface for special issue on enzyme engineering (2018). Chin J Biotech, 2018, 34(7): 1021−1023.
作为新型Ⅲ型聚酮合酶,米曲霉来源的CsyB能够依次接受一个起始单元为短链脂肪酰辅酶A、一个延伸单元为丙二酰辅酶A和另一个延伸单元为乙酰乙酰辅酶A的3个底物形成短链的csypyrone B1-3。基于CsyB的晶体结构分析,显示它的活性中心存在一个长约16 Å的能够接受脂肪酰辅酶A结合通道,这个通道很可能能够接受多种底物。为了检测该酶的底物多样性,将基因导入到存在长链脂肪酰辅酶A前体的大肠杆菌中表达。高效液相结果显示,相比对照菌株,重组菌株产生了一系列长链的csypyrone衍生物。利用紫外可见光特征吸收值和高分辨液相色谱-质谱联用仪对这些新产物作了初步分析。对3个具有羟基的csypyrone产物的结构进行了核磁共振一维谱和二维谱的详细鉴定,确定了其羟基的位置。上述结果显示,CsyB具有广泛的底物特异性,不但可以接受多种长链饱和或不饱和脂肪酰辅酶A,还可以接受具有羟基修饰的长链脂肪酰辅酶A作为底物。
Ⅲ型聚酮合酶,CsyB,底物多样性,体内合成,csypyrone
聚酮类化合物是一类化学结构复杂多样、生物活性良好的天然产物。它广泛存在于自然界中,具有抗菌、抗肿瘤、抗寄生虫和免疫抑制等药理活性[1-5]。常见的聚酮类药物有洛伐他汀、阿霉素、红霉素、四环素、两性霉素等。尽管它们在结构上是多样的,但在其生物合成途径中,其核心结构均由聚酮合酶 (Polyketide synthase, PKS) 催化合成。根据聚酮合酶的结构及其他性质,其被分为模块型的Ⅰ型PKS、迭代型的Ⅱ型PKS和查尔酮型的Ⅲ型PKS三大类[6-9]。
由于独特的结构、作用机制及催化活性,Ⅲ型PKS的研究备受瞩目。Ⅲ型PKS和其他两种PKS迥然不同,它在不需要酰基载体蛋白 (Acyl carrier protein, ACP) 的情况下直接催化丙二酰辅酶A间的缩合,主要负责单环或双环芳香类聚酮化合物的生物合成,如白藜芦醇、花青素、查耳酮、二苯乙烯、苯甲酮、吖啶酮、间苯三酚、吡喃酮和苯并-γ-吡喃酮等含有黄酮类骨架结构的化合物。这些化合物使植物拥有抗氧化、抗诱变、抗紫外辐射、抗病害侵扰等抵御外界胁迫的能力。目前研究表明,它们与人类的健康也有密切的关系,具有抗炎、抗癌、保护心脑血管系统等多种药理作用[10-12]。Ⅲ型聚酮合酶的查尔酮合酶超家族 (CHS; EC 2.3.1.74) 是一类蛋白质分子量为40–45 kDa的酶,一般是形成简单的同源二聚体,以具有芳香基团或脂肪链基团的辅酶A为底物,连续地催化和丙二酰辅酶A的脱羧缩合反应,形成一系列具有生物活性的次级代谢产物[13-15]。尽管Ⅲ型聚酮合酶主要分布在植物中,但在微生物中也发现了它的存在,不仅有细菌还有像真菌这样的真核微生物[16-18]。
来源于米曲霉的CsyB是一个独特的Ⅲ型聚酮合酶,它存在于csypyrone B1-3的生物合成途径中。CsyB是第一个Ⅲ型PKS的双功能酶,不仅具有常规Ⅲ型PKS催化脱羧克莱森缩合反应的能力,而且可以催化非脱羧的克莱森缩合反应,一步法缩合两个二酮辅酶A单元[19-23]。具体来说,CsyB能够接受脂肪酰辅酶A、丙二酰辅酶A和乙酰乙酰辅酶A为底物。首先,将脂肪酰辅酶A与丙二酰辅酶A脱羧缩合产生β-酮酰基二缩酮 (β-ketoacyl-diketide),然后,再与乙酰乙酰辅酶A完成非脱羧的缩合反应,产生3-乙酰基-4-羟基-6-烷基-α-吡喃酮 (3-acetyl-4- hydroxy-6-alkyl-α-pyrone),一个假定的csypyrone B的前体 (图1A,1)。此外,CsyB可以催化两个乙酰乙酰辅酶A分子或两分子β-酮酰基二缩酮中间体的缩合反应,分别产生脱氢乙酸 (3-acetyl- 4-hydroxy-6-methyl-α-pyrone) 和3-乙酰基-4-羟基- 6-烷基-α-吡喃酮 (图1A,2)[23]。
为了更好地了解该酶的催化特性,我们对该酶的晶体结构进行了解析,得到了1.7 Å的晶体[24]。对晶体结构的详细分析显示,它是一个典型的硫解酶结构,如图1B所示,活性口袋由三部分构成:一是从活性中心一直延伸到酶表面的、狭长的接受起始单元脂肪酰辅酶A的结合通道;二是酶的催化中心,由三联催化中心 (Cys-His-Asn,CHN) 所构成,负责延伸单元与起始单元的碳碳键的形成;三是能够容纳乙酰基或者更长侧链的一个额外的底物接纳口袋。虽然在米曲霉中,基因表达后的产物是接受四碳或者五碳的酰基辅酶A所产生的,但CsyB存在一个长约16 Å的底物起始单元结合通道,显然从该通道的长度和大小上分析,可以容纳更大的底物,这一特点与另外两个接受长链底物的酶极为相似,即粗糙脉孢霉的2′-氧烷基雷琐酸合成酶 (2′-oxoalkylresorcylic acid synthase,ORAS) 和米曲霉的CsyA[25]。这两个酶的产物分别是五缩酮烷基间苯二酚酸(pentaketide alkylresorcylic acid)和二羟基苯甲酸(dihydroxybenzoic acid)。另外,CsyB与ORAS和米曲霉来源的CsyA分别具有37%和45%的氨基酸序列一致性。因此,有理由相信CsyB可以接受更多复杂长链的底物。为了进一步研究CsyB的底物多样性,我们将CsyB在大肠杆菌中进行异源表达,希望借助大肠杆菌这个具有长链脂肪酸前体的系统,对CsyB的底物耐受性进行更为全面的考察。
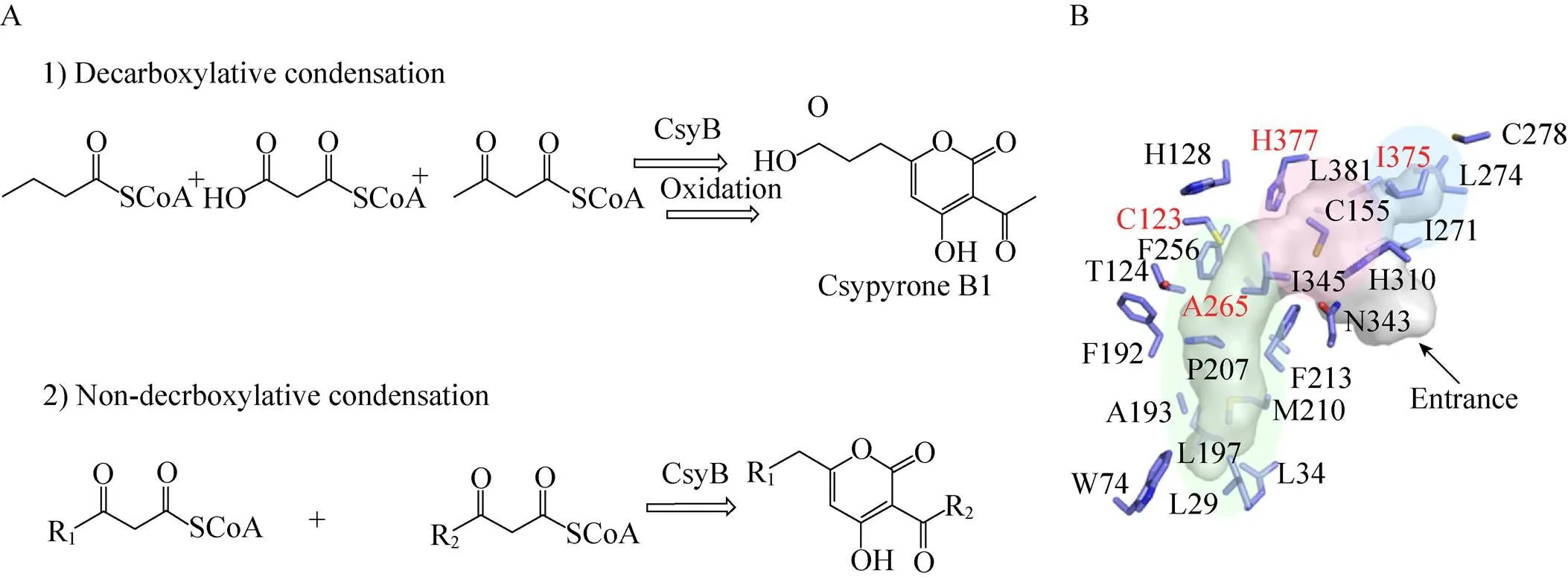
图1 CsyB催化的酶反应(A) 及其蛋白质晶体结构(B)
1 材料与方法
1.1 材料
表达质粒pET28a和大肠杆菌BL21 (DE3) 购自美国NOVAGEN公司。氯化钠、琼脂、卡那霉素、异丙基硫代半乳糖苷 (Isopropyl β-D- thiogalactoside, IPTG) 购自上海生物工程有限公司;乙腈、乙酸乙酯购自阿拉丁试剂 (上海) 有限公司;蛋白胨、酵母提取物购自英国OXOID公司;氘代氯仿、NMR核磁管购自美国NORELL公司;脱氢乙酸购自Sigma-Aldrich公司。培养基为Luria-Bertani (LB) 培养基。活化固体培养基:蛋白胨20 g,酵母提取物10 g,氯化钠10 g,琼脂10 g,去离子水定容至1 L。活化液体培养基:蛋白胨20 g,酵母提取物10 g,氯化钠10 g,去离子水定容至1 L。发酵培养基:蛋白胨10 g,酵母提取物5 g,氯化钠10 g,去离子水定容至1 L。
1.2 方法
1.2.1 表达质粒pET28a-CsyB的构建
以米曲霉全基因组为模板,使用带有限制性酶切位点Ⅰ的正向引物 (5′-CCTATCGAACCGTTACCGAC-3′) 和带有Ⅰ的酶切位点的反向引物 (5′-AATTTATGC GTGCAGATACGAGC-3′)为引物,对基因进行扩增。扩增程序如下:预热95 ℃ 2 min;95 ℃ 30 s,54 ℃ 30 s,72 ℃ 1 min,循环30次;最后,延伸72 ℃ 10 min。使用适量的快速限制性内切酶FastDigestⅠ和Ⅰ,对质粒和PCR产物进行双酶切 (37 ℃ 3 h),然后将酶切产物进行胶回收,在16 ℃下连接3 h,再将连接产物转化到大肠杆菌感受态细胞DH5a,挑取转化子进行菌体PCR,对阳性克隆进行酶切和测序验证,对测序正确的转化子,扩大培养,提取其质粒,转化至表达宿主BL21 (DE3) 中待用。
1.2.2 重组大肠杆菌发酵液的制备
将保藏在–20 ℃冰箱甘油中的含有表达载体pET28-CsyB的大肠杆菌BL21 (DE3) 活化,使用接种环将甘油保藏的菌种在含有卡那霉素抗性 (50 μg/mL) 的活化固体培养基平板上划线,置于37 ℃培养过夜。用灭菌牙签挑取生长良好的单菌落,转接到含有卡那霉素抗性 (50 μg/mL) 的液体活化液体培养基5 mL中,置于37 ℃培养7–8 h,然后,按照0.1%的接种率接种到含有卡那霉素抗性 (50 μg/mL) 的液体发酵培养基中,待培养基600值达到0.6时,将诱导剂IPTG的终浓度设为 0.5 mmol/L进行诱导,在25 ℃条件下培养2–3 d。
1.2.3 重组大肠杆菌发酵提取物的提取与粗分
发酵培养结束后,使用德国BECKMAN COULTER J-26 XP离心机,5 000 r/min离心20 min,将菌体与菌液分离,取上层菌液,使用分液漏斗,以乙酸乙酯为萃取溶剂,按体积比1∶1萃取3次,收集乙酸乙酯层,使用EYELA旋转蒸发仪将其减压浓缩获得粗浸膏,通过减压硅胶柱层析,以氯仿甲醇 (体积比分别为10%、30%、50%、70%、90%和100%) 为流动相进行梯度洗脱,获得6个组分。将主要含有新产物的3号和4号组分,使用Agilent 1260的半制备液相色谱仪进行分离,使用的是反相柱Agilent ZORBAX SB-C18 5 μm (9.3 mm× 250 mm),程序为:20%乙腈,洗脱5 min;20%–55%乙腈,洗脱5 min;55%–65%乙腈,洗脱5 min;65%–100%乙腈,洗脱8 min;最后,100%乙腈洗脱10 min,紫外检测器的吸收为310 nm。流动相分别为A:超纯水 (0.05%三氟乙酸 (Trifluoroacetic acid,TFA));B:乙腈 (0.05% TFA),以3 mL/min的流速进行分离,将收集的各个产物旋转蒸发干,放于4 ℃冰箱保存。
1.2.4 重组大肠杆菌发酵提取物组分的HPLC以及LC-HRMS的分析
对粗浸膏以及划段后的6个组分进行HPLC分析,使用分析液相色谱仪Ultimate 3000 Thermo Scientific Dionex公司进行分析,使用的是菲罗门反相柱Gemini®5 μm C18 (4.6 mm×250 mm),程序:20%乙腈,洗脱5 min;20%–55%乙腈,洗脱 5 min;55%–65%乙腈,洗脱5 min;65%–100%乙腈,洗脱8 min;最后,100%乙腈洗脱10 min。紫外检测器的吸收为310 nm,流动相分别为A:超纯水 (0.05% TFA);B:乙腈 (0.05% TFA),以1 mL/min的流速进行分析。
对分离的新产物进行LC-HRMS分析,使用Water UPLC-QTOF-MS/MS型号液质进行产物的分析,使用的是高效反相柱ACQUITY UPLC HSS T3 C18 (2.1 mm×100 mm,1.8 μm),程序为:20%乙腈,洗脱5 min;20%–55%乙腈,洗脱5 min;55%–65%乙腈,洗脱5 min;65%–100%乙腈,洗脱8 min;最后,100%乙腈洗脱10 min。紫外检测器的吸收为310 nm,流动相分别为A:超纯水 (0.5%乙酸);B:乙腈 (0.5%乙酸),以0.5 mL/min的流速进行分析。离子扫描模式为:ESI-正离子模式;扫描范围:100–1 200 Da。
1.2.5 重组大肠杆菌发酵提取物组分的NMR分析
将分离得到的新产物称重,溶解在200 μL的氘代氯仿中,用移液器小心地转移入核磁管中,在800兆美国Agilent 1001核磁共振仪中进行一维核磁共振谱氢谱和碳谱,以及二维核磁共振谱COSY谱、HMQC谱和HMBC谱的测定。
2 结果与分析
2.1 高效液相的结果分析
从高效液相结果可以看出,与对照菌株相比,在含有CsyB基因的重组大肠杆菌的发酵液中,明显出现了12个新峰,而且这些新峰的特征紫外吸收峰和之前报道的csypyrone B1-3化合物的紫外吸收峰完全一致,都出现在310 nm处,说明这些产生的新峰形成的都是csypyrone的结构类似物。因此,这些产物都是CsyB在大肠杆菌中表达后,利用大肠杆菌中自身的代谢物中间体为底物所形成的新产物。为了便于说明,将其命名为化合物1–12 (图2)。从这12个化合物的分布看,化合物1出峰时间较早,在11.6 min出峰,而其他11个产物均在大约30–40 min出峰。这说明化合物1的极性较大,而其他11个产物的极性相对较小,因此,化合物1可能是短链的产物,而其他产物可能是脂肪酸链较长的产物。从产量看,化合物1的产量明显高于其他11个产物,而且呈现出一种逐渐递减的趋势,原因可能是酶的底物特异性所引起的或者是反应前体在大肠杆菌中的产量差异所引起的。
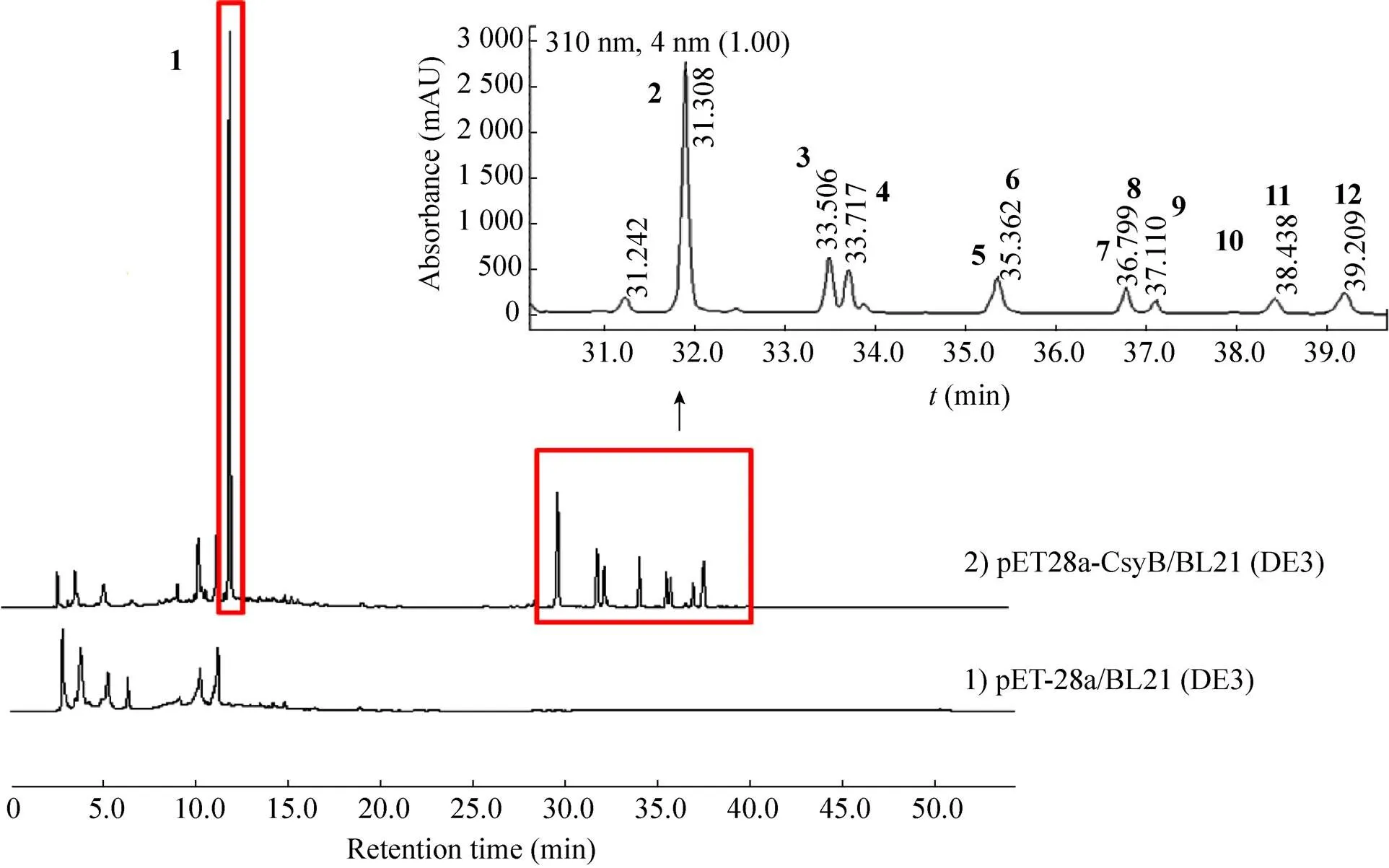
图2 含表达质粒pET28a-CsyB的重组大肠杆菌的高效液相图
2.2 LC-HRMS的结果分析
根据之前HPLC分析的结果,对重组大肠杆菌发酵提取物组分中的化合物1–12进行高分辨率分子量的测定。将分析的结果汇总在表1中,并根据csypyrone类化合物推测了这些化合物的分子式。化合物1的实测分子量是168.043 7,与预测分子量168.042 3符合,根据csypyrone的骨架可以判断,它是由两分子乙酰乙酰辅酶A自身缩合而成的最小的csypyrone化合物脱氢乙酸,通过与该已知化合物的标准品比较,1和脱氢乙酸完全一致。化合物2、7、9和11的实测分子量分别是280.165 3、308.196 4、336.231 2和364.263 2,与预测分子量280.167 5、308.198 8、336.230 1和364.261 4相符合,与脱氢乙酸相差分子量分别为112、140、168和196,因此,这4个化合物与脱氢乙酸分别相差4、5、6和7个碳二单元 (C2H4),说明这4个化合物是在脱氢乙酸的基础上,起始单元更加长的产物。根据分子量大小,可以推测化合物2、7、9和11是由起始单元分别为癸酰辅酶A、月桂酰辅酶A、肉蔻酰辅酶A以及软脂酰辅酶A为起始底物合成出来的,它们都是饱和长链的csypyrone化合物。
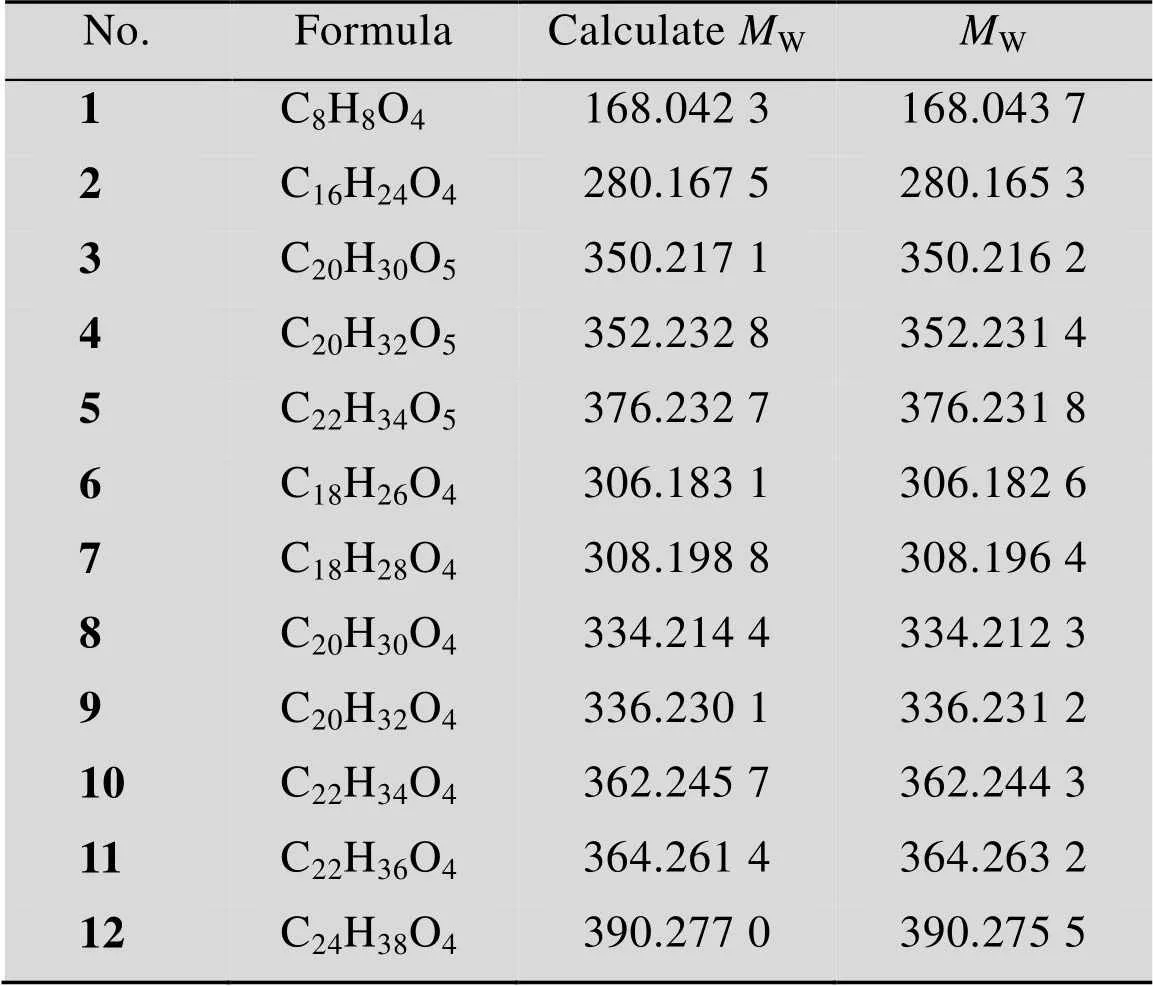
表1 Csypyrone衍生物的分子量
化合物6、8和10的实测分子量分别是306.182 6、334.212 3和362.244 3,与预测分子量306.183 1、334.214 4和362.245 7相符合,比7、9和11分子量少2,因此,6、8和10分别比7、9和11多出一个双键结构。另外,化合物12的实测分子量是390.275 5,与预测分子量390.277 0相符合,分子量比11多出28,也就是说比化合物11多出一个碳二单元。值得一提的是,化合物12的起始底物是硬脂酰辅酶A,碳链长度达到18,是目前CsyB可以接受的最长起始底物。因此,化合物6、8、10和12都是由含一个双键的癸酰辅酶A、月桂酰辅酶A、肉蔻酰辅酶A以及软脂酰辅酶A为起始底物合成出来的,它们都是含有一个双键的不饱和长链的csypyrone化合物。
化合物3、4和5的实测分子量分别为350.216 2、352.231 4和376.231 8,与预测分子量350.217 1、352.232 8和376.232 7相符合。化合物3、4和5分别比化合物8、9和10的分子量多出16,推测化合物3、4和5是化合物8、9和10的羟基化的产物。因此,化合物3和5是一类羟基化的饱和长链的csypyrone化合物,化合物4是一个羟基化的含有双键的不饱和长链的csypyrone化合物。
2.3 NMR的结果分析
我们选取产量相对较高、结构最为复杂的含有羟基的2个csypyrone化合物3和4进行了详细的结构鉴定,完成了氢谱、碳谱、COSY谱、HMBC谱以及HMQC谱的测定,结果见表2。化合物3和4推测的结构如图3所示,3和4的结构是非常接近的,在氢谱上,3出现13个信号,4出现9个信号,吡喃酮环上的碳2、3、4、6位的氢没有信号,碳5位分别在化学位移6.02和6.03处出现特征信号峰,在乙酰基上的7位碳没有信号,从而确定了csypyrone的基本骨架。在3的氢谱中,化学位移5.32和5.36的位置多出两个多重峰,这说明在3中存在一个双键,而在4的氢谱中,由于没有双键,在化学位移1.25左右处出现了严重的重叠,另外,3和4分别在化学位移4.06、4.04处出现了信号,是连接羟基的特征峰。在碳谱上,它们都出现21个信号,化合物3的碳14位和15位的化学位移分别是128.95和130.98,这些结果都说明3在碳14位和碳15位之间比4多出一个双键。另外,3和4的碳10位的化学位移分别出现在69.09和69.42,说明这个位置的碳原子可能连上了一个羟基基团。
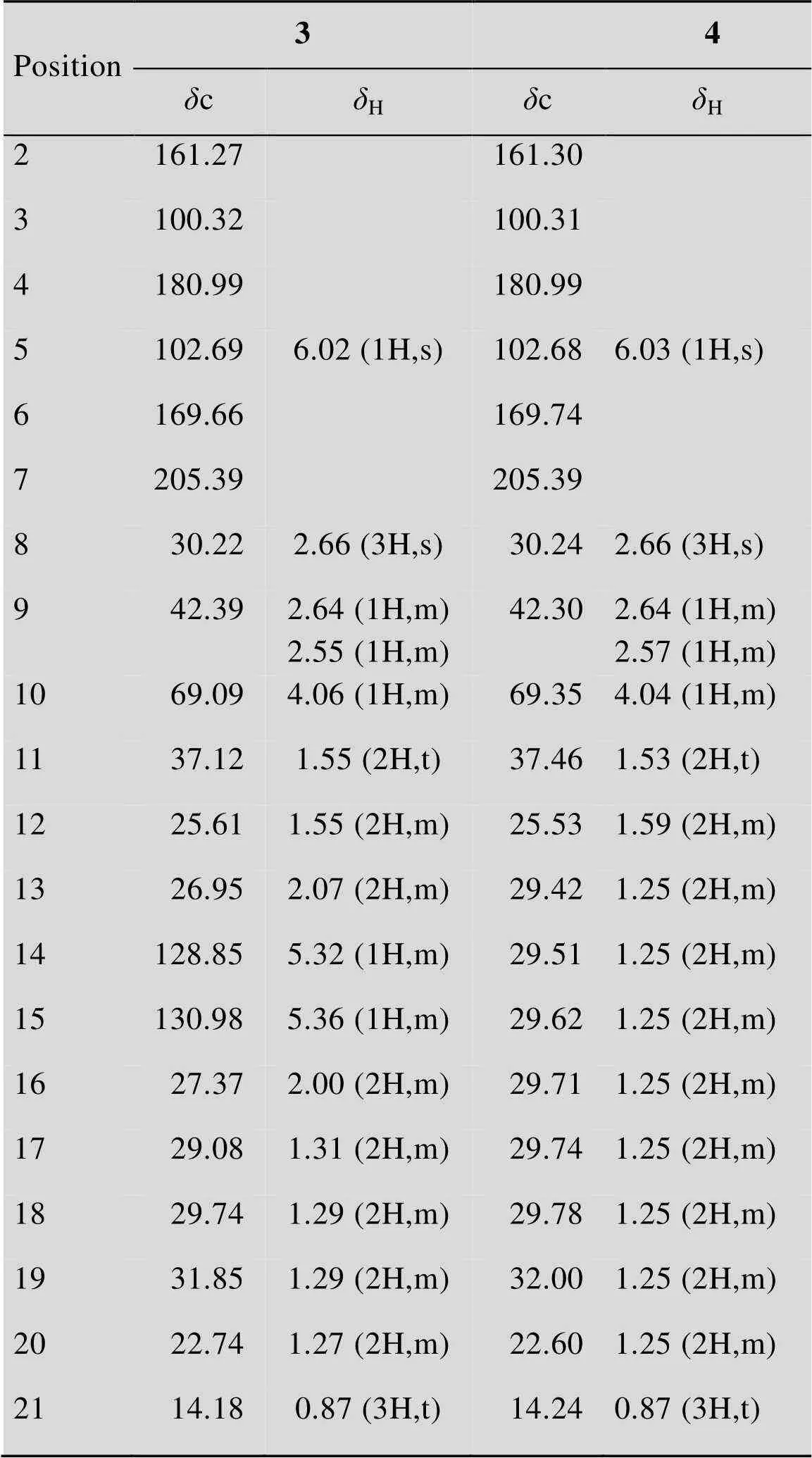
表2 3和4的氢谱和碳谱数据
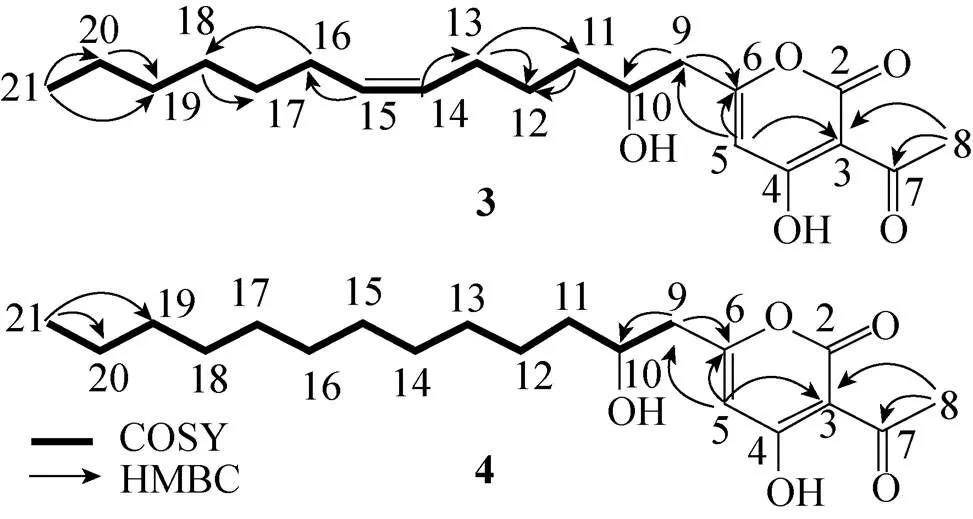
图3 3和4的1H-1H COSY谱和HMBC谱中的相关
在二维谱COSY谱中,各相邻近碳之间的氢发生相关,吡喃酮环上的数据和已经报道的csypyrone化合物完全相同。在长链部分,可以检测到明显的1H-1H化学相关 (图3),如9和10、11和12、12和13、13和14、15和16、16和18、18和19、19和20、20和21。HMBC的信号也较为清楚 (图3),可以看到相关发生在3和5、3和8、5和6、5和9、6和9、7和8、9和10、11和12、12和13、11和13、13和14、15和16、16和18、17和19、19和20、19和21、20和21。另外,HMQC的结果也归纳在表2中。这样,化合物3和4的结构就完成了解析。
2.4 新产物结构及其生物合成途径的推测
根据化合物3和4的化学结构以及前面各化合物的高分辨分子量的结果,可以对CsyB在大肠杆菌中表达所产生的新产物1–12的结构作出推测,将这些结构归纳到图4中。化合物1是CsyB通过两分子乙酰乙酰辅酶A一步缩合而成的,或者是由一分子乙酰辅酶A、一分子丙二酰辅酶A和一分子乙酰乙酰辅酶A反应所得。化合物2、7、9和11是分别通过一分子不同的起始单元癸酰辅酶A、月桂酰辅酶A、肉蔻酰辅酶A或者软脂酰辅酶A,然后接受一分子丙二酰辅酶A和一分子乙酰乙酰辅酶A缩合而成的。化合物6、8、10和12同样是先由不同的起始单元,即都含有一个双键的月桂酰辅酶A、肉蔻酰辅酶A、软脂酰辅酶A或者硬脂酰辅酶A,然后接受一分子丙二酰辅酶A和一分子乙酰乙酰辅酶A缩合而成的。化合物4的起始单元换为一个含有羟基的肉蔻酰辅酶A,然后,接受一分子丙二酰辅酶A和一分子乙酰乙酰辅酶A缩合而成的。化合物3和5的起始单元换为一个既含有羟基又含有双键的肉蔻酰辅酶A和软脂酰辅酶A,然后接受一分子丙二酰辅酶A和一分子乙酰乙酰辅酶A缩合而成的。这些中间体都是来源于大肠杆菌的脂肪酸合成途径,尽管对大肠杆菌的这些中间体研究较少,但是,通过本实验的结果和之前的研究[26-28],更加明确了大肠杆菌脂肪酸合成途径和合成过程中的一些中间体。在这个脂肪酸合成的过程中,脂肪酸的延伸是通过众多的Fab酶完成的,如FabH、FabB/F、FabG等。每次链伸长一个碳二单元要经历循环往复的过程,缩合一个来源于丙二酰辅酶A的含有羰基的碳二单元,然后还原羰基基团为羟基基团,接着,脱水去掉羟基基团形成双键,最后,还原双键形成单键,完成碳二单元的延伸。在这个过程中会出现含有双键、羟基或同时含有双键和羟基的中间体。这些与ACP蛋白连接的脂肪酰中间体通过与丙二酰辅酶A或者丙二酰ACP反应,形成长链的二酮的中间体,这个中间体和乙酰乙酰辅酶A或乙酰乙酰ACP发生非脱羧的克莱森缩合反应,生产最终csypyrone产物。
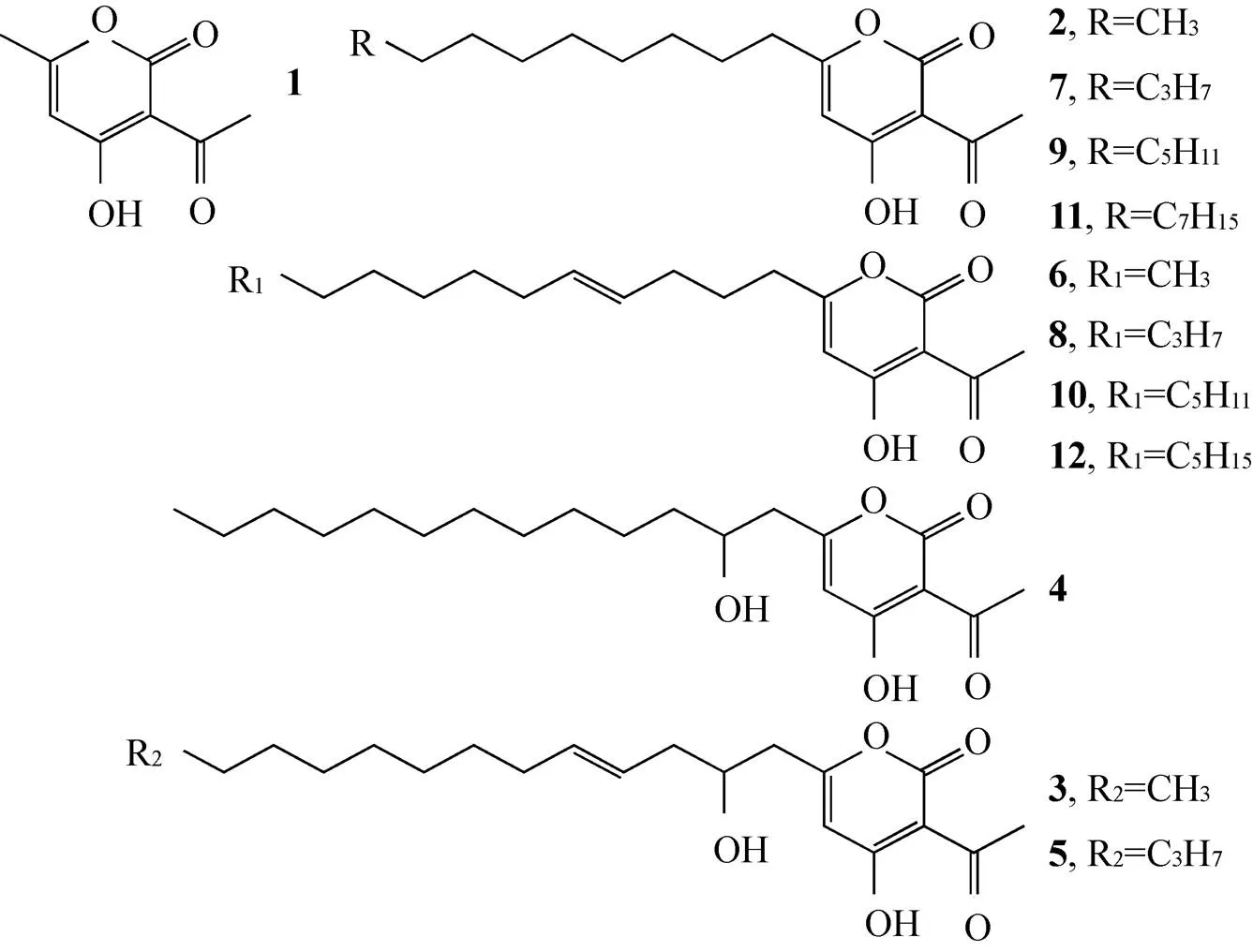
图4 推测的化合物1–12的结构
3 讨论
米曲霉来源的CsyB是一个多功能的Ⅲ型PKS,它与结核分枝杆菌烷基吡喃酮合成酶 PKS18具有26%的序列一致性,与粗糙脉孢菌2′-氧烷基雷琐酸合成酶 ORAS具有37%的同源性,它们通过脂肪酰辅酶A与丙二酰辅酶A的迭代缩合,产生长链的烷基吡喃酮和烷基雷琐酸。PKS18和ORAS的晶体学研究发现了一个狭长的绑定脂肪酰基辅酶A的疏水通道,研究表明这个通道具有广泛的底物多样性。在CsyB的晶体结构中同样发现了这样的一个通道,这条通道从酶的活性中心延伸到蛋白质的表面,使得蛋白质可以接受多种不同类型的脂肪酰辅酶A作为底物。因此,Ⅲ型PKS酶功能的多样性被认为是和其起始底物的兼容性密切相关。
本研究通过在大肠杆菌体内表达CsyB蛋白,得到了一系列csypyrone的衍生物,这一类化合物具有乙酰基吡喃酮的碳骨架,它的催化过程是由连续的两步缩合反应完成,先接受一分子起始单元,然后,缩合一分子丙二酰辅酶A,最后,缩合一分子乙酰乙酰辅酶A。CsyB对起始单元的特异性上,表现出很宽的范围和很强的容忍度,最短可以接受碳链长度为2的乙酰辅酶A,最长可以接受碳链长度为18的硬脂酰辅酶A。除了可以接受饱和脂肪酰辅酶A外,还可以接受不饱和脂肪酰辅酶A。另外,还可以接受3个具有羟基修饰的脂肪酰辅酶A。归根到底,这些特点都与其特有的结构密不可分,长且宽大的底物结合通道使其具有了可以跟更多底物结合的可能性。进而,在大肠杆菌体内完成了csypyrone类化合物的合成,得到了一系列新的产物。同时,在这个过程中,对大肠杆菌中的脂肪酸代谢中的中间体也有了更加全面的了解。
[1] Gokhale RS, Sankaranarayanan R, Mohanty D. Versatility of polyketide synthases in generating metabolic diversity. Curr Opin Struct Biol, 2007, 17(6): 736–743.
[2] Quadri LEN. Biosynthesis of mycobacterial lipids by polyketide synthases and beyond. Crit Rev Biochem Mol Biol, 2014, 49(3): 179–211.
[3] KuzmaKuzniarska M, Cornell HR, Moneke MC, et al. Lovastatin-mediated changes in human tendon cell. J Cell Physiol, 2015, 230(10): 2543–2551.
[4] Maksimenko A, Dosio F, Mougin J, et al. A unique squalenoylated and nonpegylated doxorubicin nanomedicine with systemic long-circulating properties and anticancer activity. Proc Natl Acad Sci USA, 2014, 111(2): E217–E226.
[5] Zhou H, Li YR, Tang Y. Cyclization of aromatic polyketides from bacteria and fungi. Nat Prod Rep, 2010, 27(6): 839–868.
[6] Staunton J, Weissman KJ. Polyketide biosynthesis: a millennium review. Nat Prod Rep, 2001, 18(4): 380–416.
[7] Hertweck C. The biosynthetic logic of polyketide diversity. Angew Chem Int Ed, 2009, 48(26): 4688–4716.
[8] Jing SS, Zhao SJ. A comprehensive overview of type III polyketide synthases from plants: molecular mechanism and application perspective-a review. Chin J Biotech, 2009, 25(11): 1601–1607 (in Chinese).晶生书, 赵树进. 植物Ⅲ型聚酮合酶的分子机制与应用前景. 生物工程学报, 2009, 25(11): 1601–1607.
[9] Su SY, Teng C, Zhang W, et al. Progress on type III polyketide synthase from bacteria. J Agric Sci Technol, 2013, 15(6): 119–129 (in Chinese).苏世友, 滕超, 张维, 等. 细菌Ⅲ型聚酮合酶研究进展. 中国农业科技导报, 2013, 15(6): 119–129.
[10] Jang MS, Cai LN, Udeani GO, et al. Cancer chemopreventive activity of resveratrol, a natural product derived from grapes. Science, 1997, 275(5297): 218–220.
[11] Frankel EN, German JB, Kinsella JE, et al. Inhibition of oxidation of human low-density lipoprotein by phenolic substances in red wine. Lancet, 1993, 341(8843): 454–457.
[12] Frankel EN, Waterhouse AL, Kinsella JE. Inhibition of human LDL oxidation by resveratrol. Lancet, 1993, 341(8852): 1103–1104.
[13] Abe I, Morita H. Structure and function of the chalcone synthase superfamily of plant type III polyketide synthases. Nat Prod Rep, 2010, 27(6): 809– 838.
[14] Austin MB, Noel JP. The chalcone synthase superfamily of type III polyketide synthases. Nat Prod Rep, 2003, 20(1): 79–110.
[15] Katsuyama Y, Horinouchi S.Microbial type III polyketide synthases//Mander LM, Liu HW. Comprehensive Natural Products II: Chemistry and Biology,Volume 1: Natural Products Structural Diversity-I Secondary Metabolites: Organization and Biosynthesis. Oxford: Elsevier, 2010: 147–170.
[16] Sankaranarayanan R, Saxena P, Marathe UB, et al. A novel tunnel in mycobacterial type III polyketide synthase reveals the structural basis for generating diverse metabolites. Nat Struct Mol Biol, 2004, 11(9): 894–900.
[17] Goyal A, Saxena P, Rahman A, et al. Structural insights into biosynthesis of resorcinolic lipids by a type III polyketide synthase in. J Struct Biol, 2008, 162(3): 411–421.
[18] Lv YY, Xiao J, Pan L. Type III polyketide synthase is involved in the biosynthesis of protocatechuic acid in. Biotechnol Lett, 2014, 36(11): 2303–2310.
[19] Seshime Y, Juvvadi PR, Kitamoto K, et al. Identification of csypyrone B1 as the novel product oftype III polyketide synthase CsyB. Bioorg Med Chem, 2010, 18(12): 4542–4546.
[20] Hashimoto M, Ishida S, Seshime Y, et al.type III polyketide synthase CsyB uses a fatty acyl starter for the biosynthesis of csypyrone B compounds. Bioorg Med Chem Lett, 2013, 23(20): 5637–5640.
[21] Hashimoto M, Koen T, Takahashi H, et al.CsyB catalyzes the condensation of two β-ketoacyl-CoAs to form 3-acetyl-4-hydroxy-6-alkyl-α-pyrone. J Biol Chem, 2014, 289(29): 19976–19984.
[22] Hashimoto M, Nonaka T, Fujii I. Fungal type III polyketide synthases. Nat Prod Rep, 2014, 31(10): 1306–1317.
[23] Mori T, Yang DF, Matsui T, et al. Structural basis for the formation of acylalkylpyrones from two β-ketoacyl units by the fungal type III polyketide synthase CsyB. J Biol Chem, 2015, 290(8): 4214–5225.
[24] Yang DF, Mori T, Matsui T, et al. Expression, purification and crystallization of a fungal type III polyketide synthase that produces the csypyrones. Acta Crystallogr Sect F Struct Biol Cryst Commun, 2014, 70(6): 730–733.
[25] Seshime Y, Juvvadi PR, Kitamoto K, et al.type III polyketide synthase CsyA is involved in the biosynthesis of 3,5-dihydroxybenzoic acid. Bioorg Med Chem Lett, 2010, 20(16): 4785–4788.
[26] Feng SX, Ma JC, Yang J, et al.fatty acid composition is determined by interaction of two 3-ketoacyl-acyl carrier protein reductases encoded on separate replicons. BMC Microbiol, 2015, 15: 223.
[27] Brock DJH, Bloch K. Control of the synthesis of long-chain fatty acids and triacetic acid in. Biochem Biophys Res Commun, 1966, 23(5): 775–780.
[28] Kass LR, Bloch K. On the enzymatic synthesis of unsaturated fatty acids in. Proc Natl Acad Sci USA, 1967, 58(3): 1168–1173.
(本文责编 郝丽芳)
synthesis of csypyrone derivatives by exploring the substrate diversity of start units of type Ⅲ polyketide synthase CsyB
Lixia Pan1, Jing Zhu1, Qingyan Wang1, Naikun Shen1, Yi Li1, and Dengfeng Yang1,2
1 Guangxi Key Laboratory of Biorefinery, Guangxi Biomass Industrialization Engineering Institute, National Engineering Research Center of Non-food Biorefinery, State Key Laboratory of Non-Food Biomass and Enzyme Technology, Guangxi Academy of Sciences, Nanning 530007, Guangxi, China 2 Guangxi Key Laboratory of Marine Natural Products and Combinatorial Biosynthesis Chemistry, Guangxi Beibu Gulf Marine Research Center, Guangxi Academy of Sciences,Nanning 530007, Guangxi, China
As a novel fungal type Ⅲ polyketide synthase, CsyB fromcan sequentially accept one molecular short chain fatty acyl CoA as start unit, one molecular malonyl-CoA and one molecular acetoacetyl-CoA as extend unit to produce the short chain csypyrone B1-3. On the basis of crystal structure of CsyB, a fatty acyl CoA binding tunnel of a length of about 16 Å is located in its active center that is proposed to accept diversified start units. In order to examine the substrate diversity of CsyB,gene was introduced and expressed inthat contained a number of precursors of long chain fatty acyl CoA. The results of HPLC revealed that a series of long chain csypyrone derivatives were detected in the recombinant strain in comparison with the control strain. These new csypyrone compounds were preliminarily analyzed by UV-visible spectroscopy and LC-HRMS. Three hydroxylated csypyrones were intensively determined by 1D and 2D NMR experiments, especially the position of the hydroxyl group in these compounds. These results demonstrate that CsyB exhibits a broad substrate specificity, which not only can accept the long chain saturated or unsaturated fatty acyl CoA as substrate, but also accept hydroxylated long chain fatty acyl CoA.
type Ⅲ polyketide synthase, CsyB, substrate diversity,synthesis, csypyrone
October 29, 2017;
December 11, 2017
National Natural Science Foundation of China (No. 31660251), Natural Science Foundation of Guangxi Province, China (No. 2017GXNSFAA198010).
10.13345/j.cjb.170426
Dengfeng Yang. Tel: +86-771-2503973; E-mail: yangdengfeng@gxas.cn
国家自然科学基金 (No. 31660251), 广西自然科学基金 (No. 2017GXNSFAA198010) 资助。
