产β-D-葡萄糖苷酶乳酸菌的 筛选、鉴定及系统发育分析
2018-07-11夏秀东李亚辉董明盛周剑忠
周 俊,夏秀东,李亚辉,王 英,董明盛,周剑忠,*
(1.南京农业大学食品科技学院,江苏南京 210095;2.江苏省农业科学院农产品加工研究所,江苏南京 210014)
β-D-葡萄糖苷酶(β-D-Glucosidase,EC 3.2.1.2 1)又称纤维二糖酶,可以水解β-D-葡萄糖苷键,常作用于β-D-(1,4)、β-D-(1,6)、β-D-(1,2)、β-D-(1,3)键,是纤维素酶系中一种重要的水解酶。果酒香气成分复杂,包含游离的挥发性风味物质和大量以β-D-葡萄糖苷形式存在的非挥发性风味前体。β-D-葡萄糖苷酶可以催化风味前体糖苷配基(烃基或芳基)与糖基之间的糖苷键水解,从而产生芳香物质达到增香效果[1-3]。β-D-葡萄糖苷酶在自然界中广泛分布,其中植物来源的β-D-葡萄糖苷酶非常丰富,但活性低、不稳定,且产量不高,因此微生物来源逐渐成为主要研究对象。曲霉来源的β-D-葡萄糖苷酶在葡萄酒中具有良好活性,因而被制成酶制剂用于增强果酒风味[4]。但由于曲霉在食品安全卫生方面存在隐患,本文选择具有高安全性的乳酸菌作为产酶菌株。
筛选高产菌株和检测β-D-葡萄糖苷酶活的方法,通常归纳为分光光度法、荧光法、PNPG法、电化学法等。PNPG法灵敏度高且重现性好,是目前实验室最常用的方法。然而该方法最主要的局限性在于色差不明显,需要通过测定其水解产物的吸光度才能确定菌株的酶活性,对于进行大规模的菌株筛选而言,步骤繁琐、成本太高[5]。选择七叶苷显色平板筛选产酶菌株,只需将待筛菌株划接种于显色平板,无需扩大培养即可通过颜色快速筛选出酶活性显著的菌株,同时获取单菌落,避免杂菌污染。
本文利用七叶苷显色平板法明显的色差对比,快速、高效地从实验室保藏菌株中筛选出β-D-葡萄糖苷酶高产菌株。以对硝基苯基-β-D-吡喃葡萄糖苷(PNPG)为底物进行酶活初步定位,验证菌株酶活的同时,确定菌株产酶的主要部位[6-7]。利用生理生化实验、糖发酵实验和16S rDNA系统发育树对菌株进行鉴定,并分析菌株的遗传多样性[8],为后续应用于果酒增香提供理论基础。
1 材料与方法
1.1 材料与仪器
63株待筛选乳酸菌 于江苏省农科院农产品加工所226实验室-20 ℃冰箱保存。
过氧化氢、蛋白胨、酵母提取物、盐酸、氢氧化钠、明胶、半胱氨酸、乙酸铅试纸以及各种糖化合物水解实验材料 南京寿徳有限公司;基因组提取试剂盒 天根生化科技(北京)有限公司;Taq DNA聚合酶 TaKaRa公司;DNA Maker,琼脂糖,PCR引物合成,基因测序 北京擎科生物技术有限公司;Goldview I型核酸染色剂 北京索莱宝科技有限公司;琼脂糖凝胶DNA回收试剂盒 Axygen公司。
DSX-280B型式压力蒸汽灭菌器 上海申安医疗;AG PB-10型pH计 德国Sartorius;SW-CJ-2G型超净工作台、LRH-150型生化培养箱 苏州净化设备;DHG-9146A型电热鼓风干燥箱 上海精宏实验设备;3K15型高速冷冻离心机 美国Sigma;WH-3型漩涡混合仪 上海跃进医疗;PCR仪,PRO-3型垂直平板电泳仪,GelDocEQ型凝胶成像仪 BIO-RAD公司;超纯水机 力康HealForce公司;制冰机IMS-40 常熟市雪科电器;恒温水浴锅 常州国华;分光光度计One Drop OD-2000 南京五义科技;全温振荡器THZ-C-1 太仓市实验设备;分析电子天平FA2004 上海良平仪器仪表;超声波细胞破碎仪 南京先欧仪器。
MRS液体培养基:牛肉膏10 g/L,、酵母膏5 g/L、蛋白胨10 g/L、葡萄糖20 g/L、乙酸5 g/L、磷酸氢二钾2 g/L、柠檬酸二氨2 g/L、硫酸镁0.2 g/L、硫酸锰0.05 g/L、吐温80 1 mL/L。调节pH至7.0,121 ℃高压蒸汽灭菌20 min。主要用于乳酸菌的活化和培养。
MRS固体培养基:在MRS液体培养基的基础上添加1.8%的琼脂,121 ℃高压蒸汽灭菌20 min。主要用于乳酸菌的分离和纯化。
七叶苷显色平板:在MRS固体培养基上添加0.5%的七叶苷和0.025%的柠檬酸铁,主要用于筛选产β-D-葡萄糖苷酶的乳酸菌。
1.2 实验方法
1.2.1 七叶苷显色平板筛选法产酶菌的筛选 具有β-D-葡萄糖苷酶活性的菌株能够水解底物七叶苷(6,7-2-羟基-香豆素-β-D-葡萄糖苷),其产物七叶素(6,7-2-羟基-香豆素)与柠檬酸铁中的三价铁离子反应能产生棕黑色物质,形成明显的色差便于肉眼观察,极大提高了分离工作的效率[7]。
将63株乳酸菌分别划线接种于MRS固体培养基上,置于37 ℃培养箱培养16~18 h,挑取单菌落将其接种于七叶苷显色平板培养基上,置于37 ℃培养箱中避光培养24 h后观察其颜色变化,若在菌落周围出现了棕黑色物质,则表明该菌株具有β-D-葡萄糖苷酶活性[9-10]。
1.2.2β-D-葡萄糖苷酶的初步定位研究 根据酶在生物体内存在的部分,可分为胞内酶和胞外酶。胞外酶作用于细胞外,利用离心方法即可获得;而胞内酶作用于细胞内,需破碎细胞后获取。对比菌株各个组分的酶活,可确定β-D-葡萄糖苷酶在细胞中的存在部位,为后续获取目的蛋白酶采取何种纯化方案提供了理论基础。
对1.2.1中筛选出的产β-D-葡萄糖苷酶的菌株进行其产酶部位的定位。β-D-葡萄糖苷酶的初步定位研究参照相关文献[11],结合实验室条件参数调整如下:选取平板颜色有明显变化的菌株接种于MRS液体培养基中过夜培养18 h,取30 mL菌液冷冻离心(8000 g,4 ℃10 min)得菌体及菌液上清液,取1 mL菌液上清液用于酶活性测定,菌体用0.85% NaCl溶液(w/V)洗涤两遍,然后悬浮于30 mL PBS 1×buffer(KCl 2.7 mmol/L;NaCl 140 mmol/L;Na2HPO410 mmol/L;KH2PO41.8 mmol/L,pH7.4)。取1 mL菌体悬浮液(作为完整细胞)用于酶活性测定,向剩余菌液中加入溶菌酶(1 mg/mL),并于37 ℃孵育1 h后在冰浴条件下用超声波细胞粉碎机破碎20 min(功率25%,超声1 s,间停3 s),取1 mL细胞破碎液直接用于酶活性测定,另取10 mL破碎液离心(8000×g,4 ℃,10 min),得破碎上清液和细胞碎片。取1 mL破碎上清液直接用作酶活性测定。细胞碎片用0.85% NaCl溶液洗涤两次,然后悬浮于10 mL PBS 1×buffer溶液中,取1 mL菌体悬浮液(作为细胞碎片)用作酶活性测定。经过上述实验步骤,总共获得5个样品:菌液上清、完整细胞、细胞破碎液、破碎上清和细胞碎片。最后按照以下酶活性测定方法对样品进行酶活性测定,所有样品重复测定三次。
实验中改进了Grimaldi等[4]所用的β-D-葡萄糖苷酶活性测定方法,反应在96孔板中进行反应,并使用酶标仪测定吸光值。反应体系(80 μL)包括:50 μL磷酸氢二钠-柠檬酸缓冲液(0.1 mol/L 柠檬酸,0.2 mol/L K2HPO4,pH4.5),10 μL 20 mmol/L PNPG溶液,20 μL菌液,于37 ℃培养箱中反应30 min后,立即加入120 μL的1 mol/L Na2CO3溶液终止反应,混匀后静置一会,于405 nm下测定吸光值(OD),测定3个重复。空白对照为未加入乳酸菌的反应体系,阴性对照为用无菌水代替PNPG溶液,其他处理相同。
1.2.3 产β-D-葡萄糖苷酶乳酸菌的鉴定
1.2.3.1 菌体形态和生理生化实验 将 1.2.1中筛选出的产β-D-葡萄糖苷酶的菌株接种于MRS琼脂平板上,置于37 ℃培养18 h,观察菌落形态,并进行革兰氏染色后观察菌体形态。各项生理生化实验指标参照《乳酸细菌分类鉴定及实验方法》[12]、《乳酸菌:生物学基础及应用》[13]及《伯杰细菌鉴定手册》[14]。凡革兰氏染色呈阳性,过氧化氢酶实验、硫化氢产生实验为阴性且在pH4.5条件下可以生长的杆状无内生芽孢菌株可初步认定为是乳杆菌。再进行其他生理生化实验:明胶液化实验、吲哚实验和硝酸盐还原实验证是否符合乳杆菌属的生物特性。根据菌种对单糖、多糖以及糖醇类等碳水化合物的利用情况可以粗略划分乳杆菌种[15],若出现与发酵群种的生理生化特征相矛盾的情况可增补其他鉴别特征:所产乳酸的异构体、肽聚糖类型等。
1.2.3.2 16S rDNA PCR 将筛选得到的具有β-D-葡萄糖甘酶活性的菌株接种于液体MRS培养基中,培养16~18 h至对数期后制成乳酸菌菌体悬浮液,按照细菌基因组提取试剂盒操作说明提取菌株的基因组DNA。将提取得到的基因组DNA溶液用One Drop OD-2000和琼脂糖凝胶电泳进行检测,测定其OD260、OD280及其比值[16],以确定DNA浓度和纯度是否达到后续实验要求。剩余DNA溶液放置于-20 ℃冰箱冻藏。
以提取得到的基因组DNA作为PCR扩增的模板。反应体系为50 μL,扩增反应体系见表1。PCR扩增反应条件:98 ℃预变性5 min,98 ℃变性1 min,55 ℃退火1 min,72 ℃延伸1 min,共进行30个循环,最后在72 ℃下延伸10 min。将PCR扩增后的产物进行琼脂糖凝胶电泳,电压为120 V,电泳结束后用凝胶成像仪观察条带,将观察到的目的条带进行切胶处理,用DNA胶回收试剂盒进行切胶回收,并由北京擎科生物股份有限公司完成测序。

表1 16S rDNA PCR扩增体系Table 1 PCR amplification system of 16S rDNA PCR
1.2.3.3 16S rDNA序列同源性分析 用DNAStar软件的SeqMan对测序结果进行双向拼接后,通过NCBI网站上的BLAST程序(http://www.ncbi. nlm.nih.gov/)对筛选菌株的16S rDNA基因的核苷酸序列进行分析和确定。一般来讲,在种分类等级上,如果2个分类单位间的16S rDNA序列同源性大于97.5%,则认为属于同一个种[17]。根据序列同源性在标准菌株网站(http://www.straininfo.net/)中选取不同的模式菌株,使用MEGA 5.0软件将模式菌株序列、Blast获取的高同源性菌株序列以及测定菌株序列以Clustal W方法进行多序列比对。删除两端未对齐的碱基,使用NJ法[18]构建进化树,采用自举分析进行置信度检测,自举数据集为1000次[19]。
1.3 数据处理
利用Excel和Prism 7软件进行数据处理及作图分析。
2 结果与分析
2.1 平板筛选
为凸显颜色差异,选择将具有活性的与部分无活性菌株重新接种于同一块显色平板上,结果如图1所示。从筛选结果可以看出,编号为CM3、Hsb、FL12、T61的菌株具有β-D-葡萄糖苷酶活性,且根据平板颜色的深浅,可以初步判定菌株T61的酶活最高,菌株CM3、Hsb、FL12的酶活接近。相比于从平板中挑取单菌落扩大培养再测定酶活的方法,七叶苷显色平板法避免了繁琐的实验步骤,而且灵敏度高,重现性好,是大规模筛选产β-D-葡萄糖苷酶菌株的优先选择[20]。

图1 七叶苷显色平板筛选结果Fig.1 Detection of enzymes on the Esculin color-developing plate
2.2 β-D-葡萄糖苷酶的初步定位
设定CM3的完整细胞为最高酶活100%,计算其他组分的相对酶活。图2为4株菌株所产β-D-葡萄糖苷酶初步定位的结果。有下图可知,4株菌均具有β-D-葡萄糖苷酶活性,其中T61的酶活明显要低于其他菌株,这与七叶苷显色平板法的结果相矛盾。对比4个菌株培养基的菌液浊度可知,菌株T61的菌体浓度明显低于其他菌株,对比该菌株在MRS固体培养基中的生长状态,推测该菌株为兼性厌氧型菌株,前期有氧条件可以促进生长,后期生长需要尽量保持低氧环境。
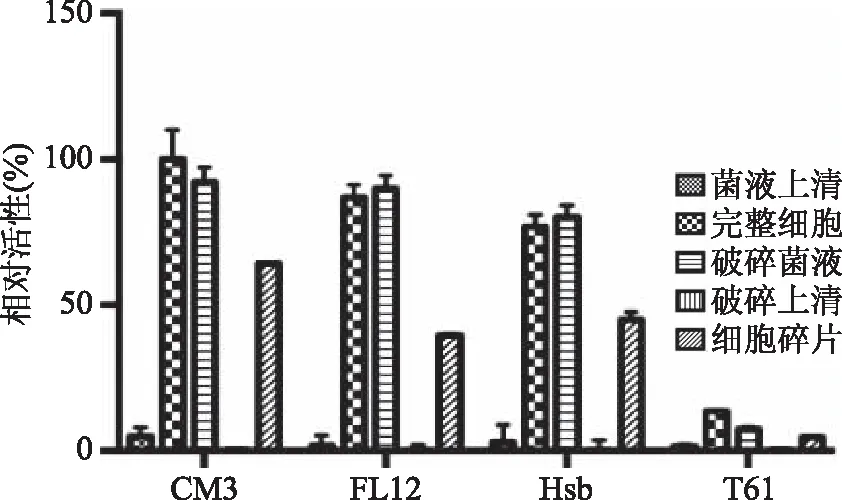
图2 β-D-葡萄糖苷酶的初步定位Fig.2 β-glucosidase activity of different fractions from four strains
菌株CM3、FL12、Hsb各组分的酶活情况相似:菌液上清几乎无酶活,而完整细胞具有酶活,说明菌株所产的β-D-葡萄糖苷酶并没有分泌到细胞外,属于胞内酶[21]。完整细胞和破碎菌液酶活相接近,说明细胞破碎并不会显著影响酶活性。破碎上清(可溶性酶)无酶活而细胞碎片(细胞膜部分)约保留40%~60%酶活性,推测细胞中含有某些物质能促进酶催化反应,因而细胞碎片酶活降低。并且菌株所产的β-D-葡萄糖苷酶与细胞膜紧密结合,形成不可溶状态。
虽然这与大部分胞外及胞内β-D-葡萄糖苷酶的研究结果存在差异[22-24],但仍有前人报道过类似情况[23]。产生这种情况的原因可能与酶的结构和特性有关。目的蛋白酶与细胞膜结合的特性,使得无法通过硫酸铵沉淀、凝胶过滤层析等传统纯化方式获得纯酶。除了考虑表面活性剂等材质处理目标蛋白酶,还可以通过修饰目的基因片段,添加标签,将目的蛋白通过外源表达方式获取[5,25]。
2.3 产β-D-葡萄糖苷酶乳酸菌的鉴定
2.3.1 形态学观察 菌株菌落形态基本相似,为生长状况良好、边缘光滑整齐、乳白色、凸起的圆形菌。T61的菌落为灰白色,中间有深色突起的褐色小点。4株菌经染色后,在油镜下观察均呈现蓝紫色,为革兰氏染色阳性菌,无芽孢、无鞭毛。细胞形态都为杆状,有长杆短杆,短杆菌成单个或成对出现,一般形成链。符合乳杆菌的形态特征。
2.3.2 生理生化实验 将选出的4株疑似乳杆菌菌株进行一系列生理生化鉴定实验,结果如表2所示。4株菌株均为过氧化氢酶实验阴性、硫化氢实验阴性、明胶液化阴性、硝酸盐还原阴性。在耐酸碱实验中,pH4.5时,菌株都能生长,表明菌株CM3、Hsb、FL12、T61都耐酸,T61在pH为2.5时仍能生长,可见耐酸性能最强。随着生长环境pH的升高菌种生长状况逐渐变好,在pH6.5时长势最好,在中性或初始碱性pH条件时,生长情况开始下降[8,26]。筛选获取的4株菌株初步判断为乳杆菌。
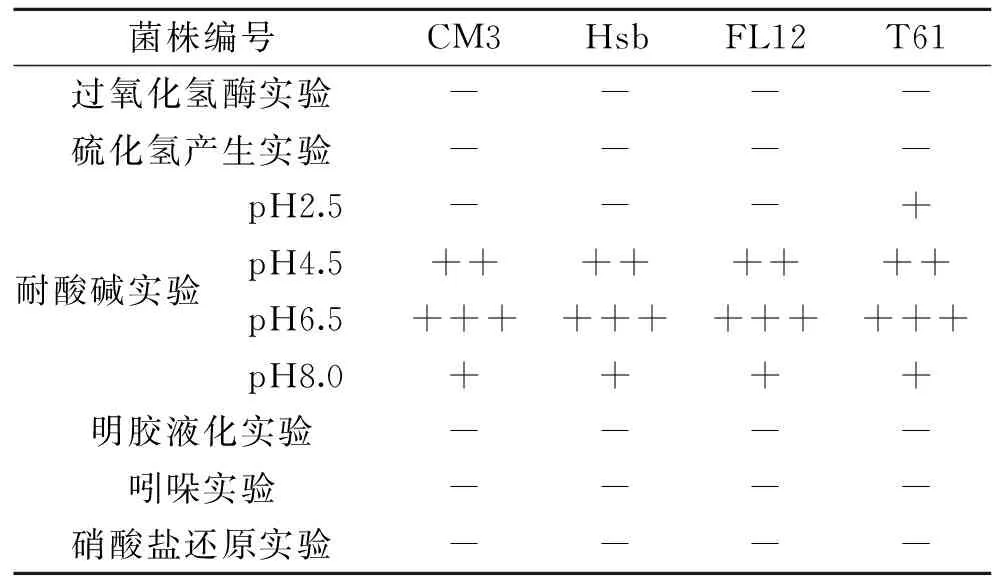
表2 生理生化实验结果Table 2 Physiological and biochemical result of the strains
糖类发酵实验可划分菌种,由表3可知,CM3、Hsb、FL12菌株生长情况相近,利用多糖的情况基本相同,T61菌株利用木糖和甘露糖的能力不是很强,不能利用海藻糖和棉籽糖。将生理生化、糖发酵结果与《乳酸菌分类鉴定及实验方法》中乳杆菌的特征对照,基本符合乳杆菌的理化特性。
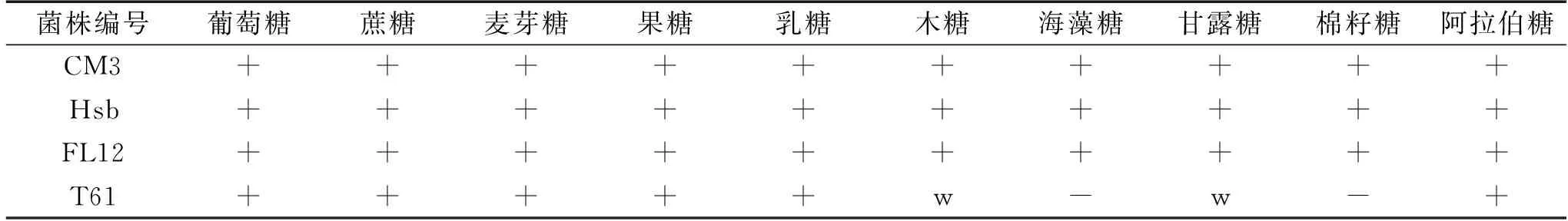
表3 糖发酵实验结果Table 3 Results of sugar fermentation
2.3.3 16S rDNA序列分析及鉴定结果 用One Drop OD-2000和1%的琼脂糖凝胶电泳检测CM3、Hsb、FL12、T61菌株提取的基因组DNA。确保各菌株提取的基因组DNA纯度较高,符合PCR扩增的要求。再将经过16S rDNA扩增后的PCR产物用1%的琼脂糖凝胶电泳对其进行电泳检测,在大约1500 bp处获得荧光条带(图3),且电泳图无杂带确保其完整性。
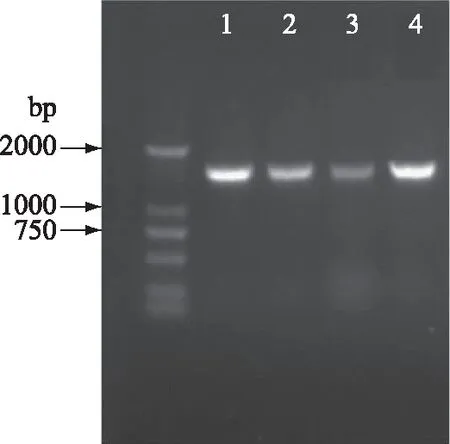
图3 乳酸菌16Sr DNA片段PCR扩增产物凝胶电泳图Fig.3 Agarose gel electrophoresis pattern of PCR amplified products of 16S rDNA fragment注:1.CM3;2.Hsb;3.FL12;4.T61。
在NCBI网站中使用BLAST软件对测序结果进行同源性分析,结果见表4。CM3菌株和戊糖乳杆菌(Lactobacilluspentosus)同源性最高,Hsb、FL12菌株和植物乳杆菌(Lactobacillusplantarum)同源性最高,T61菌株和短乳杆菌(Lactobacillusbrevis)同源性最高。所有菌种的同源性、覆盖率均达到99%以上,E值都为0.0。
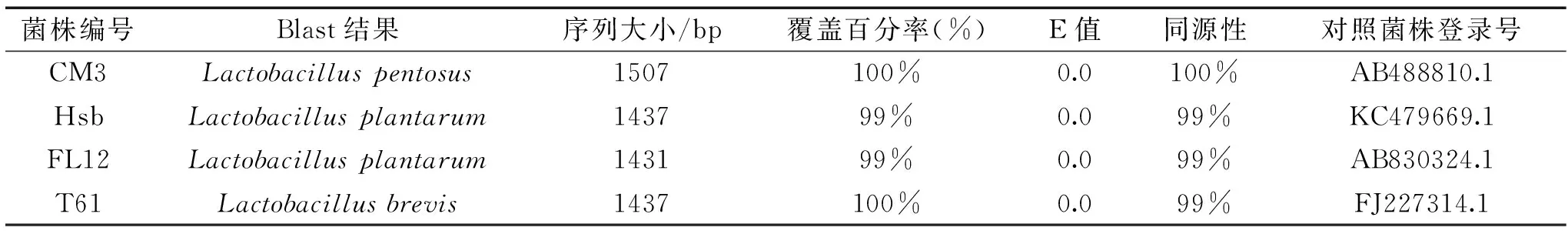
表4 菌株的16S rDNA序列同源性比对结果Table 4 Homologous alignment analysis of 16S rDNA sequences from isolates
2.3.4 16S rDNA 序列系统发育树 以4株乳酸菌测序后拼接的序列、与其高同源性菌株序列以及标准菌株序列为对象,构建进化树。
如图4所示,系统发育树呈现3个较大分支。其中CM3、Hsb和FL12处于一个较大分支,Hsb、FL12和植物乳杆菌模式菌株(L.plantarumAB830324.1)自聚为一小分支,CM3与戊糖乳杆菌模式菌株(L.pentosusD79211.1)处于一个大分支;T61和短乳杆菌模式菌株(L.brevisEU194349.1)处于另一个大分支。一般来说,如果低自展值靠近分支末端,可能是由于相似度太高难以区分;如果低自展值靠近根,可能是由于相似度太低[27]。系统发育树各分支自展值均高于50,其分支结构符合上表4序列同源性比对结果,综合所有实验数据可以判定:Hsb和FL12为植物乳杆菌,CM3为戊糖乳杆菌,T61为短乳杆菌。同时因为各个菌株的亲缘关系与它们在进化树中的距离呈正比,表明戊糖乳杆菌和植物乳杆菌的亲缘关系较近。
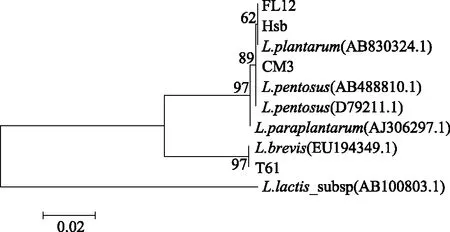
图4 基于16S rDNA序列对乳酸杆菌进行构建系统发育树Fig.4 Phylogenetic tree for lactic acid bacteria based on their 16S rDNA sequences
3 结论
以实验室保藏的63株菌为对象,通过七叶苷显色平板法共筛出4株可高产β-D-葡萄糖苷酶的乳酸菌疑似菌株。通过酶定位实验,确定了菌株所产β-D-葡萄糖苷酶为胞内酶,并且超声破碎后上清不存在活性,不利于天然蛋白的纯化。通过镜检、过氧化氢酶实验初步判定为乳酸菌菌株,再通过生理生化实验、糖发酵实验进一步验证,推测4株菌株都为乳杆菌,对其进行测序、同源性分析和系统发育树的构建,4株代表性菌株中,FL12、Hsb为植物乳杆菌(Lactobacilluspentosus),CM3为戊糖乳杆菌(Lactobacilluspentosus),T61为短乳杆菌(Lactobacilluspentosus)。
由于无法通过收集破碎上清液的方式获取纯酶,后续研究将尝试使用分子生物学技术对β-D-葡萄糖苷酶基因进行克隆、表达纯化,以探究乳酸菌来源的β-D-葡萄糖苷酶对风味物质的影响。
