陕南水稻根际细菌多样性变化趋势
2018-07-06王梦姣
王梦姣
(1.陕西理工大学陕西省资源生物重点实验室,陕西 汉中 723000; 2.陕西理工大学陕西省食药用菌工程技术研究中心,陕西 汉中 723000; 3.陕西理工大学生物科学与工程学院,陕西 汉中 723000)
植物根际微生物是根际微生态系统的重要组成部分,能够直接影响土壤物理结构,对土壤肥力、植物营养转化起决定性作用,是衡量土壤肥力的重要指标[1-2]。对农作物根际微生物结构组成、空间分布、多样性变化趋势的分析,有助于从微生物角度改善土壤环境和土壤结构,对进一步研究根际微生物与农作物的互作关系,提高陕南农作物产量和质量具有重要的理论和实践意义。
目前,人们对小麦(TriticumaestivumL.)、玉米(ZeamaysLinn.)等作物的根际微生物多样性进行了许多研究,均发现根际微生物多样性与作物的生长发育密切相关[3-4]。例如,胡桂萍等[5]针对水稻内生菌和根际微生物群落多样性进行共同研究发现,不同水稻品种其根际微生物群落多样性具有显著差异;借助根际微生物群落多样性的平台,刘波等[6]进行新的微生物分离方法的研究,这为从不同发育时期分离不同微生物,并对其功能研究提供了新的思路;张仕颖等[7]利用丁草胺污染对水稻及水稻根际微生物区系作用的试验阐明了外界影响能够对水稻根际微生物群落特征有显著影响,这也反应了在“根际微生物-土壤-作物”这个微生态系统中,根际微生物的变化能够直接关联作物的生长发育;化感水稻根际微生物的生物学特性研究[8],可以使我们了解特定水稻品种的根际微生物的生态学特性。这些研究结果都证明,水稻根际微生物与水稻生长发育密切相关,根际微生物多样性与作物的生长发育存在紧密的互作和联系[9-10]。
陕西南部地区是陕西省水稻的主产区,也是中国重要的水稻主产区[11],该地区的水稻种植方式属于轮作制[12]。前人研究还未涉及到陕南这一具有油菜-水稻轮作制耕种体系的根际微生物多样性分析,本研究旨在利用培养法研究陕南水稻-油菜轮作区的2个水稻品种在其五叶期、分蘖期、孕穗期[13-14]的根际细菌多样性组成,拟从土壤细菌层面探讨水稻生长发育与根际细菌之间的关系,为进一步指导实际生产提供理论依据。
1 材料与方法
1.1 采样地及水稻概况
本试验在陕西南部地区水稻主要种植区汉滨、城固和宁强[15-16]进行,这3个采样地气候分别属于亚热带大陆性季风气候、北亚热带湿润季风区和暖温带山地湿润季风气候,降水集中在9月,经度分别为109°01′ E、107°13′ E、105°60′ E,纬度分别为32°42′N、32°75′N、32°55′N,海拔高度分别为260 m、480 m、820 m,供试土壤均为潴育性水稻土,灌溉水源均为河水,使用肥料均为尿素、复合肥(N∶P∶K=15∶15∶15,质量比)。前茬作物为油菜。
水稻K优082为平原地区主栽品种[16],3个采样地产量分别是10 062.0 kg/hm2、10 207.5 kg/hm2、9 100.5 kg/hm2,千粒质量分别为31.9 g、31.2 g、31.6 g,单株有效穗数分别为17.3、16.7、16.9,每穗粒数分别为180.8、160.6、161.4,结实率分别为84.3%、85.8%、82.6%。先丰优901为山区地区主栽水稻品种[17],3个采样地产量分别是9 762.0 kg/hm2、9 906.0 kg/hm2、9 252.0 kg/hm2,千粒质量分别为29.7 g、29.2 g、29.4 g,单株有效穗数分别为18.1、17.4、16.2,每穗粒数分别为182.5、170.0、169.1,水稻结实率分别为86.9%、84.7%、80.7%。
1.2 水稻根际采集时间及采集方法
采集土样时间为2015年4月至8月,具体采样时间见表1。采用五点采样法进行根际样品采集,将水稻根系从土壤中挖出,用抖落法[17]抖掉与根系松散结合的土体,然后将土壤连带植物组织包裹带回实验室后进行充分混匀,过2 mm筛。

表1 采样时间表
1.3根际微生物的分离计数及纯培养
称取10 g水稻根际放入装有100 ml无菌水的三角瓶中,150 r/min振荡10 min,吸取5 ml至装45 ml有无菌水的三角瓶中,制成1∶100的稀释液,再依次制备成10-1、10-2、10-3梯度稀释液。吸取各稀释液100 ml至牛肉膏蛋白胨培养基的培养皿中,涂布均匀,37 ℃培养 14~18 h。每个稀释梯度做3个重复。利用菌落计数器对每一个平板上的土壤细菌进行单菌落计数,计算分离出来的土壤中的细菌数[18]。
菌落数=菌落平均数×稀释倍数/干土质量×100%
1.4 细菌16S rDNA的扩增及序列分析
用天根细菌基因组DNA提取试剂盒将细菌基因组DNA提取后,用通用引物27F(5′-AGAGTTTGATCCTGGCTCAG-3′)和1492R(5′-GGTTACCTTGTTACGACTT-3′)进行16S rDNA的序列扩增。PCR反应体系(50.00 μl)为:10×Buffer 5.00 μl、2.5 mmol/L Dntp 5.00 μl、上下游引物各2.00 μl、模板1.00 μl、Taq酶0.25 μl,加水至50.00 μl。PCR反应条件为:94 ℃预变性5 min;94 ℃变性30 s、55 ℃退火30 s、72 ℃延伸90 s,35个循环;72 ℃延伸5 min,终止PCR反应。采用0.8%琼脂糖对PCR扩增产物进行电泳检测,并送上海英骏测序公司测序。
测序完成后,对所有菌株进行 16S rDNA序列测定。测序结果利用BLAST软件(http://www.ncbi.nlm.nih.gov/blast/Blast.cgi)与GenBank 数据库中的序列进行比对分析,初步确定分离得到菌的种属。
1.5 系统发育树的构建
选取同源性最高且有效发表的菌株序列,利用 MEGA5.1 软件(http://www.megasoftware.net/mega5.1.html)进行分析,用 Clustal W 按照最大同源性的原则进行排序,采用Kimura-2 计算核苷酸差异值,最后用邻接法(Neighbor-Joining method)[19]构建系统发育树。
2 结果与分析
2.1 水稻根际细菌的数量变化
对3个地区根际细菌菌落数的统计(图1)发现,在溶液稀释10倍和100倍的条件下,汉滨和城固地区K优082的根际细菌菌落数大于先丰优901,宁强地区则相反。这可能是由于平原主栽品种K优082在平原地区(汉滨和城固)生长状态更好,与根际微生物互作关系更强,能够被分离得到更多的细菌,而山地主栽品种先丰优901则在山地(宁强)地区更有生长优势。这说明,水稻品种及地理位置对水稻根际细菌数量有显著影响,这与黄柳琴等[20]、厉桂香等[21]的研究结果较为一致。稀释1 000倍的土壤溶液中可培养的细菌数量均较小,不能进行比较和分析。
分析曲线变化可以发现,在五叶期的10倍和100倍根际土壤稀释液中分离得到了较多的细菌菌落,且其数量较其他两个时期有显著差异。这一现象均出现在3个采样地的2个水稻品种根际土壤细菌分离过程中。可以初步推断,水稻可能在五叶期与微生物互作更为紧密,分蘖期次之,孕穗期最差。这可能与水稻生长特性相关,随着水稻的生长发育,土壤含水量逐渐升高,导致细菌数量下降[22]。
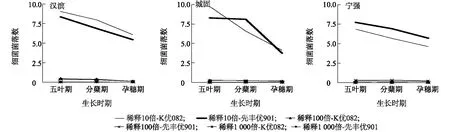
图1 细菌菌落数变化趋势图Fig.1 The variation tendency of bacterial colony
2.2 水稻根际土壤细菌组成
对分离获得具有明显差异的单菌落进行DNA提取及16S rDNA扩增,每个采样地的每个水稻品种3个时期的根际细菌单菌落均筛选出 70株细菌。从图2中可以看出,提取的DNA较为完整,扩增后得到约为1 500 bp的细菌16S rDNA条带,可保证后续测序结果的完整性。
每个采样地的每个品种在每个时期均成功进行了62个测序,通过对测序结果的分析发现,水稻根际中共检测出4个门,分别为拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、变形菌门(Proteobacteria)和厚壁菌门(Firmicutes)。其中,厚壁菌门为优势菌种,在每个采样点所占比例均超过70%,在汉滨采样地的水稻分蘖期先丰优901的根际细菌均属于厚壁菌门。从图3中还可以发现,随着水稻的生长发育,厚壁菌门所占比例也逐渐上升,在分蘖期达到最高峰,又在孕穗期下降。变形菌门在所有的五叶期和孕穗期的根际中均能发现。

A图和B图中M代表Maker,用来指示条带长度,为康为世纪的100 bp ladder,货号为CW0636M。A图中1~6代表6个不同的细菌菌液提取出来的基因组DNA,B图中1~6代表6个细菌基因组DNA经过PCR扩增出来的16S DNA条带。图2 水稻根际细菌基因组DNA提取(A)及细菌16S rDNA扩增图(B) Fig.2 Results of DNA(A) and 16S rDNA(B) on soil bacteria form rice rhizosphere

a:K优082-五叶期;b:先丰优901-五叶期;c:K优082-分蘖期;d:先丰优901-分蘖期;e:K优082-孕穗期;f:先丰优901-孕穗期。图3 水稻根际细菌组成Fig.3 The group of bacteria in rice rhizosphere soil
进一步从属的角度分析,从图4中可以看出:首先,在分析的所有根际样本中,共有26个属,1~26分别为芽孢杆菌属(Bacillussp.)、假单胞菌属(Pseudomonassp.)、葡萄球菌属(Staphylococcussp.)、节杆菌属(Arthrobactersp.)、根瘤菌属(Rhizobiumsp.)、产碱杆菌属(Alcaligenessp.)、肠杆菌属(Enterobactersp.)、泛菌属(Pantoeusp.)、不动杆菌属(Acinetobactesp.)、短杆菌属(Brevibacteriumsp.)、嗜冷杆菌属(Psychrobactersp.)、鞘氨醇菌属(Sphingobacteriumsp.)、Lysinibacillusvarians、弗拉托氏菌属(Frateuriasp.)、Fictibacillus、黄单胞菌属(Xanthomonassp.)、巴斯德菌属(Pasteurellasp.)、噬氢菌属(Hydrogenophagaatypica)、梭菌属(Fusiformis)、Terribacillus、丛毛单胞菌属(comamonas sp.)、寡养单胞菌属(Stenotrophomonas)、土壤杆菌属(Agrobacteriumsp.)、阪崎肠杆菌属(Cronobactersp.)、气单胞菌属(Aeromonassp.)和嗜氮根瘤菌属(Azorhizophilussp.)。其中,枯草芽孢杆菌属为优势菌,其在除汉滨地区K优082的五叶期根际样本(枯草芽孢杆菌属细菌占总菌数的64.52%)以外的所有根际样本中所占比例均超过80.00%,最高甚至达到98.39%(宁强采样地的K优082在孕穗期的根际样本)。
其次,3个采样地的2个水稻品种在五叶期的根际样本中细菌丰度最好,这一结论也与本研究关于细菌计数及细菌组成分布的结论一致。因此,五叶期水稻与土壤及根际微生物互作最为显著。
2.3 系统进化树的构建
根据测序结果对所有分离得到的细菌进行了统一归类,对获得的细菌DNA序列与GenBank数据库中的序列进行比对,选择一些与测定序列同源性较高的抑制种序列作为参考序列,构建系统发育树。结果(图5)表明,在所有土壤样本中,共得到66个不同的细菌种,枯草芽孢杆菌属为优势细菌类群。

图4 3个采样地不同水稻生长时期根际细菌多样性变化趋势Fig.4 The diversity trends of bacteria in rhizosphere soil in different rice growth period
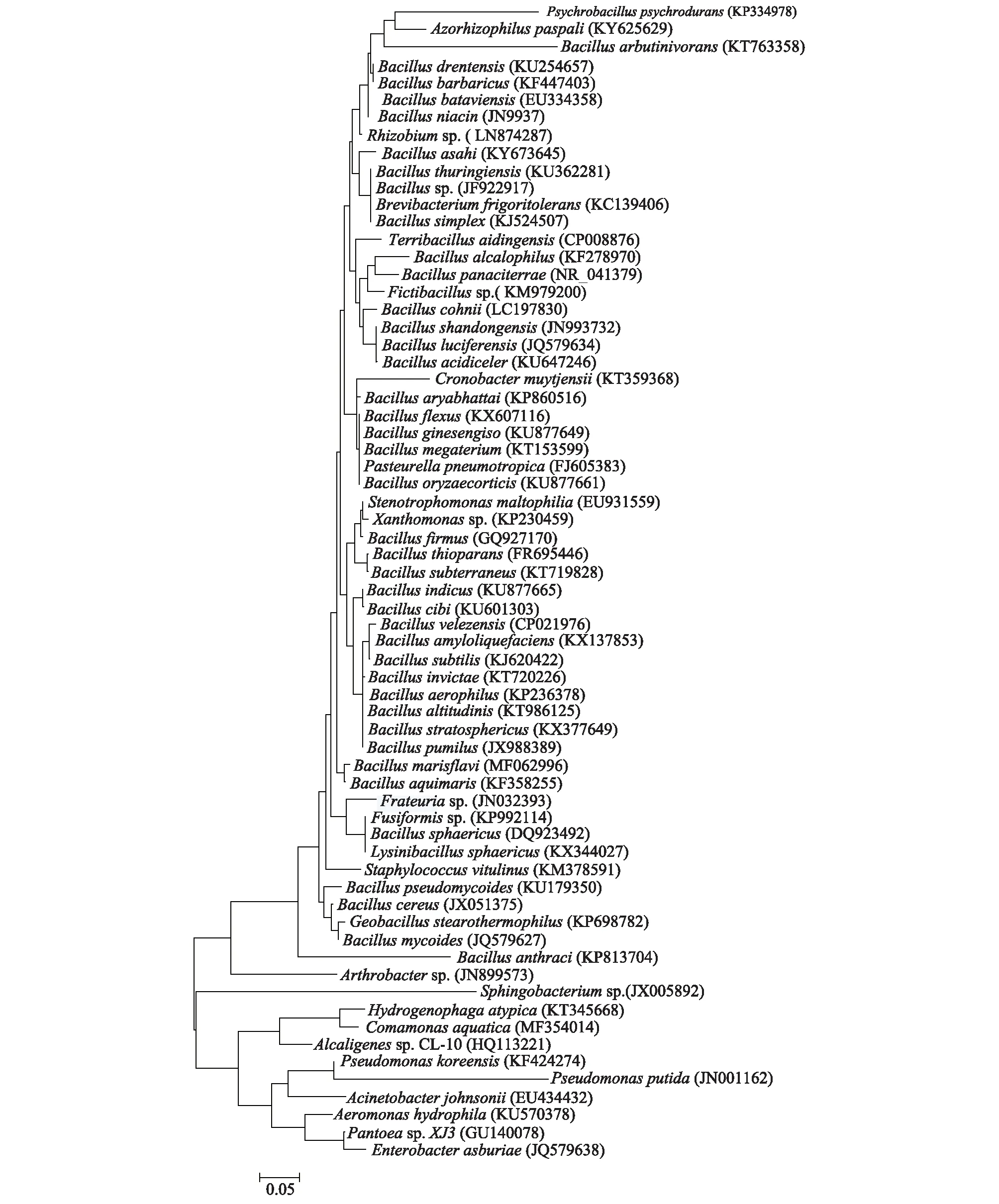
图5 基于16S rDNA的水稻根际细菌系统发育树Fig.5 Phylogenetic tree of bacteria in rice rhizosphere soil based on 16S rDNA
3 讨 论
本研究主要针对西北水稻主产地——陕西南部地区,对不同生长发育时期,2个水稻品种的根际微生物组成进行了分析。结果表明,从所有根际样本中共分离得到了26个属的细菌,分别划归于4个门。其中厚壁菌门的细菌占大多数,芽孢杆菌属为优势属。本研究数据对构建陕南土壤微生物数据库提供了基础试验数据。同时,试验还发现了一些其他具有一定研究价值的菌属,比如前人研究较少,具有良好抗盐效果的Terribacillus属[23],将会在后续研究中继续进行探索。
本研究还对不同生长阶段的细菌多样性变化趋势进行分析,结果发现,2个水稻品种根际细菌均在五叶期的多样性最强,随后逐渐降低,但分蘖期和孕穗期差异不大。这说明,与分蘖期和孕穗期相比,五叶期水稻与根际细菌互作更为频繁。这就意味着,在下一步研究轮作制状态下水稻与根际微生物互作关系时选择五叶期更为合适。
本研究仅采用牛肉膏蛋白胨单一培养基分别对水稻根际细菌进行分离研究,初步揭示了陕南油菜-水稻轮作制下,水稻根际特有生境中微生物种群的组成特征,但仍然存在一定的局限性。有必要采用不依赖纯培的分子生物学方法,以获取更全面的水稻根际微生物多样性信息。
参考文献:
[1] ASEMOLOYE M D, AHMAD R, JONATHAN S G.Synergistic action of rhizospheric fungi with Megathyrsus maximus root speeds up hydrocarbon degradation kinetics in oil polluted soil[J]. Chemosphere, 2017, 187:1-10.
[2] 段红平,张乃明,李进学,等. 高产水稻根基微生物类群数量初探[J]. 中国农学通报,2007,23(2):285-289.
[3] ROSIER A, BISHNOI U, LAKSHMANAN V, et al. A perspective on inter-kingdom signaling in plant-beneficial microbe interactions[J]. Plant Mol Biol, 2016, 90(6):537-548.
[4] LAKSHMANAN V, SELVARAJ G, BAIS H P.Functional soil microbiome: belowground solutions to an aboveground problem[J]. Plant Physiol, 2014, 166(2):689-700.
[5] 胡桂萍. 水稻内生菌及其根系土壤微生物群落多样性的研究[D]. 福州:福建农林大学,2010.
[6] 刘 波,胡桂萍,郑雪芳,等. 利用磷脂脂肪酸(PLFAs)生物标记法分析水稻根际土壤微生物多样性[J]. 中国水稻科学,2010,24(3):278-288.
[7] 张仕颖,夏运生,肖 炜,等. 丁草胺污染对高产水稻土微生物区系的影响[J]. 生态环境学报,2014,23(4):679-684.
[8] 林文雄. 化感水稻抑草作用的根际生物学特性与研究展望[J]. 作物学报,2013,39(6):951-960.
[9] KUMAR A S, BAIS H P.Wired to the roots: impact of root-beneficial microbe interactions on abovegroundplant physiology and protection[J]. Plant Signal Behav, 2012, 7(12):1598-1604.
[10] LAKSHMANAN V, RAY P, CRAVEN KD.Toward a resilient, functional microbiome: drought tolerance-alleviating microbes for sustainable agriculture[J]. Methods Mol Biol, 2017, 1631:69-84.
[11] 马 欣,吴绍洪,戴尔阜,等. 气候变化对我国水稻主产区水资源的影响[J]. 自然资源学报,2011,26(6):1052-1063.
[12] YU L, ZHU J, HUANG Q, et al. Application of a rotation system to oilseed rape and rice fields in Cd-contaminated agricultural land to ensure food safety[J]. Ecotoxicol Environ Saf, 2014,108:287-293.
[13] 汤 洁,梁 爽,张 豪,等. 吉林省西部盐水田水稻生长不同时期土壤有机碳和碱解氮的动态变化特征[J]. 中国科技论文,2015,10(9):1053-1057.
[14] 朱海平,李贵勇,夏琼梅,等. 不同时期干旱胁迫对水稻产量及生长特性影响[J].中国稻米,2017,23(4):135-138.
[15] 张选明,冯志峰,李 勤,等. 陕西汉中优质稻生产现状及思考[J].陕西农业科学,2010, 56(3):126-128.
[16] 乔 帅,王梦姣,邓百万,等. 轮作区水稻根际土壤钙镁离子含量、含水量和酸碱度变化趋势[J].江苏农业科学,2017,45(5):284-288.
[17] FUJII Y, AKIHIRO F, SYUNTARO H. Rhizosphere soil method: a new bioassay to evaluate allelopathy in the field [C]//TUPPER G, WILKES S, FLYNN H, et al. 3rd Australian New Zealand Soils Conference: Novel Approaches. Australia: Charles Sturt University Press, 2004: 490-492.
[18] 岳 辉,李志真,钟炳林. 水土流失区芒箕生长与土壤微生物区系研究[J].林业科技,2009,34(4):22-26.
[19] TAMURA K,PETERSON D,PETERSON N,et al. MEGA5:molecular evolutionar y genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J]. Molecular Biology & Evolution,2011,28(10):2731-2739.
[20] 黄柳琴. 我国部分地球关键带中氨氧化菌群分布及其对环境的响应[D]. 北京:中国地质大学,2015.
[21] 厉桂香,马克明.土壤微生物多样性海拔格局研究进展[J]. 生态学报,2018,38(5):1521-1529.
[22] 杨瑞红,赵成义,王新军,等.梭梭和怪柳土壤微生物多样性初步分析[J]. 土壤,2016,48(6):1120-1130.
[23] LIU W Y, JIANG L L, GUO C J, et al.Terribacillusaidingensissp. nov., a moderately halophilic bacterium[J].International Journal of Systematic and Evolutionary Microbiology, 2010, 60:2940-2945.
