基于深度学习的乳腺癌病理图像自动分类
2018-06-26何雪英韩忠义魏本征
何雪英,韩忠义,魏本征
山东中医药大学 理工学院,济南 250355
1 引言
癌症是威胁人类健康和生命的主要杀手之一,其中乳腺癌是女性中最常见的癌症,据世界卫生组织(WHO)下属的国际癌症研究中心(IARC)2012年公布的数据显示[1],乳腺癌是造成女性死亡的第二大病症,且其发病率呈逐年上升和年轻化的趋势。临床上,相对于X-ray、钼靶、核磁共振等图像,病理图像是医生最后确诊乳腺癌的重要标准,而对病理图像进行准确地分类是医生制订最佳治疗方案的重要依据。目前基于人工的病理图像分类,不仅耗时、费力,而且诊断结果容易受到诸多主观人为因素的影响,借助于计算机辅助诊断(Computer-Aided Diagnosis,CAD),自动将病理图像进行良、恶性分类,不仅可以提高诊断效率,还能为医生提供更加客观、准确的诊断结果,因而具有重要的临床应用价值。
乳腺癌病理图像自动分类是一项非常有挑战性的工作。一是由于病理图像自身的特点:图像之间细微的差异性、细胞重叠现象、颜色分布不均匀等,给图像分类工作带来了很大的困难。二是由于缺乏大型公开的、已标记的数据集,给算法研究带来一定的困难。尽管如此,在乳腺癌病理图像的自动分类方面,学者们已经进行了很多研究,并取得了一系列重要的研究进展。主要集中在以下两个方面:
(1)基于人工特征提取和传统机器学习算法的乳腺癌病理图像分类。Kowal等人[2]采用不同的细胞核分割算法,在500幅乳腺癌病理图像上的识别率为96%~100%。Zhang等人[3]提出了一种基于人工设计特征的单类核主成分分析方法,对361幅乳腺癌病理图像分类,识别率为92%。基于颜色空间下4个形状特征和138个纹理特征,Wang等人[4]使用支持向量机算法对68幅乳腺癌病理图像的分类准确率为96.19%。Spanhol等人[5]公开了乳腺癌病理图像数据集BreaKHis,基于该数据集,采用了局部二值模式(LBP)、灰度共生矩阵(GLCM)等6种特征描述子和支持向量机、随机森林等不同的分类算法进行分类,准确率达到80%~85%。值得注意的是,上述分类算法大都是在小型数据集上开展,算法之间缺乏统一的对比标准,准确率之间不具有可比性。更重要的是,这些算法采用基于人工的特征提取方法,不仅需要专业领域知识,耗费大量的时间和精力完成,关键是要提取出有区分性的高质量特征也往往存在一定的困难。严重制约了传统机器学习算法在乳腺癌病理图像分类中的应用。
(2)基于深度学习的乳腺癌病理图像分类。一方面,深度学习能够自动从数据中学习特征,避免了传统算法中人工设计、提取特征的复杂性和局限性,另一方面,卷积神经网络(Convolutional Neural Networks,CNN)在自然语言处理、物体识别、图像分类识别等领域获得了广泛应用[6-7],为CNN在乳腺癌病理图像中的应用奠定基础[8-9]。在BreaKHis数据集[5]上,Spanhol等人[10]使用AlexNet网络,采用不同的融合策略分类,其识别率比传统机器学习算法高出6%。Bayramoglu等人[11]同样在BreaKHis数据集上,采用基于放大倍数独立的深度学习方法分类,其识别率约83%。然而,这些方法的识别率都有待进一步提高,以充分发挥CAD的作用,更好地满足临床应用。
针对上述问题,本文采用一种更深、更复杂的深度学习模型,来避免人工提取特征的复杂性和局限性,实现乳腺癌病理图像的自动分类;同时采用先进的数据增强方法和迁移——微调学习来防止深度学习算法在小样本集上训练易出现的过拟合,提高图像的识别率,以适应高标准的临床需求。
2 方法
深度学习可看作是多层的人工神经网络[12],通过构建含有多个隐层的神经网络模型,将低层特征通过逐层非线性特征组合变换,形成更加抽象的高层特征表达,以发现数据的分布式特征表示[13]。卷积神经网络作为最常使用的深度学习模型之一,以2D或3D图像直接作为网络的输入,避免了传统机器学习算法中复杂的特征提取过程,相比全连接的神经网络,其局部连接、权值共享和下采样的特性减少了网络参数的数量,降低了计算复杂度,同时对图像的平移、缩放、旋转等变化具有高度不变性。图1显示了深度卷积神经网络的特征学习过程,模型从原始图像仅能学习到一些边缘、纹理、颜色等低层特征,随着层数的加深,模型可以学习到更抽象的包含丰富语义信息的高层特征。

图1 卷积神经网络特征学习的可视化流程
对比目前常用的深度卷积神经网络模型LeNet[14]、AlexNet[15]、VGG[16]等,选用ILSVRC14竞赛中获得冠军的GoogLeNet[17]作为本文模型的基础架构,该模型共计22层,主要包括输入层、2个普通卷积层、9个Inception层和若干池化层。其中,每个Inception层包含6个卷积层和1个池化层。包括Inception结构在内的所有卷积都使用ReLU函数。模型通过Inception结构,在增加网络宽度的同时,减少了参数的数量,降低了计算复杂度;通过在不同深度处增加两个loss来保证梯度回传的消失。
2.1 深度卷积神经网络架构
2.1.1 网络架构设计
输入层:负责载入图像,产生一个输出向量作为卷积层的输入。本文模型加载整幅3通道乳腺癌病理图像,图像自动缩减为224×224大小,以适应卷积层。
卷积层:负责特征的学习。由一组特征map组成,同一个特征map共享一个卷积核,卷积核实际上是一组权重,也称作滤波器。一个可学习的卷积核与前一层若干个特征map作卷积,将对应元素累加后再加一个偏置,传给一个非线性激活函数,如ReLU函数、Sigmoid函数,得到一个特征map,即实现了一个特征的提取。计算公式如式(1)所示,多个不同的卷积核实现多个特征的提取。

其中,l表示层数,kij表示连接第l层的特征mapj与第l-1层的特征mapi的卷积核,Ml-1表示第l-1层选择的输入特征maps,∗表示卷积操作,b表示偏置,表示非线性激活函数。
本文模型中普通卷积层的卷积核大小分别设为7×7和3×3,步长(stride)为2,各Inception层卷积核的大小、stride值依模型具体情况设定,所有卷积都使用ReLU函数。
池化层:负责相邻区域特征的聚合统计。通过取区域最大值或平均值对卷积层的map做下采样,以减小下一层的输入大小,从而减少网络的参数个数和计算量。本文模型中除最后一个池化层采用Mean-pooling方式,感受野设为7×7,stride为1,其余池化层均采用Max-pooling方式,全部感受野设为3×3,stride为2。
此外,学习率为0.01,最大迭代次数为55 620。
2.1.2 Softmax分类器
本文采用Softmax分类器进行特征识别。Softmax分类器是logistic模型在多分类问题上的推广,将目标变量分为多类的算法。假设有N幅输入图像每幅图像的标记{yi∈{1 ,2,…,k},k≥2} ,共k类,本文中k=2。对于给定的测试图像xi,用假设函数估计出其属于每个类别 j的概率值 p(yi=j|xi)。则假设函数hθ(xi)为:
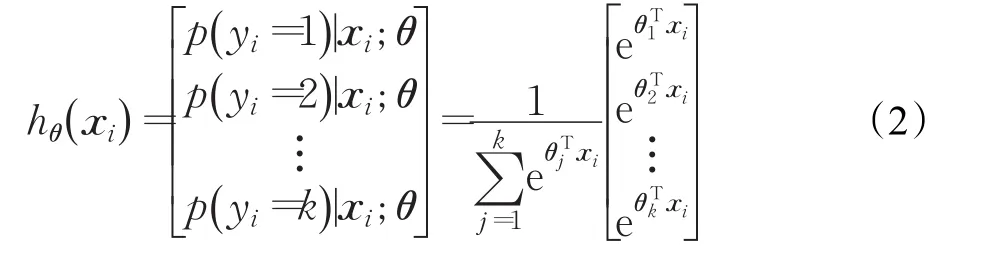
式中,代表对概率分布进行归一化,即全部概率之和为1。θ表示Softmax分类器的参数。
Softmax分类器的损失函数为:
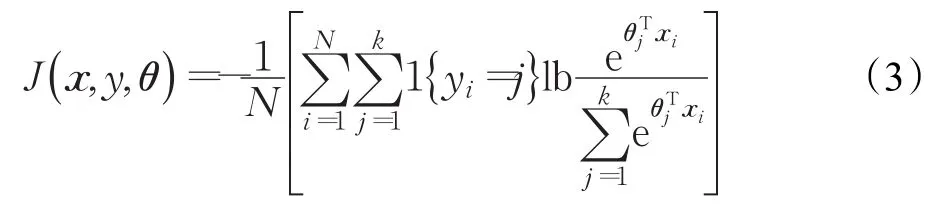
其中1(yi=j)为指示性函数。其取值规则为:1{值为真的表达式}=1,1值为假的表达式=0。最后通过随机梯度下降法最小化误差函数。
2.2 数据增强与迁移学习
缺乏大规模的训练数据,是将CNN应用于医学图像分类面临的主要挑战之一,因为CNN参数的训练需要大规模的有标记样本,才能防止过拟合,而通常要获取大规模的医学图像是困难、且代价昂贵的,尤其是经过专业医生标注的医学图像。本文在乳腺癌病理图像数据集BreaKHis[5]基础上,采用了两种解决方案:
一是数据增强[12]。采用仿射变换方法对BreaKHis数据集进行增强。主要通过将图像旋转90o/180o/270o、按照0.8的比例缩放、在水平和垂直方向作镜像,以及这些操作的组合操作,将数据集扩充了14倍。
二是迁移学习[18]。通过在现有的大规模数据集上进行预训练,得到模型的初始化参数,再迁移到目标数据集上进行微调训练。迁移学习可以在预训练数据集上学到一些有助于目标数据集分类的最基本特征,比如颜色、边缘特征等,从而提高了模型的分类性能。本文在ImageNet(包含120余万幅自然图像和1 000多种不同的类别)上对模型进行预训练,然后将模型参数迁移到BreaKHis上进行微调训练。
3 实验及结果分析
3.1 数据集
本文采用公开数据集BreaKHis[5],该数据集包含来自于82位患者的7 909幅已标注的乳腺癌病理图像,其中良性肿瘤图像2 480幅,恶性肿瘤图像5 429幅。每幅病理图像均采用4种不同的放大倍数(40X、100X、200X、400X),固定大小为700×460像素,模式为RGB三通道图像(24位颜色,每个通道8位)。表1是不同放大倍数的良、恶性肿瘤图像的具体分布情况[5]。
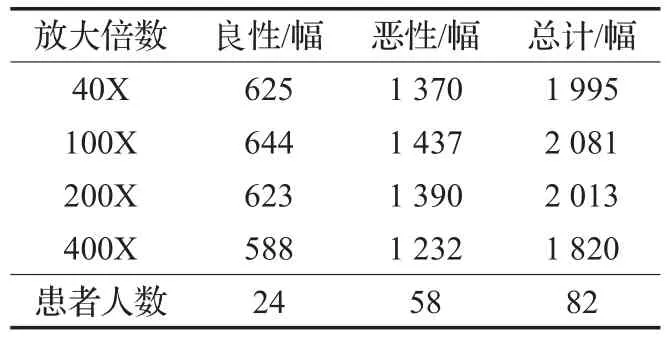
表1 不同放大倍数的良、恶性肿瘤图像的具体分布
增强后的数据集被扩充了14倍,约含11万余幅乳腺癌病理图像。为了充分保留图像轮廓和全局等特征信息,将整幅切片图像作为网络的输入。
3.2 评价标准
对于医学图像的分类,可以从两个方面评价模型的分类性能:患者级别和图像级别。从患者的层面计算识别率,令Nnp为每位患者病理图像的数量,Nrp为每位患者被正确分类的图像数量,Np为患者总数量,有:

则患者级别的识别率可表示为:

不考虑患者层面,而仅从图像级别来计算识别率时,令Nall代表验证集和测试集中病理图像的数量,Nr代表其中被正确分类的图像数量,则图像级别的识别率可表示为:

3.3 训练策略
增强前和增强后的数据集均被随机分成三部分:训练集50%、验证集25%和测试集25%。其中,训练集用于模型训练和参数学习;验证集用于优化模型,在训练过程中对模型进行测试,根据测试结果自动微调参数;测试集用于测试模型的识别和泛化能力。为保证模型对未知数据的泛化能力,数据集之间互不交叉。结果为10次随机分配数据集实验的平均值。
为验证迁移学习的有效性,采用两种训练策略:随机初始化训练和迁移——微调训练。为验证数据增强的有效性,将两种训练策略分别应用在增强前和增强后的数据集上。
3.4 实验工具和时间消耗
本文模型在Lenovo ThinkStation,Intel i7 CPU,NVIDIA Quadro K2200GPU上训练,使用Caffe框架[19],平均训练时间在增强前的数据集上约50 min,在增强后的数据集上约10 h16 min,单张测试时间平均约0.053 s。数据增强算法由Matlab2016a实现。
3.5 实验结果及分析
为检验本文方法的性能,选择与本文应用在同一数据集BreaKHis[5]上的方法进行对比,实验结果见表2。该表选用对应文献中的最优结果,分别从图像级别和患者级别两个方面进行了对比。表中,AlexNet是Spanhol等人在文献[10]中采用的网络模型,结果为采用不同融合策略后的最优分类结果;PFTAS+QDA/SVM/RF是文献[5]中采用的方法,先用PFTAS(Parameter-Free Threshold Adjacency Statistics)方法进行特征提取,然后分别使用三种机器学习方法QDA(Quadratic Discriminant Analysis)、SVM 和 RF(Random Forests)进行分类;Single-Task CNN是文献[11]中采用的基于放大倍数独立的CNN算法。由表2可知:无论在图像级别还是患者级别,本文方法的识别率均高于其他方法的识别率,并且,在不同放大倍数的图像上,分类结果几乎相同,约91%。表明本文方法能够提取更具区分性的特征,因而识别率更高,且具有较好的鲁棒性和泛化性。
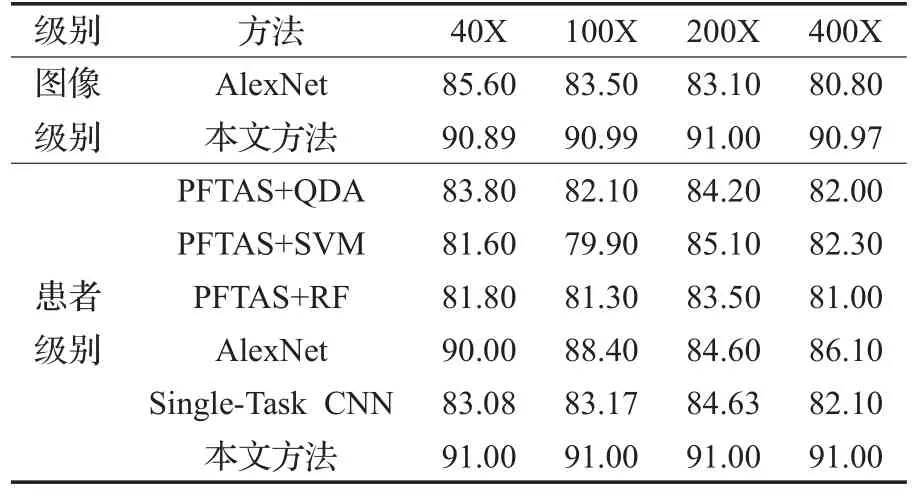
表2 不同放大倍数下患者级别和图像级别的识别率与其他方法的对比结果%
在增强前和增强后的数据集上分别进行随机初始化训练和迁移——微调训练,实验结果如图2所示,从图像级别和患者级别两个方面进行了对比。图2结果显示:图像级别和患者级别的结果差异甚微;数据增强后,无论是在随机初始化训练时还是迁移——微调训练时,识别率均得到了大幅提升(见图2中蓝色和红色曲线),证实了数据增强方法的有效性;采用迁移学习后,无论在元数据上还是增强后的数据上,识别率也均得到了提升(见图2中蓝色和绿色曲线),证实了迁移学习的有效性。表明本文方法有效避免了由于训练样本集不足而导致的过拟合问题,模型在ImageNet上预训练时学到了一些特征,而这些特征有助于BreaKHis上图像的分类,因而具有更高的识别率。
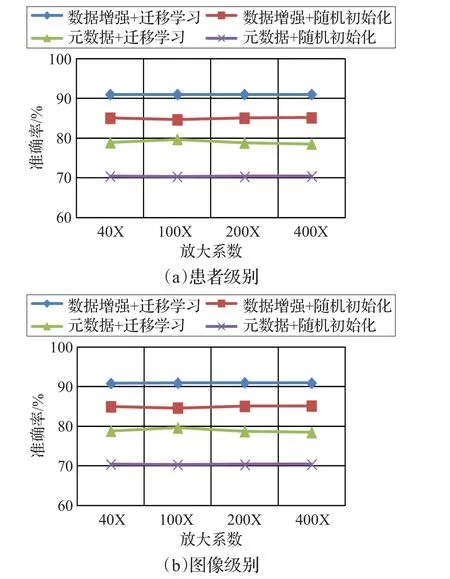
图2 数据增强前和增强后分别采用两种训练策略的分类结果
4 结束语
本文研究了利用深度学习方法实现乳腺癌病理图像的自动分类。采用的深度卷积神经网络模型具有更深、更复杂的结构,而参数更少,准确率更高,避免了人工提取特征的复杂性和局限性。采用的数据增强和迁移学习方法有效避免了深度学习算法在样本量不足时易出现的过拟合问题,实验证明本文方法提高了识别率,具有较好的鲁棒性和泛化性,在一定程度上满足了更高要求的临床需求。
[1]Stewart B,Wild C P.World cancer report 2014[M]//International Agency for Research on Cancer.[S.l.]:World Health Organization,2014.
[2]Kowal M,Filipczuk P,Obuchowicz A,et al.Computer-aided diagnosis of breast cancer based on fine needle biopsy microscopic images[J].Computers in Biology and Medicine,2013,43(10):1563-1572.
[3]Zhang Y,Zhang B,Coenen F,et al.One-class kernel subspace ensemble for medical image classification[J].EURASIP Journal on Advances in Signal Processing,2014,(1):17.
[4]Wang P,Hu X,Li Y,et al.Automatic cell nuclei segmentation and classification of breast cancer histopathology images[J].Signal Processing,2016,122:1-13.
[5]Spanhol F A,Oliveira L S,Petitjean C,et al.A dataset for breast cancer histopathological image classification[J].IEEE Transactions on Biomedical Engineering,2016,63(7):1455-1462.
[6]LeCun Y,Bengio Y,Hinton G.Deep learning[J].Nature,2015,521(7553):436-444.
[7]He K,Zhang X,Ren S,et al.Deep residual learning for image recognition[C]//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition,2016:770-778.
[8]Chen H,Dou Q,Wang X,et al.Mitosis detection in breast cancer histology images via deep cascaded networks[C]//ProceedingsoftheThirtieth AAAIConference on Artificial Intelligence,2016:1160-1166.
[9]Wang D,Khosla A,Gargeya R,et al.Deep learning for identifying metastatic breast cancer[J].arXiv preprint arXiv:1606.05718,2016.
[10]Spanhol F A,Oliveira L S,Petitjean C,et al.Breast cancer histopathological image classification using ConvolutionalNeuralNetworks[C]//2016 InternationalJoint Conference on Neural Networks(IJCNN),2016:2560-2567.
[11]Bayramoglu N,Kannala J,Heikkilä J.Deep learning for magnification independent breast cancer histopathology image classification[C]//International Conference on Pattern Recognition(ICPR),2016:2441-2446.
[12]Shen D,Wu G,Suk H I.Deep learning in medical image analysis[J].Annual Review of Biomedical Engineering,2017,19(1).
[13]Bengio Y,Delalleau O.On the expressive power of deep architectures[C]//InternationalConference on Algorithmic Learning Theory.Berlin Heidelberg:Springer,2011:18-36.
[14]LeCun Y,Bottou L,Bengio Y,et al.Gradient-based learning applied to document recognition[J].Proceedings of the IEEE,1998,86(11):2278-2324.
[15]Krizhevsky A,Sutskever I,Hinton G E.Imagenet classification with deep convolutional neural networks[C]//Advances in Neural Information Processing Systems,2012:1097-1105.
[16]Simonyan K,Zisserman A.Very deep convolutional networks for large-scale image recognition[J].arXiv preprint arXiv:1409.1556,2014.
[17]Szegedy C,Liu W,Jia Y,et al.Going deeper with convolutions[C]//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition,2015:1-9.
[18]Shin H C,Roth H R,Gao M,et al.Deep convolutional neural networks for computer-aided detection:CNN architectures,dataset characteristics and transfer learning[J].IEEE Transactions on Medical Imaging,2016,35(5):1285-1298.
[19]Jia Y,Shelhamer E,Donahue J,et al.Caffe:Convolutional architecture for fast feature embedding[C]//Proceedings of the 22nd ACM International Conference on Multimedia,2014:675-678.
