貂熊亚科线粒体基因组及系统发育研究进展
2018-06-26蔡莹莹
蔡莹莹 李 波,2*
(1.东北林业大学野生动物资源学院,哈尔滨,150040;2.国家林业局动植物检测中心,哈尔滨,150040)
貂类或貂属(Martes)动物在传统的分类上属于食肉目(Carnivora)鼬科(Mustelidae)貂亚科(Martinae)。它是一类中小型食肉动物,广泛分布于北半球的森林中。通常认为该属动物包括8个现存物种[1]。Anderson(1970)根据动物体型大小和裂齿上内叶小尖的形状将其细分为3个亚属(subgenera):渔貂亚属(Pekania),现存单一种渔貂(M.pennanti);黄喉貂亚属(Charronia),包括黄喉貂(M.flavigula,模式物种)和格氏貂(M.gwatkinsii);以及真貂亚属(Martes),包括石貂(M.foina,模式物种)、美洲貂(M.americana)、欧洲松貂(M.martes)、日本貂(M.melampus)和紫貂(M.zibellina)[1]。
貂熊(Gulo gulo)与貂属动物形态上差异显著,长期以来认为二者的亲缘关系较远,被单独列为一属——貂熊属(Gulo)。分子系统学研究表明貂熊与貂属动物同属一个单系类群,而且渔貂位于貂属-貂熊进化枝外面[2-6]。这证明了貂熊与貂属动物亲缘关系较近。由此有学者提出将渔貂亚属升格为独立的属[3,6-7]。另一个与貂属 -貂熊进化枝亲缘关系较近的是狐鼬属(Eira) 的狐鼬(E.barbara)[2-4,8-9]。多位学者建议将貂属-貂熊-渔貂-狐鼬进化枝列为一个亚 科, 称 为 貂 亚 科[3,5,9]或 貂 熊 亚 科 (Guloninae)[4,6,10-11]。在国际动物命名法规中貂熊亚科(Guloninae Gray,1825)出现的时间早于貂亚科(Martinae Wagner,1841)[10],因此更多的学者使用貂熊亚科来代表该进化枝。貂熊亚科动物的分类和地理分布详见表1。

表1 貂熊亚科动物的分类和地理分布Tab.1 Taxonomy and distribution of Guloninae species

续表1
1 貂熊亚科线粒体基因组
1.1 研究概况
动物线粒体基因组具有无组织特异性、呈母性遗传、快速率的碱基替换、高保守的基因等特点,目前已经被广泛应用于分子系统学和种群遗传学研究。随着第二代测序技术的成熟,动物线粒体基因组测序变得日益简便。目前已经发表的貂熊亚科的线粒体基因组涵盖现存10个貂熊亚科物种中的8个,不包括狐鼬和格氏貂(表2)。基因组序列长度为16288~16549 bp。线粒体基因组序列长度差异显著的物种为黄喉貂,2个测通的基因组大小分别为16467 bp和16549 bp。
另外,该亚科动物线粒体基因组的测序方法也存在较大差异。例如,日本貂线粒体基因组的测序采用的是长和精确的聚合酶链反应(long and accurate polymerase chain reaction,LA-PCR)结合产物直接测序的方法[12]。渔貂线粒体基因组测序采用的是LA-PCR结合多重大规模平型测序的方法(multiplexed massively parallel sequencing)[13]。欧洲松貂线粒体基因组测序采用的是LA-PCR结合Illumina高通量测序的方法(Illumina HiSeqTM2000 sequencing system)[7]。前者采用的引物步移(primer walking)的测序策略,后两者采用的鸟枪法(shotgun)的测序策略。相比较而言,鸟枪法比引物步移更有优势,因为遇到控制区(control region或D-Loop)中的重复序列区时引物步移法通常难以测通。但鸟枪法有时也可能产生缺口,为消除缺口需要重新设计引物扩增和测序该区域。
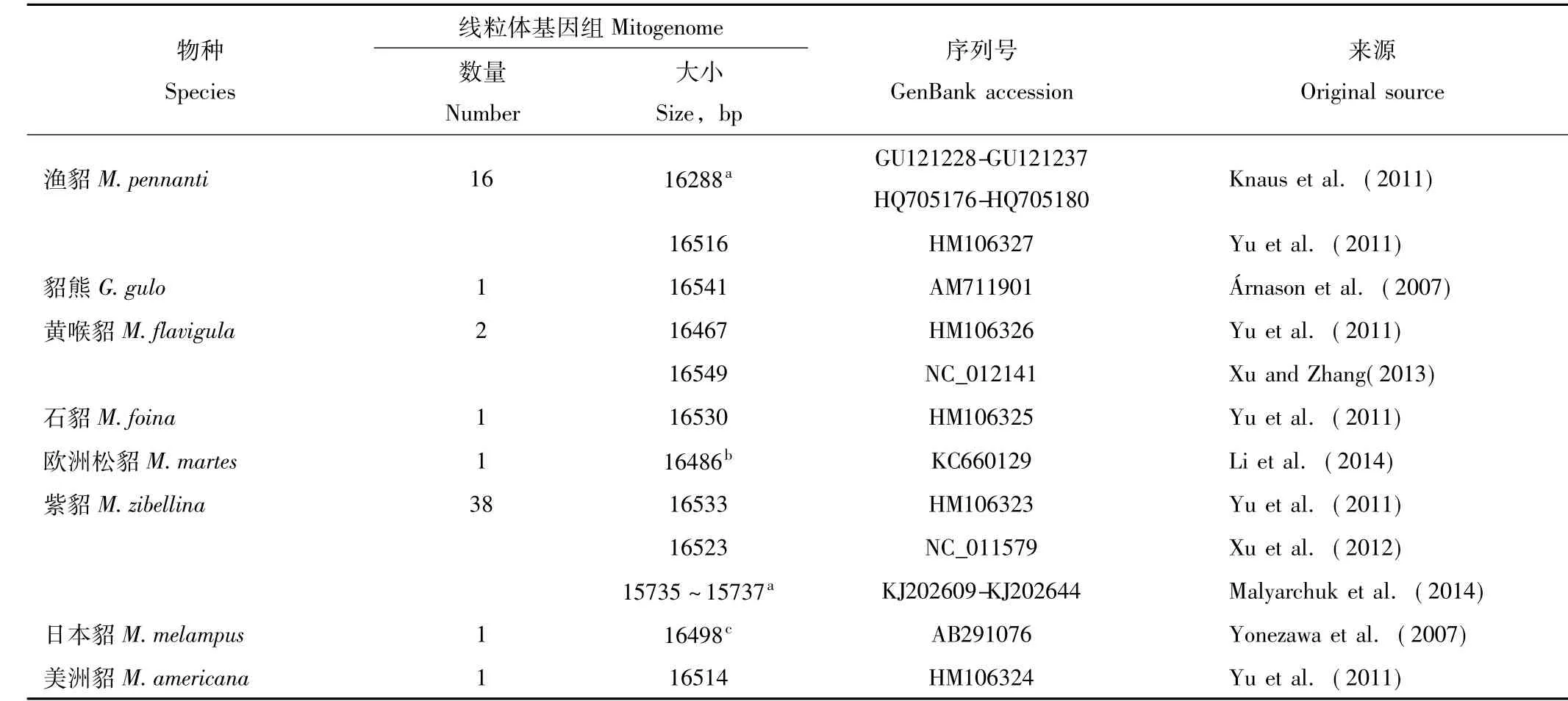
表2 已发表的貂熊亚科动物线粒体基因组Tab.2 The detail information of published mitogenomes among Guloninae species
1.2 结构特点
与其他脊椎动物的线粒体基因结构相似,貂熊亚科动物线粒体基因组也是由2个非编码区和37个基因构成。2个非编码区是控制区和L-链的复制起点(origin of L-strand replication,OL)。37个基因是2个rRNA基因(12S和16S),22个tRNA基因(tRNA-Phe、tRNA-Val、 tRNA-Leu、 tRNA-Ile、 tRNA-Gln、 tRNAMet、 tRNA-Trp、 tRNA-Ala、 tRNA-Asn、 tRNA-Cys、tRNA-Tyr、 tRNA-Ser、 tRNA-Asp、 tRNA-Lys、 tRNAGly、 tRNA-Arg、 tRNA-His、 tRNA-Ser、 tRNA-Leu、tRNA-Glu、tRNA-Thr、tRNA-Pro),以及13个蛋白质编码基因(COX1、COX2、COX3编码3个细胞色素氧化酶亚基,ATP6和ATP8编码2个ATP酶亚基,Cytochrome b(Cyt b)编码细胞色素b,ND1、ND2、ND3、ND4、ND4L、ND5、ND6编码7个NADH氧化还原酶亚基)。其中有1个蛋白质编码基因(ND6)和8个tRNA 基因 (tRNA-Gln、tRNA-Ala、tRNA-Asn、tRNACys、tRNA-Tyr、tRNA-Ser、tRNA-Glu、tRNA-Pro) 位于L-链,其余28个基因均位于H-链。貂熊亚科动物线粒体基因组碱基组成表现为A、T丰富,A+T含量比G+C含量高,碱基含量大小排列是A>C>T>G(表3)。
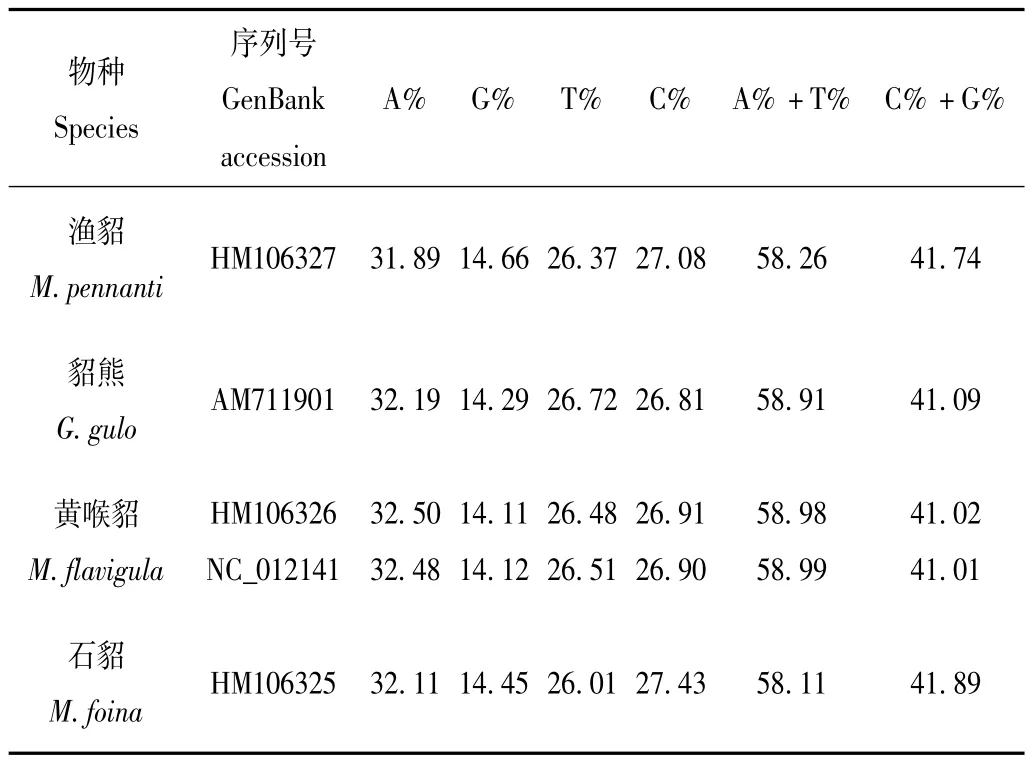
表3 貂熊亚科动物线粒体基因组碱基组成Tab.3 Base composition of mitogenomes among Guloninae species

续表3
貂熊亚科动物线粒体基因组上基因排列极为紧凑,除了D-Loop控制区外,几乎没有内含子序列。在编码区的37个基因之间,基因间隔区约占30 bp。并有多个基因间具有重叠现象,其中最多的是ATP6和ATP8间重叠43 bp,显著多于鸟类ATP6和ATP8间重叠10 bp。貂熊亚科动物线粒体基因组的13个蛋白质编码基因中,最长的是ND5(貂熊为1836 bp、在13543 bp后有6 bp插入序列,剩余物种均为1830 bp),最短的是ATP8(均为204 bp)。在起始密码子的使用上,已测序的8个物种的ND3和ND5基因均是以ATA为起始密码子,渔貂(HQ705180)和紫貂(HM106323)的ND2基因以ATC为起始密码子,剩余物种的ND2基因以ATT为起始密码子,剩余蛋白质编码基因均是以ATG为起始密码子。在终止密码子的使用上,已测序的8个物种的ND1均是以TA为终止密码子,ND2、COX3和ND4基因均是以T为终止密码子,Cyt b基因以AGA为终止密码子,剩余蛋白质编码基因均是以TAA为终止密码子。
貂熊亚科动物线粒体控制区位于氨基酸tRNA-Pro和tRNA-Phe之间,包含启动子、复制和转录调控序列,无结构基因。其序列长度是1021~1107 bp,通常可划分为3个结构域:延长终止序列区(extended termination associated sequence,ETAS)、中央保守区(central conserved domain)和保守序列区(conserved sequence block,CSB)[7,14]。后 者 可 以 进 一 步 细 分 为CSB-1、CSB-2和CSB-3,且在CSB-1与CSB-2之间存在不同重复数目的重复序列区(repetitive sequence,RS)。重复序列区的基本单元为AC,并插入若干个GT或AT。该亚科不同物种间RS重复次数差异显著,例如黄喉貂(NC_012141)控制区ACACGT重复了37次,紫貂(NC_011579)控制区AC重复了114次[14],欧洲松貂控制区AC重复了70次[7]。这也是不同物种线粒体基因组序列长度差别的主要来源。
2 貂熊亚科系统发育
2.1 起源
貂熊亚科动物在形态上变异较大、地理分布较广,具有复杂的起源和进化历史。Anderson认为最早的貂类化石是德国出土中新世早期(约18百万年前)的M.laevidens[15]。它的下颌骨左右各具4枚裂齿,上裂齿内叶还没有显著增大,下裂齿具不完全凹陷。由于其头骨特征与现存貂类差异明显,Sato等将它排除在貂类之外[16]。最早的貂熊亚科动物化石应是在北美发现、距今7.05~7.3百万年的Pekania occulta[17]。其他近缘物种的化石断定的年限也比它晚。例如,亚洲出土的上新世早期Eirictis pachygnatha化石距今4.5~5.3百万年[18]。最早的貂熊属物种化石是在尼泊尔发现的G.minor,距今3.1~3.6百万年[19]。
目前,公认最早的貂属化石是在波兰发现的M.wenzensis,距今3.3~4.0百万年[16]。其下颌具有4枚裂齿,上裂齿内叶增大,下颌第4枚裂齿具明显的小附尖,下臼齿表明平坦。这与松貂化石形态相似,但其体型与现存的渔貂相近[20]。此外,在德国发现的貂属动物M.vetus Kretzoi(1942)的化石距今1.75~2.0百万年[7,21]。其体型与欧洲松貂相近,又具有石貂(裂齿和臼齿的大小,以及颌骨宽度)和欧洲松貂(下裂齿具3个小尖,裂齿长度与其宽度相同,下颌骨较宽)二者共有的形态特征。结合其地理分部信息,Anderson推测M.vetus很可能是欧洲松貂和石貂的共同祖先,但并不认为它是起源于M.wenzensis[15]。在真貂亚属中,北美发现最早的美洲貂化石距今0.6~1.0百万年[22]。而最早的欧洲松貂化石距今0.18~0.24百万年,该亚属其他物种化石年限都比较晚,包括紫貂的化石。总之,根据现有发现的貂熊亚科动物化石,我们可以初步描述其起源进化的轮廓。但一些关键化石具体年限间存在断层,这也限制了进一步揭示其更为清晰的进化历史和系统发育关系。
2.2 系统发育关系
包括貂熊亚科在内的鼬科动物是渐新世(oligocene)以后物种快速进化辐射和发生近期物种形成事件的典型代表[23-24]。近20年来,貂熊亚科内部的系
统发育关系受到很多学者的关注[2-7,10,16,23,25-26]。在一些分析中貂熊亚科内的各属的关系已经非常明确。Anderson根据形态学分析将貂属划分为3个亚属分别为渔貂亚属、黄喉貂亚属、真貂亚属[1]。随着分子遗传学研究的深入,单纯的形态学的分类方法受到质疑,其中不乏学者应用分子遗传学的手段对貂熊亚科动物进行重新分类和系统发育分析。基于mtDNA或nDNA部分序列的分析认为貂熊与貂属(真貂亚属、黄喉貂亚属)亲缘关系非常近并聚为一个分支形成姐妹群,而渔貂位于貂属 -貂熊进化枝外面[2-5,27]。由此部分学者建议将渔貂从貂属中划分出来,单独列为一个属。关于貂熊属的分类地位,部分学者将貂熊属与黄喉貂亚属化为一支[4,6,16,25,27-28],还有学者将貂熊与真貂亚属化为一支[9,16,27]。狐鼬属的系统发育研究结果为狐鼬属与渔貂属首先聚为一支,并与貂熊与貂属分支形成姐妹群[2-3]。
关于真貂亚属内物种的进化关系,早期的形态学研究表明松貂、紫貂、日本貂、美洲貂的亲缘关系较近,而与石貂的亲缘关系较远[1,29-30],这一结果得到了 nDNA-mtDNA 序列分析的高度支持[3-4,6]。然而松貂、紫貂、日本貂和美洲貂的关系在形态分类和分子研究方面一直存在很大争议。一些来自单个mtDNA基因(Cyt b或ND)产生的拓扑结构为紫貂与松貂首先聚为一支,再与日本貂聚合,最后与美洲貂形成并系群[3-4,6,16,24-26,31-32]。另外一些基于线粒体相同序列或者更小序列的数据[16,26,31,33-36]或者来自 nDNA <5.5 kb的数据[9,10,27]分析则产生不同的拓扑结构。利用 nDNA和mtDNA>8 kb的数据对其亚属内物种进化关系的研究采用不同的分析方法也会产生不同拓扑结构[3 -4,6]。
2.3 线粒体基因组的应用
利用部分mtDNA、nDNA或者nDNA-mtDNA序列对貂熊亚科的系统发育研究得到了一些共同的结果,例如发现貂熊属与貂属的亲缘关系较近,且渔貂属位于貂熊属与貂属进化支之外,但对真貂亚属各物种间亲缘关系的研究结果存在不同程度的不一致。由于数据的不完整性可能导致系统发育分析中的随机错误[4]。针对貂熊亚科内部分物种间系统发育存在的争议,有学者利用线粒体全基因组对其进行了全面的分析[7]。结果表明基于线粒体全基因组、不包含控制区的线粒体基因组和线粒体13个蛋白编码基因3种序列数据集的多种分析方法构建的系统发育关系获得了一致的拓扑结构且每个节点都具有高度的支持率。综合的结果进一步证实渔貂属位于貂熊属与貂属进化支的外侧,貂属内的真貂亚属与黄喉貂亚属分别作为单系存在,真貂亚属内各物种的亲缘关系是:石貂首先分化出来;紫貂与松貂首先聚为一支,再与日本貂聚合,最后与美洲貂形成并系群(图1)。较之前基于部分mtDNA或nDNA-mtDNA关于真貂亚属内物种系统发育的研究,基于线粒体全基因组的研究具有更高的可信度。
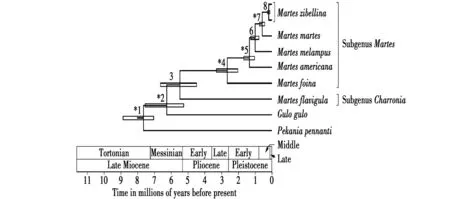
图1 貂熊亚科系统发育与分化[7]Fig.1 Dated phylogeny of Martes,Gulo,and Pekania
貂熊亚科动物具有较为复杂的起源及进化历史,越来越多的学者结合化石和分子生物学方法对貂熊亚科内部物种的分化时间进行研究,在利用线粒体全基因组进行对其进行分化时间估计之前,大部分学者利用部分mtDNA或nDNA-mtDNA序列对其进行了研究,如表4。利用新的化石记录和线粒体基因组对貂熊亚科内各物种的分化时间估计为:貂熊属与渔貂属的平均分化时间为7.65百万年,貂熊属与貂属的平均分化时间为6.28百万年,黄喉貂亚属与真貂亚属的分化时间为5.48百万年,石貂与其他真貂亚属物种(美洲貂、日本貂、松貂、紫貂)的分化时间为2.67百万年,美洲貂与其他真貂亚属物种(日本貂、松貂、紫貂)的分化时间为1.35百万年,日本貂与松貂和紫貂的分化时间为1.01百万年,松貂与紫貂的分化时间约为0.58百万年。基于线粒体全基因组分析的渔貂属、貂熊属、黄喉貂亚属、真貂亚属的分歧时间略早于其他基于nDNA、mtDNA和nDNA-mtDNA的分化时间估计,而关于美洲貂、日本貂、紫貂、松貂支系间估计的分化时间略晚于其他分析,并且其95%置信区间的范围与其他基于nDNA和nDNA-mtDNA的分析范围较为接近,甚至范围更窄,因此基于线粒体基因组对于貂熊亚科的分化时间估计更为可靠。
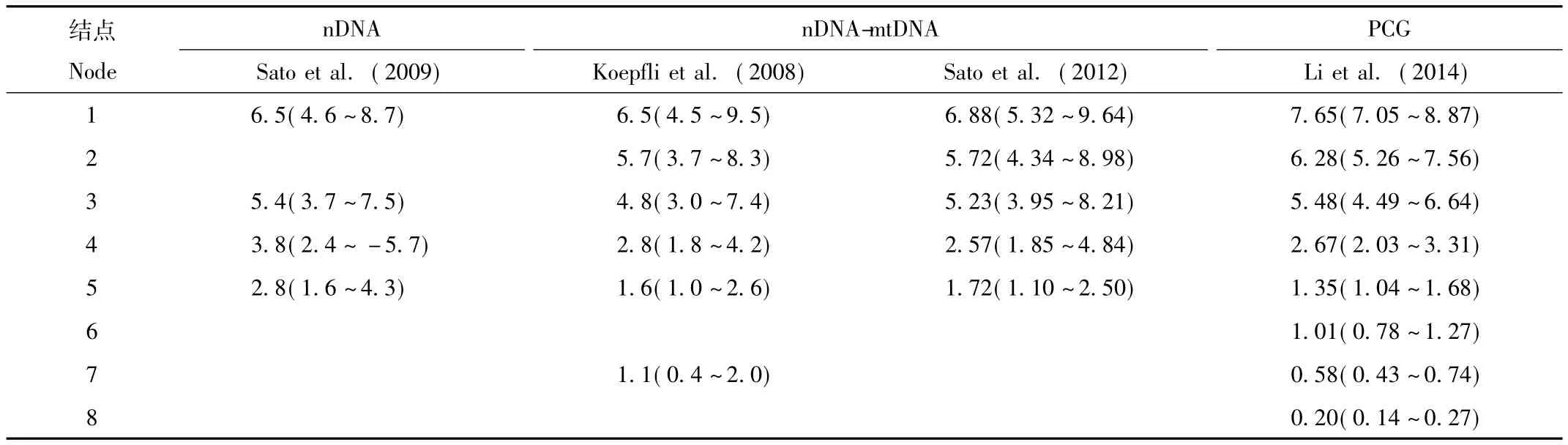
表4 基于多基因序列的貂属、貂熊属、渔貂属的分化时间估计[3,6,7,10]Tab.4 Previous estimates of Martes,Gulo,and Pekania divergence times from multigene sequence data
3 展望
线粒体基因组对貂熊亚科的系统发育关系重建起到了重要作用,但部分该亚科物种缺乏相应的数据,例如狐鼬和格氏貂的线粒体基因组尚未见报道,某种程度上阻碍了全面揭示该亚科的系统发育关系。另外,随着测序技术的成熟,分子生物学已经发展到了宏基因组时代。有学者指出宏基因组在构建物种系统发育的应用效果比线粒体基因组更为合适[37]。目前,貂熊亚科中已经完成宏基因组测序的是紫貂(张洪海,2017,个人通讯,未发表)。相信不久的将来,随着其他貂熊亚科物种宏基因组测序的完成,貂熊亚科物种间的系统发育关系及其进化历史最终将得到全面解决。
[1] Anderson E.Quaternary evolution of the genus Martes(Carnivora,Mustelidae) [J].Acta Zoologica Fennica,1970,130:127 -132.
[2] Koepfli K P,Wayne R K.Type I STS markers are more informative than cytochrome b in phylogenetic reconstruction of the Mustelidae(Mammalia:Carnivora) [J].Systematic Biology,2003,52(5):571-593.
[3] Koepfli K P,Deere K A,Slater G J,et al.Multigene phylogeny of the Mustelidae:resolving relationships,tempo and biogeographic history of a mammalian adaptive radiation [J].BMC Biology,2008,6(1):10.
[4] Wolsan M,Sato J J.Effects of data incompleteness on the relative performance of parsimony and Bayesian approaches in a supermatrix phylogenetic reconstruction of Mustelidae and Procyonidae(Carnivora)[J].Cladistics,2010,26(2):168 -194.
[5] Yu Li,Peng Dan,Liu Jiang,et al.On the phylogeny of Mustelidae subfamilies:analysis of seventeen nuclear non-coding loci and mitochondrial complete genomes[J].BMC Evolutionary Biology,2011,11(1):92.
[6] Sato J J,Wolsan M,Prevosti F J,et al.Evolutionary and biogeographic history of weasel-like carnivorans(Musteloidea) [J].Molecular Phylogenetics and Evolution,2012,63(3):745-757.
[7] Li Bo,Wolsan M,Wu Dan,et al.Mitochondrial genomes reveal the pattern and timing of marten(Martes),wolverine(Gulo),and fisher(Pekania)diversification[J].Molecular Phylogenetics and Evolution,2014,80:156-164.
[8] Flynn J J,Finarelli J A,Zehr S,et al.Molecular phylogeny of the Carnivora(Mammalia):assessing the impact of increased sampling on resolving enigmatic relationships [J].Systematic Biology,2005,54(2):317-337.
[9] Fulton T L,Strobeck C.Molecular phylogeny of the Arctoidea(Carnivora):effect of missing data on supertree and supermatrix analyses of multiple gene data sets[J].Molecular Phylogenetics and Evolution,2006,41(1):165-181.
[10] Sato J J,Wolsan M,Minami S,et al.Deciphering and dating the red panda's ancestry and early adaptive radiation of Musteloidea [J].Molecular Phylogenetics and Evolution,2009,53(3):907-922.
[11] Wolsan M.Family Mustelidae:weasels,polecats,otters,Ratel and allies[J].Mammals of Africa,2013,5:82-84.
[12] Yonezawa T,Nikaido M,Kohno N,et al.Molecular phylogenetic study on the origin and evolution of Mustelidae [J].Gene,2007,396(1):1-12.
[13] Knaus B J,Cronn R,Liston A,et al.Mitochondrial genome sequences illuminate maternal lineages of conservation concern in a rare carnivore[J].BMC Ecology,2011,11(1):10.
[14] Xu Chunzhu,Zhang Honghai,Ma Jianzhang,et al.The complete mitochondrial genome of sable,Martes zibellina [J].Mitochondrial DNA,2012,23(3):167-169.
[15] Anderson E.Evolution,prehistoric distribution,and systematics of Martes[M] ∥Buskirk S W,Harestad A S,Raphael M G,et al.Martens,sables,and fishers:biology and conservation.Cornell U-niversity Press,Ithaca,NY,1994:13 -25.
[16] Sato J J,Hosoda T,Wolsan M,et al.Phylogenetic relationships and divergence times among Mustelids(Mammalia:Carnivora)based on nucleotide sequences of the nuclear interphotoreceptor retinoid binding protein and mitochondrial cytochrome b genes[J].Zoological Science,2003,20(2):243 -264.
[17] Samuels J X,Cavin J.The earliest known fisher(Mustelidae),a new species from the rattlesnake formation of Oregon[J].Journal of Vertebrate Paleontology,2013,33(2):448 -454.
[18] Wang Xingming,Tseng Z J,Takeuchi G T,et al.Zoogeography,molecular divergence,and the fossil record-the case of an extinct fisher,Pekania palaeosinensis(Mustelidae,Mammalia),from the late Miocene Baogeda Ula Formation,Nei Mongol[J].Vertebrata Palasiatica,2012,50(3):293-307.
[19] Wolsan M,Sotnikova M.Systematics,evolution,and biogeography of the Pliocene stem meline badger Ferinestrix(Carnivora:Mustelidae) [J].Zoological Journal of the Linnean Society,2013,167(1):208-226.
[20] Kurtén,Bjorn.Pleistocene mammals of Europe [J].Journal of Mammalogy,1968,50(2):387.
[21] Wolsan M.Phylogeny and classification of early European Mustelida(Mammalia:Carnivora) [J].Acta Theriologica,1993,38(4):345-384.
[22] Hughes S S.Synthesis of Martes evolutionary history[M] ∥Aubry K B,Zielinski W J,Raphael M G,et al.Biology and conservation of martens,sables,and fishers:a new synthesis.Ithaca,NY:Cornell University Press,2012:3-22.
[23] Koepfli K P,Wayne R K.Phylogenetic relationships of otters(Carnivora:Mustelidae)based on mitochondrial cytochrome b sequences[J].Journal of Zoology,1998,246(4):401 -416.
[24] Marmi J,López-Giráldez J F,Domingo-Roura X.Phylogeny,evolutionary history and taxonomy of the Mustelidae based on sequences of the cytochrome b gene and a complex repetitive flanking region[J].Zoologica Scripta,2004,33(6):481 -499.
[25] Hosoda T,Suzuki H,Harada M,et al.Evolutionary trends of the mitochondrial lineage differentiation in species of genera Martes and Mustela[J].Genes& Genetic Systems,2000,75(5):259 -267.
[26] Stone K D,Cook J A.Molecular evolution of Holarctic martens(genus Martes,Mammalia:Carnivora:Mustelidae) [J].Molecular Phylogenetics and Evolution,2002,24(2):169-179.
[27] Sato J J,Wolsan M,Suzuki H,et al.Evidence from nuclear DNA sequences sheds light on the phylogenetic relationships of Pinnipedia:single origin with affinity to Musteloidea [J].Zoological Science,2006,23(2):125-146.
[28] Zhang Honghai,Xu Chunzhu,Ma Jiangzhang.Structure of the mtDNA control region and phylogeny of the Mustelidae species [J].Acta Ecologica Sinica,2009,29:3585 -3592.
[29] Wolsan M.Zmiennosc uzebienia w rodzaju Martes(Mustelidae,Carnivora) [D].Cracow:Jagiellonian University,1987.
[30] Wolsan M.Dental polymorphism in the genus Martes(Carnivora:Mustelidae)and its evolutionary significance.Acta Theriologica,1989,34(40):545-593.
[31] Carr S M,Hicks S A.Are there two species of marten in North A-merica genetic and evolutionary relationships within Martes[M] ∥Proulx G,Bryant H N,Woodard P M.Martes:taxonomy,ecology,techniques,and management.Provincial Museum of Alberta,Edmonton,1997:15-28.
[32] Sato J J,Hosoda T,Kryukov A P,et al.Genetic diversity of the sable(Martes zibellina,Mustelidae)in Russian Far East and Hokkaido inferred from mitochondrial NADH dehydrogenase subunit 2 gene sequences[J].Mammal Study,2011,36(4):209-222.
[33] Rozhnov V V,Meshcersky I G,Kholodova M V.Molecular genetic study of marbled polecat(Vormela peregusna,Carnivora:Mustelidae) [J].Doklady Biological Sciences,2006,407(1):169 -172.
[34] Agnarsson I,Kuntner M,May-Collado L J.Dogs,cats,and kin:a molecular species-level phylogeny of Carnivora[J].Molecular Phylogenetics and Evolution,2010,54(3):726-745.
[35] Xu Chunzhu,Zhang Honghai,Ma Jianzhang.Organization of the complete mitochondrial genome and its evolution in sable[J].Journal of Beijing Forestry University,2010,32(1):82-88.
[36] Hosoda T,Sato J J,Lin L K,et al.Phylogenetic history of mustelid fauna in Taiwan inferred from mitochondrial genetic loci[J].Canadian Journal of Zoology,2011,89(6):559-569.
[37] Springer M S,DeBry R W,Douady C,et al.Mitochondrial versus nuclear gene sequences in deep-level Mammalian phylogeny reconstruction [J].Molecular Biology and Evolution,2001,18(2):132-143.
