长白山北部林区野猪局域种群的遗传多样性研究
2018-06-26葛宝庆胡素贤孙铁铎张崇颖姜广顺
葛宝庆 胡素贤 孙铁铎 张崇颖 姜广顺
(1.国家林业局猫科动物研究中心;东北林业大学野生动物资源学院,哈尔滨,150040;2.东北林业大学图书馆,哈尔滨,150040;3.吉林省林业勘察设计研究院,长春,130021;4.吉林省天桥岭林业局,汪清,133204)
野猪(Sus scrofa),隶属于哺乳纲、偶蹄目(Artiodactyla)、猪科(Suidae)、猪属动物,共有8属22种[1],其分布范围广泛,以欧洲、亚洲与非洲分布最广,在半干旱气候至热带雨林、温带林地、草原等都有其分布,但是在极为干旱、海拔极高与寒冷的地区难以发现其踪迹。除戈壁沙漠与青藏高原外,在中国境内均有野猪分布,野猪主要集中分布在东北三省、云贵地区、福建、广东地区。其中分布在中国东北的是乌苏里野猪(S.s.ussuricus)。近年来,由于栖息地质量下降与人为猎杀,野猪种群数量在逐渐减少[2]。
对野猪种群数量和种群特征的监测是野猪种群资源管理的重要内容。传统的野生动物种群监测和管理技术,包括直接观察、个体计数、足迹识别、测量跟踪和记录猎物[3]。但这些传统技术几乎是不可能为精确管理方法,如性别比例、个体间关系、遗传多样性、种群分化、亚种群间基因流和近亲繁殖或远亲繁殖水平,提供所需的关键信息。从而,野生动物的保护研究逐渐从宏观的研究手段过渡到微观手段,来揭示野生动物种群资源动态的特征。
遗传多样性能够反应遗传变异的水平,是衡量种群生存、进化以及对环境条件适应的关键指标。栖息地质量的下降以及强烈的人为活动会在分子、细胞与个体水平上对动物产生影响,造成动物遗传变异水平下降。遗传多样性水平的下降将会削弱动物对环境的适应能力,导致动物扩散迁移能力降低,近交程度增加,动物个体生存能力下降。目前使用分子标记技术研究野猪的种群遗传学还较少。如兰宏等利用线粒体DNA序列分析了西南地区家猪和野猪遗传多样性[4],Vernesi等使用微卫星标记分析了在种群数量减少和重引入对野猪的遗传影响[5],李崇奇采用线粒体变异探讨了野猪系统地理学和家猪起源[6],张晨岭使用微卫星标记探讨了中国大陆地区野猪的遗传多样性及遗传结构,表明了中国大陆地区的野猪较国外野猪有更高的遗传多样性[7]。同样,霍金龙等使用76个微卫星标记对云南南部地区野猪群体的遗传多样性进行评估,也表明该野猪群体遗传多样性较丰富[8]。韩春梅等使用mtDNA控制区序列评估了新疆野猪遗传多样性,阐明了新疆野猪与亚洲野猪和国内地方猪种之间的遗传关系较近[9]。然而,关于东北地区野猪种群遗传多样性的研究较少。本研究通过采集长白山北部5个林区的野猪粪便样本,结合微卫星标记技术,评估长白山北部区域野猪种群的遗传多样性水平和近交程度,为制定野猪种群保护和利用策略提供遗传基础。
1 材料与方法
1.1 样品采集及保存
2014~2016年期间,在长白山北部的5个采样地点采用系统布样的设计方法,分别在汪清自然保护区共布设19条样线,张广才岭南部林区共布设25条样线,珲春自然保护区共布设18条样线,天桥岭自然保护区共布设20条样线,穆棱林区中共布设19条长度大于5 km的样线,寻找野猪足迹。
在样线上发现新鲜的野猪足迹后,沿着足迹链跟踪,保证跟踪距离达到3 km以上。在跟踪足迹的过程中,为避免重复取样,在一条野猪足迹链上只采取一堆粪便样本。用一次性PE手套将粪便样品装入封口袋中,并记录采样时间、地点、足迹链编号、经纬度及采集人。每次收集粪便时要更换PE手套和收集袋,避免交叉污染。由于冬天研究地区的温度大多低于-20℃,所以收集到的粪便样品可直接放在室外阴暗处保存。粪便样品从野外采集结束后,将粪便样品送回实验室,在-20℃的低温冰箱中冷冻保存。
在5个地点共收集疑似野猪粪便样品140份,其中汪清(WQ)共计36份,珲春(HC)共计35份,张广才岭南(ZGCLN)共计23份,天桥岭(TQL)共计27份,穆棱(ML)共计19份。
1.2 DNA提取
DNA Stool Mini Kit试剂盒(产自德国QIAGEN公司)对粪便样本提取DNA。使用苯酚-氯仿抽提法提取野猪肌肉样本 DNA。在 4℃或者 -20℃下保存DNA。
1.3 特异性引物及微卫星位点信息
SSR(simple sequence repeats,简单重复序列),又称微卫星DNA(microsatellite DNA),是指含有1~6个碱基对的短串联重复DNA序列,常见的是双核苷酸重复,如(CA)n和(TG)n。微卫星DNA重复单位数目为10~60个,重复单元数目的差异和重复程度的不同造成微卫星DNA片段长度差异,差异使得同一基因座位在一物种中存在很多的等位基因,表现出较高的多态性[10]。微卫星DNA由于其多态性高,信息量大,实验步骤少且简单,实验结果稳定,DNA需求量少等优点,广泛应用于遗传学分析。本研究采用世界粮农组织(FAO)推荐的适于野猪遗传学分析的9个微卫星标记。
使用野猪特异性引物进行物种鉴定,上游引物序列为5'-GACTAGGAACCATGAGGTTGCG-3',下游引物序列为5'-AGCCTACACCACAGCCACAG-3',扩增片段大小为134 bp,PCR扩增时退火温度为63℃[11]。微卫星位点信息见表1。所有引物都由上海生工生物有限公司合成。

表1 微卫星位点信息Tab.1 Microsatellite markers information
1.4 PCR扩增及电泳检测
PCR扩增的反应体系为 Buffer 10 μL、dNTPs 4 μL、Taq 酶 0.3 μL、上游引物 0.75 μL、下游引物0.75 μL、DNA 1 μL,用 ddH2O 补足体系到 20 μL。
PCR循环参数为:94℃,预变性2 min后;98℃,变性20 s;在各引物退火温度下,退火30 s;68℃ 延伸20 s;扩增35个循环后,最后以68℃延伸10 min。4℃保存。扩增产物在含有核酸染料Golden View的1%琼脂糖凝胶中,以120 V的电压电泳30 min。最后在UVP凝胶成像系统上紫外透射观察,照相,保存。每个样本进行PCR扩增3~5次,以确保实验结果的准确性。
使用ABI3730X1测序仪对所有微卫星位点荧光标记下的PCR扩增产物进行基因型分型。基因型判读时,若3次PCR扩增中,出现3次复等位基因则判断为杂合子。若不能判断为杂合子,则再进行PCR扩增,根据多次结果,判断为纯合子还是杂合子。若多次扩增之后,仍无法判断,则舍弃样本。
1.5 统计分析
使用Excel的Microsatellite Toolkit进行个体鉴定。使用Gimlet软件计算微卫星位点的个体联合判别率。使用GenAIEx计算如下遗传参数:等位基因频率、有效等位基因数(Ne),Hardy-Weinberg平衡检验、杂合度、多态信息含量(PIC)、Shannon指数和F-统计值。
2 结果
2.1 个体判别率
使用Gimlet软件,得到9个微卫星位点的个体联合判别率,结果见表2。如图1所示,当联合使用前5个微卫星位点分析时,就可以进行无偏差个体判别。当使用前7个微卫星位点分析时,就可以达到全同胞个体判别的要求。本文使用的9个微卫星位点对样本进行个体鉴别时,全同胞基因型相似概率为0.0001415。研究采用的9个微卫星位点能满足个体判别的需要。
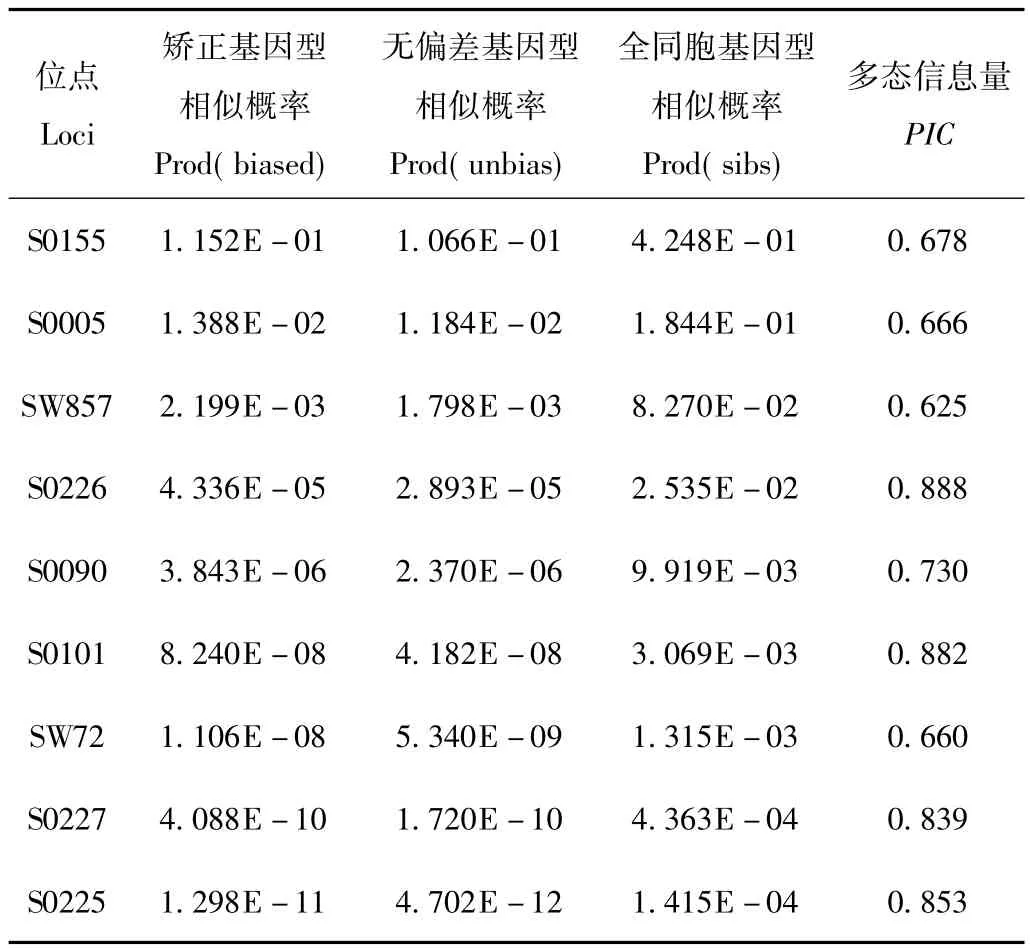
表2 微卫星位点个体判别率和多态信息含量(PIC)Tab.2 PI-idiotype similar probability and polymorphism information content(PIC)
9个微卫星位点PIC值结果见表2。微卫星位点PIC最小值为0.625(SW857),最大值为0.888(S0226)。所有微卫星位点的PIC值均大于0.5,表明本研究使用的微卫星位点都为高度多态性位点,可以满足实验要求。
2.2 物种鉴定和个体数量
使用物种特异性引物对收集到的140份粪便样本进行物种鉴定时,只有32号、62号粪便样本在琼脂糖凝胶电泳实验时没有检测到有效的条带。利用微卫星引物进行PCR扩增时,有些样本无法得到有效的扩增片段,故舍弃掉这些样本不用于接下来的遗传分析。
使用Excel中的Microsatellite Toolkit对101份野猪DNA进行个体鉴定。经鉴定,珲春有23个野猪个体,张广才岭南有14个野猪个体,汪清有19个野猪个体,天桥岭有13个野猪个体,穆棱有16个野猪个体。
2.3 等位基因数、有效等位基因数(Ne)和基因频率
9个微卫星标记得到的等位基因数及各种群实际等位基因数(Na)和有效等位基因数(Ne)结果见表3。
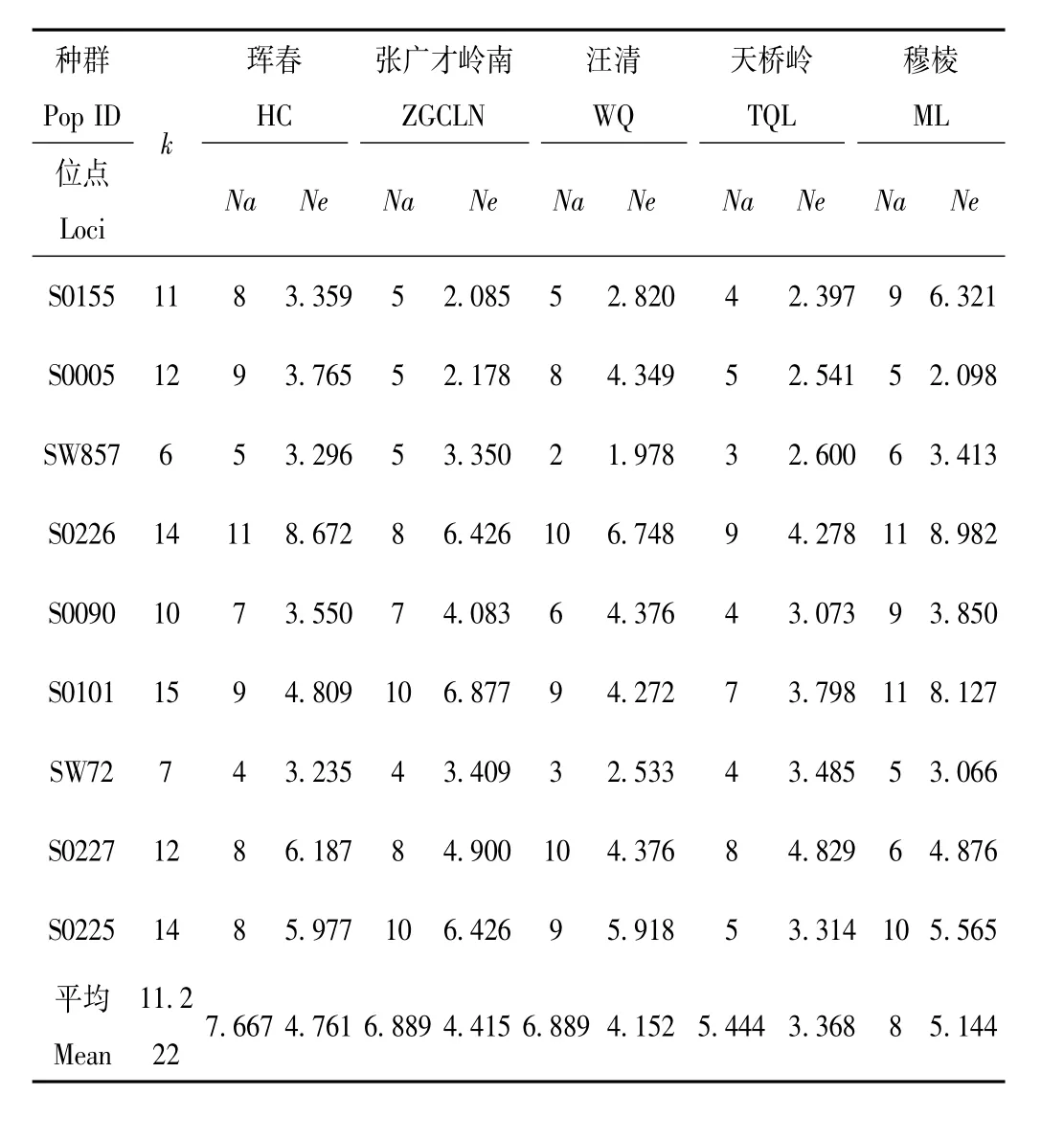
表3 各种群9个微卫星位点的实际等位基因数(Na)、有效等位基因数(Ne)和等位基因数目(k)Tab.3 The number of actual alleles(Na),effective alleles(Ne)for different combinations of locus-population and for each population,number of alleles for each loci
9个微卫星标记得到的等位基因数变化范围为6~15个,SW857位点的等位基因数最少,S0101位点的等位基因数最大。S0005位点在222 bp上的等位基因频率最高,为0.65625。整体来看,所有种群的平均实际等位基因数量中,天桥岭种群的平均实际等位基因数最小,为5.444个,穆棱种群的平均等位基因数最大,为8个。所有种群的平均有效等位基因数量中,天桥岭种群的平均有效等位基因数最小,为3.368个,穆棱种群的平均有效等位基因数最大,为5.144个。
本研究中的5个种群中,珲春种群Ne与Na之间差异最大的位点为S0005位点(5.624),张广才岭南种群中Ne与Na之间差异最大的位点为S0227位点(3.574),汪清种群中Ne与Na之间差异最大的位点为SW72(5.624),天桥岭种群中Ne与Na之间差异最大的位点为S0226位点(4.721),穆棱种群中Ne与Na之间差异最大的位点为S0090位点(5.150)。
2.4 杂合度
各种群9个微卫星位点的表观杂合度、期望杂合度及平均杂合度,结果见表4。在5个种群中,天桥岭种群平均期望杂合度最小,为0.687,珲春种群平均期望杂合度最大,为0.766。张广才岭南种群平均表观杂合度最小,为0.762,穆棱种群平均表观杂合度最大,为0.813。平均表观杂合度均大于平均期望杂合度。
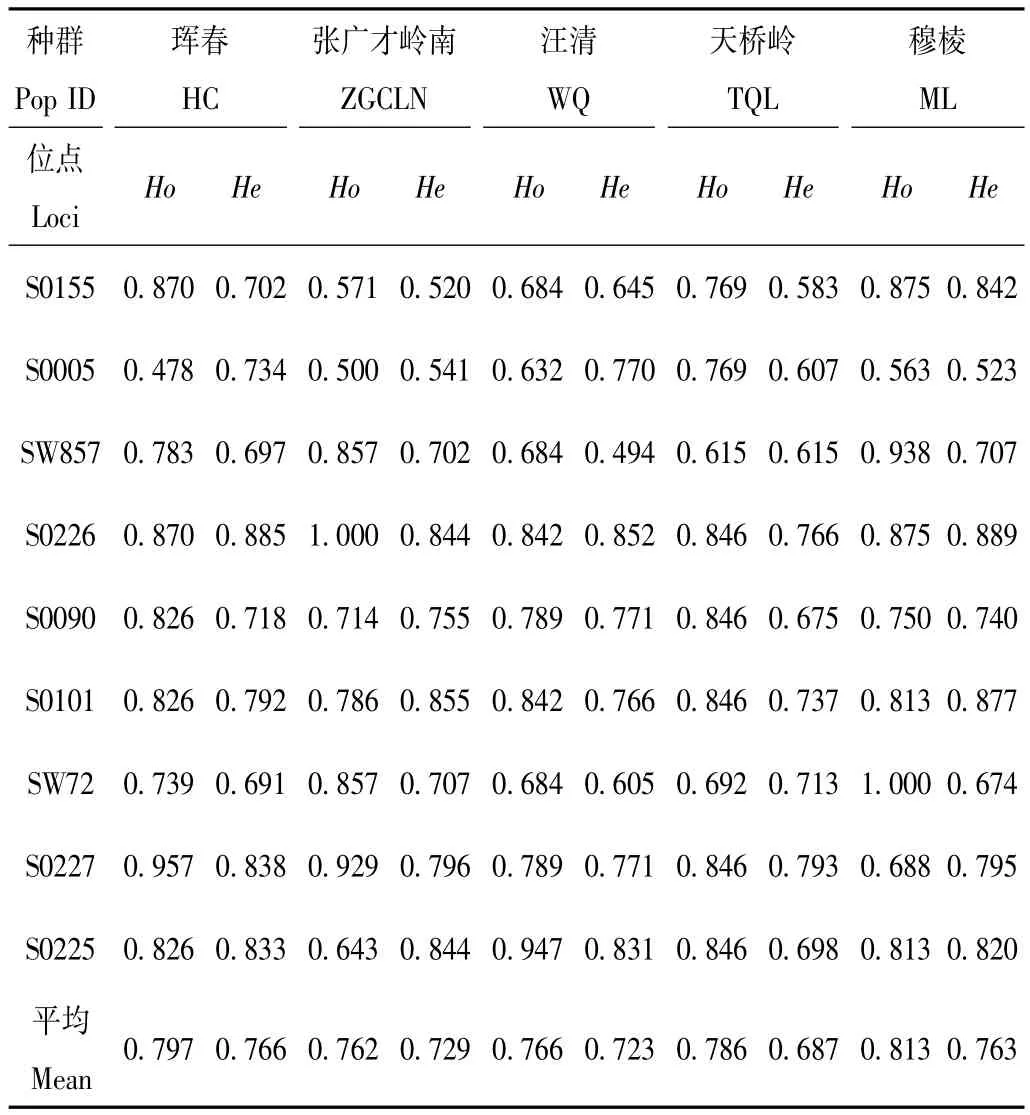
表4 各种群9个微卫星位点的表观杂合度(Ho)和期望杂合度(He)及平均杂合度Tab.4 The observed heterozygosity(Ho),the expected heterozygosity(He)and mean heterozygosity different combinations of locus-population and for each population
2.5 Hardy-Weinberg平衡检验
各种群每个位点Hardy-Weinberg平衡检验的P值及显著性结果见表5。9个微卫星位点Hardy-Weinberg平衡检验中,珲春种群的S0226位点,P值为0.011(<0.05);张广才岭南种群的S0101位点,P值为0.023(<0.05);汪清种群的S0005位点和S0101位点,P值都为0.000(<0.001);天桥岭种群的SW72位点,P值为0.020(<0.05);穆棱种群的S0225位点,P值为0.034(<0.05),这些位点显著偏离Hardy-Weinberg平衡。各种群的其余位点未显著偏离Hardy-Weinberg平衡(P>0.05)。5个种群的45个Hardy-Weinberg平衡检验中,共有6个(约13.3%)显著偏离Hardy-Weinberg平衡。
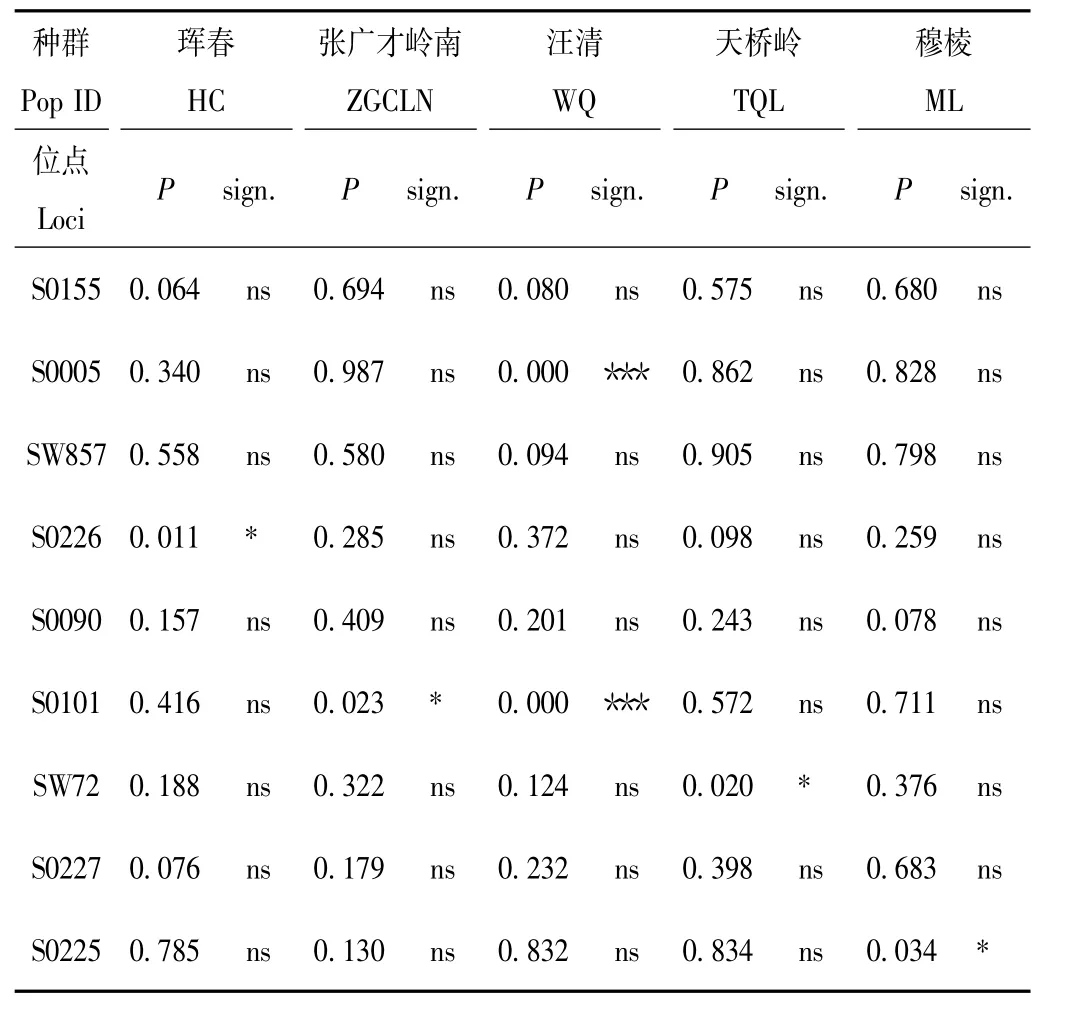
表5 各种群9个微卫星位点的Hardy-Weinberg平衡卡方检验的P值及显著性Tab.5 P value and significance of Hardy-Weinberg equilibrium
2.6 Shannon指数
各种群9个位点Shannon指数的平均值中,天桥岭种群的平均Shannon指数最小,为1.359,穆棱种群的平均Shannon指数最大,为1.729,珲春种群平均Shannon指数为1.688,张广才岭南种群平均Shannon指数为1.580,汪清种群平均 Shannon指数为1.532。
2.7 F-统计值
9个微卫星位点的F-统计值(Fis、Fit、Fst)结果见表6。在9个位点中,Fis值的范围在-0.206到0.074之间,只有S0005微卫星位点的Fis值大于0,其余8个微卫星位点的Fis值均小于0。
3 讨论
多态信息含量(PIC)作为衡量微卫星位点多态性的重要指标。本研究采用的9个微卫星位点的PIC值变化范围在0.625(SW857)~0.888(S0226),PIC值均大于0.5。根据Botstein等的建议,当PIC>0.5时,位点为高度多态性位点[12]。因此,本研究所采用的9个微卫星位点都为高度多态性位点,这就满足了遗传多样性研究要求,研究结果更为可靠。同时,每一个位点的等位基因数目的大小也能表明所筛选的微卫星位点是否适合研究该物种的遗传多样性。本研究中的9个微卫星位点,等位基因数目的变化范围在6(SW857)~15(S0101)个。9个微卫星位点得到的等位基因数目比较多,表明了所选位点能正确评价群体间的遗传关系,所有的微卫星位点都适合于分析野猪的遗传多样性。然而,霍金龙等[8]评估云南南部地区野猪遗传多样性时,采用76个微卫星标记探测到等位基因数目在3~9个之间,微卫星标记的PIC值在0.3701~-0.8624,小于本研究的9个微卫星标记的PIC值及等位基因数目。
有效等位基因数(Ne)是一个衡量遗传变异程度的指标。Ne与Na之间的差异,能说明某等位基因在群体内分布的均匀程度。当Ne与Na之间的差异越小,表明该等位基因在群体内分布的均匀度越好。珲春种群的S0005位点、张广才岭南种群的S0227位点、汪清种群的SW72位点、天桥岭种群的S0226位点和穆棱的S0090位点,等位基因的均匀度较其他的8个微卫星位点较差。
杂合度是衡量遗传多样性的一个重要指标。本研究中,各微卫星位点的表观杂合度在0.478~1.000之间,期望杂合度在0.494~0.889之间。这与霍金龙等[8]得到的76个微卫星标记的表观杂合度在0.409~1.000之间和期望杂合度在0.406~0.8765之间相一致。本研究的5个种群的平均表观杂合度都大于平均期望杂合度。这表明种群杂合子比例较高,发生近交的可能性少,遗传一致性较低,群体遗传多样性较高。
F-统计量是用来衡量种群遗传差异程度的指标。Fis是指亚群体的固定指数,Fit是指总群体的固定指数,Fst是指亚群体间的基因分化率。本研究中的9个微卫星位点的Fis值变化范围为-0.206(SW857)到0.074(S0005)。所有微卫星位点的Fis值均小于Fit值。9个微卫星位点的平均Fis值为-0.073,平均Fit值为-0.007,平均Fst值为0.062。这就表明总群体比亚群间更稳定,即5个种群之间存在一定程度的分化。同时,Fis也是衡量杂合子水平的指数。Fis值的变化范围在-1到1之间。当Fis大于0时,表明群体内的杂合子水平较低,纯合子水平较高;当Fis小于0时,表明群体内的杂合子水平较高,纯合子水平较低。张晨岭[7]使用11个微卫星标记计算得中国6个区域野猪种群的Fis值在0.06~0.20之间,野猪群体内部的纯合子多,杂合子少。而本研究Fis值大部分为负值,表明了没有呈现杂合子缺乏的情况,近交程度较低。
Shannon指数是反映群体离散程度的一个重要指标。Shannon指数普遍变化范围为1.5到3.5[13]。本研究中种群Shannon指数平均值最大的为1.729(穆棱),平均值最小的为1.359(天桥岭)。Shannon指数越大,表明群体的离散程度越高,群体遗传多样性越高。这就表明,从Shannon指数来看,群体遗传多样性从高到低,依次为穆棱、珲春、张广才岭南、汪清、天桥岭。总体来说,5个种群的Shannon指数都较高,表明受近交的影响较小。
根据Hardy-Weinberg平衡定律,其假设的群体是一个理想群体,该理想群体在随机交配的过程中,种群中的基因频率和基因型频率不变。本研究中,Hardy-Weinberg平衡的卡方检验结果可以看出,各种群的9个微卫星位点,只有少部分微卫星位点处于偏离Hardy-Weinberg平衡状态。偏离Hardy-Weinberg平衡状态很可能是群体并不符合随机交配,受到物种突变、迁移等影响。
4 结论
本研究初步筛选出了长白山北部野猪遗传多样性的微卫星标记。所筛选的9个微卫星位点在遗传学分析应用中获得的结果可靠。通过使用所筛选的微卫星位点对长白山北部野猪种群进行遗传多样性分析,表明5个区域的种群均具有较丰富的遗传多样性,近交程度低。这对今后进行野猪种群保护和管理策略的制定有重要意义。而且,野猪作为东北虎的主要捕食对象,保持野猪种群较高的多样性,对东北虎保护和管理也有积极意义。
冠心病是一种临床多因素疾病,多因冠状动脉粥样硬化导致心血管疾病的发生。根据流行病学调查结果显示,冠心病具有较高的发病率与死亡率,随着人口老龄化的加剧,冠心病发病率呈逐渐增长趋势[1]。对于冠心病高危人群进行早期诊断与早期干预对于冠心病的发生具有降低作用,可使患者的危害有效降低。本次研究对血清胆红素与尿酸在冠心病患者的临床检测价值进行对比分析,现报告如下。
致谢:感谢国家林业局野生动物与自然保护区管理司“虎、东北豹资源调查技术研究”和“东北虎、东北豹种群及栖息地调查评估标准制定及信息汇总”项目的资助;感谢浙江大学方盛国教授给予实验室分子生物学技术的咨询和帮助;感谢国家林业局猫科动物研究中心对野外样本与数据信息的提供。
[1] 马逸清.黑龙江省兽类志[M].哈尔滨:黑龙江科学技术出版社,1986:390-395.
[2] 周绍春,张明海,王双玲.完达山林区森林采伐和非采伐区马鹿、狍子对冬季生境因子选择的比较[J].动物学研究,2006,27(6):575-580.
[3] Riordan P.Unsupervised recognition of individual tigers and snow leopards from their footprints[J].Animal Conservation Forum,1998,1(4):253-262.
[4] 兰宏,王文,施立明.西南地区家猪和野猪mtDNA遗传多样性研究 [J].遗传学报,1995,22(1):28-33.
[5] Vernesi C,Crestanello B,Pecchioli E,et al.The genetic impact of demographic decline and reintroduction in the wild boar(Sur scrofa):a microsatellite analysis[J].Molecular Ecology,2003,12(3):585-595.
[6] 李崇奇.基于线粒体序列变异探讨野猪系统地理学及家猪起源[D].南京:南京师范大学,2005.
[7] 张晨岭.微卫星变异探讨中国大陆地区野猪遗传多样性、种群结构和种群动态[D].南京:南京师范大学,2006.
[8] 霍金龙,苗永旺,霍海龙,等.云南南部地区野猪群体的遗传多样性 [J].动物学杂志,2008,43(6):137-146.
[9] 韩春梅,高庆华,赵书红,等.新疆野猪mtDNA控制区序列遗传多样性分析 [J].安徽农业科学,2008,36(8):3142-3143,3245.
[10] Woodruff D S.Non-invasive genotyping of primates[J].Primates,1993,34(3):333-346.
[11] Walker J A,Hughes D A,Anders B A,et al.Quantitative intrashort interspersed elements PCR for species-specific DNA identification[J].Analytical Biochemistry,2003,316(2):259-269.
[12] Botstein D,White R L,Skolnick M,et al.Construction of a geneticlink age map in man using restriction fragment length polymorphisms[J].American Journal of Human Genetics,1980,32(3):314-331.
[13] Magurran A E.Biological diversity [J].Current Biology,2005,15(4):116-118.
