两种盐生植物根际土壤细菌多样性和群落结构
2018-06-22杨晓东吕光辉何学敏张雪妮
李 岩,杨晓东,秦 璐,吕光辉,何学敏, 张雪妮
1 新疆大学干旱生态环境研究所, 乌鲁木齐 830046 2 绿洲生态教育部重点实验室, 乌鲁木齐 830046 3 新疆大学资源与环境科学学院, 乌鲁木齐 830046 4 新疆环境保护科学研究院, 乌鲁木齐 830011 5 新疆大学生态学博士后科研流动站, 乌鲁木齐 830046
土壤微生物是陆地土壤系统中极为重要和最为活跃的组成成分,在土壤的形成与发育、有机质转化、生态系统平衡、土壤环境净化、生物修复等方面起着不可替代的作用[1- 2]。微生物-植物-土壤之间存在着复杂的相互作用,根际是直接受植物根系和分泌物影响的土壤区域,是土壤微生物与植物相互作用的重要场所。根际土壤微生物,特别是一些对植物生长具有促进作用的细菌和真菌,对植物生长和抗逆性,如耐盐性,发挥着重要作用[3- 7]。同时,根际土壤微生物多样性和群落结构也受到植物的影响,表现出一定的植物种类特异性和生长发育阶段的特异性[8- 9]。植物活动改变了根际土壤微环境,使得根际土壤与非根际土壤微生物的群落结构和组成存在明显差异[10- 11]。
土壤微生物群落结构也受土壤理化性质、营养状况、水分等因子的影响[12- 13]。盐碱环境条件下的土壤含盐量和pH比非盐碱土高,土壤盐分对土壤微生物群落的活性、多样性和结构存在显著的影响[13- 14]。作为生长在盐碱地中的植物类群,盐生植物在长期的适应和进化过程中,形成了独特的耐盐机制,以应对盐碱对自身的不利影响,除植物本身的应对机制外,根际微生物以及一些内共生菌对植物的耐盐性也起到了一定的作用[4, 14]。开展盐生植物根际微生物多样性和群落结构研究有助于深入理解盐生植物的耐盐性及其与根际土壤微生物间的相互作用。在过去10年间,人们对非盐生植物在正常条件或盐胁迫条件下的根系微生物多样性开展了较多研究,然而,缺少对盐生植物根系土壤微生物群落组成的研究[7]。
黑果枸杞(Lyciumruthenicum)和里海盐爪爪(Kalidiumcaspicum)是两种分布于我国西北干旱区的盐生植物,其中里海盐爪爪在国内仅分布于新疆。这两种植物多见于荒漠地区的盐渍化土壤中,均具有很强的耐盐碱性。国内学者从生理生态方面对它们的耐盐碱机理开展过研究[15- 17],但关于它们的根际土壤微生物多样性以及根际土壤微生物与耐盐性之间的关系方面还缺少深入研究。本研究利用高通量测序技术,分析黑果枸杞和里海盐爪爪根际与非根际土壤细菌的群落结构,以及根际土壤中细菌群落之间的相似性和差异性,从而揭示它们根际土壤细菌的多样性和群落结构,为进一步揭示根际土壤细菌与两种盐生植物耐盐性之间的关系提供理论基础。
1 材料与方法
1.1 样品采集
调查区位于新疆精河县西北部的准噶尔盆地西南的艾比湖湿地国家级自然保护区。该区气候干燥、降雨少、地表蒸发强烈;土壤盐分高、碱性强,浅层(0—10 cm)土壤的平均电导率为5.41 ms/cm,pH值为8.77;土壤平均容重约1.38 g/cm3,含水量平均为7.19%。该区分布有胡杨(Populuseuphratica)、柽柳(Tamarixramosissima)、梭梭(Haloxylonammodendron)、盐穗木(Halostachyscaspica)、盐节木(Halocnemumstrobilaceum)、碱蓬(Suaedaglauca)、盐爪爪(Kalidiumfoliatum)、里海盐爪爪(K.caspicum)、黑果枸杞(Lyciumruthenicum)等多种耐旱、耐盐植物[18]。
土壤样品于2016年8月采自精河县艾比湖湿地国家级自然保护区的盐碱地。里海盐爪爪群落和黑果枸杞群落相距大约2 km, 分别在里海盐爪爪、黑果枸杞居群中选择生长状况相近的健康个体(具有相近遗传背景,个体高度、冠幅基本一致),里海盐爪爪采集3个个体,黑果枸杞5个个体,个体之间距离大约30—50 m。将植株根系挖出,抖去根部附着比较疏松的土壤,留下与根系结合比较紧密的土壤(厚度约1 mm),装入50 mL无菌离心管中,于冷藏条件带回实验室。加入适量无菌PBS溶液(137 mmol/L NaCl, 2.7 mmol/L KCl, 8.5 mmol/L Na2HPO4, 1.5 mmol/L KH2PO4, pH 7.3), 漩涡震荡10 min,弃去根系,土壤溶液即为植株根际土壤溶液,储存于4℃备用。非根际土壤与根际土壤取自同一地点和同一时间,撇去表层土,收集0—30 cm土层的土壤,装于无菌保鲜袋中, 于冰上带回实验室,储存于4℃,备土壤总DNA提取。
1.2 研究方法
1.2.1 土壤理化性质分析
土壤pH和电导率(EC)分别采用pH计和电导率仪测定,土壤有机碳(TOC)和有机质(SOM)采用重铬酸钾容重法进行测定,全氮(TON)采用重铬酸钾-硫酸消化法进行测定。
1.2.2 土壤微生物基因组DNA的提取
提取基因组DNA时,吸取1 mL根际土壤溶液至2 mL无菌离心管中,于4℃、10000g离心30 s,弃上清,所得沉淀用于根际土壤微生物基因组DNA提取;对非根际土,用无菌药匙取大约0.2 g土壤,加入2 mL无菌离心管中,用于提取基因组DNA。利用OMEGA试剂盒E.Z.N.ATM Mag-Bind Soil DNA Kit试剂盒(OMEGA)提取基因组DNA,利用琼脂糖凝胶检测DNA的长度和完整性,利用NanoDrop检测DNA的浓度和纯度。
1.2.3 DNA扩增及测序
采用两轮法对细菌16S rDNA V3-V4区进行扩增, 第一轮扩增利用融合了barcode序列的通用引物进行,上游引物341F:ccctacacgacgctcttccgatctg (barcode) cctacgggnggcwgcag,下游引物805R: gactggagttccttggcacccgagaattccagactachvgggtatctaatcc,反应体系30 μL,包含15 μL 2×Taq master Mix(Thermo),上下游引物(10 μmol/L)各1 μL,模板DNA 20 ng。扩增条件:94℃ 3 min,94℃ 30 s,45℃ 20 s,65℃ 30 s,扩增5个循环;94℃ 20 s,55℃ 20 s,72℃ 30 s,扩增20循环,72℃延伸5 min。第二轮扩增使用Illumina桥式PCR兼容引物,反应体系同上,扩增条件:95℃ 30 s,95℃ 15 s,55℃ 15 s,72℃ 30 s,扩增5个循环,72℃延伸5 min。PCR产物经磁珠法回收后,送至生工(上海)生物工程有限公司进行测序,重复样品的PCR产物等量混合,每个混合样品包含所有重复,共测序4个样品,分别为两种植物的根际和非根际土壤样品。
1.3 数据分析
测序原始数据去除引物接头序列、去除各低质量碱基(Phred Quality Score=20)后进行拼接,舍弃长度短于200 bp的序列,去除非特异性扩增序列及嵌合体后,得到每个样本的有效序列数据。采用Mothur 1.30软件包以97%为划定阈值,对16S序列划分操作分类单元(operational taxonomic unit,OTU)。采用RDP classifier 2.12 对97%相似度水平的OTU代表序列进行分类学分析,得到每个OTU对应的物种分类信息,并在界、门、纲、目、科、属水平上统计各个样品的细菌群落组成。基于物种分类分析,绘制物种分类条形图和物种丰度热图。利用QIIME 1.8软件进行样品Alpha多样性分析,计算ACE、Chao1、Shannon、Simpson等物种多样性指数。基于Alpha多样性指数进行稀释性分析。通过unifrac分析,评估样本的Beta多样性,根据样本之间weighted unifrac距离,绘制样本聚类树。采用CCA分析影响细菌物种群落结构的主要环境因子。
2 结果与分析
2.1 土壤理化性质
土壤样品理化性质测定结果见表1。两种盐生植物的根际土壤有机碳(TOC)、有机质(SOM)、总氮(TON)含量均高于非根际土壤,其pH值均低于非根际土壤。黑果枸杞群落的非根际土壤的TOC、SOM和TON含量、电导率均高于里海盐爪爪群落的土壤,但其pH低于后者;里海盐爪爪的根际土壤的TOC、SOM和TON含量、电导率则高于黑果枸杞根际土,pH则相反。黑果枸杞根际土壤电导率低于非根际土,而里海盐爪爪根际土电导率略高于非根际土壤。
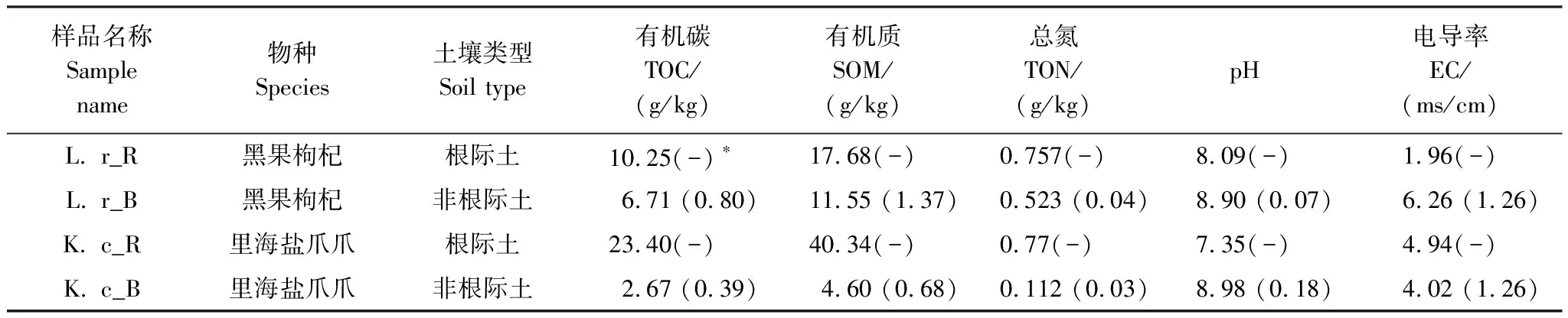
表1 根际土壤与非根际土壤的理化性质
数值格式:平均值(标准差)Mean (Std. Deviation);*:由于根际土样品量太少,将每个重复的根际土壤混合后进行测定,只有一个值,未计算偏差,以“-”表示
2.2 细菌多样性及群落结构
2.2.1 测序数据分析
研究测得的有效序列长度在400—440 bp之间, 平均长度420 bp左右。经土壤DNA基因组序列分析,从黑果枸杞和里海盐爪爪的根际土壤和非根际土壤中测定和分析出的序列数分别为27501条、18801条、21198条和35367条,有效OTU数分别为1081、1591、1788和1294(表2)。基于Shannon指数的稀释曲线(图1)表明,样品稀释曲线均基本趋于平缓,说明取样基本合理,能够比较真实地反映土壤样本的细菌群落,另外,测序覆盖率均高于90%,也表明本次测序深度合理,基本能代表样本的真实情况(表2)。
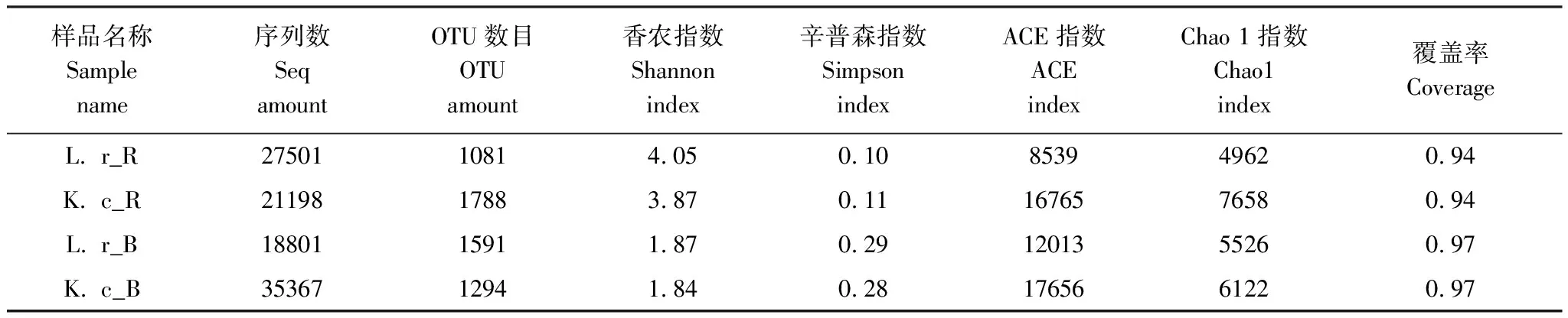
表2 土壤样品基因组DNA测序数据统计及alpha多样性分析

图1 相似度为0.97条件下各土壤样品中细菌群落的alpha指数(Shannon)稀释曲线Fig.1 Rarefaction curves of soil bacterial community (at 97% identity) constructed with Shannon indexL. r_R:黑果枸杞根际土壤细菌,bacterial community in rhizosphere soil of L. ruthenicum; L. r_B: 黑果枸杞非根际土壤细菌,bacterial community in bulk soil of L. ruthenicum;K.c_R:里海盐爪爪根际土壤细菌,bacterial community in rhizosphere soil of K. caspicum;K.c_B:里海盐爪爪非根际土壤细菌,bacterial community in bulk soil of K. caspicum
2.2.2 细菌多样性
测定的盐生植物根际和非根际土壤细菌的Alpha多样性结果见表2。Shannon指数和Simpson指数表明,根际土壤细菌多样性显著高于非根际土壤(P<0.01);黑果枸杞根际土壤细菌多样性高于里海盐爪爪,但差异不显著(P>0.05),但其ACE、Chao1指数低于里海盐爪爪,说明黑果枸杞根际土壤细菌丰度低于里海盐爪爪;两种植物的非根际土壤细菌多样性无显著差异(P>0.05)
2.2.3 细菌群落组成和结构
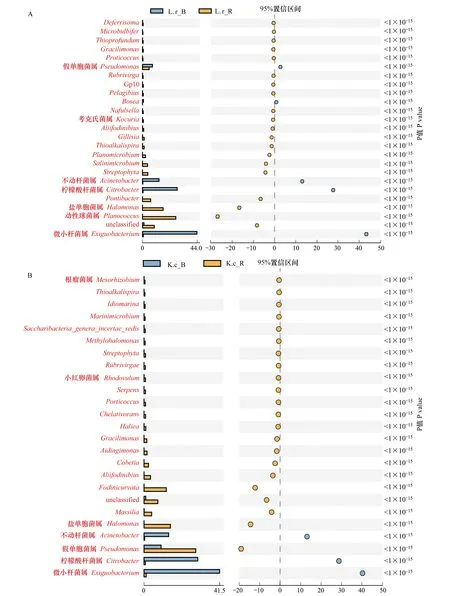
根据土壤基因组DNA序列的分类学分析结果,从所有供试土壤样品中共检测出细菌30门、55纲、88目、202科、672属。优势门(丰度>1%)数量少,非优势门数量多(图2)。在检测到的30个门中,变形菌门(Proteobacteria)和厚壁菌门(Firmicutes)在所有土壤样品中均为优势门,丰度分别为37.26%—81.65%和1.73%—45.67%。从黑果枸杞和里海盐爪爪根际土壤中分别检测出细菌21和22门,从非根际土壤中分别检测出28和24门。根际土壤细菌优势门数量多于非根际土壤,在根际土壤细菌中共检测出6个优势门,其中黑果枸杞根际土壤细菌优势门有6个,丰度从大到小分别为变形菌门(37.26%)、厚壁菌门(33.07%)、拟杆菌门(Bacteroidetes)(16.08%)、放线菌(Actinobacteria)(5.27%)、蓝细菌门(Cyanobacteria)(4.32%)、浮霉菌门(Planctomycetes)(1.07%),其他16门的丰度均低于1%,其中Deinococcus-Thermus等10个门的丰度低于0.1%;里海盐爪爪根际土壤细菌中的优势门有变形菌门(81.65%)、厚壁菌门(1.73%)、拟杆菌门(8.55%)和放线菌门(4.33%)4个门, 其他18门的丰度均低于1%,其中Parcubacteria等12个门的丰度低于0.1%;从非根际土壤细菌中检测出2个优势门,即变形菌门和厚壁菌门,占非根际土壤细菌总数的90%以上,在黑果枸杞的非根际土壤中的丰度分别为52.68%和45.67%,在里海盐爪爪非根际土壤中丰度分别为55.4%和43.22%,而其他门的丰度均少于1%,其中丰度低于0.1%的各有23和19个。
在属分类水平上,从黑果枸杞和里海盐爪爪两种盐生植物的根际土壤中分别检测出289和304个属,而在非根际土壤中分别检测出285和336个属,优势属占比很低;在根际和非根际土壤中的优势属数量和组成也不同,其中在根际土壤中的优势属数量多于非根际土壤。在黑果枸杞和里海盐爪爪根际土壤中的细菌优势属分别有10个和9个,而在非根际土壤中的细菌优势属有4个,仅假单胞菌属(Pseudomonas)在根际和非根际土壤中均为优势属(图2)。在黑果枸杞根际土壤中的10个优势属按丰度顺序依次为:动性球菌属(Planococcus,26.92%)、盐单胞菌属(Halomonas,16.7%)、Pontibacter(6.58%)、假单胞菌属(5.34%)、Streptophyta(4.32%)、Salinimicrobium(4.1%)、Planomicrobium(2.45%)、芽孢杆菌属(Bacillus, 1.77%)、Gillisia(1.4%)、Thioalkalispira(1.37%);在里海盐爪爪根际土壤中的9个优势属按丰度顺序依次为动性球菌属(28.39%)、盐单胞菌属(14.47%)、Fodinicurvata(12.24%)、Massilia(4.19%)、Aliifodinibius(3.56%)、Cobetia(2.45%)、Aidingimonas(1.63%)、Gracilimonas(1.51%)、微小杆菌属(Exiguobacterium,1.17%);在两种供试盐生植物非根际土壤中的4个优势属依次为微小杆菌属(44.01%、41.45%)、柠檬酸杆菌属(Citrobacter,28.07%、29.49%)、不动杆菌属(Acinetobacter,13.3%、13.51%)、假单胞菌属(8.09%、9.33%),其中微小杆菌属、不动杆菌属和柠檬酸杆菌属在根际土壤细菌中的丰度均显著降低(0.25%—1.17%),而一些低丰度属(如动性球菌属、盐单胞菌属、Pontibacter、Salinimicrobium等)则在根际土壤中显著增高,成为优势属,表明黑果枸杞和里海盐爪爪两种植物的根际土壤细菌的群落组成和丰度与非根际土壤存在较大的差异。

图2 门和属水平上的土壤样品细菌群落结构Fig.2 The bacterial community structures of soil samples at levels of phylum and genus 门图例(自上而下、自左至右,仅列出有对应中文名称的门):变形菌门Proteobacteria;厚壁菌门Firmicutes;拟杆菌门Bacteroidetes;放线菌门Actinobacteria;蓝细菌门Cyanobacteria;浮霉菌门Planctomycetes;分类未定unclassified;酸杆菌门Acidobacteria;疣微菌门Verrucomicrobia;绿弯菌门Chloroflexi;异常球菌-栖热菌门Deinococcus-Thermus;芽单胞菌门Gemmatimonadetes;硝化螺旋菌门Nitrospirae;热脱硫杆菌门Thermodesulfobacteria;衣原体门Chlamydiae;热袍菌门Thermotogae;螺旋体门Spirochaetes;梭杆菌门Fusobacteria;绿菌门Chlorobi;纤维杆菌门Fibrobacteres; 属图例(自上而下、自左至右,仅列出有对应中文名称的属):微小杆菌属Exiguobacterium;柠檬酸杆菌属Citrobacter;假单胞菌属Pseudomonas;不动杆菌属Acinetobacter;盐单胞菌属Halomonas;动性球菌属Planococcus;分类未定unclassified;芽孢杆菌Bacillus;小红卵菌属Rhodovulum;考克氏菌属Kocuria;根瘤菌属Mesorhizobium;氮单胞菌属Azomonas
2.2.4 细菌群落相似性及差异性分析
植物的根际土壤与非根际土壤细菌群落之间,以及不同植物的根际土壤细菌在群落组成和丰度上都存在显著差异。在门水平上,拟杆菌门、放线菌门、蓝细菌门、浮霉菌门、疣微菌门(Verrucomicrobia)、绿弯菌门(Chloroflexi)在根际土壤中的丰度显著高于非根际土壤,而厚壁菌门的丰度则低于非根际土壤(图2, 附图1A, B)。变形菌门、厚壁菌门、拟杆菌门、蓝细菌门、酸杆菌门丰度在两种植物的根际土壤中的丰度也具有显著的差异(图2, 附图1C),而非根际土壤细菌丰度在门水平上差异较小。在属水平上的细菌多样性丰度图(图3)显示,与根际土壤细菌群落相比,非根际土壤中的4个优势属组成相同,而且丰度也基本一致,除假单胞菌属外,不动杆菌属、柠檬酸杆菌属、微小杆菌属 3个优势属的丰度在根际土壤细菌群落中显著降低(附图2A, B)。另外,多个非优势属的丰度也在根际土壤中下降,甚至消失,如氮单胞菌属(Azomonas)、Brevundimonas、Enterococcus、Kluyvera等;与之相反,许多属的丰度在根际土壤中则显著增加,如盐单胞菌属、Streptophyta、Aliifodinibius、Gracilimonas等;动性球菌属、Pontibacter、Thioalkalispira、Cobetia和Aidingimonas是根际土壤中的特有属,在非根际菌群中未检测到(图3)。

图3 各土壤样品中在属水平上的细菌群落结构热度图及聚类图 Fig.3 The heatmap and clustering map of bacterial community structures of soil samples at level of genus热度图列代表样本,行代表群落结构,颜色块代表相对物种丰度值(由蓝到红,相对丰度逐渐升高),只显示丰度最高的前50个属,剩余的合并成other; 热图上方的分支结构为样品聚类树
通过对两种植物根际土壤细菌群落结构的比较,发现假单胞菌属和盐单胞菌属两属为这两种植物根际土壤中的共有优势属(图2),假单胞菌属在黑果枸杞根际土壤的丰度(5.34%)显著低于里海盐爪爪根际土壤(28.39%)。动性球菌属、Planomicrobium、Pontibacter、Salinimicrobium、芽孢杆菌属、Gillisia及Thioalkalispira是黑果枸杞根际土壤中的优势属,丰度为1.37%—26.92%,但在里海盐爪爪根际土壤中的丰度为0—0.49%;Aliifodinibius、微小杆菌属、Gracilimonas、Massilia、Fodinicurvata、Cobetia及Aidingimonas为里海盐爪爪根际土壤中的优势属,丰度为1.17%—12.23%,但在黑果枸杞根际土壤中的丰度为0.07%—0.95%,它们在根际和非根际土壤中的丰度具有显著差异(附图2C)。黑果枸杞和里海盐爪爪两种植物的遗传背景不同,其根系细菌群落结构组成和丰度也不同,表明不同植物即使在同一种盐碱环境条件下,它们的根际细菌群落结构和组成明显不同,植物种类的不同是影响其根际土壤细菌多样性的重要因子。
黑果枸杞和里海盐爪爪的根际土壤细菌群落之间的unifrac距离为0.44,而二者的非根际土壤细菌群落之间的距离为0.04,二者根际土壤与非根际土壤细菌群落间的距离分别为0.69和0.65。由聚类图(图3)可以看出,黑果枸杞和里海盐爪爪的根际土壤细菌群落聚为一个分支,二者的非根际土壤细菌群落聚为一个分支,表明两种植物的根际土壤细菌群落之间的相似性大于根际土壤和非根际土壤细菌群落间的相似性。
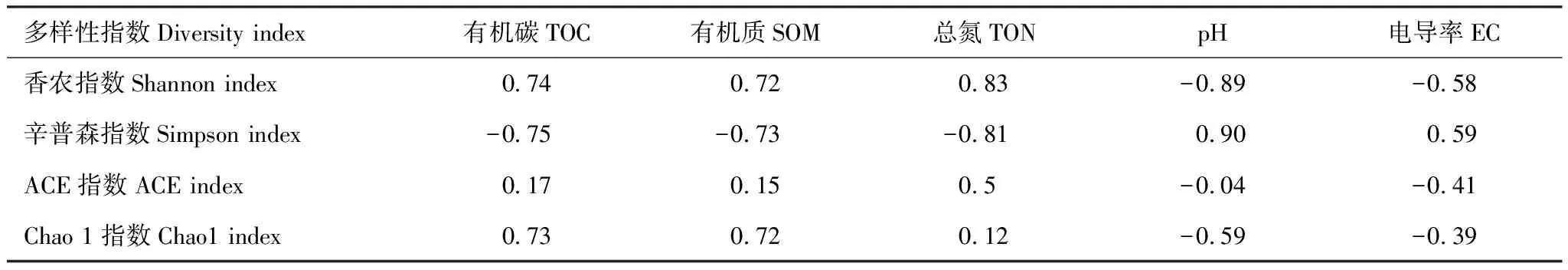
表3 两种植物根际土壤细菌多样性指数与土壤理化性质之间的相关性
2.2.5 细菌多样性和群落结构与土壤理化性质的关系

图4 两种植物根际土壤细菌群落组成与土壤理化因子的CCA分析图Fig.4 CCA analysis of two plants′ rhizosphere soil bacterial community composition and soil chemical factors
通过对二种植物根际土壤细菌多样性与土壤理化因子之间相互关系的分析,发现根际土壤细菌多样性的Shannon、ACE、Chao 1指数与TOC、SOM和TON呈正相关关系(表3),但与pH和EC呈负相关,但相关性均未达到显著水平(P>0.05)。通过对二种植物根际土壤细菌群落(物种)组成与土壤理化因子CCA间的关系分析,发现电导率和pH,TOC和TON分别是非根际土壤,根际土壤细菌群落组成的主要影响因子(图4)。
3 讨论
植物对根际土壤细菌群落结构具有主动选择性[19],植物通过根系活动改变根际环境,从而有选择性地提高或降低某些根际土壤细菌类群的丰度或多样性,使有益于植物自身生长的细菌种类的丰度增加,而不利于自身生长的细菌种类减少甚至消失,从而形成有益于自身的根际细菌群落结构,导致不同植物具有不同的根际细菌组成结构[8]。 如Comamonadaceae、Flavobacteriaceae、Rhizobiaceae 是大麦根际土壤细菌的主要类群[20];变形菌门、放线菌门、拟杆菌门和酸杆菌门是菊芋根际土壤中的优势类群[21];厚壁菌门、变形菌门、放线菌门是西瓜根际土壤中的优势类群[22]。
黑果枸杞和里海盐爪爪的根际土壤与非根际土壤细菌群落的多样性和群落组成具有显著差异。Alpha多样性分析表明,植物根际土壤细菌多样性高于非根际土壤,这与其他植物的相关研究成果[23- 24]相一致。变形菌门和厚壁菌门在植物根际和非根际土壤中均为优势类群,但变形菌门的丰度更高于厚壁菌门,变形菌门和厚壁菌门细菌是我国许多盐渍化土壤中的优势菌群[25],这两个门细菌在多种植物根际土壤中也是优势类群,如菊芋[21]、葡萄[26]、红芸豆[27]、Stellerachamaejasme[28]。变形菌门包括多样的病原菌和固氮菌,厚壁菌门细菌可发生于多种环境中,其中许多细菌可耐受极端条件,如干旱、盐碱等环境,常与植物的抗耐逆性有关,厚壁菌门在黑果枸杞和里海盐爪爪根际土壤中的丰度在30%以上,高于葡萄[26]、野燕麦[11]等非盐生植物,表明该类群细菌适宜盐碱环境的生长和繁殖,具有较强的抗耐盐性。非根际土壤中的大部分细菌门的丰度较低(<0.1%),然而拟杆菌门、蓝细菌门、疣微菌门、放线菌门在根际土壤中的丰度均有提高。
聚类分析表明, 两种植物根际细菌群落的相似性高于根际土壤与非根际土壤之间的相似性,暗示在盐碱化胁迫下两种植物趋向于选择和形成相似的根际土壤细菌结构,可能是盐生植物在长期的盐碱胁迫下,形成的一种对根际细菌群落的趋同性选择机制。两种植物根际土壤中的优势细菌属的数量多于非根际土壤,非根际土壤的优势属有3个,不动杆菌属、柠檬酸杆菌属、微小杆菌属,但它们在根际土壤中的丰度均显著降低;而在非根际土壤中的低丰度属,如动性球菌属、盐单胞菌属、Salinimicrobium等,在根际土壤中则为优势属。与非根际土壤细菌群落结构相比,根际土壤细菌丰度增加的种类大多具有抗耐盐性,微小杆菌属中有些种类具有耐盐性(可耐受NaCl浓度为13%),适生pH 5—11[29];盐单胞菌属为嗜盐菌,可以在5%—25% NaCl环境中生长[30];Salinimicrobium分布于盐渍化土壤中,可以耐受含1%—3% NaCl胁迫[31];Fodinicurvata是一类嗜盐菌,可在盐分含量5%—20%的土壤中生存[32]。假单胞菌属是盐碱土壤中的优势菌群,是一种耐盐碱菌群, 大多数种不需要有机生长因子,同时还可降解环境中广泛存在的多种有机分子[33]。
两种植物根际细菌群落组成具有相似性,同时也具有一定的差异,表现为优势菌群相对丰度不同,低丰度菌群组分不同, 这反映了根际群落结构的植物特异性。每种植物有自己的遗传背景和物质代谢过程,两种植物根际分泌物的种类、数量不同,根际土壤微环境不同,如根际土壤理化性质(表1)所示,SOM、pH、EC差异显著。黑果枸杞根际土壤中的细菌优势门数多于里海盐爪爪,虽然从两种植物根际土壤中检测出的细菌属总数和优势属数目几乎相同,但其具体种类和丰度均具有很大差异,仅假单胞菌属和盐单胞菌属是两种植物根际土壤共有的优势属,且假单胞菌属在黑果枸杞根际土壤中的丰度显著低于里海盐爪爪根际土壤。动性球菌属、Pontibacter等是黑果枸杞根际土壤中的细菌优势属,它们的丰度明显高于里海盐爪爪根际土壤中的丰度;而Aliifodinibius、微小杆菌属、Gracilimonas等是里海盐爪爪根际土壤中的细菌优势属,它们的丰度明显高于黑果枸杞根际土壤中的丰度。无论是优势属,还是低丰度属,它们在植物根际土壤和非根际土壤中均存在显著差异,尤其在两种植物的根际土壤中存在的差异更大,说明低丰度细菌类群比高丰度类群细菌对土壤环境更敏感[25]。
在盐渍化地区,含盐量是土壤微生物群落结构的重要限制因子,对土壤细菌群落结构和多样性具有显著影响[21]。干旱区盐碱化土壤养分含量低,且盐分含量高、碱性高,不利于土壤微生物的生长和繁殖。盐分胁迫会降低植物根际土壤细菌的多样性[34-35],然而,对耐盐植物而言,盐胁迫可增加根际土壤细菌多样性和生物量[36],这可能是由于盐分的升高促进了耐盐或嗜盐细菌的增殖[21]。本研究发现,黑果枸杞和里海盐爪爪两种植物的根际土壤的有机碳、有机质、总氮含量均高于非根际土壤,根际土壤细菌多样性亦高于非根际土壤,而相关性分析也证明土壤细菌的多样性与有机碳、有机质、总氮含量呈正相关,而与电导率和pH呈负相关;含盐量和pH是非根际土壤细菌群落组成的主要影响因子,而土壤有机质和总氮含量是根际土壤细菌群落组成的主要影响因子, 这与以往研究一致[37]。植物根际土壤有机质增加、pH降低、细菌丰度增加,这一结果表明耐盐植物根系可以缓解土壤盐渍化的胁迫效应,并为相应细菌生长提供更有利的微生境[25],同时,植物根际土壤细菌可以缓解土壤盐分对植物的胁迫作用[38]。
参考文献(References):
[1] Gans J, Wolinsky M, Dunbar J. Computational improvements reveal great bacterial diversity and high metal toxicity in soil. Science, 2005, 309(5739): 1387- 1390.
[2] 刘彩霞, 黄为一. 耐盐碱细菌与有机物料对盐碱土团聚体形成的影响. 土壤, 2010, 42(1): 111- 116.
[3] Palaniyandi S A, Damodharan K, Yang S H, Suh J W.Streptomycessp. strain PGPA39 alleviates salt stress and promotes growth of ′Micro Tom′ tomato plants. Journal of Applied Microbiology, 2014, 117(3): 766- 773.
[4] da Silveira Lúcio W, de Lacerda C F, Filho P F M, Hernandez F F F, Neves A L R, Gomes-Filho E. Growth and physiological responses of melon plants inoculated with mycorrhizal fungi under salt stress. Semina-Ciencias Agrarias, 2013, 34(4): 1587- 1602.
[5] Vaishnav A, Jain S, Kasotia A, Kumari S, Gaur R K, Choudhary D K. Effect of nitric oxide signaling in bacterial-treated soybean plant under salt stress. Archives of Microbiology, 2013, 195(8): 571- 577.
[6] Vaishnav A, Kumari S, Jain S, Varma A, Choudhary D K. Putative bacterial volatile-mediated growth in soybean (GlycinemaxL. Merrill) and expression of induced proteins under salt stress. Journal of Applied Microbiology, 2015, 119(2): 539- 551.
[7] Ruppel S, Franken P, Witzel K. Properties of the halophyte microbiome and their implications for plant salt tolerance. Functional Plant Biology, 2013, 40(9): 940- 951.
[8] Ziegler M, Engel M, Welzl G, Schloter M. Development of a simple root model to study the effects of single exudates on the development of bacterial community structure. Journal of Microbiological Methods, 2013, 94(1): 30- 36.
[9] Li X Z, Rui J P, Mao Y J, Yannarell A, Mackie R. Dynamics of the bacterial community structure in the rhizosphere of a maize cultivar. Soil Biology and Biochemistry, 2014, 68: 392- 401.
[10] Ciccazzo S, Esposito A, Rolli E, Zerbe S, Daffonchio D, Brusetti L. Safe-site effects on rhizosphere bacterial communities in a high-altitude alpine environment. Biomed Research International, 2014, 2014: 480170.
[11] Shi S J, Nuccio E, Herman D J, Rijkers R, Estera K, Li J B, da Rocha U N, He Z L, Pett-Ridge J, Brodie E L, Zhou J Z, Firestone M. Successional trajectories of rhizosphere bacterial communities over consecutive seasons. mBio, 2015, 6(4): e00746- 15.
[12] Ramirez K S, Craine J M, Fierer N. Consistent effects of nitrogen amendments on soil microbial communities and processes across biomes. Global Change Biology, 2012, 18(6): 1918- 1927.
[13] Bell C W, Asao S, Calderon F, Wolk B, Wallenstein M D. Plant nitrogen uptake drives rhizosphere bacterial community assembly during plant growth. Soil Biology and Biochemistry, 2015, 85: 170- 182.
[14] Chandrasekaran M, Boughattas S, Hu S J, Oh S H, Sa T M. A meta-analysis of arbuscular mycorrhizal effects on plants grown under salt stress. Mycorrhiza, 2014, 24(8): 611- 625.
[15] 王龙强. 盐生药用植物黑果枸杞耐盐生理生态机制研究[D]. 兰州: 甘肃农业大学, 2011.
[16] 姜霞. 黑果枸杞耐盐机理的相关研究[D]. 西安: 西北农林科技大学, 2012.
[17] 杨瑞瑞, 曾幼玲. 盐生植物盐爪爪的耐盐生理特性探讨. 广西植物, 2015, 35(3): 366- 372.
[18] 张雪妮, 杨晓东, 吕光辉. 水盐梯度下荒漠植物多样性格局及其与土壤环境的关系. 生态学报, 2016, 36(11): 3206- 3215.
[19] Kowalchuk G A, Buma D S, de Boer W, Klinkhamer P G L, van Veen J A. Effects of above-ground plant species composition and diversity on the diversity of soil-borne microorganisms. Antonie van Leeuwenhoek, 2002, 81: 509- 520.
[20] Bulgarelli D, Garrido-Oter R, Münch P C, Weiman A, Dröge J, Pan Y, McHardy A C, Schulze-Lefert P. Structure and function of the bacterial root microbiota in wild and domesticated barley. Cell Host & Microbe, 2015, 17(3): 392- 403.
[21] Yang H, Hu J X, Long X H, Liu Z P, Rengel Z. Salinity altered root distribution and increased diversity of bacterial communities in the rhizosphere soil of Jerusalem artichoke. Scientific Reports, 2016, 6: 20687.
[22] Ling N, Deng K Y, Song Y, Wu Y C, Zhao J, Raza W, Huang Q W, Shen Q R. Variation of rhizosphere bacterial community in watermelon continuous mono-cropping soil by long-term application of a novel bioorganic fertilizer. Microbiological Research, 2014, 169(7/8): 570- 578.
[23] Aira M, Bybee S, Domínguez J. Carnivory does not change the rhizosphere bacterial community of the plantDroseraintermedia. Applied Soil Ecology, 2015, 92: 14- 17.
[24] Cui H Y, Yang X Y, Lu D X, Jin H, Yan Z Q, Chen J X, Li X Z, Qin B. Isolation and characterization of bacteria from the rhizosphere and bulk soil ofStellerachamaejasmeL. Canadian Journal of Microbiology, 2015, 61(3): 171- 181.
[25] Yang J, Ma L A, Jiang H C, Wu G, Dong H L. Salinity shapes microbial diversity and community structure in surface sediments of the Qinghai-Tibetan Lakes. Scientific Reports, 2016, 6: 25078.
[26] Vega-Avila A D, Gumiere T, Andrade P A M, Lima-Perim J E, Durrer A, Baigori M, Vazquez F, Andreote F D. Bacterial communities in the rhizosphere ofVitisviniferaL. cultivated under distinct agricultural practices in Argentina. Antonie van Leeuwenhoek, 2015, 107(2): 575- 588.
[27] Suyal D C, Yadav A, Shouche Y, Goel R. Bacterial diversity and community structure of Western Indian Himalayan red kidney bean (Phaseolusvulgaris) rhizosphere as revealed by 16S rRNA gene sequences. Biologia, 2015, 70(3): 305- 313.
[28] Jin H, Yang X Y, Yan Z Q, Liu Q, Li X Z, Chen J X, Chen J X, Zhang D H, Zeng L M, Qin B. Characterization of rhizosphere and endophytic bacterial communities from leaves, stems and roots of medicinalStellerachamaejasmeL. Systematic and Applied Microbiology, 2014, 37(5): 376- 385.
[30] Vreeland R H, Litchfield C D, Martin E L, Elliot E.Halomonaselongata, a new genus and species of extremely salt-tolerant bacteria. Journal of Systematic Bacteriology, 1980, 30(2): 485- 495.
[31] Chen Y G, Cui X L, Zhang Y Q, Li W J, Wang Y X, Kim C J, Lim J M, Xu L H, Jiang C L.Salinimicrobiumterraesp. nov., isolated from saline soil, and emended description of the genusSalinimicrobium. International Journal of Systematic and Evolutionary Microbiology, 2008, 58(11): 2501- 2504.
[32] Infante-Dominguez C, Lawson P A, Johnson C N, Sánchez-Porro C, Ventosa A.Fodinicurvatahalophilasp. nov., a moderately halophilic bacterium from a marine saltern. International Journal of Systematic and Evolutionary Microbiology, 2015, 65: 766- 771.
[33] 李新, 焦燕, 代钢, 杨铭德, 温慧洋. 内蒙古河套灌区不同盐碱程度的土壤细菌群落多样性. 中国环境科学, 2016, 36(1): 249- 260.
[34] Ibekwe A M, Poss J A, Grattan S R, Grieve C M, Suarez D. Bacterial diversity in cucumber (Cucumissativus) rhizosphere in response to salinity, soil pH, and boron. Soil Biology and Biochemistry, 2010, 42(4): 567- 575.
[35] Bencherif K, Boutekrabt A, Fontaine J, Laruelle F, Dalpè Y, Sahraoui A L H. Impact of soil salinity on arbuscular mycorrhizal fungi biodiversity and microflora biomass associated withTamarixarticulataVahll rhizosphere in arid and semi-arid Algerian areas. Science of the Total Environment, 2015, 533: 488- 494.
[36] Nie M, Zhang X D, Wang J Q, Jiang L F, Yang J, Quan Z X, Cui X H, Fang C M, Li B. Rhizosphere effects on soil bacterial abundance and diversity in the Yellow River Deltaic ecosystem as influenced by petroleum contamination and soil salinization. Soil Biology and Biochemistry, 2009, 41(12): 2535- 2542.
[37] Pavloudi C, Oulas A, Vasileiadou K, Sarropoulou E, Kotoulas G, Arvanitidis C. Salinity is the major factor influencing the sediment bacterial communities in a Mediterranean lagoonal complex (Amvrakikos Gulf, Ionian Sea). Marine Genomics, 2016, 28: 71- 81.
[38] Borruso L, Bacci G, Mengoni A, De Philippis R, Brusetti L. Rhizosphere effect and salinity competing to shape microbial communities inPhragmitesaustralis(Cav.) Trin. ex-Steud. FEMS Microbiology Letters, 2014, 359(2): 193- 200.
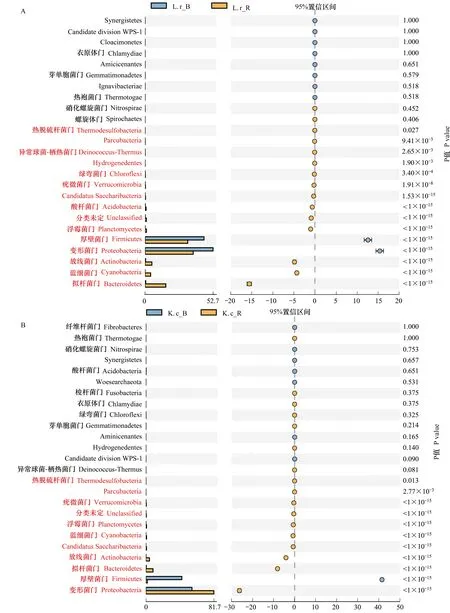
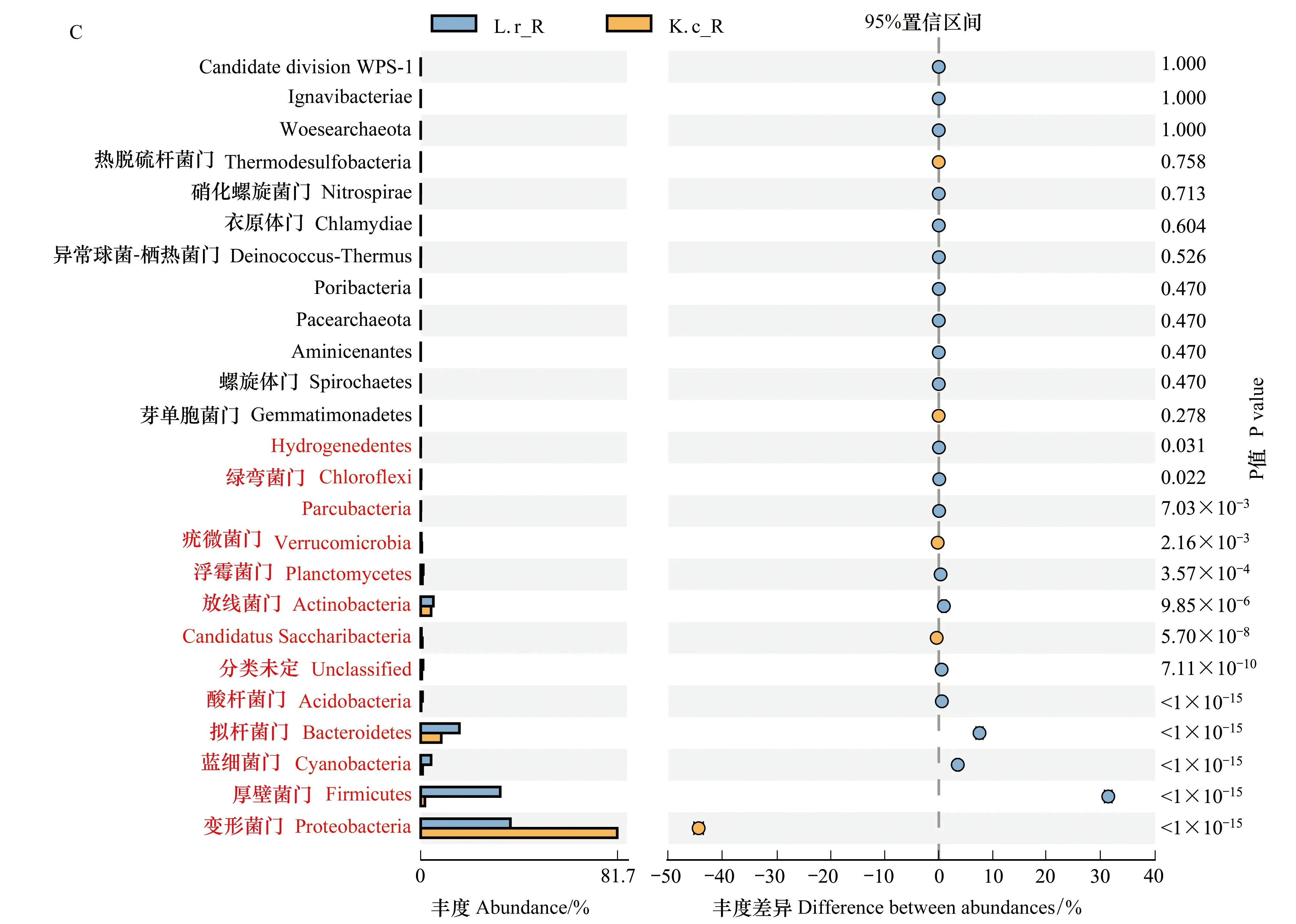
附图1 黑果枸杞和里海盐爪爪根际和非根际土壤中细菌群落组成在门水平的差异分析Fig.S1 The differential analysis of bacterial community composition between rhizosphere and bulk soil of L. ruthenicum and K. caspicum at level of phylumA:黑果枸杞根际vs非根际菌群;B:里海盐爪爪根际vs非根际菌群;C:黑果枸杞根际菌群vs里海盐爪爪根际菌群;L. r_R:黑果枸杞根际土壤细菌,bacterial community in rhizosphere soil of L. ruthenicum; L. r_B: 黑果枸杞非根际土壤细菌,bacterial community in bulk soil of L. ruthenicum;K.c_R:里海盐爪爪根际土壤细菌,bacterial community in rhizosphere soil of K. caspicum;K.c_B:里海盐爪爪非根际土壤细菌,bacterial community in bulk soil of K. caspicum
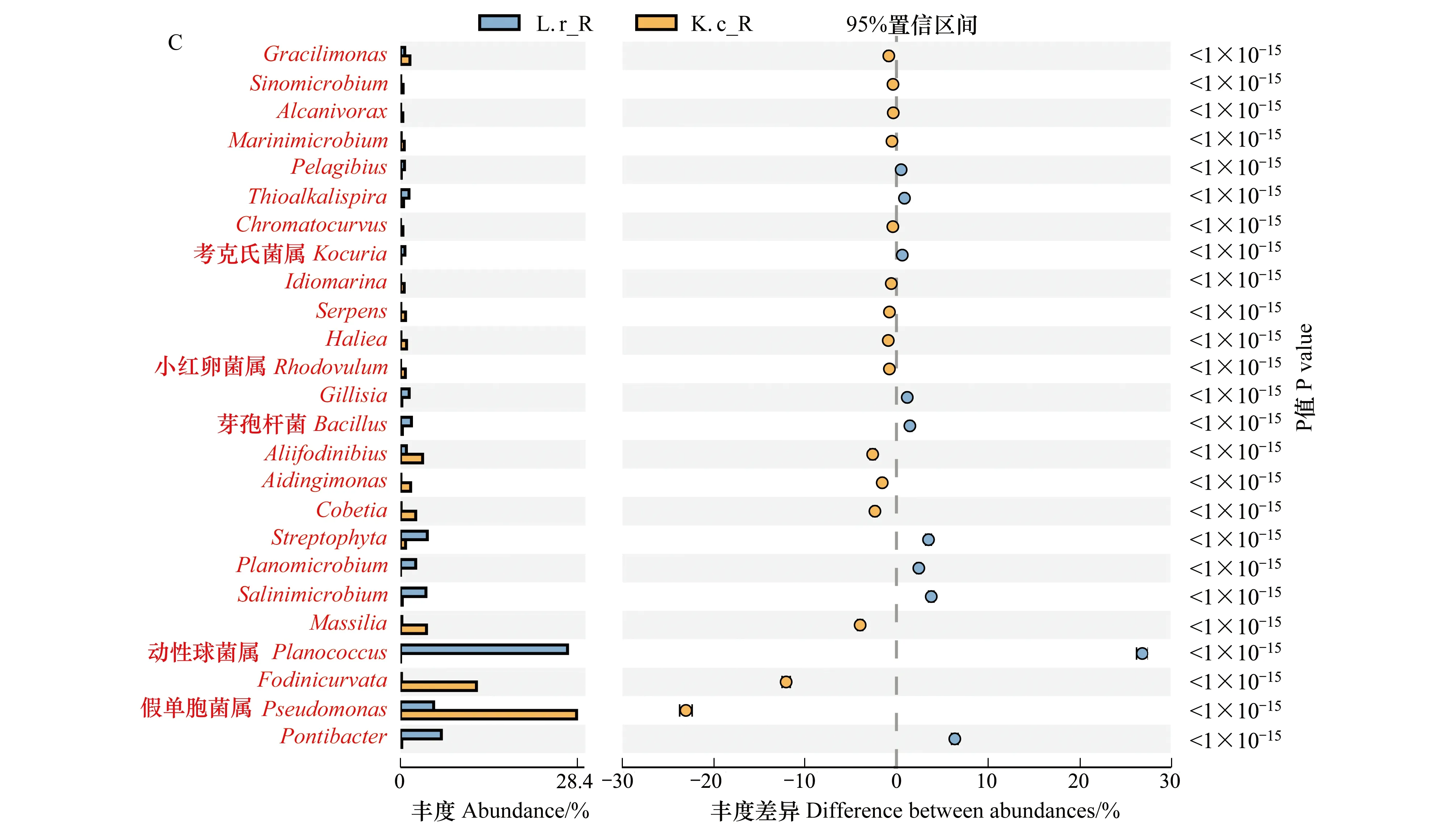
附图2 黑果枸杞和里海盐爪爪根际和非根际土壤细菌群落组成在属水平的差异分析Fig.S2 The differential analysis of bacterial community composition between rhizosphere and bulk soil of L. ruthenicum and K. caspicum at level of genusL. r_R:黑果枸杞根际土壤细菌,bacterial community in rhizosphere soil of L. ruthenicum; L. r_B: 黑果枸杞非根际土壤细菌,bacterial community in bulk soil of L. ruthenicum;K.c_R:里海盐爪爪根际土壤细菌,bacterial community in rhizosphere soil of K. caspicum;K.c_B:里海盐爪爪非根际土壤细菌,bacterial community in bulk soil of K. caspicum
