应用石斛EST序列对SNP位点开发与分析1)
2018-06-15毛立彦龙凌云檀小辉檀业维韦勇杰於艳萍宾振钧覃剑锋覃茜金刚
毛立彦 龙凌云 檀小辉 檀业维 韦勇杰 於艳萍 宾振钧 覃剑锋 覃茜 金刚
(广西壮族自治区亚热带作物研究所,南宁,530001)
单核苷酸多态性(SNP)是指基因组范围的单个核酸碱基的插入、缺失、转换、颠换等突变引起的DNA序列多态性。由于其密度高、遗传稳定性强,易于自动化分析等特点,已经成为第三代分子标记,目前已广泛应用于动植物的遗传多样性分析[1]、遗传连锁图谱构建[2]、品种鉴定[3]及重要性状的基因定位[4]等相关研究中。但SNP标记开发前期需要测序的成本费用较高导致其未能被大规模开发,因此,利用已知数据,通过生物信息学分析进行SNP标记进行前期位点开发挖掘、再通过试验进行候选SNP位点检测验证,是SNP标记降低成本的快捷高效的开发途径之一[5]。
EST(表达序列标签)是来源于功能基因表达的cDNA片段,也是识别转录区多态性的重要资源。随着研究的深入,公共数据库中EST序列以飞快的速度递增,极大地促进了以EST序列为基础的分子标记的开发,目前EST-AFLP、EST-RFLP、EST-SSR、EST-SNP等分子标记手段已经非常普遍[6]。这些基于EST序列开发出的分子标记除具有一般常用分子标记的特点之外还具有通用性好、信息量大、开发方法简单快捷、成本低等优点。特别是综合多种优点的EST-SNP,其研究结果很可能与表达基因紧密相关,可直接运用于动植物分子育种等相关研究领域的实践应用[7]。对于没有全基因序列信息的动植物来讲,利用EST序列进行候选SNP位点的挖掘具有非常重要的研究意义。
石斛属(DendrobiumSw.)是兰科植物中仅次于石豆兰属(BulbophyllumThou.)的第二大属,也是兼具较高药用价值和观赏价值的一个属,全球有1 000~1 400个种,广泛分布于亚热带及热带地区。我国是石斛的重要分布地区之一,由于品种数量庞大,对于其品种鉴定前人尝试了多种方法[8],但至今对于石斛的分子鉴定及分类还是存在极大困难,严重限制了石斛育种进程及其产业的发展,因此,对于石斛的品种鉴定、分类及遗传多样性的研究仍旧是石斛研究当务之急。利用石斛在NCBI中的dbEST数据库进行候选SNP位点的挖掘,开发石斛SNP分子标记对石斛育种、分类及遗传多样性分析都具有重要意义。本研究从NCBI的EST数据下载石斛EST序列,通过生物信息学方法筛选候选SNP位点,为石斛EST-SNP标记的开发及后续石斛的遗传多样性分析、品种鉴定、性状基因定位及分子育种等研究奠定基础。
1 材料与方法
1.1 石斛EST序列获取及聚类簇分析
从NCBI的dbEST数据库(https://www.ncbi.nlm.nih.gov/nucest/?term=Dendrobium)下载16 183条石斛EST序列,所有EST序列均以FASTA格式保存,序列信息来源见表1。序列下载后,采用DNASTAR 7.1.0(44.1)软件包中的SeqMan程序检测去除全部EST序列的载体序列,然后组装拼接叠连群。

表1 石斛EST序列来源信息
1.2 石斛EST-SNP位点筛选及分析
采用SNASTAR中的SeqMAn程序中的SNP工具对拼装好的叠连群进行候选SNP位点筛选。筛选原则:①候选SNP位点两侧至少有5 bp完全保守序列;②拼接组装含有4条(包括4条)以上EST序列的叠连群;③候选SNP位点中的次要等位基因频率至少为30%(图1)[9-10]。
SNP频率=(候选SNP数目/叠连群长度)×100%。

图1 候选SNP位点的人工筛选原则
1.3 候选SNP所在核苷酸序列同源性比对
提取筛选得到的SNP位点两侧各约50 bp的EST序列,采用NCBI上的BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastn&PAGE_TYPE=BlastSearch&LINK_LOC=blasthome)进行核酸序列比对,从比对结果中提取与比对序列相似性最高的序列注释信息,对SNP靶向基因产物及物种来源进行分析。
2 结果与分析
2.1 石斛EST序列聚类
从NCBI的dbEST数据库中下载得到石斛EST序列16 183条,参与拼接的EST序列为9 756条,拼接聚类后共得到叠连群2 267个,其中含4条及4条以上EST序列的叠连群621个,占总数的27.4%,总长度628 444 bp,未参与拼接的序列6 427条,拼接效率为60.3%。
2.2 石斛EST序列SNP频率
通过DNASTAR软件中的SeqMan程序对含有4条EST序列以上的621个叠连群进行候选SNP位点筛选,结果表明:共有342个叠连群含有候选SNP位点1 083个,平均580.28个bp含有1个SNP位点,每个叠连群含有3.25个SNP位点,SNP位点发生频率为0.17%。其中含SNP位点最多的叠连群(叠连群规模为8)共有22个SNP位点,30.70%的叠连群只含有1个SNP侯选位点,含3个以下候选SNP位点的叠连群占全部候选SNP位点的总叠连群数的69.59%,含3个以上候选SNP位点的叠连群只占总叠连群的30.41%,多数叠连群包含的SNP位点并不丰富,这可能与石斛的遗传背景紧密联系(表2)。
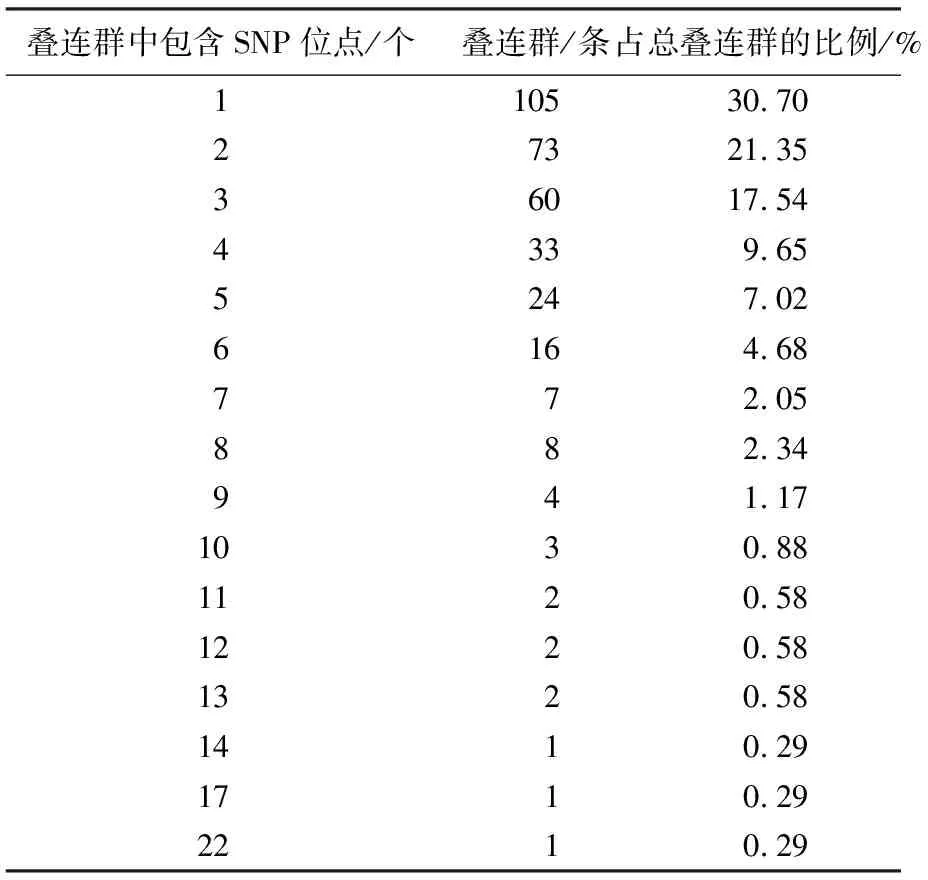
表2 包含不同数量SNP位点数的叠连群数量统计
随着叠连群规模不断增加,叠连群包含的候选SNP位点总数呈下降趋势,整体上显示小规模叠连群包含的候选SNP位点数最多,其中叠连群规模为4~6的包含的候选SNP位点数占候选SNP位点总数的42.7%,在规模为4的叠连群中包含150个SNP位点,这些位点的阳性率可能会很高[11](表3)。包含SNP候选位点的叠连群规模在4~6的占总叠连群的48.8%(表3),通过统计分析每种规模叠连群平均包含候选SNP位点数与叠连群规模之间并没有相关性,平均含量分别在叠连群规模为19时最大,15、21、43次之(表3)。综合以上数据说明虽然大规模叠连群容易筛选到更多的SNP位点,但其总量却远远不如小规格叠连群。
2.3 候选SNP位点类型
筛出的候选SNP位点有转换、颠换及插入缺失3类型,其中转换类型位点为655个,占总数的60.5%,颠换类型位点为408个,占总数的37.7%,二者比值约为1.6∶1.0,插入缺失为20个,占总数的1.8%(表4)。在转换类型中C-T转换的频率(37.0%)远远大于A-G转换频率(23.5%)。颠换类型中以AT和GT颠换类型为主,二者分别占候选SNP位点总数的10.3%和10.5%,CG和CA颠换占少数,比例分别为8.1%和8.7%。
2.4 候选SNP位点所在核酸序列同源性比对
提取筛选得到的1 063个转换和颠换SNP位点两侧各50 bp序列在NCBI核酸比对数据库中进行同源性比对,发现共有25个SNP候选位点所在的10条核酸序列无比对结果,可能是还未发现的基因,但也需要进一步验证。在具比对结果的SNP位点中有2个SNP位点所在核酸序列与梵净山石斛叶绿体DNA具99%的同源性,1个SNP位点所在核酸序列与金钗石斛叶绿体DNA具99%的同源性,3个SNP位点所在核酸序列与流苏石斛叶绿体DNA具99%的同源性,6个SNP位点所在核酸序列与小兰屿蝴蝶兰(Phalaenopsisequestris)的细胞色素蛋白具86%的同源性,另外3个SNP位点所在核酸序列与小兰屿蝴蝶兰的休眠相关蛋白同源物具87%的同源性,1个SNP位点所在核酸序列与建兰花叶病毒(Cymbidiummosaicvirus)的外壳蛋白具99%的同源性,共有1 021个SNP所在核酸序列与铁皮石斛的不同基因序列存在高度同源性(表5,表6),说明同科属植物存在较高的同源性。唯一一个SNP位点所在核酸序列与绿豆的60 S核糖体蛋白具有98%的同源性(表7)。
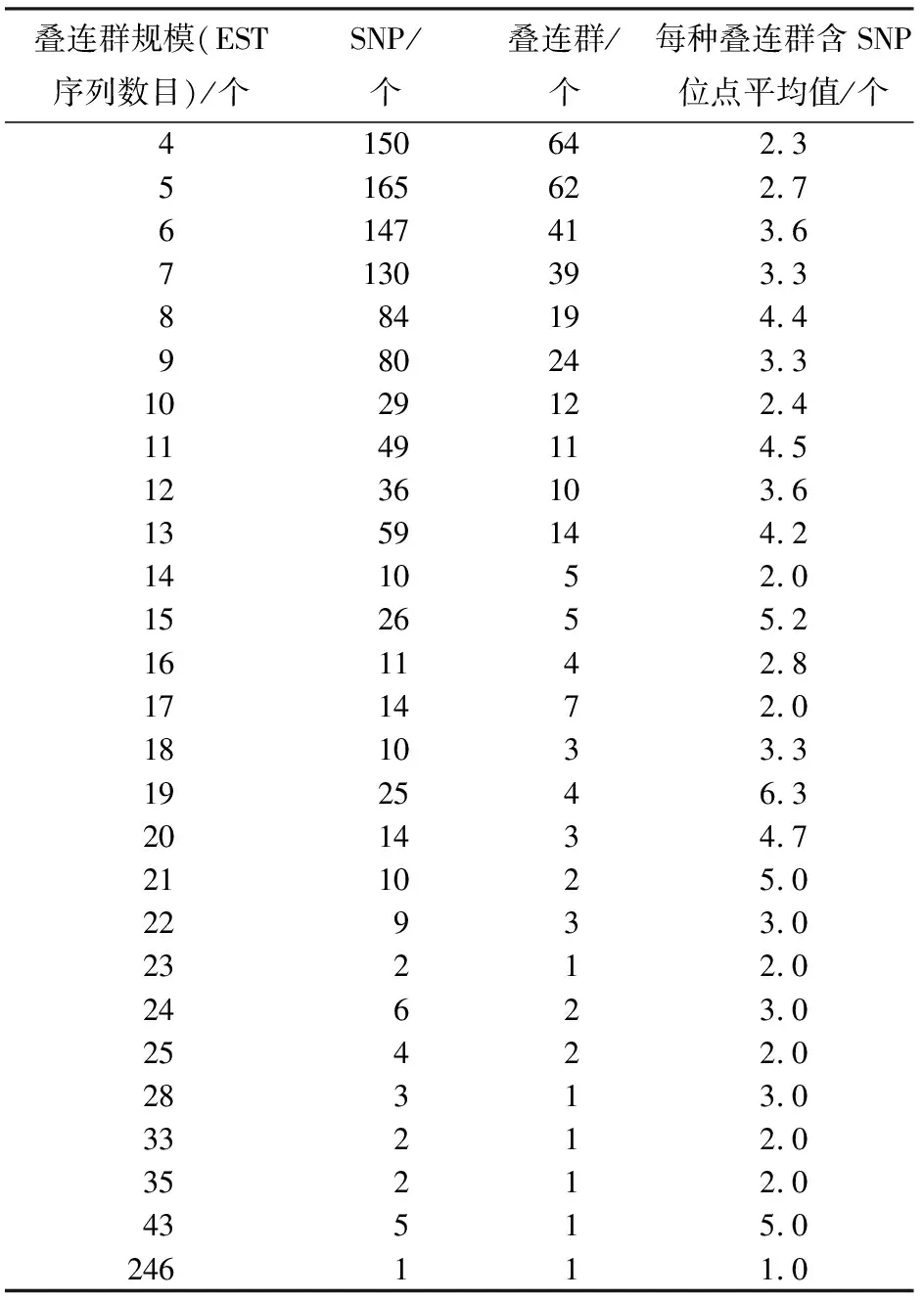
表3 叠连群规模与SNP位点数目的关系

表4 SNP候选位点类型
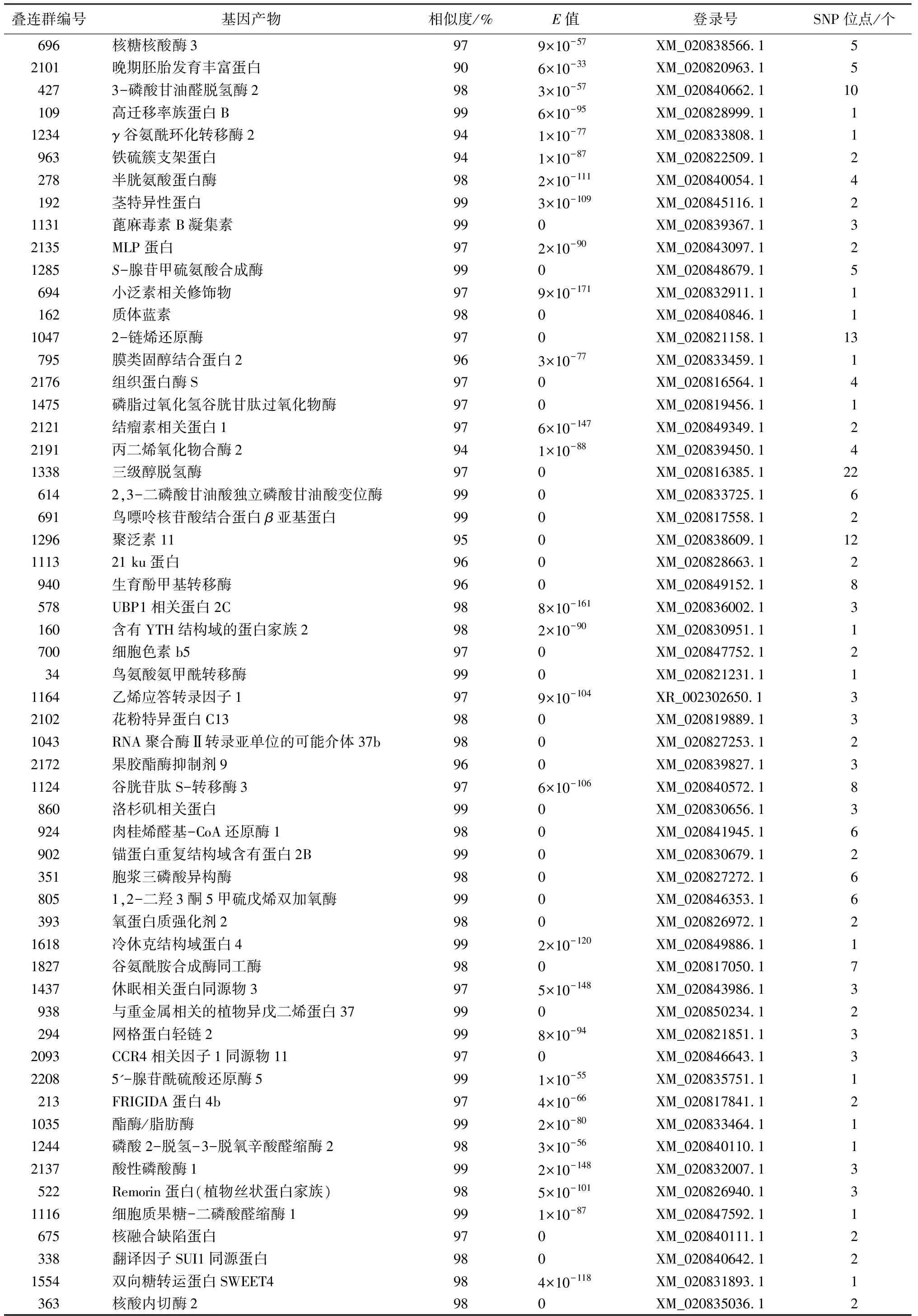
表5 同源比对位于铁皮石斛核酸序列的280个石斛SNP位点
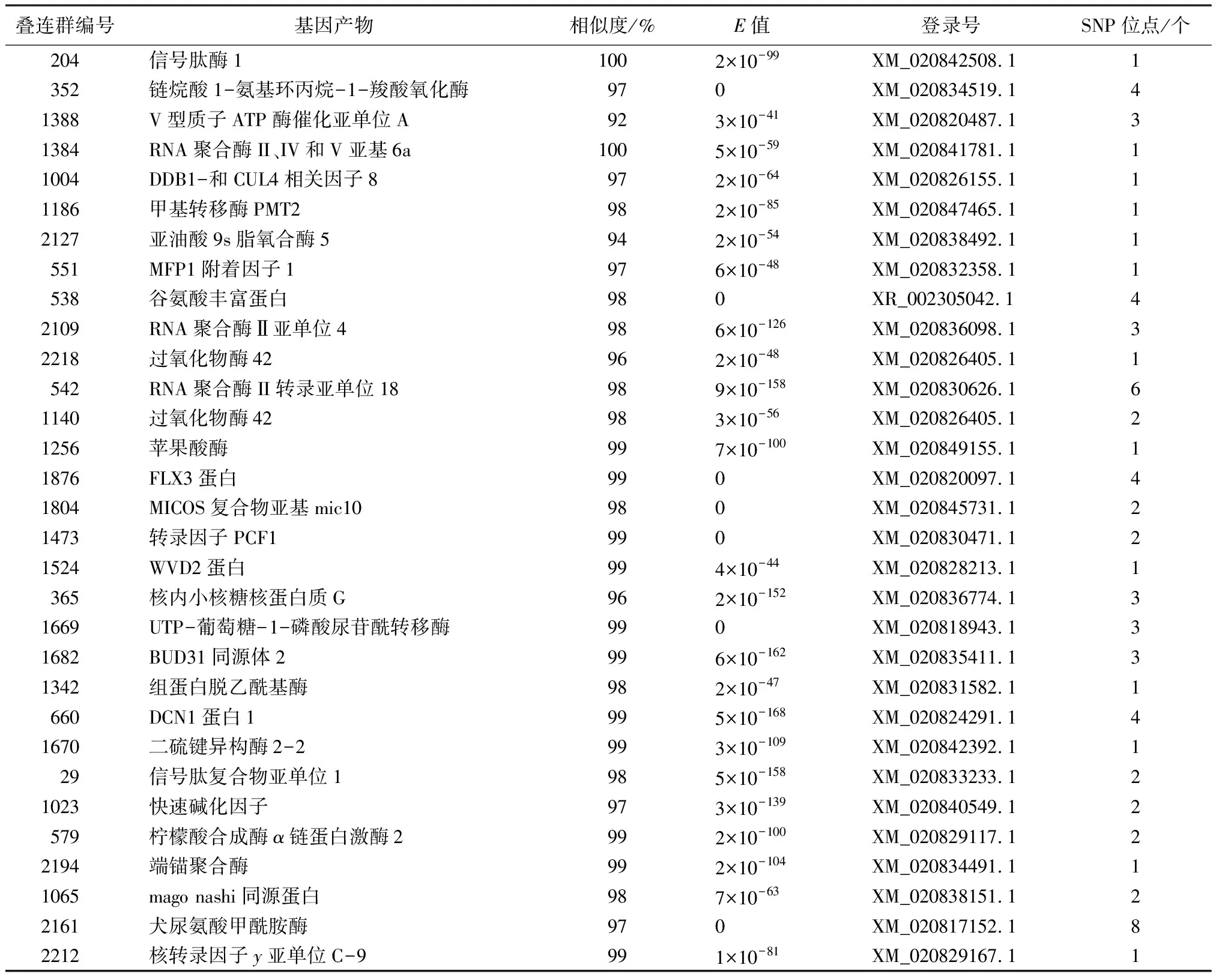
续(表5)

表6 同源比对位于铁皮石斛核酸序列的741个石斛SNP位点

续(表6)
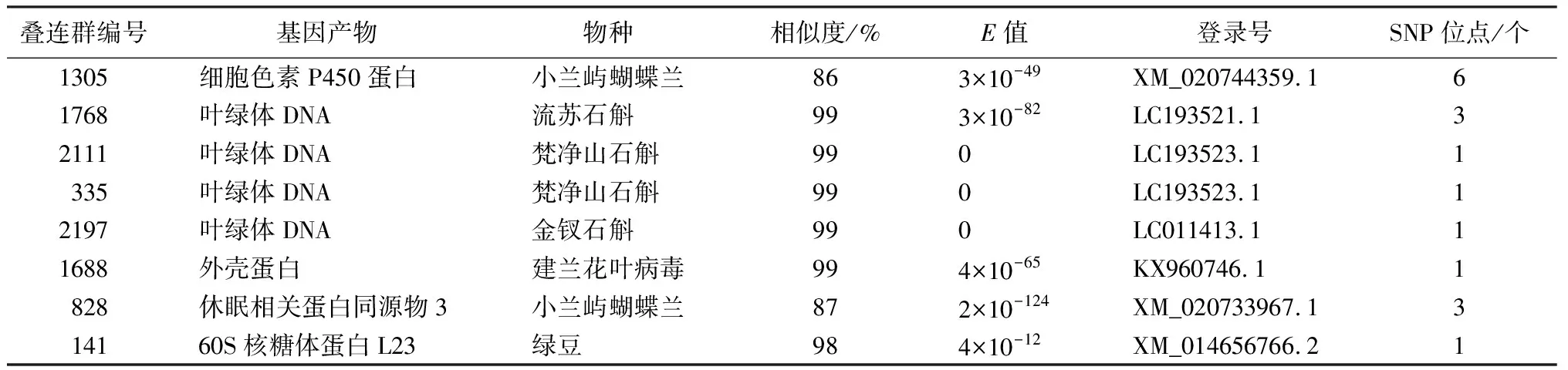
表7 不与铁皮石斛同源的SNP位点所在核酸序列比对
1 021个SNP位点所在核酸序列与铁皮石斛的302个基因同源,其中有相同基因产物或基因所在细胞位置一致的总结归纳后详见表3。经统计共有280个SNP位点分别与铁皮石斛中的88条相关蛋白基因序列存在高度同源,同源性均在90%以上,113个SNP位点所在核酸序列与25条铁皮石斛的未知功能基因序列具有较高的同源性,但基因产物还待进一步验证,有22个SNP位点所在核酸序列与铁皮石斛线粒体的6个核酸序列具有较高同源性,99个SNP位点所在核酸序列与26条叶绿体基因序列同源性较高,剩余507个SNP位点所在核酸序列分别与157条铁皮石斛中某些酶基因或代谢活动相关酶基因序列存在高度同源,同源性均在89%以上。
3 结论与讨论
SNP是动植物基因中广泛、随机分布的一种可遗传变异,具有很多独特的优点,自问世以来不断取得研究者的重视。利用公共数据库的已知EST序列进行SNP位点的开发不仅可以降低成本、快捷高效,而且开发得到的SNP位点还可能位于转录基因的功能区域,对于物种的遗传多样性分析、品种鉴定和遗传育种都具重要意义。目前利用EST序列进行SNP位点开发已经广泛应用于甘蔗(SaccharumofficinarumL.)[11]、枇杷(EriobotryajaponicaLindl.)[12]、葡萄(VitisviniferaL.)[9]、板栗(CastaneamollissimaBL.)[13]、菊花(Dendranthemamorifolium(Ramat.) Tzvel.)[14]、玫瑰(RosarugosaThunb.)[15]、梅(ArmeniacamumeSieb.)、杏(ArmeniacavulgarisLam.)、桃(AmygdaluspersicaL.)[16],芸薹属(Brassica)[17]等多种植物中,但在对石斛的EST-SNP开发方面还未见报道。本研究利用dbEST数据库中的16 183条石斛EST序列,采用SeqMan拼接,最终统计得出石斛的SNP位点平均出现频率为0.17%,SNP发生频率与菊花的SNP发生频率比较接近,与其他植物相比相对较低[11-13,15-17],这主要是因为不同植物的遗传背景差异不同造成的,遗传背景差异性越大,SNP发生频率越高[18]。另有研究表明,SNP频率与EST来源的品种数量呈相关性,开发的EST序列来源的品种数目越多,SNP位点开发频率就越高[19]。目前对石斛的基因组学研究较少,dbEST数据库中的EST序列品种来源仅有两个,与菊花EST序列来源品种一致,这也是造成在石斛中开发出的SNP频率与菊花相似的重要原因。
进行软件或人工筛选开发EST-SNP位点过程中,筛选原则是影响筛选结果准确度和候选SNP位点阳性率高低的关键因素,而EST来源的品种数量对SNP开发频率有重要影响[20]。研究者在甘蔗[11]、玫瑰[15]中对采用的葡萄SNP位点筛选原则进行了改良,筛选SNP位点的候选叠连群规模提高到20,从而提高SNP候选位点的阳性率,但他们的研究结果显示这种修改仅适于EST序列品种来源丰富的物种,且容易漏掉部分候选SNP位点。故本研究基于dbEST数据库中现有的石斛EST序列品种来源较少,EST序列较少的情况,完全参照在葡萄[9]上经试验验证的筛选原则对石斛SNP位点进行预测,在降低SNP位点假阳性率的同时提高候选SNP位点数量,以便为后期开展验证试验提供充足的可选位点。
数据统计分析显示,预测的石斛SNP候选位点含量超过3个的叠连群仅占所有获取的叠连群的30.93%,超过60%的叠连群包含的候选SNP位点不丰富,推测可能与本试验中获取的石斛EST序列的来源品种数量较少和遗传背景差异较小有关。此外,本研究发现预测的石斛SNP候选位点总数及包含SNP候选位点的相同规模叠连群数量随叠连群规模增大而呈下降趋势,这一趋势与已报道的葡萄[9]、芸薹属[17]植物中预测的SNP位点及其叠连群数量与叠连群规模的相关性一样,而与甘蔗[11]、大麦(HordeumvulgareL.)[20]等禾本科植物中的同类研究结果差异较大;获得的石斛叠连群中包含的SNP位点平均数与叠连群规模之间未显示任何相关性,该结果与玫瑰[15]的SNP位点预测分析结果相似,但有别于菊花[14]、葡萄[9]、芸薹属[17]植物的同类研究结论,故推测不同物种SNP位点在基因上的分布可能存在较大差异。
对预测的石斛候选SNP位点进行归类,结果显示预测的石斛EST-SNP以转换类型为主,颠换次之,这与葡萄[9]、甘蔗[11]、菊花[14]、玫瑰[15]等已报道候选SNP位点归类分析研究结论相似,但本研究发现预测的石斛SNP位点的插入或缺失突变率低于葡萄[9]、甘蔗[11]、菊花[14]、玫瑰[15]等,这可能与本研究所选取的石斛EST序列来源品种的数量较少、遗传背景差异不明显有关。此外,本研究结果显示石斛SNP位点的C-T转换率为37.0%,高于A-G转换频率(23.5%),推测可能是由于C在生物体中多以甲基化形式存在,容易脱氨后转换为T,进而导致了转换类型高于颠换类型[21]。
筛选的1 063个置换与颠换类型的SNP候选位点中有1 038个SNP位点被注释到398个基因上,但有25个基因属于铁皮石斛未知基因产物基因序列,需要进一步验证其功能。另外有25个SNP位点所在的10条核酸序列未被注释,需要进一步验证其全长mRNA和基因功能。
采用合理的人工筛选原则从提取的16 183条石斛EST序列得出1 083个SNP候选位点,为提高候选SNP位点的阳性率,只筛选了叠连群规模为4以上的EST序列,另外72.6%的叠连群并未参与筛选,这些叠连群中也可能存在着潜在的SNP位点,需采用测序等其他手段进行开发。接下来本研究将根据已获得的研究结果设计相应的SNP位点引物,采用测序、荧光定量或酶切手段进一步验证候选SNP位点的可靠性,为石斛的多样性分析、品种鉴定及遗传育种提供参考。
参 考 文 献
[1] 吴永升,邹成林,黄爱花,等.玉米自交系遗传关系及应用潜势分析[J].西南农业学报,2014,27(3):955-959.
[2] TREBBI D, MACCAFERRI M, DE HEER P, et al. High-throughput SNP discovery and genotyping in durum wheat (TriticumdurumDesf.)[J]. Theoretical and Applied Genetics,2011,123(4):555-569.
[3] JIANG D, YE Q L, WANG F S, et al. The mining of citrus EST-SNP and its application in cultivar discrimination[J]. Agricultural Science in China,2010,9(2):179-190.
[4] SINGH A, SINGH P K, SINGH R, et al. SNP haplotypes of the BADH1 gene and their association with aroma in rice (OryzasativaL.)[J]. Molecular Breeding,2010,26(2):325-338.
[5] KIM S, MISRA A. SNP genotyping: technologies and biomedical applications[J]. Annual Review of Biomedical Engineering,2007,9:289-320.
[6] 陈全求,詹先进,蓝家样,等.EST分子标记在基因组学中应用的研究进展[J].中国农学通报,2010,26(3):59-63.
[7] LEIN W, USADEL B, STITT M, et al. Large scale phenotyping of transgenic tobacco plants (Nicotianatabacum) to identify essential leaf functions[J]. Plant Biotechnology Journal,2008,6(3):246-263.
[8] 栗丹,李振坚,毛萍,等.基于ITS序列石斛材料的鉴定及系统进化分析[J].园艺学报,2012,39(8):1539-1550.
[9] 李猛,郭大龙,刘崇怀,等.葡萄EST-SNP位点的信息与特征[J].浙江大学学报(农业与生命科学版),2012,38(3):263-270.
[10] WANG S, SHA Z, SONSTEGARD T S, et al. Quality assessment parameters for EST-derived SNPs from catfish[J]. BMC Genomics,2008,9.Doi.10.1186/1471-2164-9-450.
[11] 檀小辉,张继,梁芳,等.基于EST序列的甘蔗SNP发掘分析[J].江苏农业科学,2016,44(7):64-67.
[12] 王俊.枇杷(EriobotryajaponicaLindl.)SNP位点筛选及遗传多样性分析[D].重庆:西南大学,2013.
[13] 刘伟,康明,黄宏文.中国板栗EST-SNP和抗栗疫病候选基因分析及同源比对[J].植物科学学报,2012,30(1):55-63.
[14] 万志兵,王言言,陈黎.菊花EST-SNP的发掘与特征分析[J].东北林业大学学报,2013,41(5):84-86,90.
[15] 梁芳,张继,吕平,等.基于EST序列的玫瑰EST-SNP位点发掘与分析[J].南方农业学报,2016,47(3):325-331.
[16] 李晓颖,王玉柱,上官凌飞,等.梅、杏、桃EST同源序列特征分析及EST-SNP发掘[J].南京农业大学学报,2012,35(4):47-53.
[17] 李雪姣,张耿,顾爱侠,等.芸薹属作物EST-SNP的发掘与分析[J].植物遗传资源学报,2010,11(6):772-776.
[18] VAN TASSELL C P, SMITH T P, MATUKUMALLI L K, et al. SNP discovery and allele frequency estimation by deep sequencing of reduced representation libraries[J]. Nature Methods,2008,5(3):247-252.
[19] DONG Q H, CAO X, YANG G, et al. Discovery and characterization of SNPs in Vitis vinifera and genetic assessment of some grapevine cultivars[J]. Scientia Horticulturae,2010,125(3):233-238.
[20] DURAN C, APPLEBY N, VARDY M, et al. Single nucleotide polymorphism discovery in barley using autoSNPdb[J]. Plant Biotechnology Journal,2009,7(4):326-333.
[21] VEZZULLI S, MICHELETTI D, RIAZ S, et al. A SNP transferability survey within the genus Vitis[J]. BMC Plant Biology,2008,8.Doi.1186/1471-2229-8-128.
