43个SNP遗传标记复合检验体系的建立及其法医学应用
2018-06-04李亚男栾晓辉徐倩男张家硕唐铭池边英男陈丽琴
李亚男 ,李 敏 ,姜 磊 ,栾晓辉 ,梁 娜 ,徐倩男 ,张家硕 ,5,唐铭池 ,边英男 ,陈丽琴
(1.内蒙古医科大学基础医学院,内蒙古 呼和浩特 010030;2.司法鉴定科学研究院 上海市法医学重点实验室 上海市司法鉴定专业技术服务平台,上海 200063;3.温州医科大学法医学系,浙江 温州 325035;4.基纳生物技术(上海)有限公司,上海 200032;5.苏州大学医学部法医学系,江苏 苏州 215123;6.哈尔滨市公安局道里分局刑事技术大队,黑龙江 哈尔滨 150070)
单核苷酸多态性(single nucleotide polymorphism,SNP)遗传标记因其突变率低、扩增子短等优势而备受法医遗传学界关注,有望在高度降解检材检验、特殊亲权关系鉴定和表型特征推断等方面发挥独特作用[1-11]。常见的SNP分型检验方法有SNaPshot微测序[12]、TaqMan 探针[13]、DNA 芯片[14]、焦磷酸测序[15]、高通量测序[16]和DNA质谱[17-18]等技术,这些方法在各具优势的同时,也存在着检测通量不足、操作步骤繁琐或者实验成本高昂等劣势。但是,DNA质谱技术因操作简便、成本低廉、结果准确等优点,被广泛应用于临床和科研领域的SNP分型检验中,并且美国Agena Bioscience公司还推出了可用于个人身份识别和样本追踪的iPLEX®Sample ID Plus Panel试剂盒。JOHANSEN等[17]的研究显示,该试剂盒具有一定的法医学应用前景。然而,该试剂盒包含的45个常染色体SNP位点尚无法满足我国法医DNA鉴定标准中对系统效能的要求[19]。为此,本研究特针对我国人群,结合实践需求,筛选并建立了基于DNA质谱技术的43重SNP复合检验体系,并对其在华东地区汉族人群中的法医学应用进行评估,旨在与iPLEX®Sample ID Plus Panel试剂盒配合使用,为法医学应用提供基础数据。
1 材料与方法
1.1 样本
本研究共收集了123例华东地区汉族健康无关个体的血液样本,其中男性36例,女性87例。所有样本的采集均遵循知情同意原则。
1.2 主要试剂及仪器
QIAamp®DNA Blood Mini试剂盒(德国QIAGEN公司),Complete iPLEX®Pro Genetyping Reagent Set 10×96试剂盒(美国Agena Bioscience公司)。
9700型PCR仪(美国AB公司),MassARRAY®飞行时间质谱仪、MassARRAY®RS1000纳升点样仪(美国Agena Bioscience公司),旋转混匀仪(杭州米欧仪器有限公司)。
1.3 实验方法
1.3.1 DNA提取
按照QIAamp®DNA Blood Mini试剂盒说明书推荐流程提取每名志愿者200μL血液样本中的DNA,并保存于-20℃备用。
1.3.2 位点选择、优化和复合扩增体系设计
根据文献[1,6,7,20-22]报道和本实验室研究积累,选择在中国汉族人群中最小等位基因频率(minor allele frequency,MAF)大于 0.2、符合 Hardy-Weinberg 平衡并且任意两标记间距离大于5 Mb的位点作为候选标记。根据美国Agena Bioscience公司MassARRAY Typer 4.0软件的设计结果,经人工调整,获得43个SNP位点的复合扩增体系(表1)。引物组合比例及反应体系均按照Complete iPLEX®Pro Genetyping Reagent Set 10×96试剂盒推荐比例配制。
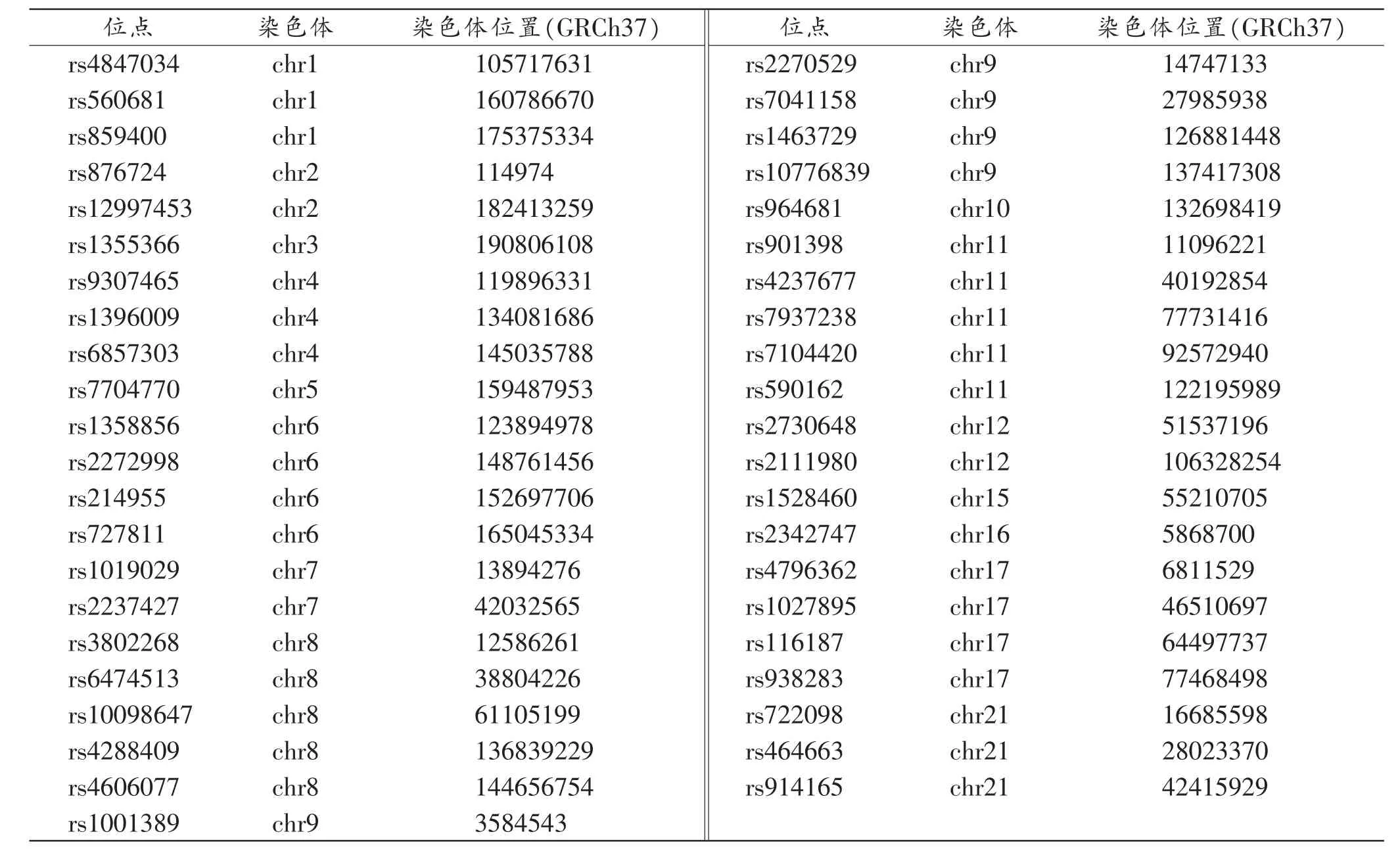
表1 复合扩增体系所包含的43个SNP位点及其染色体位置
1.3.3 PCR扩增
扩增体系:10×缓冲液 0.5 μL、25 mmol/L MgCl20.4μL、25mmol/L dNTP 0.1μL、扩增引物预混液 0.5μL、5U/μL扩增酶0.1μL和DNA模板10ng,超纯水补充至 5μL。
扩增条件:95℃预变性 2min;95℃ 30s,56℃ 30s,72℃ 60s,45个循环;72℃延伸 5min。
1.3.4 dNTP消化
反应体系:在扩增产物中加入虾碱性磷酸酶(shrimp alkaline phosphatase,SAP) 0.3 μL、SAP 缓冲液0.17μL和超纯水1.53μL。
反应条件:37℃孵育40min后,85℃热失活SAP。
1.3.5 单碱基延伸
反应体系:在消化反应产物中加入10×Complete iPLEX®Pro Buffer Plus 0.2μL、Complete iPLEX®Pro Termination Mix 0.2μL、单碱基引物预混液 0.94μL、Complete iPLEX®Pro酶0.04μL和超纯水0.62μL。
反应条件:94℃预变性 30 s;95℃ 5 s,52℃ 5 s,80℃ 5s,52℃ 5s,80℃ 5s,52℃ 5s,80℃ 5s,52℃5s,80℃ 5s,52℃ 5s,80℃ 5s,40 个循环;72℃延伸3min。
1.3.6 产物脱盐
在单碱基延伸后的反应产物中加入超纯水41μL和预先风干的洁净树脂15 mg,置于旋转混匀仪上孵育并温和混匀15min,然后以4400×g离心5min。
1.3.7 芯片上样
使用MassARRAY®RS1000纳升点样仪将脱盐后的反应产物上样至Spectro CHIP Array 96点芯片上,通过调节点样针与芯片的时间和点样针的移动速度使上样体积在8~12μL范围内。
1.3.8 飞行时间质谱检验
使用MassARRAY®飞行时间质谱仪对上样后的芯片进行检测,通过MassARRAY Typer 4.0软件查看分型结果。
1.4 数据分析
个体识别率(DP)的计算公式:

式中,Pi为该群体第i个表型的频率。
多态信息含量(PIC)的计算公式:
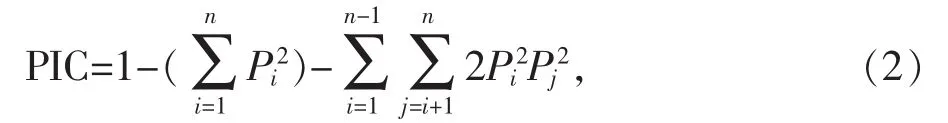
式中,n为等位基因数,Pi和Pj分别为第i个和第j个等位基因的频率。
观察杂合度(Ho)的计算公式:

预期杂合度(He)的计算公式:

式中,n为等位基因数,k为等位基因种类的数目,Pi为第i个等位基因的频率。
三联体非父排除率(PEtrio)的计算公式:
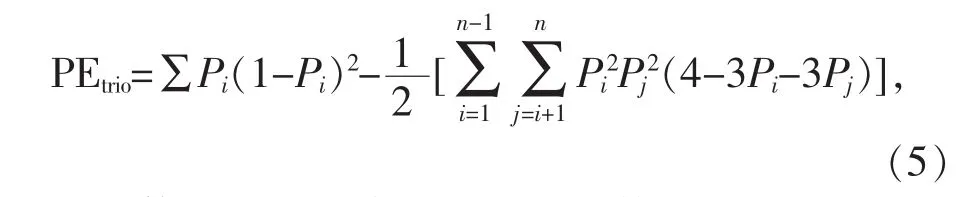
二联体非父排除率(PEduo)的计算公式:
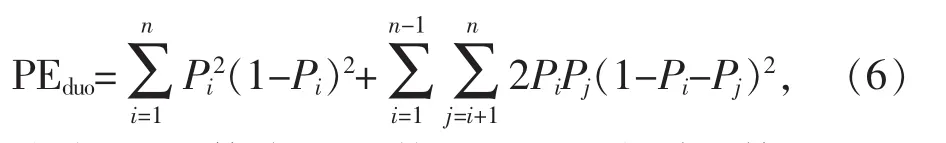
式中,n为等位基因数,Pi和Pj分别为第i个和第j个等位基因的频率。
累积非父排除率(CPE)的计算公式:

式中,PEk为第k个遗传标记的PE值。
累积个体识别率(CDP)的计算公式:

式中,Pj为第j个等位基因的Pm值,∏Pj为第k个遗传系统的总Pm值。
2 结 果
2.1 基因分型
本研究构建了包含43个SNP位点的复合检验体系。对123例样本的分型结果显示,除rs10776839位点的分型成功率不足65%以外,其余位点的分型成功率均超过97.56%。
2.2 等位基因频率及其多样性
43个SNP位点的等位基因频率分布见表2。除rs1355366、rs2270529、rs10776839 和 rs938283 外,其余39个位点的MAF值均大于0.25,其中MAF值为0.4~0.5的高多态性位点有24个。此外,rs560681、rs7041158和rs914165 3个位点的MAF值也非常接近0.4。
2.3 法医学评价
经χ2检验,123名华东地区汉族健康无关个体在43个SNP位点上均符合Hardy-Weinberg平衡(P〉0.05)。
43个SNP位点的群体遗传学参数见表3。其中DP 为 0.290 1~0.654 4,CDP 为 1-9.8×10-11,PIC 为0.1708~0.5000,Ho 为 0.1557~0.5935,PEtrio为 0.0854~0.2500,PEduo为 0.0146~0.1250。 各 SNP 位点之间相互独立,采用乘积原则计算,CPEtrio为0.999 986,CPEduo为0.9924361。
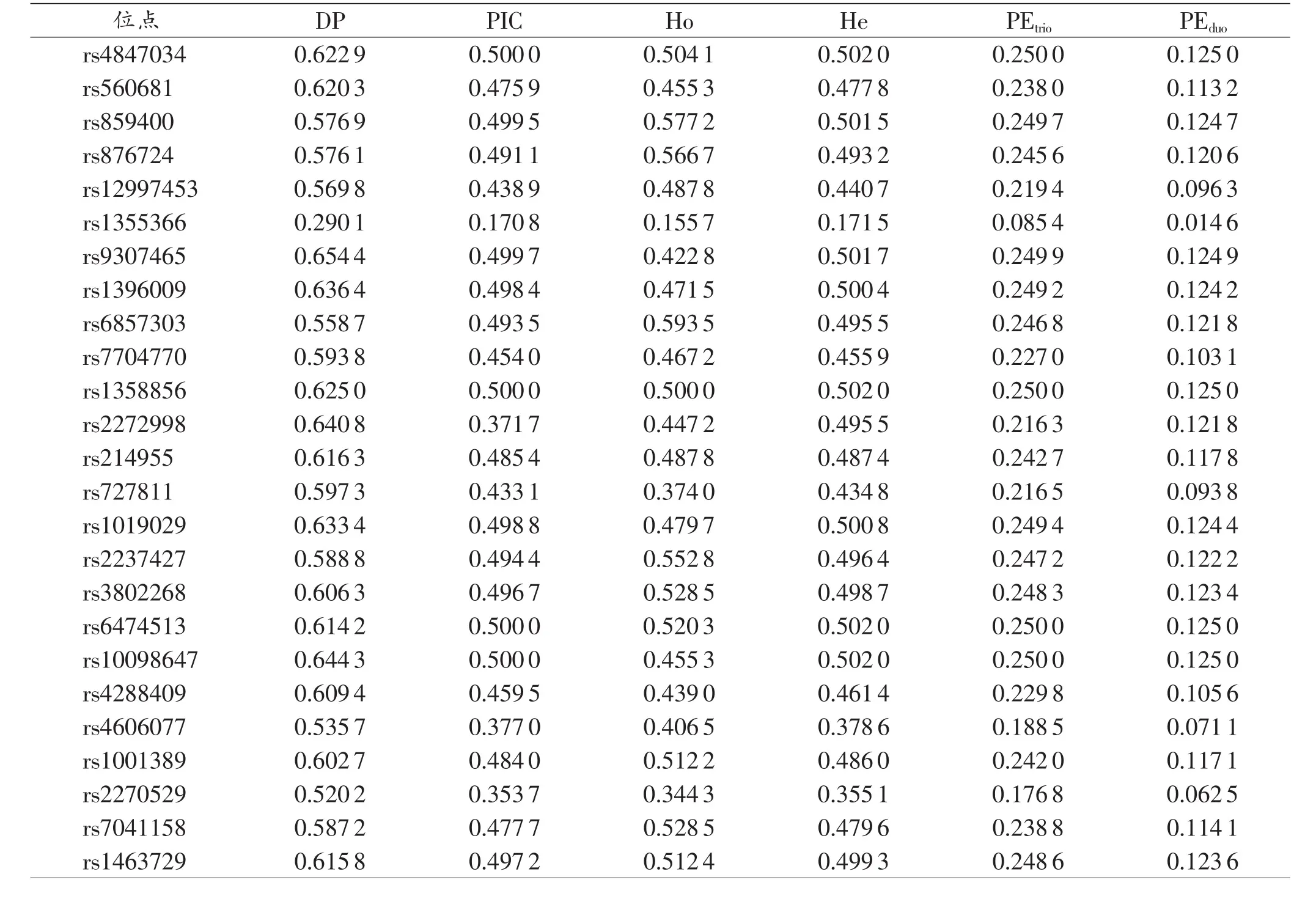
表3 43个SNP位点的群体遗传学参数 (n=123)
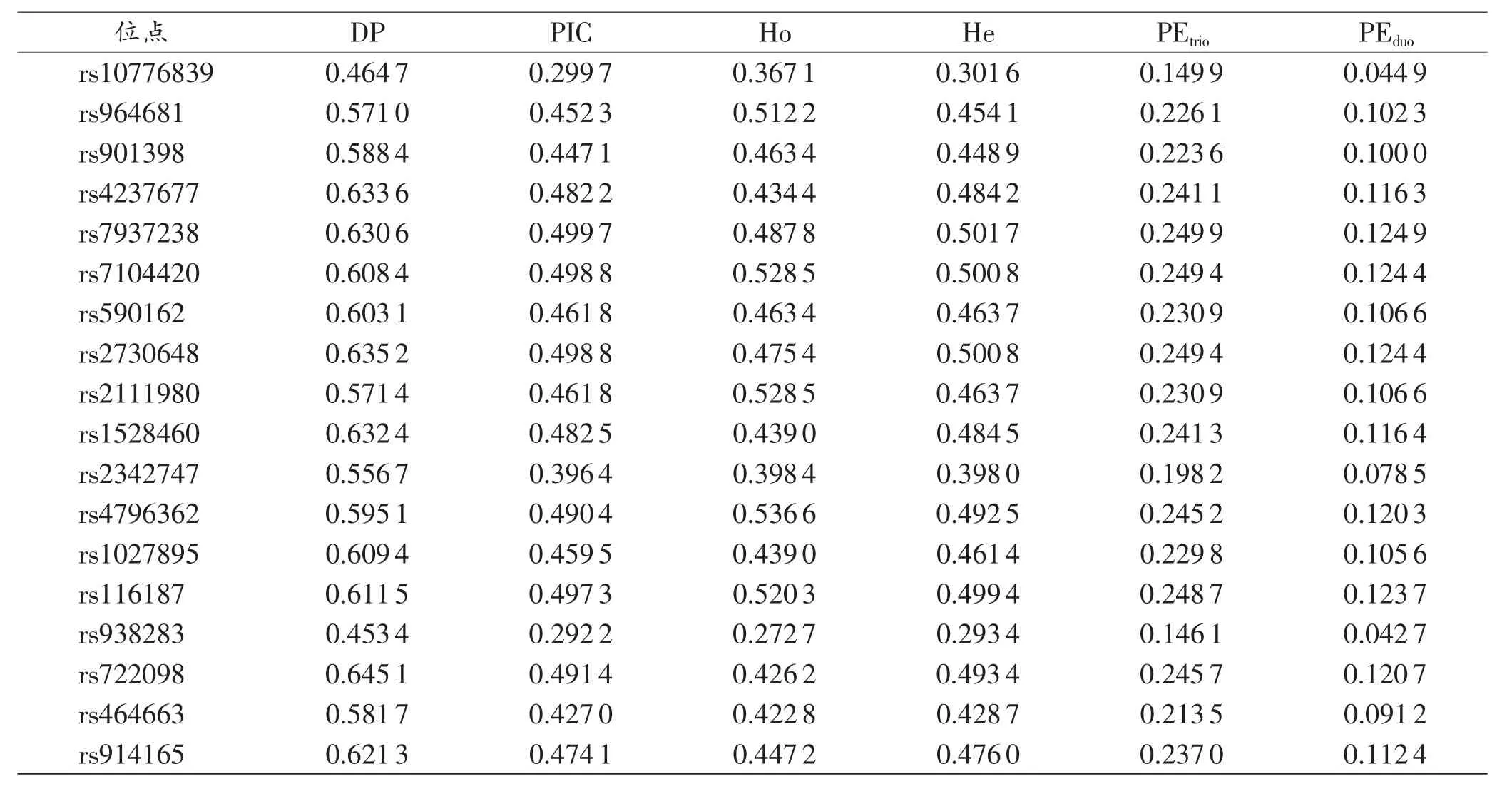
表3(续)
3 讨 论
目前,虽然短串联重复序列(short tandem repeat,STR)遗传标记被广泛应用于国内外法医物证学鉴定[23]中,但是其在实际应用中存在明显的缺陷。首先STR在遗传过程中具有较高的突变率[24-25],容易导致无法判断亲子关系或错误判断亲子关系的情况。其次,对于高度降解检材,基于毛细管电泳的STR检验可能无法获得全部基因座的完整分型结果,即使配合使用miniSTR试剂盒,也要求检材DNA长度在150 bp以上[26]。因此,针对STR遗传标记的不足,SNP遗传标记因其突变率低和检验所需扩增子短的特点,对于降解检材和亲子关系的检测具有独特的优势,正成为法医遗传学的研究热点。虽然已经有一些针对SNP遗传标记的检验系统被开发出来,但是因检测通量不足、操作步骤繁琐或实验成本高昂等原因,不能满足法医物证学鉴定需要。DNA质谱技术因操作简便、成本低廉、结果准确等优点,而被广泛应用于临床和科研领域的SNP分型检验之中,并且已经有可用于个人身份识别和样本追踪的商品化试剂盒(iPLEX®Sample ID Plus Panel),因此本研究选择了基于DNA质谱技术平台的SNP复合检验系统,筛选并建立了43个SNP遗传标记复合检验体系。复合扩增遗传标记个数是决定检验体系通量、成本和简便程度的直接指标,本研究构建了一个在同一反应池内包含43个SNP位点的复合检验体系。实验结果显示,除rs10776839外,其余位点的分型结果均稳定可靠。与此前文献[27-30]报道的SNP检验系统相比,本系统的检验通量明显提高,虽不及高通量测序[16],但是已能在相当程度上满足日常检验需要。
本研究结果显示,rs1355366、rs10776839和rs938283的MAF值在本次检验的华东地区汉族人群中均小于0.2,我们将通过对更大人群样本的研究对其进行考察。为了提高本系统在实际检案中的应用能力,我们将对系统进一步优化,并考虑使用其他位点替换分型结果不稳定的rs10776839及MAF值较低的rs1355366和rs938283,并增加性别检验基因座。
43个 SNP位点的 DP在 0.290 1(rs1355366)~0.654 4(rs9307465),其中 40 个位点的 DP 大于 0.5,24个位点的DP大于0.6,43个SNP复合检验体系的CDP为1-9.8×10-11,表明其在华东汉族人群中具有较好的个体识别能力。
国际千人基因组计划(http://www.international genome.org/)的数据显示,iPLEX®Sample ID Plus Panel试剂盒所含45个常染色体SNP位点在我国北京汉族人群中MAF值介于0.4~0.5的高多态性位点仅有18个。本研究筛选并建立的43个SNP位点中MAF值为0.4~0.5的高多态性位点有24个。经计算,iPLEX®Sample ID Plus Panel试剂盒所含45个常染色体SNP位点的CPEtrio和CPEduo分别为0.999 910 6和0.984 529 8。本研究筛选并建立的43个SNP复合检验体系的CPEtrio和CPEduo分别为0.999 986和0.992 436 1,表明其在我国人群中具有良好的法医学应用前景。
我们在系统设计过程中,特别考虑到与iPLEX®Sample ID Plus Panel试剂盒联合使用,因此选择了5个与iPLEX®Sample ID Plus Panel试剂盒重复的位点,分别为 rs876724、rs1463729、rs964681、rs901398和rs914165,作为联合使用时的质量控制位点,并在此基础上选择了38个符合乘积原则的高多态性独立位点。因此,本体系可与iPLEX®Sample ID Plus Panel试剂盒联合使用,其系统效能已经达到或接近于行业标准对个体识别和亲子鉴定的要求[19]。因此,本系统可以用于个体识别或三联体亲子关系鉴定,也可用作对STR标记系统的补充和验证,特别是对于特殊情况下的高度降解检材和出现不符合遗传学规律的亲子鉴定。
本研究针对我国人群建立了基于DNA质谱技术的43重SNP复合检验体系,并对其在华东地区汉族人群中的法医学应用进行了评估。实验结果显示,该系统具有较高的遗传多态性,既可以作为传统STR遗传标记的有效补充和验证,也可以与其他商品化试剂盒配合使用,以应对高降解检材和出现不符合遗传规律现象亲子鉴定的挑战。本课题组将在今后的工作中继续对位点进行优化并对体系极端适用条件进行测试,以期获得可用于法医物证鉴定实践的SNP检测系统。
[1]SANCHEZ J J,PHILLIPS C,BØRSTING C,et al.A multiplex assay with 52 single nucleotide polymorphisms for human identification[J].Electrophoresis,2006,27(9):1713-1724.
[2]INAGAKI S, YAMAMOTO Y, DOI Y, et al.A new 39-plex analysis method for SNPs including 15 blood group loci[J].Forensic Sci Int,2004,144(1):45-57.
[3]LEE H Y,PARK M J,YOO J E,et al.Selection of twenty-four highly informative SNP markers for human identification and paternity analysis in Koreans[J].Forensic Sci Int,2005,148(2-3):107-112.
[4]DIXON L A,MURRAY C M,ARCHER E J, et al.Validation of a 21-locus autosomal SNP multiplex for forensic identification purposes[J].Forensic Sci Int,2005,154(1):62-77.
[5]LI L, LI C T, LI R Y, et al.SNP genotyping by multiplex amplification and microarraysassay for forensic application[J].Forensic Sci Int,2006,162(1-3):74-79.
[6]PAKSTIS A J,SPEED W C,FANG R, et al.SNPs for a universal individual identification panel[J].Hum Genet,2010,127(3):315-324.
[7]LOU C, CONG B, LI S, et al.A SNaPshot assay for genotyping 44 individual identification single nucleotide polymorphisms[J].Electrophoresis,2011,32(3-4):368-378.
[8]WALSH S, LIU F, BALLANTYNE K N, et al.IrisPlex:a sensitive DNA tool for accurate prediction of blue and brown eye colour in the absence of ancestry information[J].Forensic Sci Int Genet,2011,5(3):170-180.
[9]WALSH S, LIU F, WOLLSTEIN A, et al.The HIrisPlex system for simultaneous prediction of hair and eye colour from DNA[J].Forensic Sci Int Genet,2013,7(1):98-115.
[10]RUIZ Y,PHILLIPS C,GOMEZ-TATO A,et al.Further development of forensic eye color predictive tests[J].Forensic Sci Int Genet,2013,7(1):28-40.
[11]FONDEVILA M,PHILLIPS C,SANTOS C,et al.Revision of the SNPforID 34-plex forensic ancestry test: Assay enhancements, standard reference sample genotypes and extended population studies[J].Forensic Sci Int Genet,2013,7(1):63-74.
[12]HAKENBERG J, VORONOV D, NGUYÊN V H,et al.A SNPshot of PubMed to associate genetic variants with drugs, diseases, and adverse reactions[J].J Biomed Inform,2012,45(5):842-850.
[13]DE LA VEGA F M,LAZARUK K D,RHODES M D,et al.Assessment of two flexible and compatible SNP genotyping platforms:TaqMan SNP Genotyping Assays and the SNPlex Genotyping System[J].Mutat Res,2005,573(1-2):111-135.
[14]HO C C, MUN K S, NAIDU R.SNP array technology:an array of hope in breast cancer research[J].Malays J Pathol,2013,35(1):33-43.
[15]翟仙敦,刘之江,侯一平.焦磷酸测序技术及其法医学应用[J].中国司法鉴定,2009(4):39-42,63.
[16]ZHANG S,BIAN Y,CHEN A,et al.Developmental validation of a custom panel including 273 SNPs for forensic application using Ion Torrent PGM[J].Forensic Sci Int Genet,2017,27:50-57.
[17]JOHANSEN P,ANDERSEN J D, BØRSTING C, et al.Evaluation of the iPLEX®Sample ID Plus Panel designed for the Sequenom MassARRAY®system.A SNP typing assay developed for human identification and sample tracking based on the SNPforID panel[J].Forensic Sci Int Genet,2013,7(5):482-487.
[18]宋雨桐,李莉,张立男,等.MALDI-TOF-MS对Y染色体上66个二等位基因遗传标记的检测[J].法医学杂志,2017,33(3):239-243.
[19]侯一平.法医物证学[M].2版.北京:人民卫生出版社,2004.
[20]ZHANG S, BIAN Y, ZHANG Z, et al.Parallel analysis of 124 universal SNPs for human identification by targeted semiconductor sequencing[J].Sci Rep,2015,5:18683.
[21]HWA H L, WU L S, LIN C Y, et al.Genotyping of 75 SNPs using arrays for individual identification in five population groups[J].Int J Legal Med,2016,130(1):81-89.
[22]ZENG Z,WANG L,FENG Q,et al.Evaluation of 96 SNPs in 14 populations for worldwide individual identification[J].J Forensic Sci,2012,57(4):1031-1035.
[23]CHOI J Y,SEO T S.An integrated microdevice for high-performance short tandem repeat genotyping[J].Biotechnol J,2009,4(11):1530-1541.
[24]LU D,LIU Q,WU W,et al.Mutation analysis of 24 short tandem repeats in Chinese Han population[J].Int J Legal Med,2012,126(2):331-335.
[25]BRINKMANN B,KLINTSCHAR M,NEUHUBER F, et al.Mutation rate in human microsatellites: influence of the structure and length of the tandem repeat[J].Am J Hum Genet,1998,62(6):1408-1415.
[26]BUTLER J M,SHEN Y,MCCORD B R.The development of reduced size STR amplicons as tools for analysis of degraded DNA[J].J Forensic Sci,2003,48(5):1054-1064.
[27]DE LA PUENTE M,SANTOS C,FONDEVILA M,et al.The Global AIMs Nano set:A 31-plex SNaPshot assay of ancestry-informative SNPs[J].Forensic Sci Int Genet,2016,22:81-88.
[28]BIRDSELL D N, VOGLER A J, BUCHHAGEN J,et al.TaqMan real-time PCR assays for single-nucleotide polymorphisms which identify Francisella tularensis and its subspecies and subpopulations[J].PLoS One,2014,9(9):e107964.
[29]LIU Y,LIAO H,LIU Y,et al.Developing a new nonbinary SNP fluorescent multiplex detection system for forensic application in China[J].Electrophoresis,2017,38(8):1154-1162.
[30]BERTOIN F, LETOUZÉ E, GRIGNANI P, et al.Genome-wide paternal uniparental disomy as a cause ofBeckwith-Wiedemann syndromeassociated with recurrent virilizing adrenocortical tumors[J].Horm Metab Res,2015,47(7):497-503.
