利用HiTAIL-PCR技术鉴定T-DNA在RNAi-OsRhoGAP2水稻中的插入位点
2018-05-30葛慧雯李佳佳王高华闫鑫甜安文静杜京尧彭静静王美娜梁卫红
葛慧雯, 李佳佳, 王高华, 闫鑫甜, 安文静, 杜京尧, 石 佳, 彭静静, 王美娜, 梁卫红
(河南师范大学生命科学学院,河南新乡 453007)
近年来,随着农杆菌介导的遗传转化技术的发展,T-DNA插入技术已经广泛应用于水稻等模式植物转基因研究[1]。但是,在转基因植物的研究过程中发现外源基因在转基因植物中并不都能实现预期表达,其表达的稳定性问题有待解决[2]。已有研究报道,转基因植物中外源基因能否在植物基因组中稳定表达受到多种因素的影响,如外源基因自身的结构、启动子等[3-4]。此外,也与其插入位点的侧翼序列和染色体定位有关,外源基因插入到染色体的不同位置,其表达稳定性和表达效率可能也不同[5]。因此,鉴定外源基因的插入位点,了解其侧翼序列及染色体定位对于后续的基因功能研究有重要意义。目前T-DNA插入位点侧翼序列的鉴定多采用PCR扩增的方法,主要包括热不对称交错PCR法(thermal asymmetric interlaced-PCR,TAIL-PCR)[6]、反向PCR(inverse PCR,I-PCR)[7]和PCR-walking法[8]3种。
前期研究发现水稻ROP(Rho of plant)基因OsRacD与水稻育性相关[9],为研究其功能和作用机制,利用酵母双杂交技术,以OsRacD为诱饵筛选水稻幼穗cDNA文库,获得了若干功能未知基因[10-11],其中包括OsRhoGAP2。为了鉴定OsRhoGAP2基因的功能,本研究利用Gateway克隆技术,构建了OsRhoGAP2的RNA干扰植物表达载体pANDA-OsRhoGAP2,对水稻进行遗传转化,筛选获得阳性转基因植株,并发现转基因水稻株高显著高于对照水稻。为确定鉴定外源载体的整合位点,本研究采用改良的高效热不对称交错PCR法(high-efficiency thermal asymmetric interlaced PCR,HiTAIL-PCR)对RNAi-OsRhoGAP2转基因水稻T-DNA的侧翼序列进行分析,旨在为研究RNAi-OsRhoGAP2转基因水稻表型变化的分子基础提供依据。
1 材料与方法
1.1 植物材料
本研究所用植物材料包括水稻粳稻栽培品种日本晴(OryzasativaL.)野生型(简称WT)和笔者所在的实验室前期筛选获得的T1代OsRhoGAP2干扰转基因水稻植株(简称RNAi-OsRhoGAP2),遗传背景为日本晴。
1.2 质粒和菌株
大肠杆菌(Escherichiacoli)DH5α、根癌农杆菌(Agrobacteriumtumefaciens)EHA105由笔者所在的实验室保存;pAD-OsRhoGAP2来自酵母双杂交筛选,由笔者所在的实验室保存;pENTR/D gateway入门载体购自Invitrogen公司;RNAi干扰植物表达载体pANDA由山东省农业科学院谢先芝研究员馈赠。
1.3 供试试剂
SanPrep柱式DNA胶回收试剂盒购于生工生物工程(上海)股份有限公司;2×TaqMaster Mix购于北京康为世纪生物科技有限公司;2×VazymeLAmp®Master Mix购于南京诺唯赞生物科技有限公司;高保真PrimeSTAR®PrimeSTAR聚合酶、DNA Marker购于宝生物工程(大连)有限公司。
1.4 引物设计及合成
本试验所用引物(表1)均在苏州金唯智生物科技有限公司合成。

表1 引物序列及用途
注:V=A、C或G;N=A、C、T或G。
1.5OsRhoGAP2基因的RNA干扰载体的构建及水稻的遗传转化
将OsRhoGAP2基因的cDNA序列提交NCBI(http:// www.ncbi.nlm.nih.gov/),以Blastn进行检索,并用DNAMAN软件进行序列同源性比对。根据RNAi载体设计原则,选取OsRhoGAP2基因cDNA序列的3′端261 bp的特异区段作为RNA干扰片段。
根据OsRhoGAP2基因3′端的特异序列设计引物P1和P2。以pAD-OsRhoGAP2质粒为模板,用高保真PrimeSTAR®rHS DNA聚合酶扩增OsRhoGAP2基因的RNA干扰片段。PCR产物经琼脂糖凝胶电泳检测后,PCR产物参照SanPrep柱式DNA胶回收试剂盒步骤回收定量后,与pENTR/D gateway入门载体连接,将载体命名为pENTR/D-OsRhoGAP2。
将pANDA空载体和pENTR/DNOsRhoGAP2按比例混匀,将pENTR-OsRhoGAP2和pANDA载体进行LR反应后,转到DH5α感受态细胞中,将质粒命名为pANDA-OsRhoGAP2。
采用农杆菌介导法[12]将pANDA-OsRhoGAP2转化水稻愈伤,筛选阳性愈伤,诱导分化,获得再生植株,2016年5月10日移植到河南师范大学生物实验园地,鉴定后于同年10月收获T0代种子。
1.6 水稻基因组DNA的提取
将T0代转基因水稻种子,消毒后置于27 ℃恒温培养箱中培养7 d,获得T1代转基因水稻幼苗。取其叶片,液氮冷冻后,迅速研磨至粉末,然后以CTAB法[13]提取基因组DNA,经1%琼脂糖凝胶电泳检测质量。
1.7 转基因植株的PCR鉴定
按照2×TaqMaster Mix说明书配制PCR反应体系(20 μL):2×TaqMaster Mix(10 μL)、P3(0.8 μL)、P4(0.8 μL)、基因组DNA(0.4 μL)和无菌水(8 μL)。PCR反应程序为:94 ℃,2 min;94 ℃、30 s,55 ℃、30 s,72 ℃、30 s,共35个循环;72 ℃延伸5 min。扩增产物经1%琼脂糖凝胶电泳检测。
1.8 T-DNA在水稻基因组中插入位点的鉴定
采用HiTAIL-PCR技术扩增T-DNA侧翼序列,反应体系和反应程序见表2、表3。将第3轮反应产物经1%琼脂糖凝胶电泳检测后,送至苏州金唯智生物科技有限公司测序。
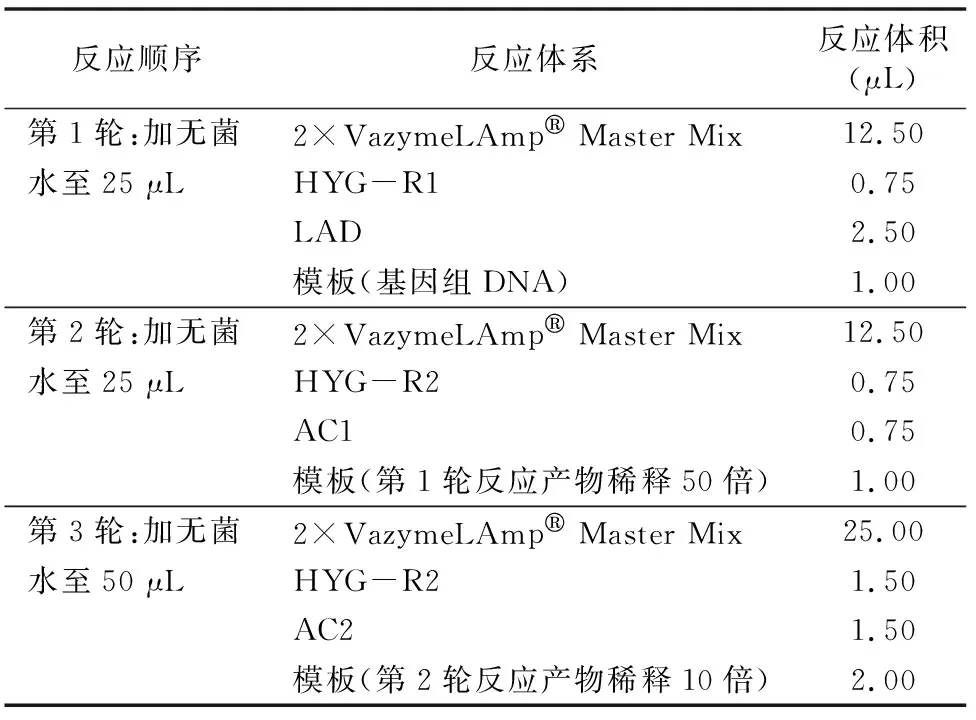
表2 HiTAIL-PCR反应体系
1.9 序列比对和分析
采用DNAMAN软件对测序结果进行序列分析,T-DNA在水稻基因组DNA上插入位点的分析采用Blast结合DNAMAN软件。
2 结果与分析
2.1OsRhoGAP2基因RNA干扰载体的构建
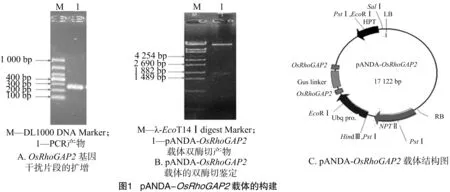
以pAD-OsRhoGAP2质粒为模板,用引物P1和P2进行PCR扩增OsRhoGAP2基因特异片段。电泳检测显示,扩增产物与预期的261 bp长度相符(图1-A),回收该扩增条带,连入pENTR/D gateway入门载体,筛选阳性克隆,并测序确认后,将其与pANDA载体进行LR反应,然后将反应产物转化到感受态细胞,筛选转化子,并从中提取重组载体。
采用KpnⅠ和SacⅠ双酶切鉴定重组载体,电泳结果显示,重组载体释放出1条1 800 bp左右的目的条带(图1-B),条带大小与预期一致,说明OsRhoGAP2基因RNA干扰载体构建成功(图1-C),进一步测序确认。

表3 HiTAIL-PCR反应程序
2.2 RNAi-OsRhoGAP2水稻的筛选及表型分析
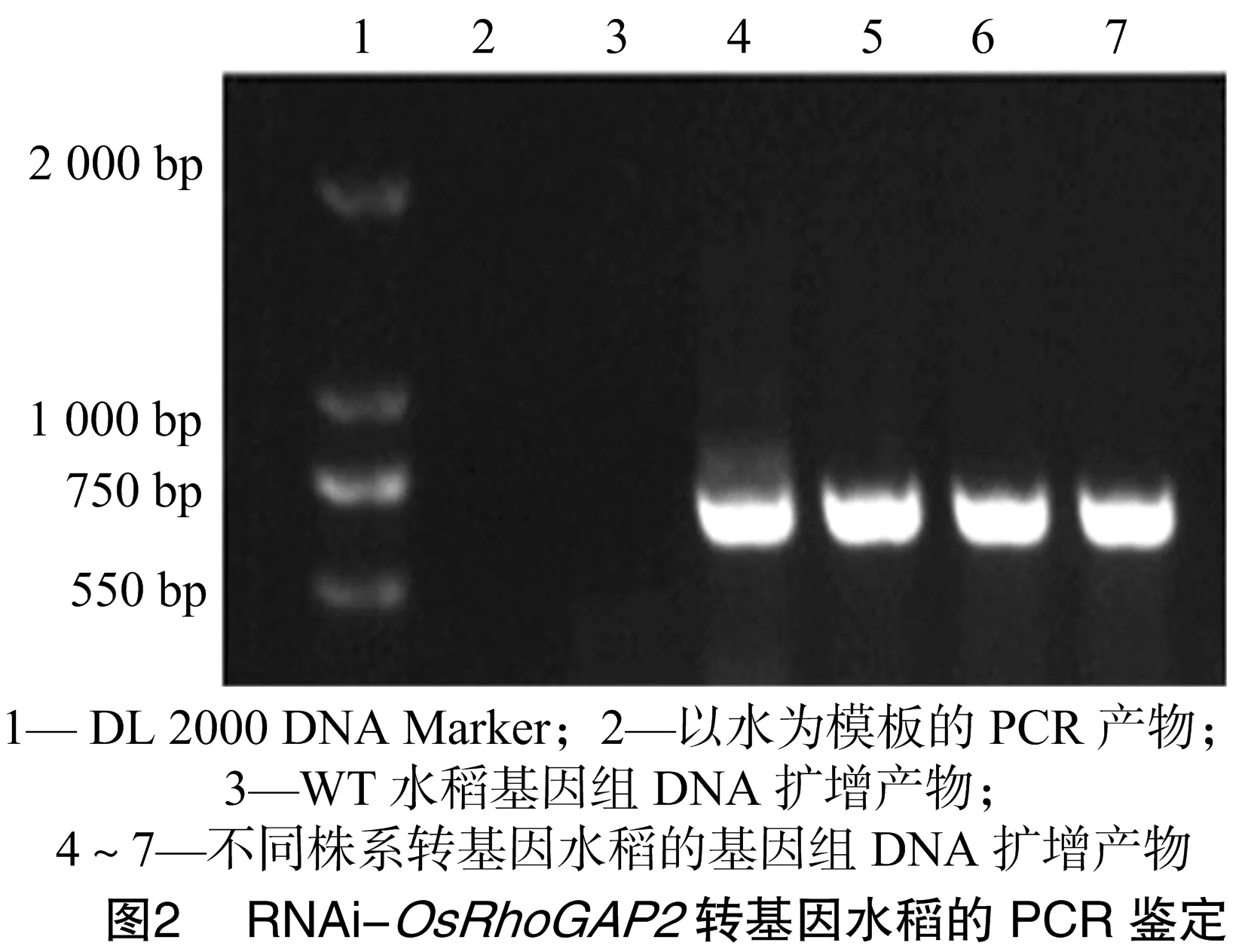
通过农杆菌介导法,将pANDA-OsRhoGAP2载体转入到水稻愈伤组织中,筛选潮霉素抗性愈伤,并进一步诱导分化为再生植株。提取再生水稻植株的基因组DNA,利用载体上序列设计的特异性引物P3和P4进行PCR扩增。电泳检测显示,所检测的4个不同株系的转基因植株均可以扩增出特异性片段,且片段长度与预期的638 bp大小相符(图2),而以水为模板的阴性对照,以及对照WT水稻中均未扩增出该条带,表明载体已经整合到水稻基因组中。比较转基因水稻和对照的表型,结果显示RNAi-OsRhoGAP2转基因植株的株高显著高于对照水稻(图3)。
2.3 RNAi-OsRhoGAP2水稻T-DNA侧翼序列的扩增
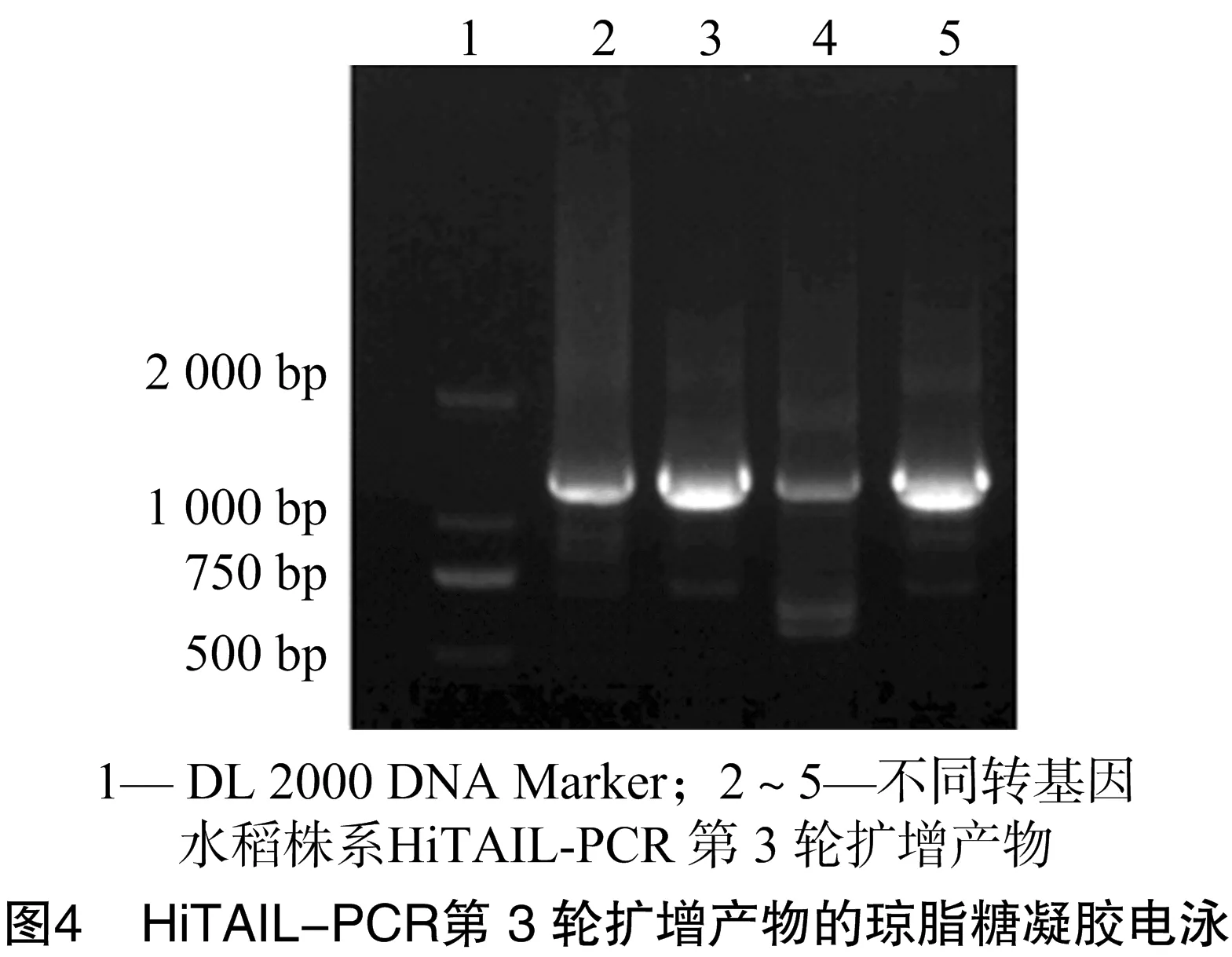
根据pANDA载体上的潮霉素基因设计3条特异性嵌套引物HYGR1、HYGR2和HYGR3,参考文献[14]的方法设计3条随机简并引物LAD、AC1和AC2, 采用HiTAIL-PCR方法扩增左侧翼序列。以提取的水稻基因组DNA为模板,用以上引物分别进行3轮扩增,取第3轮扩增产物,经1%琼脂糖凝胶电泳检测。结果显示,4个检测的株系均扩增出约1 200 bp的条带(图4),与预期的扩增产物大小相符,将这些PCR产物测序作进一步分析。
2.4 T-DNA在RNAi-OsRhoGAP2水稻插入位点的分析
使用DNAMAN软件,对4个转基因水稻株系PCR产物的测序结果进行比对,结果显示序列完全相同,表明T-DNA

插入到水稻基因组的同一位置。将测序序列与水稻OsRhoGAP2基因的基因组DNA序列、pANDA载体的左边界序列进行对比,结果显示,测序序列与水稻OsRhoGAP2基因的基因组序列、pANDA载体的左边界序列均有重合部分(图5),说明载体序列已整合到水稻基因组DNA序列中。
将测序结果经NCBI网站Blast确定RNAi-OsRhoGAP2水稻T-DNA左边界插入位点,发现T-DNA插入位于日本晴水稻第2号染色体克隆NC029257.1中,其左边界插入位点位于OsRhoGAP2基因编码区的第2 669位, 处于该基因的第5个外显子内(图6)。
3 讨论与结论
水稻OsRhoGAP2是通过酵母双杂交筛选到的与水稻ROP蛋白OsRacD相互作用蛋白的编码基因。有研究显示,OsRacD参与水稻育性调控[9]。之前的试验表明,OsRhoGAP2只与GTP结合的、处于激活形式的OsRacD发生结合,并可能是负调控OsRacD活性的蛋白,在OsRacD的信号调节通路中扮演重要角色,但其功能未知。前期试验结果显示,OsRhoGAP2在细胞分裂旺盛的幼叶、幼根、幼穗或成熟根中高表达(未发表),说明该基因可能参与了相关组织的生长分化过程。

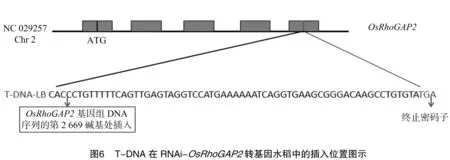
为了研究水稻OsRhoGAP2基因的功能,本研究构建了OsRhoGAP2的RNA干扰转基因水稻,并发现RNAi-OsRhoGAP2转基因水稻的株高与对照水稻相比,株高差异显著。为鉴定外源载体在转基因水稻中的插入位点,本研究采用HiTAIL-PCR方法对RNAi-OsRhoGAP2转基因水稻的 T-DNA 的侧翼序列进行扩增和分析,以期了解株高发生显著变化相关的分子信息,为后续利用转基因水稻开展对OsRhoGAP2基因的功能研究奠定基础。
热不对称交错PCR法(TAIL-PCR)最早由Liu等报道[15]。该技术可以有效地分离T-DNA侧翼序列,只须经3次PCR特异扩增过程,大大提高了目的片段的扩增效率和特异性,成为分析植物T-DNA侧翼序列的有效方法。使用TAIL-PCR方法高效地分离T-DNA的侧翼序列已有报道,例如,邵彦春等采用TAIL-PCR法成功分离了红曲霉突变株T-DNA插入位点的侧翼序列,不仅丰富了丝状真菌的生物信息学资源,也为深入研究这些序列包含的功能信息奠定了基础[16]。王海燕等利用TAIL-PCR成功扩增了小麦抗叶锈病基因同源序列RGA1侧翼序列,为克隆目的基因Lr35奠定了基础[17]。TAIL-PCR有许多优点:简单快速,不需要特殊的酶切连接甚至是转化等操作,PCR产物可以直接用于测序;具有高特异性和高灵敏性,设计的3条嵌套特异性引物,很好地利用了巢式PCR的原理来选择特异性的产物,此外通过巧妙的控制高温和低温的退火次数,让特异性产物占优势[18]。因此该技术广泛应用于许多生物的分子生物学研究之中[19],包括在拟南芥和水稻中大规模检测T-DNA和转座子插入位点,以及分离已知序列的上游(启动子)和下游序列[20-24]。不过,TAIL-PCR技术也有自身的缺陷:一是随机简并引物太多,能扩增出特异产物的数量有限,因此须要通过大量试验筛选和确定;二是结果不稳定,第3轮PCR有弥散的情况,很难得到特异性的条带,或者得到了清楚的条带,却因为太短没有测序的价值。
本试验采用的HiTAIL-PCR是Liu等针对上述问题改进后的方法[25],该方法结合了TAIL循环和抑制PCR的优点,且第2轮和第3轮PCR不再使用随机简并引物,而是利用AC引物代替了随机简并引物,这样就抑制了短的特异产物的扩增,有效扩增长片段靶序列。反应的成功率高于90%,在大多数情况下获得的主要产物的大小为1~3 kb。笔者利用HiTAIL-PCR技术扩增RNAi-OsRhoGAP2转基因水稻T-DNA的侧翼序列,结果显示T-DNA插入到水稻OsRhoGAP2基因的第5个外显子内,推测OsRhoGAP2水稻的表型改变可能是OsRhoGAP2基因的结构发生改变引起的。同时,也说明OsRhoGAP2基因可能与水稻的株高性状控制相关,值得后续深入的研究。
:
[1]Sha Y,Li S,Pei Z,et al. Generation and flanking sequence analysis of a rice T-DNA tagged population[J]. Theoretical and Applied Genetics,2004,108(2):306-314.
[2]华志华,黄大年. 转基因植物中外源基因的遗传学行为[J]. 植物学报,1999,41(1):1-5.
[3]王 槐,陈正华. 基质结合区(MARs)与转基因植物的基因表达[J]. 生命科学,1999(2):54-57.
[4]Peach C,Velten J. Transgene expression variability(position effect)of CAT and GUS reporter genes driven by linked divergent T-DNA promoters[J]. Plant Molecular Biology,1991,17(1):49-60.
[5]Fladung M. Gene stability in transgenic Aspen(Populus). Ⅰ. Flanking DNA sequences and T-DNA structure[J]. Molecular Genetics and Genomics,1999,260(6):574-581.
[6]Liu Y G,Chen Y,Zhang Q. Amplification of genomic sequences flanking T-DNA insertions by thermal asymmetric interlaced polymerase chain reaction[J]. Methods in Molecular Biology,2005,286:341-348.
[7]Kim S R,Jeon J S,An G. Development of an efficient inverse PCR method for isolating gene tags from T-DNA insertional mutants in rice[J]. Methods in Molecular Biology,2011,678:139-146.
[8]Cottage A,Yang A,Maunders H,et al. Identification of DNA sequences flanking T-DNA insertions by PCR-walking[J]. Plant Molecular Biology Reporter,2012,19(4):321-327.
[9]叶建荣,黄美娟,赵淑慧,等.OsRACD基因表达与光敏核不育水稻光周期育性转换的相关性[J]. 自然科学进展,2004,14(2):166-172.
[10]Liang W H,Tang C R,Wun A H,et al. Cloning and characterization of a new actin gene fromOryzasativaL.[J]. Progress in Natural Science,2004,14(10):867-874.
[11]梁卫红,唐朝荣,吴乃虎. 两种水稻GDP解离抑制蛋白基因的分离及特征分析[J]. 中国生物化学与分子生物学报,2004,20(6):785-791.
[12]Hiei Y,Ohta S,Komari T,et al. Efficient transformation of rice (OryzasativaL.) mediated byAgrobacteriumand sequence analysis of the boundaries of the T-DNA[J]. Plant Journal,1994,6(2):271-282.
[13]Doyle J. Isolation of plant DNA from fresh tissue[J]. Focus,1990,12:13-15.
[14]黄 磊. 稻曲病菌T-DNA插入突变体B-726、B-1015侧翼相关基因的克隆[D]. 南京:南京农业大学,2014:20-22.
[15]Liu Y G,Whittier R F. Thermal asymmetric interlaced PCR:automatable amplification and sequencing of insert end fragments from P1 and YAC clones for chromosome walking[J]. Genomics,1995,25(3):674-681.
[16]邵彦春,丁月娣,陈福生,等. TAIL-PCR法快速分离红曲霉色素突变株T-DNA插入位点侧翼序列[J]. 微生物学通报,2007,34(2):323-326.
[17]王海燕,姜亚博,杨文香,等. 小麦抗叶锈病Lr35基因同源序列RGA1侧翼序列的扩增与分析[J]. 分子植物育种,2006,4(3):329-332.
[18]郑 岑,张立平,唐忠辉,等. TAIL-PCR技术及其在植物基因中的克隆[J]. 基因组学与应用生物学,2009,28(3):544-548.
[19]焦翠翠,郭妍妍,吴均章,等. 水稻Ds标记的曲穗突变体的分子鉴定[J]. 江苏农业科学,2017,45(11):26-29.
[20]Liu Y G,Mitsukawa N,Oosumi T,et al. Efficient isolation and mapping ofArabidopsisthalianaT-DNA insert junctions by thermal asymmetric interlaced PCR[J]. Plant Journal,1995,8(3):457-463.
[21]Grossniklaus U,Vielle-Calzada J P,Hoeppner M A,et al. Maternal control of embryogenesis by Medea,a polycomb group gene inArabidopsis[J]. Science,1998,280(5362):446.
[22]Chin H G,Choe M S,Lee S H,et al. Molecular analysis of rice plants harboring anAc/Dstransposable element-mediated gene trapping system[J]. Plant Journal for Cell&Molecular Biology,1999,19(5):615-623.
[23]周延清,王婉珅,张 喻,等. 高效热不对称交互式PCR技术克隆地黄基因[J]. 河南师范大学学报(自然科学版),2015(1):100-105.
[24]裴忠有. 水稻T-DNA插入突变体筛选及其功能的初步分析[D]. 北京:中国科学院微生物研究所,2004:15-18.
[25]Liu Y G,Chen Y L. High-efficiency thermal asymmetric interlaced PCR for amplification of unknown flanking sequences[J]. Biotechniques,2007,43(5):649-656.
