天祝牦牛Fas基因的克隆与生物信息学分析
2018-05-25陈学生杨秀梅
陈学生,杨秀梅,曹 健
(甘肃省酒泉市畜牧兽医局,酒泉 735000)
细胞凋亡,也称细胞程序性死亡,是一种受遗传学机制调控的为维持体内环境稳定的细胞主动性死亡过程,为多细胞生物生命活动所必需[1]。Fas抗原,也称CD95、APO-1或TNFRSF6,是一种Ⅰ型膜蛋白,其分子大小为45 kDa,属于肿瘤坏死因子(TNF)/神经生长因子受体家族[2],Fas系统功能正常对机体自身稳定具有重要作用,其功能过强或缺陷均可导致发生疾病[3]。FasL是Fas的配体(Fas是FasL的受体),主要在被激活的T细胞和B细胞上表达。当Fas抗原和FasL结合后,可以诱发Fas表达,引发细胞凋亡[4]。Fas基因分别位于小鼠的19号和人的10号染色体上,包含9个外显子[5-6]。Fas广泛表达于各种正常的组织和细胞,如活化的T细胞和 B 细胞[7]。
牦牛是我国青藏高原及其周边高寒牧区重要的生产资源和生活资源。牦牛生长调控相关基因功能的研究将有助于逐步阐明其作用机理。为了深入探讨牦牛Fas基因的功能,本试验以天祝牦牛卵巢颗粒细胞为材料,对该基因进行了分子克隆和生物信息学分析,以期为进一步在蛋白水平上研究此基因进化机制、表达特性、编码蛋白结构及其生理功能等提供基础研究资料。
1 材料与方法
1.1 材料
在甘肃省天祝县屠宰场采集5对牦牛卵巢,放于37℃生理盐水中6 h内运回实验室,并接种于直径为60 mm的培养皿中培养3 d,待牦牛卵泡颗粒细胞的汇合度达80%~90%时进行RNA提取。
试剂:Ficoll-PaqμeTMPLUS 购自 Amersham Biosciences;Trizol试剂、GoldScript cDNA synthesis kit及 TA Cloning kit购自英骏公司;Ex Taq Mix和TaKaRa LA Taq购自宝生物;Gel Extraction Spin Kit购自GENOMED,其余试剂均为国产分析纯。
1.2 方法
1.2.1 引物设计 使用设计引物1(P1)和引物2(P2)用于天祝牦牛Fas基因克隆。引物由北京六合华大基因科技股份有限公司合成。引物1序列F:5-AGTCGGGGCTCACTACTCAC,R:5-TGGAATGGGGTCAACCTGTG;引物 2序列 F:5-TGTTCATCTTCTTGCCTGTT,R:5-TTAGGTAGTTCGGAGCATC。
1.2.2 总RNA的提取 用Trizol法[8]提取天祝牦牛卵泡颗粒细胞总RNA,检测并调整浓度后分装保存于-70℃冰箱中。
1.2.3 RT-PCR分析
1.2.3.1 cDNA的合成 用Invitrogen公司的GoldScript cDNA synthesis kit试剂盒,以oligo dT为引物,按说明进行cDNA第一链合成。得到的cDNA用作随后PCR扩增的模板。
1.2.3.2 PCR扩增 PCR反应总体积25μL,各成分用量:10×PCR 缓冲液2μL(Mg2+1.5μmol/L),DNA模板(约 50 ng/μL)0.8 μL,上下游引物(0.25 μmol/L)各 0.6 μL,dNTP(150 μmol/L)0.6 μL,Taq DNA 聚合酶(0.5 U)0.2μL,超纯水15.2μL。扩增程序:94℃预变性5min;94℃变性30 s,退火(引物1和引物2退火温度分别为60℃和 53℃)30 s,72℃延伸 50 s,35 个循环;72℃延伸10min;4℃保存。扩增产物用1%琼脂糖凝胶电泳检测。
1.2.3.3 天祝牦牛Fas基因的克隆和测序 按DNA回收试剂盒说明回收特异性条带,将纯化产物与克隆载体连接,连接产物转化至感受态细胞进行蓝白斑筛选,挑取阳性克隆接种于LB培养基培养过夜,取1.5mL阳性新鲜菌液送北京六合华大基因公司测序。
1.2.4 天祝牦牛Fas基因生物信息学分析 利用Editseq软件进行序列拼接,用Protean软件对蛋白结构特征分析,采用MegAlign软件进行同源性分析,开放阅读框用NCBI提供的ORF finder程序分析。
基因编码产物的理化特性采用DNAStar软件和ProtParam(http://expasy.org/tools/protparam.html)共同分析。蛋白质功能采用ProtFun预测(http://www.cbs.dtu.dk/services/ProtFun/)。蛋白信号肽采用SignalP预测(http://www.cbs.dtu.dk/services/SignalP-2.0/)。蛋白质结构域用Interpro预测(http://www.ebi.ac.uk/Tools/pfa/iprscan/)。功能位点用PREDICT PROTEIN预测(http://www.predictprotein.org/)。二级结构用SOPMA预测(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)。用于同源性分析的其他物种的Fas基因序列从GenBank上下载。
2 结果与分析
2.1 目的基因的PCR扩增
扩增产物用1%琼脂糖凝胶电泳检测。由图1可见,PCR扩增产物琼脂糖电泳条带清晰,特异性良好,片段大小在预期范围内,与设计扩增片段相符。

图1 天祝牦牛Fas基因RT-PCR扩增结果
2.2 天祝牦牛Fas基因开放阅读框分析
开放阅读框是证明一个新的DNA序列为特定蛋白质编码基因的部分或全部的先决条件。本研究采用NCBI的 ORFFinder程序,参照 Kozak 法则[9],在所获得的天祝牦牛Fas基因序列中寻找出一条长972 bp的ORF,起始密码子位于86 bp,终止密码子位于1 057 bp,推测编码323个氨基酸残基。
2.3 天祝牦牛Fas基因编码蛋白理化特性预测与分析
运用Expasy服务器上的ProtParam工具预测天祝牦牛Fas基因编码产物的理化性质。Fas基因编码的323个氨基酸中,Glu所占比例最高,为9%。负电荷残基总数(Asp+Glu)为 44,正电荷残基总数(Arg+Lys)为 41。其分子式为C1548H2487N459O497S28,分子质量36.37 kDa,理论等电点6.5。Fas基因编码产物的不稳定指数31.54,根据Guruprasad原则表明此编码产物相对稳定。
2.4 天祝牦牛Fas基因编码蛋白亲水性/疏水性预测与分析
疏水性/亲水性的分析可为蛋白质高级结构的预测及功能分析提供理论参考。运用Expasy服务器上的Protscale程序,对天祝牦牛Fas蛋白的氨基酸序列进行亲水性/疏水性分析,见图2。多肽链的第197位赖氨酸具有最低分值-3.567,亲水性最强;第181位的脯氨酸具有最高分值3.556,疏水性最强,整条多肽链氨基酸序列表现为亲水性。该结果与DNASTar软件分析结果基本一致。
2.5 天祝牦牛Fas基因编码蛋白信号肽预测与分析
信号肽位于分泌蛋白的N端,含有信号肽的蛋白质一般能够分泌到细胞外作为重要的细胞因子起作用[10]。运用SignalP 3.0在线软件对天祝牦牛Fas蛋白进行分析,神经网络模型(NN)预测的信号肽结果如图3所示,隐马尔可夫模型(HMM)预测的信号肽结果如图4所示。天祝牦牛Fas蛋白的分值曲线非常典型,C值为0.561,Y值为0.594,S值为0.962,基本可以判定Fas编码产物存在信号肽。
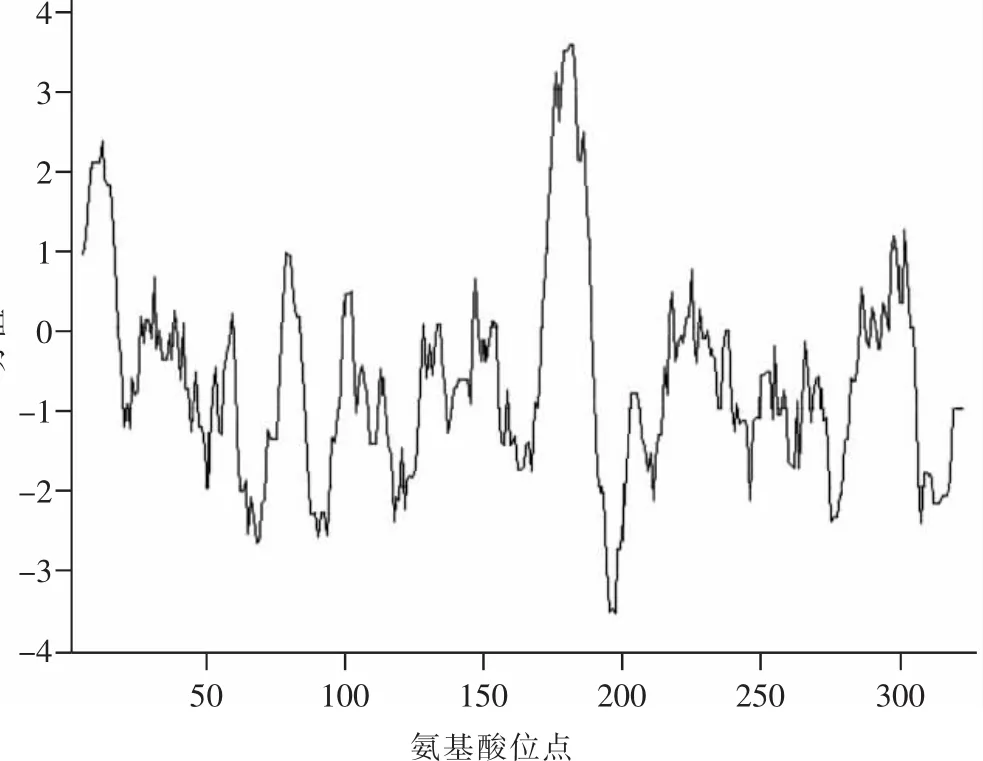
图2 天祝牦牛Fas蛋白的疏水性结果

图3 NN模型分析天祝牦牛Fas编码产物信号肽的曲线图

图4 HMM模型分析天祝牦牛Fas编码产物信号肽的曲线图
2.6 天祝牦牛Fas基因编码产物功能预测与分析
运用Protfun分析软件预测Fas编码产物的功能,从表1结果可知,该蛋白具有受体(Receptor)、转录调控(Transcription regulation)、胁迫应答(Stress response)信号转导(Signal transducer)和免疫应答(Immune response)的可能性分别为 2.772、1.194、1.147、0.920、0.575。表明Fas有可能在受体和转录调控中发挥重要作用。
2.7 天祝牦牛Fas基因编码产物结构域和蛋白质功能位点预测
用Interpro在线工具预测了天祝牦牛Fas蛋白的结构域,结果显示,该序列具有完整的Fas receotpr家族蛋白功能域(图5)。运用PREDICTPROTEIN在线服务器对天祝牦牛Fas蛋白分析可知,该蛋白含有3个糖基化位点(分别位于38、115、215位处),2个蛋白激酶C磷酸化位点(分别位于89位和95位处),8个酪蛋白激酶 II磷酸化位点(分别位于 20、44、102、135、144、207、217、227位处),3个肉豆蔻酰化位点(分别位于34、85、245位处),2个酰胺化位点(分别位于62位和277位处),1个TNFR/NGFR家族半胱氨酸富集区(位于82处)。
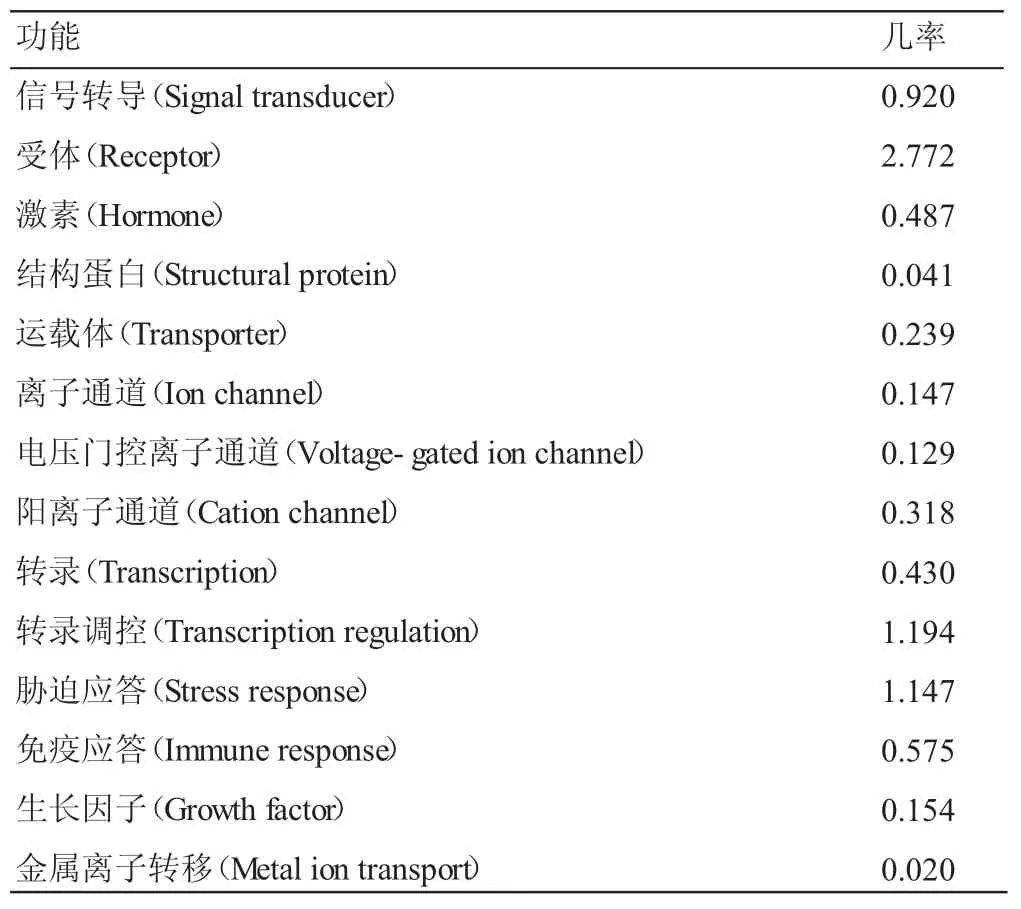
表1 天祝牦牛Fas基因编码产物功能分析结果

图5 天祝牦牛Fas基因编码产物的结构域预测
2.8 天祝牦牛Fas蛋白的结构特征分析
用Protean软件对天祝牦牛Fas蛋白的结构和特性进行了预测分析,详见图6。用Jameson-Wolf方法分析天祝牦牛Fas蛋白潜在的抗原决定簇,结果表明,该蛋白含有大量抗原指数较高的区域。利用Emini方法分析天祝牦牛Fas蛋白的表面可能性区域主要集中在63~70、85~97、116~125、305~321 和 189~202区段,其他区段的可能性较小。

图6 天祝牦牛Fas蛋白结构和特性
2.9 二级结构预测
利用SOPMA程序对天祝牦牛Fas氨基酸序列的二级结构进行预测,结果表明,天祝牦牛Fas蛋白由35.91%的α-螺旋、7.74%的延伸链、52.32%的不规则卷曲和4.02%的β-转角组成。可推断无规则卷曲、α-螺旋和延伸链是天祝牦牛Fas蛋白主要的二级结构元件。
2.10 天祝牦牛Fas氨基酸序列同源性分析
应用MegAlign软件对天祝牦牛及普通牛、绵羊、小鼠等10个物种的Fas氨基酸序列进行同源性分析(图7)。天祝牦牛与普通牛Fas氨基酸序列同源性最高,为98.8%,与绵羊、猪、人的同源性分别为91.3%、67.6%、59.7%,与其他哺乳动物的同源性也都在46.3%以上。蛋白质水平之间具有25%的同源性就可提示其功能的相似性[11]。因此,牦牛与其他物种间氨基酸序列间具有较高的保守性。

图7 10个物种的Fas基因编码产物序列差别与相似性
3 讨论
本实验克隆了天祝牦牛Fas基因2 605 bp的cDNA序列,包含969 bp的CDS,编码323个氨基酸,通过蛋白质的疏水性、信号肽预测以及蛋白结构域和蛋白功能位点分析了天祝牦牛Fas基因编码蛋白功能。并且通过同源性分析发现,天祝牦牛Fas基因与普通牛的亲缘关系最近,达到了98.8%,其次是绵羊(91.3%)。这种同源性在一定程度上代表着物种亲缘关系的远近,同时也反映了Fas基因编码产物在不同物种的结构上的稳定性对生物体功能的重要性[12]。疏水性/亲水性分析结果显示,天祝牦牛Fas蛋白整条多肽链表现为亲水性,说明该蛋白质的亲水性较好,其二级结构很容易接近水分子,为水溶性蛋白。信号肽分析结果显示,天祝牦牛Fas抗原同人、小鼠和普通牛的一样,氨基端含有长22个氨基酸残基的信号肽。Antonio等[13]发现,改造了的信号肽可大大提高外源蛋白表达量。提示通过化学修饰可以提高其在机体内的表达,从而发挥更大的生物学功能。编码蛋白结构域结果可知,Fas蛋白含有死亡结构域(Death)和富半胱氨酸的亚结构域(TNFR/NGFR cysteine),这与Adachi等[12]报道的结果基本一致。Fas抗原的胞外区含有3个富含半胱氨酸的重复区,为其配体识别位点,破坏该区域可使其失去与配体结合及传递信号的能力;胞质区域含有一段约80个氨基酸的保守序列,称为“死亡域”,与TNFα的相应片段有很高的同源性,能将胞外的凋亡信号传到胞内[14-15]。蛋白质中的功能位点可提供蛋白质的功能信息,具有重要的生物学意义。本研究共发现8个酪蛋白激酶II磷酸化位点。刘善荣等[16]发现,蛋白质(信号分子)的磷酸化是细胞生长发育和癌变过程中众多复杂的信号转导途径和信号分子参与下最基本的一种反应过程。说明该蛋白可能通过启动相关基因转录并翻译蛋白质,从而在细胞生长和癌变过程中发挥重要作用。2个蛋白激酶C磷酸化位点和3个肉豆蔻酰化位点的发现提示,该蛋白与细胞信号转导过程中的磷酸化/去磷酸化有关。天祝牦牛Fas蛋白的结构特征分析显示,该区域抗原指数较高,呈现在蛋白表面的可能性也较大,推测该区段的抗原表位应该是优势抗原表位所在。同时,功能分析预测该蛋白具有受体、信号传导等主要功能,Fas是细胞表面重要的死亡受体,其与配体FasL结合,活化并传导凋亡信号。这与王强等[17]研究结果基本一致。说明其在诱导细胞凋亡过程中发挥重要作用。
[1] Danial N N,Korsmeyer S J.Cell death:critical control points[J].Cell,2004,116(2):205-219.
[2] AdachiM,Watanabe R-Fukunaga,Nagata S.Aberrant transcription caused by the insertion of an early transposable element in an intron of the Fas antigen gene of lprmice[J].Proc Natl Acad Sci USA,1993,90(5):1756-1760.
[3] 包兆胜.Fas与FasL的分子生物学研究进展[J].生命的化学,2002,22(2):127-129.
[4] 李奎,康相涛,刘英,等.固始鸡免疫器官内细胞凋亡基因Fas和FasL 的动态表达[J].中国农业科学 2008,41(5):1489-1496.
[5] Inazawa J,Itoh N,Abe T,et al.Assignment of the human Fas antigen gene(Fas)to 10q24.1[J].Genomics,1992,14(3):821-822.
[6] Watanabe-Fukunaga R,Brannan C I,Itoh N,et al.The cDNA structure,expression,and chromosomal assignment of the mouse Fas antigen[J].JImmunol,1992,148(4):1274-1279.
[7] Suda T,Okazaki T,Naito Y,et al.Expression of the Fas ligand in cells of T cell lineage[J].JImmunol,1995,154(8):3806-3813.
[8] 魏群,崔丽华,杨淑杰,等.分子生物学实验指导[M].北京:高等教育出版社,2003:53-58.
[9] Carmi R,Rokhlina T,Kwitek-Black A E,etal.Use of a DNA pooling strategy to identify a human obesity syndrome locus on chromosome 15[J].Human Molecular Genetics,1995,4:9-13.
[10]张寒莹.长江三角洲白山羊GDF-9基因第一、第二外显子序列多态及生物信息学分析[D].南京:南京农业大学,2008.
[11]Garnier J,Gibrat JF,Robson B.GOR secondary structure prediction method version IV[J].Methods in Enzymology,1996,266:540-553.
[12]Walter J,High K A.Gene therapy for the hemophiliac[J].Adv Vet Med,1997,23(12):119-134.
[13]Antonio M R,Martinez A.Secretion of recombinant pro-and mature fungal sareinribo toxin by the methylotrophie yeast P/ch/apastoris:The Lysargmotif is required for maturation [J].Protein Expr Purif,1998,12(6):315-322.
[14]Orlinick JR,Elkon K B,Chao M V.Separate domains of the human fas ligand dictate self-association and receptor binding[J].JBiol Chem,1997,272(51):32221-32229.
[15] Orlinick J R,Vaishnaw A,Elkon K B,et al.Requirement of cysteine-rich repeats of the Fas receptor for binding by the Fas ligand[J].J Biol Chem,1997,272(46):28889-28894.
[16]刘善荣,刘厚奇,戚中田.蛋白激酶与蛋白磷酸酶在细胞增殖分化中的机制研究[J].第二军医大学学报,2004,5(5):550-552.
[17]王强,杨志雄,廖思海.Fas、FasL和Caspase-3在肺癌中的表达及其临床意义[J].现代肿瘤医学,2009,17(2):248-251.
