结肠癌差异DNA甲基化位点初筛及粪便样本细胞中RUNX3基因的异常甲基化谱构建
2018-05-23吴鹏吴平平马常兰施风
吴鹏,吴平平,马常兰,施风
(1.江苏建康职业学院 临床与护理学院,江苏 南京 210029; 2.江苏省肿瘤医院 肿瘤内科,江苏 南京 210009)
我国结肠癌的发病率呈逐年上升的趋势,是威胁国人健康的重大疾病[1- 2]。肠镜侵入检查是目前最主要的检查方法之一,但临床上常会碰到许多问题,比如取材时组织损伤或组织量少难以做出正确的预后判断;电子计算机体层摄影(CT)等影像学检查因经济或造影剂毒性而限制了使用;外周血活检也存在患者理解不足、畏于抽血而致依从性差等[3]。粪便中含有来自肠道肿瘤的脱落细胞,具有一定的特异性,是结直肠癌预后评估理想的无创检测素材,且患者接受程度高,依从性好,其将推动结直肠癌预后研究的重大变革,具有重要的科研和应用价值[4]。
表观遗传机制在基因表达调控上提供了一种高层次的调控模式,被认为是控制基因活动的“开关”,已成为后基因组时代研究的热点[5- 6]。脱氧核糖核酸(DNA)甲基化是目前发现的最主要的一种表观遗传修饰形式,其在结肠癌等多种肿瘤的研究中有了长足的进展[7]。我们前期在85例结直肠癌及45例肠道良性病变患者血清标本中,通过甲基化特异性聚合酶链反应(MSP)检测基因启动子区域甲基化,发现DLC1、p16和RUNX3基因甲基化比例分别为42.4%、44.7%和34.1%,均显著高于良性病变组(P<0.01),三者联合检测可显著提高结直肠癌检出率,并且特异性未降低[8]。甲基化的DNA片段可以在粪便中检测到,这种非侵袭性的检查方法使甲基化检测有了更好的应用前景,可成为结肠癌预后评估新突破口[9- 10]。
作者将系统性研究粪便样本细胞中异常甲基化谱,尤其是RUNX3基因的甲基化研究,有望获得一组能应用于临床的新型分子标记,为结肠癌的诊断、治疗及预后提供有力的支持。
1 资料与方法
1.1 一般资料
选择结肠癌患者的癌组织和癌旁组织的存档蜡块,用于差异DNA甲基化位点初筛。收集江苏省肿瘤医院2015年1月至2015年12月间100例结肠癌患者新鲜粪便,100例健康体检者为健康对照组。所有病例均有完整的临床及随访资料,用于异常甲基化谱的构建。
1.2 样本处理
对于癌组织和癌旁组织的存档蜡块用激光捕获显微切割技术收集细胞[11],经研磨、匀浆后提取基因组DNA。对于新鲜粪便样本,使用Qiagen QIAmp® DNA Stool Mini Kit按照产品操作规程来分离提取DNA。样本的分组情况见表1。
表1样本的分组情况

样本分组实验编号样本例数(男∶女)/例结肠癌组织T63∶3结肠癌旁组织N63∶3结肠癌患者新鲜粪便S⁃T10059∶41正常人新鲜粪便S⁃N10055∶45
1.3 差异DNA甲基化位点初筛
针对RUNX3基因,经亚硫酸氢盐修饰、扩增、断裂、沉淀、重悬等操作,在Illumina Infinium Methylation EPIC BeadChip(850 k)甲基化芯片上进行杂交、洗涤、延伸、染色、扫描等过程获得全基因组甲基化谱,通过生物信息学初步筛选差异性甲基化位点。
1.4 MSP方法
RUNX3启动子区的甲基化引物(M)和未甲基化引物(U)序列如表2所示,以亚硫酸氢盐修饰后的基因组DNA为模板,进行聚合酶链反应(PCR反应)。反应产物经含有EB的2%琼脂糖凝胶电泳、紫外观察、凝胶成像系统扫描成像后拍照,检测扩增效果。
1.5 统计学处理
在原始数据扣除背景后进行标准化处理,然后计算甲基化程度水平(Beta值),同时使用Beta混合物分位数扩张(BMIQ)的方法进行Beta值的标准化处理[12],作为衡量甲基化程度的指标,同时采用层次聚类算法对差异甲基化位点进行聚类分析。进一步进行甲基化位置变化(methylation variable position)分析,即针对差异化甲基化位点最近邻基因进行基因本体(gene ontology,GO)功能富集分析和京都基因与基因组(KEGG)功能富集分析。对于MSP结果,使用SPSS统计软件进行分析,计量资料采用t检验,计数资料采用卡方检验,以P<0.05为差异有统计学意义。
表2RUNX3启动子区MSP引物序列及反应条件
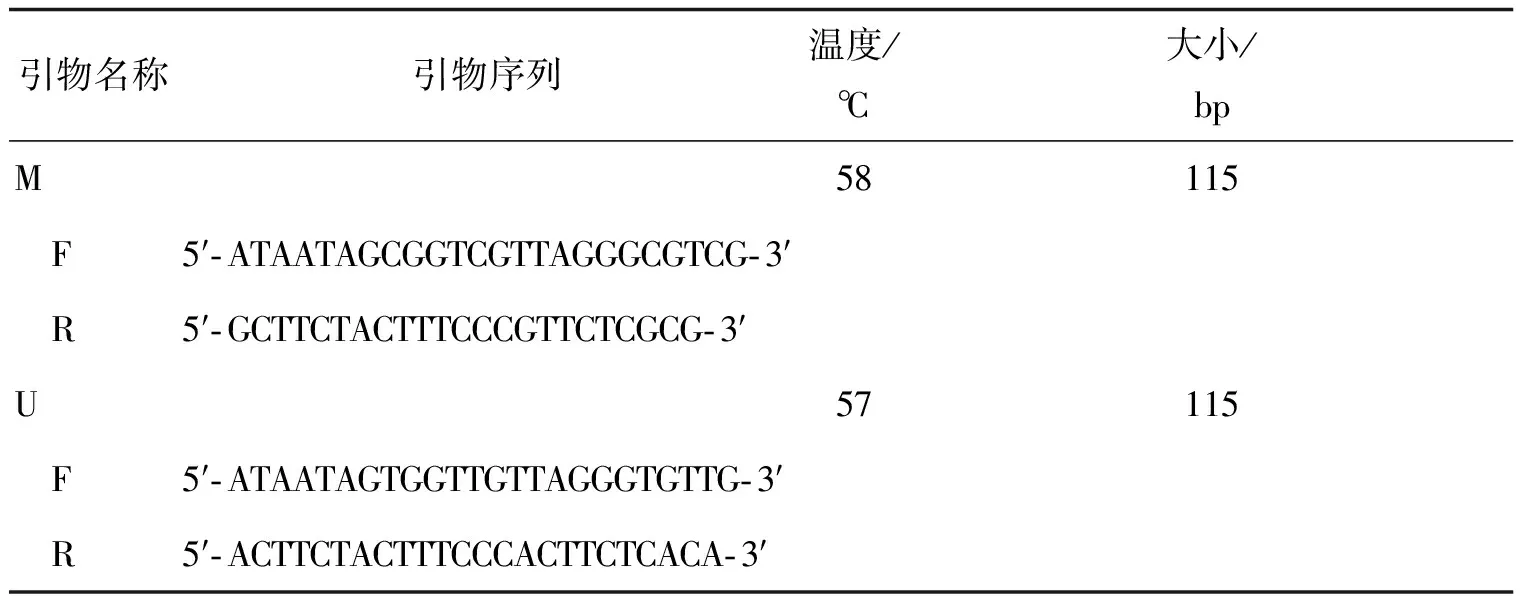
引物名称引物序列温度/℃大小/bpM58115 F5′⁃ATAATAGCGGTCGTTAGGGCGTCG⁃3′ R5′⁃GCTTCTACTTTCCCGTTCTCGCG⁃3′U57115 F5′⁃ATAATAGTGGTTGTTAGGGTGTTG⁃3′ R5′⁃ACTTCTACTTTCCCACTTCTCACA⁃3′
2 结 果
2.1 差异DNA甲基化位点分析
2.1.1 甲基化差异分析 使用Beta值的样本密度箱形图来评估甲基化芯片数据的整体分布。在箱形图(图1)中,癌组织(T)和癌旁组织(N)两组之间最大值、最小值及中位数显示两者之间正态分布差异不明显,适合聚类芯片分析。
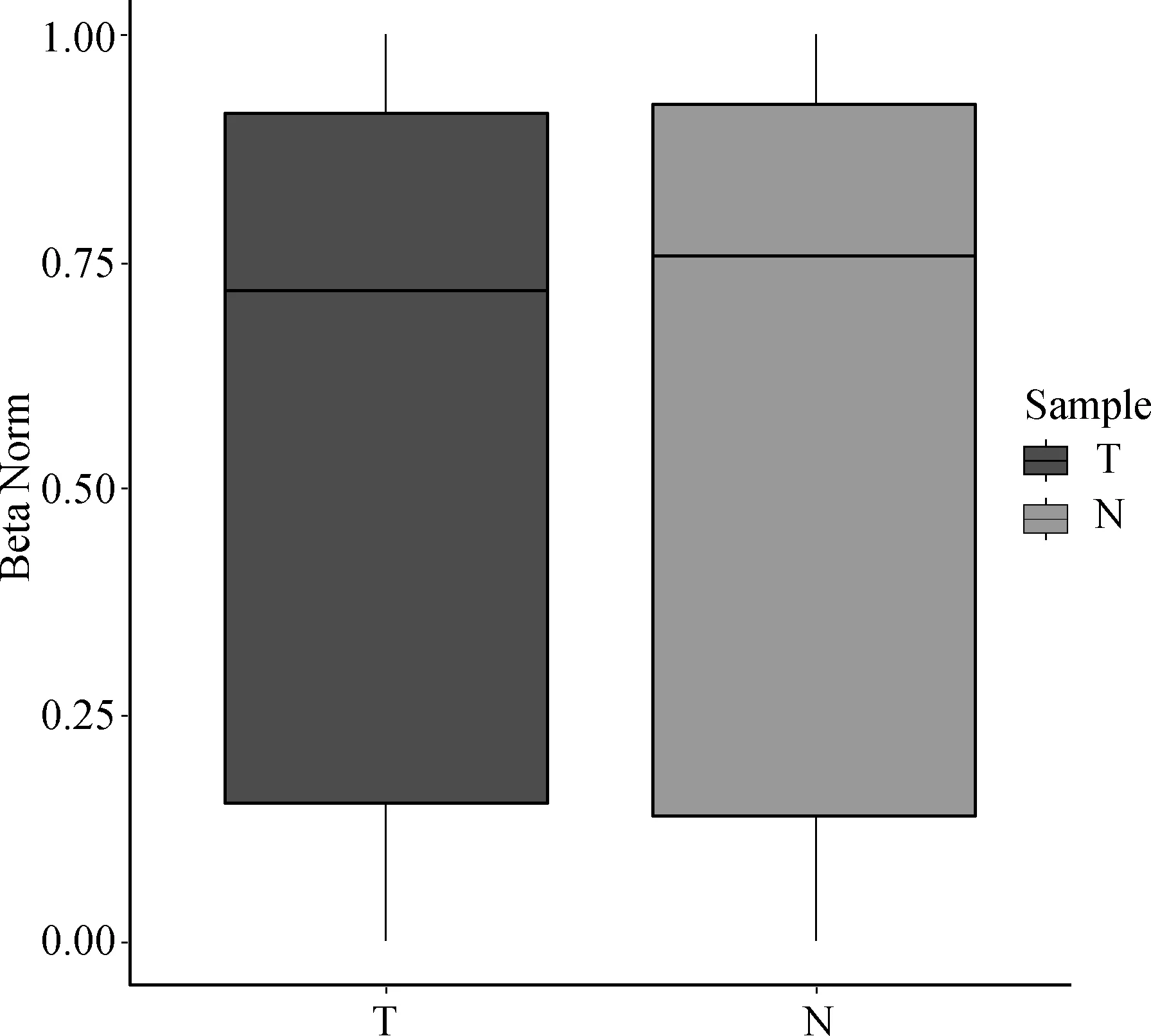
图1标准化处理后Beta值的箱式图
热图结果显示T和N两组之间甲基化位点差异性排布,颜色越深代表甲基化位点差异越明显,颜色越浅代表甲基化位点差异越不明显,因此T组中存在较多高甲基化位点,而N组中存在较多低甲基化位点(图2)。进一步通过主成分分析(PCA)发现T和N两组之间呈现组内显著富集,表明检测结果无交叉影响,结果客观可靠(图3)。

图2差异甲基化位点的热图

图3差异甲基化位点的PCA分析
甲基化位点的差异性可以使用火山图(图4)来表示,其中红点富集区域为T和N两组之间甲基化位点呈显著性差异(P<0.01),偏右区域为与N组相比甲基化位点水平升高,偏左区域为与N组相比甲基化位点水平降低;黑点富集区域为T和N两组之间甲基化位点呈不显著性差异(P>0.01)。
2.1.2 差异甲基化位点功能分析 GO功能富集分析结果表明,共有145个差异甲基化基因的功能类别(FDR<0.05),涵盖了3 155个基因,其中生物过程、细胞组成和分子功能分别含有的基因功能类别为102、22和21个。KEGG功能富集分析结果表明共有28个差异甲基化基因的功能类别(P<0.05),涵盖了697个基因。经过上述分析,共筛选出RUNX3等10个差异显著的DNA甲基化基因位点。

图4T和N两组甲基化位点差异程度的火山图红色虚线表示P=0.01
2.2 MSP检测RUNX3启动子甲基化状态
根据甲基化芯片结果选取RUNX3启动子甲基化位点,应用MSP做进一步组织样本验证及新鲜粪便样本的检测。使用MF、MR和UF、UR两对引物扩增经亚硫酸盐修饰后的DNA,若只有甲基化的引物能扩展出条带则是完全甲基化的;若只有未甲基化的引物能扩增出条带则是完全未甲基化;若两对引物均能扩增出条带则称为半甲基化,也判断为甲基化。
T和N两组的扩增条带如图5和表3所示,检测T样本6例,其中5例为甲基化,1例为非甲基化,甲基化率为83.3%;N中1例为甲基化,5例为非甲基化,甲基化率为16.7%,经卡方检验两者差异具有统计学意义(P<0.05)。新鲜粪便样本的扩增条带如图6和表3所示,对于结肠癌患者(S- T)RUNX3启动子的甲基化率为45.5%,而正常人(S- N)的甲基化率只有18.2%,经卡方检验两者差异具有统计学意义(P<0.001)。

图5两对引物在T(A)和N(B)两组中扩展出条带的电泳图
表3结肠癌和正常样本的甲基化率

样本分组RUNX3(+)(-)甲基化率/%χ2值P值T组5183.35.330.02N组1516.7S⁃T组445644.015.80<0.001S⁃N组188218.0

图6两对引物在结肠癌患者(A)和正常人(B)粪便样本中扩展出条带的电泳图
3 讨 论
本研究发现T组中存在较多高甲基化位点,而N组中存在较多低甲基化位点。GO功能富集分析结果表明共有145个差异甲基化基因的功能类别(FDR<0.05),涵盖了3 155个基因。生物过程部分包含的基因功能类别最多。本次研究表明,差异甲基化的功能基因主要集中在细胞膜、受体活性以及信号转导等方面,与肿瘤的发生、发展与转移等过程密切相关。与此同时,KEGG功能富集分析结果表明这些基因主要参与消化液分泌、神经突触信号转导、阿米巴样运动等与结肠癌高度相关的分子事件。
经过差异甲基化芯片分析,共筛选出RUNX3等10个差异显著的DNA甲基化基因位点。RUNX3基因位于人类染色体1q36.1,广泛表达于消化道上皮细胞等多种细胞中。RUNX3基因是研究比较多的抑癌基因,其编码的RUNX3蛋白是TGF- β介导的抑癌途径的重要组分,与肿瘤的发生等过程密切相关[13]。RUNX3基因的甲基化则会抑制RUNX3基因的表达,进而影响TGF- β信号转导通路,促进肿瘤的发生与发展。MSP结果表明,T RUNX3基因的甲基化水平明显高于N,差异具有统计学意义(P<0.05),进而验证了差异DNA甲基化位点分析的结果,说明RUNX3基因甲基化检测对结肠癌患者具有重要的意义。新鲜粪便样本结果中结肠癌患者RUNX3启动子的甲基化率明显大于正常人,且差异具有统计学意义,进一步证明了实验的结论。
本研究筛选出RUNX3等10个差异显著的DNA甲基化基因位点,进一步分析了T/N、S- T/S- N新鲜粪便样本细胞中RUNX3甲基化水平,结果提示RUNX3启动子的甲基化可以用于结肠癌的诊断以及疗效评价等方面。
[参考文献]
[1] CHEN W,ZHENG R,BAADE P D,et al.Cancer statistics in China,2015[J].CA:a Cancer Journal for Clinicians,2016,66(2):115- 132.
[2] 李道娟,李倩,贺宇彤.结直肠癌流行病学趋势[J].肿瘤防治研究,2015(3):305- 310.
[3] CLARKE E,WOOLSON K,SAUNDERS M,et al.PWE- 100 missed rates of colorectal cancer- diagnostic limitations[J].Gut,2016,65(Suppl 1):A187- A188.
[4] BAILEY J R,AGGARWAL A,IMPERIALE T F.Colorectal cancer screening:stool DNA and other noninvasive modalities[J].Gut and Liver,2016,10(2):204.
[5] EASWARAN H,TSAI H C,BAYLIN S B.Cancer epigenetics:tumor heterogeneity,plasticity of stem- like states,and drug resistance[J].Molecular Cell,2014,54(5):716- 727.
[6] 剧宏燕,柴秀坤,白文元.结直肠癌的表观遗传学[J].世界华人消化杂志,2014,22(15):2128- 2133.
[7] 李莉,鲁英,杨晓燕,等.新疆乳腺癌患者外周血中 p16 基因甲基化的检测意义[J].东南大学学报:医学版,2014,33(6):691- 696.
[8] 邹继红,吴平平,汤日宁,等.结直肠癌血清中 DLC1,p16 和 RUNX3 基因甲基化检测的临床意义[J].江苏医药,2011,37(24):2938- 2940.
[9] 王裴,张明鑫,张超,等.粪便 DNA 甲基化检测在结直肠癌早期诊断中的研究进展[J].现代肿瘤医学,2015,23(6):874- 880.
[10] 周婷婷,丁家华.异基因造血干细胞移植治疗骨髓增生异常综合征的时机选择[J].现代医学,2013,41(4):279- 281.
[11] JENSEN E.Laser- capture microdissection[J].The Anatomical Record,2013,296(11):1683- 1687.
[12] TESCHENDORFF A E,MARABITA F,LECHNER M,et al.A beta- mixture quantile normalization method for correcting probe design bias in illumina infinium 450 k DNA methylation data[J].Bioinformatics,2013,29(2):189- 196.
[13] MU W,WANG J,NIU Q,et al.Clinical significance and association of RUNX3 hypermethylation frequency with colorectal cancer:a meta- analysis[J].OncoTargets and Therapy,2014,7:1237- 1245.
