短扩增子对结直肠癌转移石蜡组织LncRNA定量分析的可行性
2018-05-15李佳茜孙青
李佳茜,孙青
(1 潍坊医学院临床医学院,山东潍坊261000;2 山东省千佛山医院)
筛选、鉴定与肿瘤转移相关的分子是当前肿瘤学研究的热点。长链非编码RNA(LncRNA)是一类长度大于200个核苷酸、无蛋白质编码功能的RNA。LncRNA具有数量多、类型多、作用模式多等特点,根据其生物学功能分为信号分子、诱饵分子、引导分子和支架分子。研究发现,LncRNA可通过染色体修饰、X染色体沉默、DNA甲基化、转录激活或沉默等方式,在转录或转录后以及表观遗传学水平进行调控,继而参与机体多种生理或病理过程[1,2]。近年研究发现,LncRNA在结直肠癌的发生、发展中具有重要作用[3,4]。手术切除组织或活检组织最常用的保存方式是甲醛固定后石蜡包埋[5]。但石蜡包埋组织(以下称石蜡组织)保存过程中核酸会发生部分降解,Masuda等[6]发现石蜡组织提取的mRNA片段长度平均约200 bp,故分析其LncRNA有一定困难[7]。在实时荧光定量PCR(qRT-PCR)过程中,短扩增子非常稳定且可检测,可对石蜡组织来源的LncRNA进行有效扩增[8~12]。但其对LncRNA定量分析的可行性仍待进一步验证。本研究筛选出与结直肠癌转移相关的LncRNA,在石蜡组织中通过PCR扩增短扩增子,旨在探索短扩增子用于定量分析石蜡组织LncRNA的可行性。
1 材料与方法
1.1 材料 选择2012年1月~2015年1月山东省千佛山医院病理科保存的结直肠癌石蜡组织55例份,2015年1月~2017年1月保存的结直肠癌冰冻组织40例份,每份标本包含结直肠癌组织、癌旁正常组织(距肿瘤边缘>5 cm)和转移淋巴结组织,均经组织病理检查证实。标本来源患者均为首次手术治疗,术前未行任何抗肿瘤治疗,预计生存期≥6个月,排除既往存在其他部位恶性肿瘤史、有远处转移及合并重要脏器严重疾病者。本研究经山东省千佛山医院医学伦理委员会批准。
1.2 结直肠癌转移相关LncRNA筛选 本课题组前期通过高通量LncRNA芯片进行差异筛选,确定了4条与结直肠癌转移相关LncRNA,即LncRNA-Enst00000554679、LncRNA-Enst00000550851、LncRNA-Enst00000497498、LncRNA-Enst00000513016。
1.3 LncRNA表达检测 选择冰冻组织25例份,采用qRT-PCR法分别检测结直肠癌组织、癌旁正常组织、转移淋巴结组织上述与结直肠癌转移相关的4条LncRNA表达。①RNA提取:取3种冰冻组织各100 mg,匀浆后按RNAsimple Total RNA Kit总RNA提取试剂盒(离心柱型)说明提取组织总RNA,通过gDNA Eliminator column去除基因组DNA。经NanoDrop@ND.1000微量核酸定量仪定量,OD260/OD280为1.8~2.1,说明提取的总RNA纯度合格,可用于后续实验。②逆转录反应:按Revert Aid First Strand cDNA Synthesis Kit说明将总RNA逆转录为cDNA,- 20 ℃保存。逆转录反应体系共20 μL:总RNA 10 μL,Random Hexamer 1 μL,Nuclease-Free Water 1 μL,5× Reaction Buffer 4 μL,Ribo Lock RNAse inhibitor 1 μL,10 mmol/L dNTP Mix 2 μL,Revert Aid Reverse Transcriptase 1 μL。③引物设计合成:所有引物由上海生工生物工程股份有限公司设计合成,并通过高亲和力纯化进行片段提纯。按一般原则设计4对长引物用于扩增LncRNA内的长片段。LncRNA-Enst00000554679:上游引物5′-AAAGAAGTAAACACAAGAC-3′,下游引物5′-AAAATGTTACATCAGGGAG-3′;LncRNA-Enst-00000550851:上游引物5′-ACCGTCTGCAATCAGTTAC-3′,下游引物5′-ATCATGTCAGAGAGCCTGG-3′;LncRNA-Enst00000497498:上游引物5′-CTGAGCAGAAACCAAGTTG-3′,下游引物5′-CTAAAACAG-CTTCACATGG-3′;LncRNA-Enst00000513016: 上游引物5′-TTGAGAAGATGTTGATGAC-3′,下游引物5′-TTATAGCCCTTTTACATTG-3′;内参β-actin:上游引物5′-GCGTGACATTAAGGAGAAGC-3′, 下游引物5′-TCTCTTGCTCGAAGTCCAGG-3′。④PCR扩增:PCR反应体系共20 μL:SYBR qPCR Mix 10 μL,50×Rox reference dye 0.4 μL,Water nuclease-free 3.6 μL,上下游引物各2 μL,cDNA 2 μL;反应条件:95 ℃预变性60 s,95 ℃ 15 s、60 ℃ 15 s、72 ℃ 45 s共40个循环。每个样本设3个复孔。以β-actin为内参,采用2-ΔΔCt法计算目的基因相对表达量。实验重复3次,取平均值。
1.4 短扩增子和长扩增子扩增效率评估 采用qRT-PCR法。选择冰冻组织和石蜡组织各5例份,采用1.2中方法提取结直肠癌组织总RNA。为消除个体差异的干扰,将从冰冻组织和石蜡组织提取的结直肠癌组织总RNA混合,分别使用配对的长、短扩增子扩增,绘制4条LncRNA和内参β-actin的标准曲线。根据标准曲线计算扩增子的扩增效率和测定系数(R2)。设计4对短引物,每条LncRNA与1.3中各LncRNA长引物共享上游引物序列。LncRNA-Enst00000554679(short-A):下游引物5′-AATCAAGCAAGTGGAAATG-3′,LncRNA-Enst00000550851(short-A):下游引物5′-ATGGAAGTTGCCTGTTTAC-3′,LncRNA-Enst00000497498(short-A):下游引物5′-ACCCAAACAACTCACAACG-3′,LncRNA-Enst00000513016(short-A):下游引物5′-TATAGACCTTTATCCTTCC-3′。内参β-actin的上下游引物同1.3。其余步骤同1.3。
1.5 短扩增子和长扩增子定量一致性评估 选择冰冻组织和石蜡组织各10例份,采用qRT-PCR法检测短扩增子和长扩增子扩增后靶LncRNA表达,具体步骤同1.3、1.4。分析结直肠癌组织与癌旁正常组织、淋巴结转移组织与结直肠癌组织靶LncRNA扩增循环数(Ct)的比值,>2.0为靶LncRNA表达上调,<0.5为靶LncRNA表达下调,0.5~2.0为靶LncRNA表达无明显变化。分析冰冻组织中短扩增子和长扩增子的定量一致性以及冰冻组织和石蜡组织短扩增子的定量一致性。
1.6 短扩增子对石蜡组织中LncRNA定量分析的可靠性评估 分别为4条LncRNA设计3对短扩增子引物(short-A、Short-B、Short-C),short-A同1.4。另外2个短扩增子引物序列:LncRNA-Enst0000055-4679:short-B上游引物5′-CTCATGGAAACAAGCAC-TG-3′,下游引物5′-TGCCAGATACCTGTCATTG-3′;short-C上游引物5′-TGAATGCTACAGCCCTATG-3′,下游引物5′-GCCAGCAGTAACATTGTTG-3′。LncRNA-Enst00000550851:short-B上游引物5′-GACTTTAACTGGAGCACAG-3′,下游引物5′-TGATACGCACAGCACTTGG-3′;short-C上游引物5′-GATCCTGGTAACAAATGGC-3′,下游引物5′-TTTTGGCATCCATA-TTGGG-3′。LncRNA-Enst00000497498:short-B上游引物5′-CCCTATTTTAAGCCATGTG-3′,下游引物5′-TACGTGTCCTAACCATGTG-3′;short-C上游引物5′-ACGTTTGAAATGTGCGCTC-3′,下游引物5′-AGGTAAGTTTGCATGACAC-3′。 LncRNA-Enst00000513016:short-B上游引物5′-TTTCTTCTGTCATACACTG-3′,下游引物5′-ACACTCAAATCACTCATTC-3′;short-C上游引物5′-TTCTGTCACATACACTCCC-3′,下游引物5′-CTATTAGGGTAACTGCTTC-3′。内参β-actin的上下游引物同1.3。选择40例份石蜡组织,采用qRT-PCR法检测3个短扩增子扩增后靶LncRNA表达,具体步骤同1.3、1.4。分析石蜡组织中3个短扩增子的定量一致性,方法同1.5。
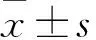
2 结果
2.1 不同冰冻组织4条LncRNA表达比较 见表1。
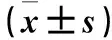
表1 不同冰冻组织4条LncRNA相对表达量比较
注:与癌旁正常组织比较,*P<0.05;与结直肠癌组织比较,#P<0.05。
2.2 冰冻组织与石蜡组织扩增效率评估 在冰冻组织中,4条LncRNA的短扩增子扩增效率为(101.0±3.1)%,达到最佳扩增效率(90%~110%);长扩增子扩增效率为(176.0±30.9)%,未达到最佳扩增效率;短扩增子的R2为0.998±0.004,长扩增子为0.831±0.101,二者比较P<0.05。在石蜡组织中,4条LncRNA的短扩增子扩增效率为(102.0±3.7)%,达到最佳扩增效率;长扩增子扩增效率为(329.0±192.0)%,未达到最佳扩增效率;短扩增子的R2为0.987±0.013,长扩增子为0.607±0.221,二者比较P<0.05。
2.3 冰冻组织与石蜡组织短扩增子和长扩增子对LncRNA定量一致性评估 见表2、3。经分析,在冰冻组织中短扩增子和长扩增子的定量一致性为62.5%;冰冻组织与石蜡组织短扩增子的定量一致性仅为25.0%。
2.4 石蜡组织中短扩增子对LncRNA定量分析的可靠性评估 经分析发现,4条LncRNA中3个短扩增子Ct变化趋势一致率为37.5%,2个短扩增子Ct变化趋势一致率达到100%。
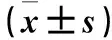
表2 冰冻组织4条LncRNA短扩增子和长扩增子Ct比较
注:与短扩增子比较,*P<0.05。
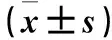
表3 冰冻组织与石蜡组织4条LncRNA短扩增子Ct比较
注:两种组织短扩增子Ct比较P均>0.05。
3 讨论
随着我国经济的发展,居民膳食结构和生活方式的变化,近年来结直肠癌的发病率逐年上升,其发病率和病死率分别居所有恶性肿瘤的第3位和第4位[13]。虽然目前对结直肠癌发生、发展机制的研究已经取得了许多新进展,但其预后差仍是当前面临的严峻问题。侵袭和转移是结直肠癌患者预后不良的主要原因[14]。因此,探索结直肠癌侵袭和转移的机制,对其早期诊断及靶向治疗至关重要。
LncRNA是一类长度大于200 bp、不具备翻译成蛋白质能力但可调控基因表达的一类RNA。一直以来,LncRNA被认为是进化过程中累积的“垃圾序列”,不具备任何生物学功能。随着对整个转录组的分析发现,LncRNA可能在肿瘤的发生、发展过程中具有重要作用[15,16]。有研究证实,LncRNA可在染色体修饰、转录前后多个层面上对基因表达、剪接等进行调控,进而参与结直肠癌的侵袭和转移[17~19]。此外,LncRNA的组织特异性使其在诊断中的敏感性明显高于蛋白质编码的RNA、DNA或蛋白质标记物[20]。本课题组前期利用高通量基因芯片技术,筛选出4条与结直肠癌转移相关的LncRNA,并发现其在转移淋巴结组织中相对表达量明显升高。据此推测这4条LncRNA可能作为癌基因参与结直肠癌的发生、发展。
石蜡组织是生物医学研究的重要资源,在医学研究中具有不可替代的作用。但石蜡组织中分离的核酸通常会发生化学结构断裂或化学性质改变[21,22],加上固定液的化学修饰以及各种酶的破坏等,故用长扩增子进行PCR定量时效果欠佳。近年研究发现,短扩增子也可用于PCR定量[12]。石蜡组织中LncRNA发生不同程度降解的情况是无法得到纠正的,这种降解削弱了扩增子定量的可靠性。如果基因断裂发生在扩增子序列或引物序列中则会导致扩增失败,并使定量的可靠性进一步降低。因此,无论使用一个长或短扩增子,在冰冻组织和石蜡组织中都不能准确地验证LncRNA表达。本研究我们采用3个非重叠的短扩增子进行qRT-PCR检测,从石蜡组织中获取LncRNA的可靠定量数据,从而使在石蜡组织中定量分析LncRNA成为可能。
扩增效率是显示扩增子是否有效的重要指标。前期研究证实,扩增子的长短可影响LncRNA定量分析[8]。本研究在引物设计方面,让短扩增子和相应的长扩增子共享引物序列,即短扩增子就是长扩增子的一部分片段。通常认为,PCR扩增的最佳效率为90%~110%。如果扩增子的扩增效率较低,表明相应的引物几乎不起作用;而扩增效率较高,则表明可能存在PCR抑制[23]。本研究结果显示,长扩增子在冰冻组织和石蜡组织的扩增效率均较差,但短扩增子可达到最佳扩增效率。参数R2表示扩增效率的可重复性,本研究短扩增子的R2明显高于长扩增子。进一步证实短扩增子的扩增效率稳定。
通常认为,冰冻组织中LncRNA比石蜡组织保存更好。本研究结果发现,短扩增子在冰冻组织、石蜡组织中Ct比较并无统计学差异。表明使用短扩增子在石蜡组织中对LncRNA定量分析是有效的。进一步分析发现,短扩增子和长扩增子在冰冻组织中的定量一致性为62.5%,而冰冻组织和石蜡组织短扩增子的定量一致性仅为25.0%。表明用单个短扩增子进行定量分析结果并不可靠。为此,我们选择了3个短扩增子对石蜡组织的LncRNA进行定量分析,结果发现4条LncRNA中至少2个短扩增子Ct变化趋势一致率达到100%。证实使用3个非重叠的短扩增子在石蜡组织中进行LncRNA定量分析是可行的。
综上所述,筛选的4条LncRNA在结直肠癌转移组织中表达升高;在标准化保存的石蜡组织中,使用短扩增子对LncRNA进行定量分析是可行的。但LncRNA在结直肠癌转移中的具体作用机制仍需进一步研究。
参考文献:
[1] Mercer TR, Dinger ME, Mattick JS. Long non-coding RNAs: insights into functions[J]. Nat Rev Genet, 2009,10(3):155-159.
[2] Wilusz JE, Sunwoo H, Spector DL. Long noncoding RNAs: functional surprises from the RNA world[J]. Genes Dev, 2009,23(13):1494-1504.
[3] Wang KC, Chang HY. Molecular mechanisms of long noncoding RNAs[J]. Mol Cell, 2011,43(6):904-914.
[4] Li CH, Chen Y. Targeting long non-coding RNAs in cancers: progress and prospects[J]. Int J Biochem Cell Biol, 2013,45(8):1895-1910.
[5] Bonin S, Petrera F, Niccolini B, et al. PCR analysis in archival postmortem tissues[J]. Mol Pathol, 2003,56(3):184-186.
[6] Masuda N, Ohnishi T, Kawamoto S, et al. Analysis of chemical modification of RNA from formalin-fixed samples and optimization of molecular biology applications for such samples[J]. Nucleic Acids Res, 1999,27(22):4436-4443.
[7] Carninci P. Molecular biology: the long and short of RNAs[J]. Nature, 2009,457(7232):974-975.
[8] Ren S, Wang F, Shen J, et al. Long non-coding RNA metastasis associated in lung adenocarcinoma transcript 1 derived mRNA as a novel plasma-based biomarker for diagnosing prostate cancer[J]. Eur J Cancer, 2013,49(13):2949-2959.
[9] Wang S, Wang L, Zhu T, et al. Improvement of tissue preparation for laser capture microdissection: application for cell type-specific miRNA expression profiling in colorectal tumors[J]. BMC Genomics, 2010(11):163.
[10] Wang S, Wang L, Bayaxi N, et al. A microRNA panel to discriminate carcinomas from high-grade intraepithelial neoplasms in colonoscopy biopsy tissue[J]. Gut, 2013,62(2):280-289.
[11] Huang W, Hu J, Yang DW, et al. Two microRNA panels to discriminate three subtypes of lung carcinoma in bronchial brushing specimens[J].Am J Respir Crit Care Med, 2012,186(11):1160-1167.
[12] Kong H, Zhu M, Cui F, et al. Quantitative assessment of short amplicons in FFPE-derived long-chain RNA[J]. Sci Rep, 2014(4):7246.
[13] Torre LA, Bray F, Siegel RL, et al. Global cancer statistics, 2012[J]. CA Cancer J Clin, 2015,65(2):87-108.
[14] Chen W, Zheng R, Baade PD, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin, 2016,66(2):115-132.
[15] Xiong XD, Ren X, Cai MY, et al. Long non-coding RNAs: an emerging powerhouse in the battle between life and death of tumor cells[J]. Drug Resist Updat, 2016(26):28-42.
[16] Zhang R, Xia LQ, Lu WW, et al. LncRNAs and cancer[J]. Oncol Lett, 2016,12(2):1233-1239.
[17] Gutschner T, Diederichs S. The hallmarks of cancer: a long non-coding RNA point of view[J]. RNA Biol, 2012,9(6):703-719.
[18] Yoon JH, Abdelmohsen K, Gorospe M. Functional interactions among microRNA and long noncoding RNA[J]. Semin Cell Dev Biol, 2014(34):9-14.
[19] Qi P, Xu MD, Ni SJ, et al. Down-regulation of ncRNA, a long non-coding RNA, contributes to colorectal cancer cell migration and invasion and predicts poor overall survival for colorectal cancer patients[J]. Mol Carcinog, 2015,54(9):742-750.
[20] Slaby O. Non-coding RNAs as biomarkers for colorectal cancer screening and early detection[J]. Adv Exp Med Biol, 2016(937):153-170.
[21] Xi Y, Nakajima G, Gavin E, et al. Systematic analysis of microRNA expression of RNA extracted from fresh frozen and formalin-fixed paraffin-embedded samples[J]. RNA, 2007,13(10):1688-1674.
[22] Li J, Smyth P, Flavin R, et al. Comparison of miRNA expression patterns using total RNA extracted from matched samples of formalin-fixed paraffin-embedded (FFPE) cells and snap frozen cells[J]. BMC Biotechnol, 2007(7):36.
[23] Krohn S, Bohm S, Engelmann C, et al. Application of qualitative and quantitative real-time PCR, direct sequencing, and terminal restriction fragment length polymorphism analysis for detection and identification of polymicrobial 16S rRNA genes in ascites[J]. J Clin Microbiol, 2014,52(5):1754-1757.
