高温条件下桑螟肠道微生物的多样性分析
2018-05-11梁燕婷刘云财高云王华兵徐豫松
梁燕婷,刘云财,高云,王华兵,徐豫松
(浙江大学动物科学学院,杭州310058)
昆虫肠道是一个重要的消化器官,其中栖息着大量的微生物。在长期进化过程中,宿主昆虫与肠道微生物相互合作、相互影响、协同进化,形成了不可分割的共生关系[1]。昆虫肠道的微生物群落从简单到复杂,对宿主的营养和消化、病原体的抗性、植物代谢物的解毒以及繁殖等方面起着重要作用[2-3]。正常的肠道菌群甚至被视为一种独立的“器官”而增加机体的适应性[4]。肠道细菌与其宿主之间的关系可以从共生到致病,从兼性到专性[5-6]。在病原体作用的特定时间中,肠道细菌与宿主可以被描述为互惠共生体,但在极端情况下,当适合的环境条件出现时,共生细菌可能会转变成病原体[7]。在正常情况下,不同来源的微生物在昆虫肠道内形成相对稳定的微生物环境,但当微生物群落的组成或位置发生变动时可能使宿主致病甚至死亡。当病原菌入侵昆虫时,会引起系统感染,昆虫肠道中的有益微生物对病原菌亦能产生拮抗作用,帮助昆虫抵御外来病原微生物。
传统的微生物培养是分析肠道微生物群落多样性和鉴定肠道微生物种类最经典的方法[8]。常规的分离培养方法会对微生物起筛选作用,其结果是在特定生境中微生物多样性可能偏离实际情况,如一些低丰度的细菌在合适的条件下迅速生长,并成为优势菌;反之,一些本来高丰度的微生物由于不适应人工培养条件而不能被分离鉴定出来。因此,常规分离鉴定方法不能真正显示肠道中微生物的多样性及种群结构。此外,在环境中约有90%的微生物不能培养[9],并且菌株培养鉴定过程烦琐且容易出现偏差。随着分子生物学的发展,基于核酸序列分析的现代分子生物学技术已广泛应用到昆虫的肠道微生物学研究中。细菌核糖体16SrDNA基因可变区是此类生物物种的特征核酸序列,可用来进行细菌的分类鉴定[10]。利用焦磷酸测序技术检测细菌16SrRNA基因序列的方法分析家蚕雌、雄幼虫中肠内微生物的多样性发现,雌蚕与雄蚕肠道微生物类群的组成和所占比率存在明显差异[11]。聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reaction-denaturing gradient gel electrophoresis,PCR-DGGE)和16SrDNA文库序列分析表明,家蚕(Bombyx mori)幼虫的肠道细菌在不同发育阶段存在差异,而肠道特殊菌群的出现与食性有关,饲料改变会引起肠道微生态平衡发生变化[12]。对小菜蛾(Plutella xylostella)敏感品系幼虫中肠微生物16S rRNA基因V6高变区进行测序分析发现,中肠细菌的组成和结构能够影响其对农药的抗性[13]。
目前,有关昆虫肠道细菌种群的研究多数集中在肠道细菌种群的结构、多样性和肠道细菌组成的相互关系方面,有关环境胁迫对肠道细菌菌群结构的影响及其协同作用机制的研究还较少。桑螟(Glyphodes pyloalis Walker)属鳞翅目螟蛾科昆虫,是桑树的主要害虫。桑螟通过食用桑叶,使桑叶产量和品质下降,其携带的病原可传播至家蚕而危害蚕桑产业[14]。桑螟对高温高湿的环境适应能力较强,夏秋季高温多湿环境易导致桑螟的爆发。本研究利用16SrDNA高通量测序技术对桑螟高温处理前后肠道菌落的多样性和结构进行分析,解析不同温度对桑螟肠道微生物多样性变化和菌群结构的影响,为研究逆境昆虫与肠道微生物的协同进化提供线索,也为从肠道微生物的角度研究害虫防治提供参考。
1 材料与方法
1.1 试虫来源及处理
试验材料桑螟幼虫采自浙江省杭州市浙江大学紫金港校区的桑园内。收集的桑螟在室温(25±1)℃、相对湿度85%、光周期16 h/8 h(昼/夜)条件下用桑叶饲养。选取饲养获得的桑螟五龄幼虫作为供试昆虫,分别将其放在25℃和37℃条件下处理12 h。用75%乙醇溶液对幼虫体表进行消毒,在无菌操作条件下解剖,取出完整肠道,用无菌水缓慢清洗后取其肠道内容物,放入灭菌后的2 mL离心管中,-80℃保存,用于DNA提取。每个样品取8头以上桑螟幼虫,并设3次重复试验。样品分别命名为常温样品(CT)和高温样品(HT)。
1.2 DNA抽提、扩增、纯化及测序
按从土壤中提取DNA试剂盒(OMEGA公司,美国)说明书抽提桑螟肠道内容物总基因组DNA,用1%琼脂糖凝胶电泳检验DNA质量,用紫外分光光度计(Thermo Fisher科技公司,美国)测定DNA浓度。16SrDNA基因V4区PCR扩增体系为:Taq DNA聚合酶25μL,正向和反向引物各2.5μL(10 μmol/L),ddH2O 20μL。PCR反应条件为:95℃预变性50 s;94 ℃变性30 s,58 ℃退火25 s,72 ℃延伸50 s,35个循环;72℃延伸10 min。PCR引物为V4通用引物(上游:5'-AAATTTTTTTTCCCCCCCGG-3';下游:5'-GGGCCCCCTTTAAAAAAAAAC-3'),扩增产物用凝胶回收试剂盒[购自生工生物工程(上海)股份有限公司]进行纯化,并委托华大基因进行Illumina测序。
1.3 测序序列分析
将原始数据中质量较低的序列(reads)滤除,得到较高质量的干净序列(clean reads);通过序列(reads)之间的重叠区域将其拼接成预聚类后的序列(tags);在97%相似度条件下将tags聚类形成操作分类单元(operational taxonomic units,OTU),根据所得OTU与各大数据库比对,并对其进行注释;基于注释结果对样品间微生物结构及不同处理间注释的差异进行分析。去除低质量序列(reads)需要符合以下几点:1)移除最终序列(read)长度低于原始长度75%的序列(reads);2)去除接头污染的序列(reads);3)去除含N的序列(reads);4)去除低复杂度的序列(reads)。
1.4 数据分析
将相似度大于97%的序列(reads)聚类到1个OTU中,使用Mothur软件及贝叶斯分类器,根据规范化的核糖体数据集将不同OTU的代表序列分类到属水平上。利用Mothur软件分析不同样品的α多样性,用丰度稀释曲线评估物种的丰度。采用艾斯(Ace)指数、观察到的物种数、赵氏(Chao)指数、香农(Shannon)指数以及辛普森(Simpson)指数等来评价样品中的物种数。其中,前面4个指数越大,最后1个指数越小,说明测得的样品微生物种类越丰富。利用MEGA 5.0软件构建系统进化树,利用SPSS19.0软件绘制样品中菌属分布柱形图。
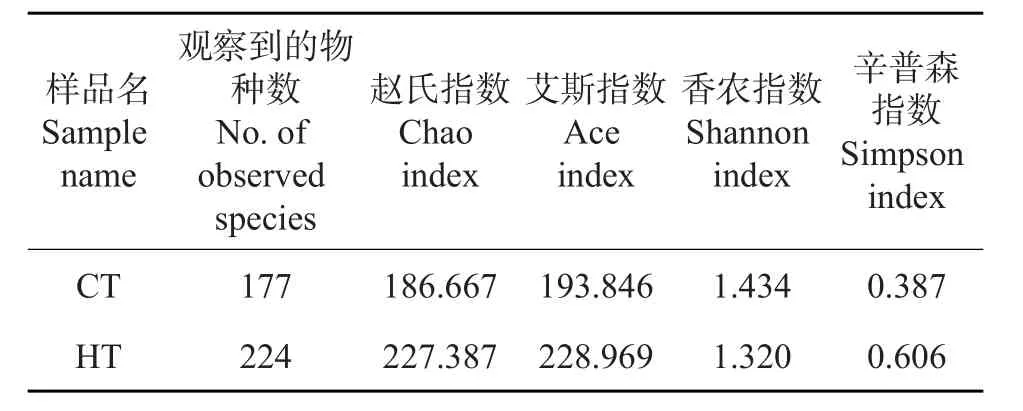
表1 样品中微生物丰富度及多样性指数Table1 Abundances and diversity indexes of microorganism in two samples
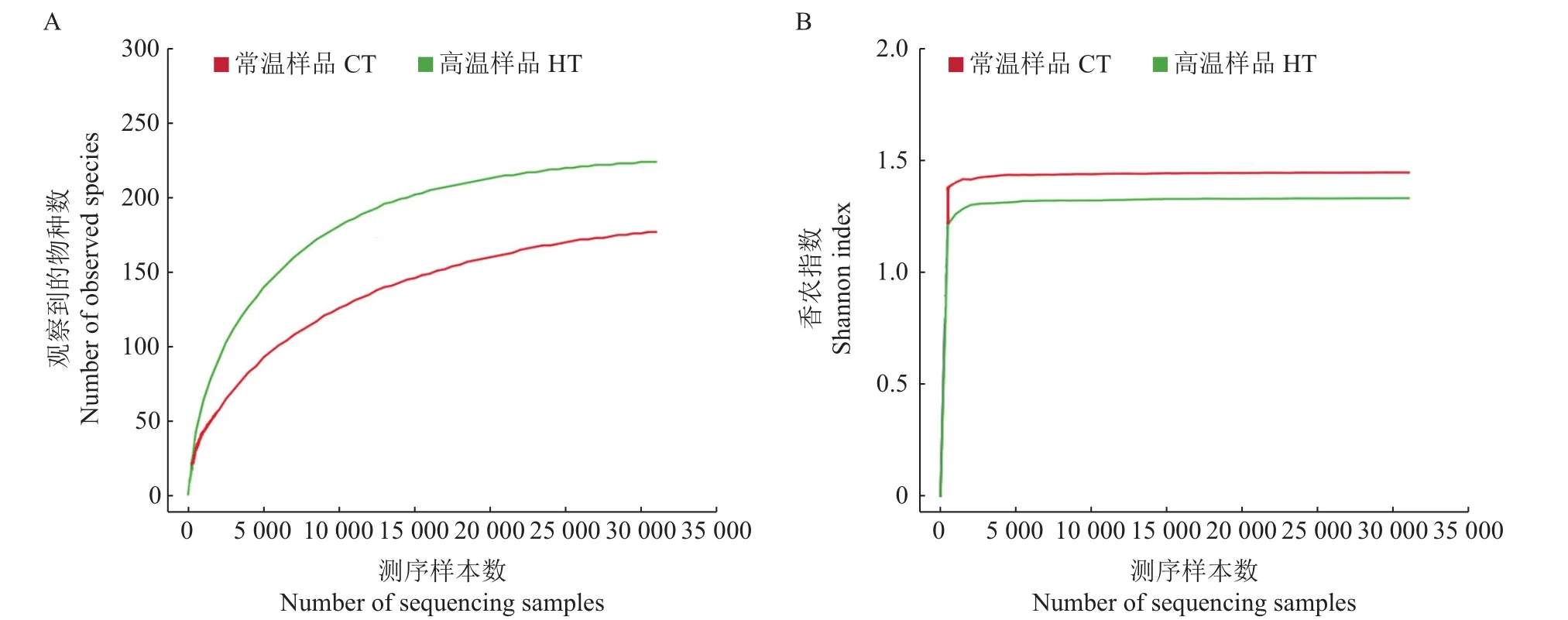
图1 桑螟肠道细菌的丰度稀疏曲线(A)及香农曲线图(B)Fig.1 Abundancerarefaction curve(A)and Shannon curve(B)of intestinal bacteriain G.pyloalis
2 结果与分析
2.1 桑螟肠道菌群结构
利用计算所得的OTUs、Chao指数、观察到的物种数、Ace指数,Shannon指数和Simpson指数来评价克隆文库的大小及内生细菌的多样性,结果如表1所示。从中可知:对于Chao指数、观察到的物种数、Ace指数、Shannon指数及Simpson指数,前面4个指数较大,最后1个指数较小,说明样品中的物种比较丰富;且HT的Ace指数和Chao指数均大于CT,说明高温下桑螟肠道菌群丰富度显著大于正常温度下的肠道菌群。从图1可以发现,丰度稀疏曲线和Shannon指数都达到了平稳状态,说明微生物多样性测序深度覆盖了样品中大部分微生物。
从图2中可以看出:在桑螟的肠道菌群门水平上,优势菌门为变形菌门和拟杆菌门;酸杆菌门(Acidobacteria)在高温处理的样品中没有检测到;而梭杆菌门(Fusobacteria)、浮霉菌门(Planctomycetes)、软壁菌门(Tenericutes)为高温处理后新增的。分析桑螟在正常条件下的优势菌门可以发现,变形菌门(Proteobacteria)占绝大部分(57.52%),其次为蓝藻菌门(Cyanobacteria)(35.30%),拟杆菌门(Bacteroidetes)(3.55%),厚壁菌门(Firmicutes)(1.80%),放线菌门(Actinobacteria)(1.73%),疣微菌门(Verrucomicrobia)(0.04%),未分类门(0.04%)以及酸杆菌门(0.006 424%)。已有研究表明,鳞翅目昆虫肠道中的细菌主要为变形菌门、厚壁菌门和放线菌门[8],本试验结果与其相符。在门水平上比较HT与CT肠道微生物的结构可以说明,这些优势菌门可能在桑螟的生长繁殖及抵御非生物胁迫过程中发挥着重要作用。
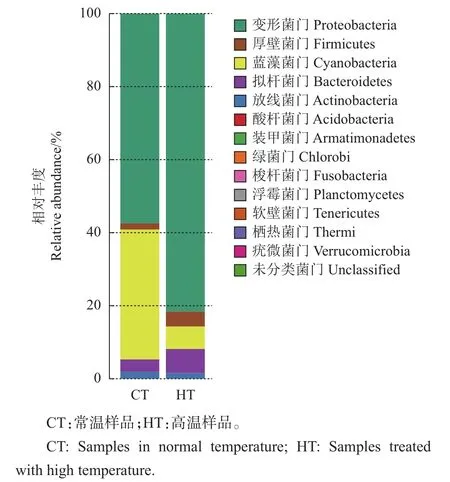
图2 在门水平上HT与CT肠道微生物的结构比较Fig.2 Structural comparison of gut microbiota in HTand CTat phylum level
从图3中可以看出,在属水平上,CT和HT样品中优势菌属所占比例分别为:沃尔巴克菌属(Wolbachia)占51.16%和77.54%,未分类属占38.87%和9.55%,节细菌属(Arthrobacter)占1.54%和0.89%,多形杆状菌属(Bacteroides)占1.48%和2.60%,埃希菌属(Escherichia)占1.31%和0.18%,吉氏副拟杆菌属(Parabacteroides)占1.40%和0.16%,交替假单胞菌属(Pseudoalteromonas)占0.91%和 0.53%,葡萄球菌属(Staphylococcus)占0.56%和0.52%,噬纤维素菌属(Cellulophaga)占0.19%和0.14%,以及普雷沃菌属(Prevotella)占0.10%和3.00%。其中,沃尔巴克菌属和普雷沃菌属的相对丰度变化比较大,说明它们对高温比较敏感。
2.2 桑螟肠道菌群聚类分析
为了研究高温对桑螟肠道优势种群的影响,选取相对丰度前15的菌属进行聚类分析,比较正常和高温处理桑螟的肠道菌属。结果(图4)显示:高温处理变化最显著的是假单胞菌属(Pseudomonas),其丰度为正常饲养时的48.74倍;其次为普雷沃菌属、普拉梭菌属(Faecalibacterium)和帕拉普菌属(Paraprevotella),其丰度分别为正常处理的29.24、22.78和6.64倍;多形杆状菌、沃尔巴克菌在高温时的丰度分别为正常状态下的1.75倍和1.52倍;节细菌属、噬纤维素菌属、埃希菌属、吉氏副拟杆菌属在高温处理后丰度下降,分别是对照的58%、70%、14%和11%。这些结果表明,高温抑制了杆菌的繁殖,促进了其他菌的繁殖。

图3 在属水平上HT与CT肠道微生物的结构比较Fig.3 Structural comparison of gut microbiota in HTand CTat genuslevel
2.3 桑螟肠道微生物进化分析
根据桑螟肠道中代表性微生物序列构建系统进化树,对桑螟肠道优势菌群进行进化分析。从图5中可以看出,桑螟肠道中的微生物主要分为5大类(Ⅰ、Ⅱ、Ⅲ、Ⅳ和Ⅴ)。Ⅰ类主要由放线菌门组成,分为4个亚类,分别为Ⅰ-Ⅰ(主要为产气柯林斯菌属),Ⅰ-Ⅱ(主要为棒状杆菌属),Ⅰ-Ⅲ(主要为丙酸杆菌属),Ⅰ-Ⅳ(主要为短杆菌属)。Ⅱ类主要由厚壁菌门组成,分为4个亚类,分别为Ⅱ-Ⅰ(主要为普拉梭菌属),Ⅱ-Ⅱ(主要为Dehalobacterium),Ⅱ-Ⅲ(主要为葡萄球菌属),Ⅱ-Ⅳ(主要为梭状芽孢杆菌属)。Ⅲ类主要由变形菌门和拟杆菌门组成,分为4个亚类,分别为Ⅲ-Ⅰ(主要为沃尔巴克菌属),Ⅲ-Ⅱ(主要为甲基杆菌属),Ⅲ-Ⅲ(主要为帕拉普菌属),Ⅲ-Ⅳ(主要为弓形杆菌属)。Ⅳ类主要由变形菌门组成,分为2个亚类,分别为Ⅳ-Ⅰ(主要为寡养单胞菌属)和Ⅳ-Ⅱ(主要为Massilia)。Ⅴ类主要为Arabidopsis属。
3 讨论
昆虫在长期进化过程中,肠道微生物发挥着重要作用。昆虫通常从环境和食物中获得各类微生物,经过长期进化,有些微生物群落经过肠道环境选择后与昆虫共生,这些肠道微生物通过参与昆虫营养、消化和防御从而影响其发育。当昆虫处在不利于进食的环境中时,肠道内的酶可消化吸收昆虫肠道微生物中的营养成分,有利于昆虫的生长发育。
桑螟作为一种对蚕桑业危害严重的害虫,长期生存在外界环境中,不断受到非生物胁迫,经过自然选择,桑螟抗逆性不断增强,但目前对于桑螟肠道菌群结构的研究还比较少。在本研究中,我们利用16SrDNA高通量测序技术探究了高温对桑螟肠道菌群结构的影响,并使用多种方法分析了桑螟肠道菌群的组成变化与高温胁迫耐受性之间的关联性。

图4 在属水平上HT和CT肠道优势菌群的聚类分析Fig.4 Cluster analysisof dominant intestinal florain HTand CTat genuslevel
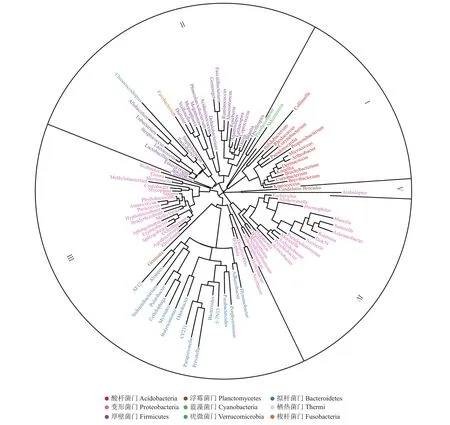
图5 桑螟肠道优势菌群进化分析Fig.5 Evolutionary analysis of dominant bacterial community in the gut of G.pyloalis
在桑螟肠道中,高温处理后节细菌属、埃希菌属、葡萄球菌属等的丰度明显降低。节细菌属广泛分布于环境中,有的种的杆状细胞好氧、不抗酸、不生孢。已有研究表明,节细菌属能够有效提高对有机磷类农药甲基对硫磷的分解能力[15]。埃希菌属以周生鞭毛运动或不运动,兼有发酵和呼吸3种代谢方式。研究表明,埃希菌属最适生长温度为37℃,能够使葡萄糖和其他糖类发酵并产生丙酮酸,再进一步转化为甲酸、乙酸和乳酸,甲酸又会被分解为等量的二氧化碳和氢气[16]。而对于葡萄球菌属,其中的很多菌种能够分解麦芽糖、蔗糖和葡萄糖,产酸不产气[17]。葡萄球菌丰度降低,则桑螟体内糖含量增高,机体渗透压升高以维持机体稳定。此外,葡萄球菌的致病性菌株能分解甘露醇。桑螟肠道中这些菌属在高温时的比例降低,可能的原因有:1)较高的温度不适合这些菌生存;2)可能与桑螟在高温条件下的耐热性有关;3)高温可能通过改变其他菌落使机体内环境改变,进而导致这些菌的丰度下降。
而在桑螟肠道中,普雷沃菌属、普拉梭菌属、帕拉普菌属、多形杆状菌属和沃尔巴克菌属的丰度都明显升高。普雷沃菌属的主要发酵产物是琥珀酸、少量异丁酸和乙酸,研究认为:最适合普雷沃菌生长的温度是37℃,在20%胆汁酸和6.5%NaCl作用下其生长会被抑制;普雷沃菌在宿主的代谢过程中发挥着重要作用,它不能还原硝酸盐,且利用氨基酸的能力低,会产生神经鞘脂[18]。对于普拉梭菌,它能够通过释放丁酸和某种尚不明确的物质产生抗炎作用,从而纠正肠道菌群失调[19]。对沃尔巴克菌的研究结果显示,给斯氏按蚊添加沃尔巴克菌,能使斯氏按蚊与沃尔巴克菌形成稳定的共生关系,从而使这种蚊具有抵抗疟原虫的免疫能力,并能将这种免疫能力传给后代[20]。此外,沃尔巴克菌也可阻止登革热病毒在蚊子体内复制增殖。因此,推测桑螟肠道中这些菌的丰度升高与桑螟较强的适应性有关;并且可以认为,肠道细菌的差异是在长期进化过程中肠道微生物协同进化、生态适应的结果。
昆虫肠道微生物丰富多样,许多菌群与昆虫长期进化维持共生关系,在昆虫生命活动中起着重要的作用。研究昆虫肠道微生物有利于全面了解昆虫的生命活动过程,也为基于肠道微生物开发新的绿色防控技术奠定基础。本研究首次利用16S rDNA高通量测序技术获得了桑螟肠道微生物相关信息,分析了桑螟经高温处理后肠道微生物的结构变化与多样性。但其变化的原因及耐高温的调节机制仍有待进一步研究。
4 结论
本研究高温处理试验分析表明,在高温下,桑螟肠道微生物在多样性和结构上均有明显的变化,这些变化的菌落多与消化吸收及免疫防御相关。在正常条件下,桑螟肠道的优势菌落在门水平上为变形菌门和蓝藻菌门;高温处理后,优势菌群为变形菌门,其次则为拟杆菌门,没有检测到酸杆菌门,但新增了梭杆菌门、浮霉菌门和软壁菌门。从属的水平上分析,在正常温度下,优势菌群为沃尔巴克菌属、节细菌属和多形杆状菌属;高温处理后则为沃尔巴克菌属和普雷沃菌属。在丰度上,高温处理后假单胞菌属的变化最显著,为正常饲养时丰度的48.7倍,其次为普雷沃菌属。这些变化在一定程度上增加了桑螟的抗逆性,使其更好地适应高温环境。
参考文献(References):
[1] 相辉,黄勇平.肠道微生物与昆虫的共生关系.昆虫知识,2008,45(5):687-693.
XIANG H,HUANGY P.Symbiosis between gut microbiota and insects.Chinese Bulletin of Entomology,2008,45(5):687-693.(in Chinese with English abstract)
[2]HANDELSMAN J,ROBINSON C J,RAFFA K F.Microbial Communities in Lepidopteran Guts:From Models to Metagenomics.Cambridge,UK:Cambridge University Press,2005:143-168.
[3]LILBURN T G,KIM K S,OSTROM N E,et al.Nitrogen fixation by symbiotic and free-living spirochetes.Science,2001,292(5526):2495-2498.
[4]O’HARAA M,SHANAHAN F.The gut flora as a forgotten organ.EMBO Reports,2006,7(7):688-693.
[5] KIKUCHI Y.Endosymbiotic bacteria in insects:Their diversity and culturability.Microbes and Environments,2009,24(3):195-204.
[6]TADA A,KIKUCHI Y,HOSOKAWA T,et al.Obligate association with gut bacterial symbiont in Japanese populations of the southern green stinkbug Nezara viridula,(Heteroptera:Pentatomidae).Applied Entomology and Zoology,2011,46(4):483-488.
[7]THAKUR A,DHAMMI P,SAINI H S,et al.Pathogenicity of bacteria isolated from gut of Spodoptera litura(Lepidoptera:Noctuidae)and fitness costs of insect associated with consumption of bacteria.Journal of Invertebrate Pathology,2015,127:38-46.
[8] 陈勃生,鲁兴萌,邵勇奇.鳞翅目昆虫肠道微生物的多样性及其与宿主的相互作用.昆虫学报,2017,60(6):710-722.
CHEN B S,LU X M,SHAO Y Q.Diversity of the gut microbiota in Lepidopteran insects and their interaction with hosts.Acta Entomologica Sinica,2017,60(6):710-722.(in Chinese with English abstract)
[9]AMANN RI,LUDWIG W,SCHLEIFERK H.Phylogenetic identification and in situ detection of individual microbial cells without cultivation.Microbiological Reviews,1995,59(1):143-169.
[10]CLARRIDGE J E.Impact of 16S rRNA gene sequence analysis for identification of bacteria on clinical microbiology and infectious diseases.Clinical Microbiology Reviews,2004,17(4):840-862.
[11]许刚,孙振丽,胡小龙,等.基于16SrRNA基因序列分析家蚕肠道细菌的多样性.蚕业科学,2015,41(4):641-649.
XU G,SUN Z L,HU X L,et al.Analysis on intestinal bacterial diversity in silkworm(Bombyx mori)based on 16S rRNA gene sequences.Science of Sericulture,2015,41(4):641-649.(in Chinesewith English abstract)
[12]相辉,李木旺,赵勇,等.家蚕幼虫中肠细菌群落多样性的PCR-DGGE和16SrDNA文库序列分析.昆虫学报,2007,50(3):222-233.
XIANG H,LIM W,ZHAOY,et al.Bacterial community in midguts of the silkworm larvae estimated by PCR/DGGEand 16S rDNA gene library analysis.Acta Entomologica Sinica,2007,50(3):222-233.(in Chinesewith English abstract)
[13]夏晓峰.小菜蛾中肠微生物多样性及其功能研究.福州:福建农林大学,2014.
XIA X F. Organizational diversity and functional characterization of microbiota in the midgut of diamondback moth,Piutella xylostella(L.).Fuzhou:Fujian Agriculture and Forestry University,2014.(in Chinesewith English abstract)
[14]LIU Y C,SU H,LI R Q,et al.Comparative transcriptome analysis of Glyphodes pyloalis Walker (Lepidoptera:Pyralidae)reveals novel insights into heat stress tolerance in insects.BMCGenomics,2017,18(1):974.
[15]解秀平,闫艳春,刘萍萍,等.降解甲基对硫磷的节杆菌(Arthrobacter sp.)L4菌株的分离和降解特性研究.环境科学学报,2006,26(10):1637-1642.
XIE X P,YAN Y C,LIU P P,et al.Isolation,degradation and characterization of methylparathion degradative strain L4.Acta Scientiae Circumstantiae,2006,26(10):1637-1642.(in Chinese with English abstract)
[16]王立志,徐谊英.圈养大熊猫粪便中微生物多样性的研究.四川动物,2016,35(1):17-23.
WANG L Z,XU Y Y.Diversity of microorganism in the feces of captive giant pandas.Sichuan Journal of Zoology,2016,35(1):17-23.(in Chinese with English abstract)
[17]姜常青,赵永峰,刁非非,等.兔源金黄色葡萄球菌的分离鉴定.山东畜牧兽医,2015,36(9):10-11.
JIANG C Q,ZHAO Y F,DIAO F F,et al.Isolation and identification of Staphylococcus aureus from rabbits.Shandong Journal of Animal Science and Veterinary Medicine,2015,36(9):10-11.(in Chinese)
[18]MATISOO-SMITH E,ROBERTSK,WELIKALA N,et al.Recovery of DNA and pollen from New Zealand lake sediments.Quaternary International,2008,184(1):139-149.
[19]张明明,于成功.普拉梭菌与炎症性肠病关系的研究进展.中华消化杂志,2012,32(11):789-792.
ZHANG M M,YU C G.Progress on the relationship between Faecalibacterium prausnitzii and inflammatory bowel disease.Chinese Journal of Digestion,2012,32(11):789-792.(in Chinese)
[20]BIAN G W,JOSHI D,DONG Y M.Wolbachia invades Anopheles stephensi populations and induces refractoriness to Plasmodium infection.Science,2013,340(6133):748-751.
