大豆转录因子GmWRKY58 亚细胞定位及在非生物胁迫下的表达分析
2018-05-07张红梅刘晓庆陈华涛袁星星崔晓艳张智民黄中文
张红梅,刘晓庆,陈华涛,袁星星,崔晓艳,张智民,黄中文,陈 新
(1.江苏省农业科学院 经济作物研究所,江苏 南京 210014;2.河南科技学院 农学系,河南 新乡 453003)
干旱、高盐、低温、高温等极端条件,是植物生长过程中所面临的主要逆境胁迫因子。在逆境条件下,与植物抗逆相关基因被诱导表达,从而产生胁迫响应。植物响应非生物胁迫的基因可分为两类:①功能基因,如编码与胁迫有关的LEA蛋白、抗冻蛋白、渗透调节因子合成酶以及逆境胁迫产生的ROS等清除系统的相关酶基因[1];②编码与抗逆相关参与信号传递蛋白激酶和调控基因表达的转录因子,如MYB、bZIP、WRKY、AP2/EREBP和NAC等[2]。WRKY转录因子是植物中发现最早的一类转录调控因子,根据WRKY结构域的数目和锌指纹结构的类型,WRKY蛋白可分成3组[3]:第Ⅰ组含2个WRKY结构域,锌指纹结构模式为C-X4-5-C-X22-23H-X-H,如SPF1、ABF1、PcWRKY1和AtWRKY33[4-7]等;第Ⅱ和Ⅲ组均含1个WRKY 结构域,第Ⅱ组锌指纹结构模式与第Ⅰ组相同,如AtWRKY18(NP_567882)等;第Ⅲ组仅在组成锌指纹的氨基酸有变化,为C-X7-C-X22-23-H-X-C,如AtWRKY70(NP_191199)等。
Ishiguro 和Nakamura[4]在甘薯中发现了第一个WRKY转录因子SPF1。随后,其他研究者又相继在水稻、拟南芥、棉花、烟草、大麦、小麦和豆类[8-14]等植物中克隆得到WRKY转录基因。WRKY转录因子不仅参与植物抗病与非生物胁迫的响应,而且在植物生长发育、次生代谢等过程中起重要作用[15-20]。拟南芥AtWRKY22和AtWRKY29是MAPK通路中重要的下游组成成分[21],对拟南芥抵抗真菌和细菌等抗性具有调节作用。李立芹[22]研究发现,烟草WRKY12基因受4 ℃低温胁迫诱导表达。在拟南芥中过表达AtWRKY25和AtWRKY33 基因,能够显著增强转基因植株对高温和高盐的耐受性[23-25];过表达OsWRKY47能提高水稻的抗旱性[26]。拟南芥AtWRKY28基因受ABA、高盐、机械损伤和低磷胁迫诱导表达[27-28]。大豆基因组中大约有300个WRKY基因(http://planttfdb.cbi.pku.edu.cn/index.php?sp=Gma),但目前仅有少数第Ⅱ组成员的功能被鉴定,如GmWRKY111[29]、GmWRKY13、GmWRKY21和GmWRKY54[15]参与盐、干旱和冷害等多种非生物逆境胁迫的应答反应,它们过量表达,能增强植物对盐、干旱和冷害的适应性。
大豆(Glycinemax)是我国最重要的油料作物之一,干旱、盐害、低温和低磷等非生物胁迫严重影响大豆的产量和品质。已有研究表明,大豆很多WRKY参与非生物胁迫的转录调控,然而关于WRKY Group Ⅲ成员参与非生物胁迫响应及功能分析的研究较少。Yang等[30]研究发现,大豆WRKY家族第Ⅲ组的GmWRKY58和GmWRKY76在大豆发育过程中具有重要作用。过量表达GmWRKY58和GmWRKY76基因的拟南芥比野生植株开花早,这种提早开花的表型与几个促进开花基因的表达增加有关;病毒诱导大豆GmWRKY58和GmWRKY76基因沉默导致植物叶片扩展减少和茎生长停止等不良现象,但GmWRKY58在响应非生物逆境胁迫的表达模式还未见报道。本研究采用荧光定量方法,分析不同非生物逆境胁迫下GmWRKY58基因的表达水平,明确其响应的非生物逆境胁迫因子,为进一步探究GmWRKY58基因在非生物胁迫应答中的功能奠定基础。
1 材料和方法
1.1 试验材料及处理
本研究所用大豆材料为科丰1号,由江苏省农业科学院豆类作物研究室提供。采用改良的Zhang等[31]的方法于2015年6月大豆长到两叶一心时,取整齐度一致的幼苗放置在1/2 Hoagland营养液中生长2 d,换至正常营养液中生长,营养液pH调至6.0。2 d以后将大豆幼苗分别于含200 mmol/L NaCl 和2% PEG8000的Hoagland 营养液中进行高盐、干旱胁迫处理,分别在0,6,12,24,48 h取根、茎、叶样;于4 ℃培养箱进行低温处理,用2 mmol/L水杨酸(SA)喷施,分别取0,6,9,12,24,48 h叶片样;低N(75.8 mmol/L)、低P(5 μmol/L)、低K(96.5 mmol/L)和缺Fe(0 mmol/L)处理0,6,12,24,48 h取根、叶样,对照组分别采用正常营养液。
挑选籽粒饱满、大小一致的科丰1号种子种植于土壤中,待真叶自然展开后,于2015年5月19日取根、茎、叶,2015年6月19日取花,2015年7月3日取幼荚,2015年8月8日取幼嫩种子。每处理3 次重复。将样品迅速置于液氮中,-80 ℃保存备用。
1.2 大豆总RNA 提取与cDNA合成
采用RNA提取试剂盒(RNA plant plus Reagent,TIANGEN)提取大豆不同组织、不同处理时间点样品的总RNA,按照反转录试剂盒(M-MLV reverse transcriptase,TaKaRa)操作说明书合成cDNA第一链[32]。
1.3 基因克隆
从Phytozome(http://www.phytozome.net/)网站下载大豆GmWRKY58 核酸序列,采用Premier 5 设计特异引物,上游引物为GmWRKY58 F:5′-ATGA
GTATTCTCTTCCCAAGAAG-3′,下游引物为GmWRKY58 R:5′-CTAAAGCAATTGGCTTTCATC-3′。
以大豆cDNA为模板进行PCR扩增,扩增条件:94 ℃预变性3 min;94 ℃ 变性30 s,55 ℃退火1 min,72 ℃延伸2 min,35个循环;最后72 ℃再延伸10 min。25 μL的PCR反应体系中包括10×Taq缓冲液3.0 μL,Taq聚合酶0.5 μL,2.5 μL的dNTPs (2.5 mmol/L),MgCl2(25 mmol/L) 2.0 μL,cDNA模板2.0 μL,上下游引物各1 μL (10 pmol/L),最后用ddH2O补至25 μL。PCR 产物电泳后,切胶纯化回收。回收片段与pGEM-T easy载体连接,连接产物进行热激转化到大肠杆菌DH5α感受态细胞。经鉴定后的阳性克隆送Invitrogen公司测序。
1.4 生物信息学分析
使用在线软件Compute pI/Mwtool、ExPASy (http://expasy.org/tools/pi_tool.html)预测蛋白的分子量和等电点;通过SignalP (http://cbs.dtu.dk/services/SignalP/)在线分析GmWRKY58蛋白质的信号肽;由DNAMAN软件完成多序列比对;以MEGA 6 软件构建进化树;选取GmWRKY58 基因ATG上游1.5 kb的碱基序列,在 PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)网站预测顺式作用元件。http://www.genscript.com/wolf-psort.html进行亚细胞定位分析。二级结构预测使用http://www.biogem.org/tool/chou-fasman/完成。
1.5 实时荧光定量(qRT-PCR)分析
以单链cDNA序列设计实时荧光定量PCR引物:F:5′-CATGCATGGAGAAAGTACGG-3′;5′-GTTG
CACTTGTTTGGTTGCT-3′。以大豆组成型表达β-Tubuin基因(GenBank 登录号为AK286413)为内参基因(F:5′-GGAGTTCACAGAGGCAGAG-3′;R:5′-C
ACTTACGCATCACATAGCA-3′)。采用ABI PRISM7500实时荧光定量系统进行基因表达分析。反应体系为:模板cDNA 1.0 μL(10 ng/μL),2×SYBR Premix Ex Taq(TaKaRa)10.0 μL,Rox Reference Dye Ⅱ 0.4 μL,cDNA 1.0 μL,Primer F 0.8 μL、Primer R 0.8 μL,补ddH2O至20.0 μL。反应程序为:95 ℃ 1 min;95 ℃ 10 s,60 ℃ 30 s,72 ℃ 34 s,40个循环。采用2-ΔΔCT[33]法分析数据,计算GmWRKY58基因相对表达量。每个取样点设3次生物学重复,qRT-PCR为3次技术重复。
1.6 亚细胞定位
以GmWRKY58基因的ORF正确的克隆提取质粒,经NcoⅠ和SpeⅠ双酶切,同时pCambia1302质粒用NcoⅠ和SpeⅠ双酶切,分别回收小片段和大片段,将二者用T4-DNA连接酶进行连接,从而得到35S∷GmWRKY58∷GFP融合表达载体,转入DH5α大肠杆菌。用菌落PCR、酶切方法筛选阳性克隆,并测序验证。
获得的重组质粒35S∷GmWRKY58∷GFP转入TOP10克隆菌株。参照文献[34],在3.0 mL LB液体培养基(含50 mg/LKam,17 mg/LRif )中培养含双元载体的农杆菌,28 ℃,200 r/min 过夜培养,室温下3 000 r/min离心10 min后收集菌体,加1.0 mL注射培养基悬浮菌体,室温3 000 r/min离心10 min后弃上清,重复一次,最后用注射培养基悬浮。测600 nm吸光值(OD),当农杆菌菌液浓度(OD600)为0.1~0.2时,用无针头的注射器将农杆菌菌液注射到3~4周龄的本氏烟叶片。然后在温室中培养(27 ℃光照16 h,22 ℃黑暗8 h) 24~72 h,在激光扫描电镜下观察荧光蛋白的发光情况。
2 结果与分析
2.1 GmWRKY58 克隆及序列分析
从Phytozome(http://www.phytozome.net/)网站下载大豆GmWRKY58 核酸序列,根据CDS序列设计引物,以科丰1号大豆叶片cDNA为模板进行PCR,扩增出单一的条带。将测序得到的结果与大豆基因组数据库(http://www.phytozome.net/)进行序列比对,结果显示与大豆Williams 82.a2.v1的Glyma.04G223300(NP_001237638)基因序列完全一致 (图1)。目的片段大小为954个核苷酸,具有一个完整的开放阅读框。序列分析发现,氨基酸序列N端具有WRKYGQK的保守结构域,C端具有C-X7-C-X22-23-H-X-C保守结构域(图1),系WRKY家族基因GroupⅢ成员。

方框为WRKY结构域和锌指蛋白结构域。Boxes stands for the WRKY domain and zinc finger protein domain,respectively.
2.2 氨基酸序列及理化性质分析
GmWRKY58基因编码一个含有317个氨基酸,分子量为35.855 6 ku蛋白质,等电点为5.46。GmWRKY58为稳定在组织中的表达特性蛋白,无信号肽。GmWRKY58编码蛋白的整条肽链都位于细胞核。二级结构预测结果表明,在GmWRKY58蛋白中,螺旋结构占60.60%,肽链占59.0%,无规则卷曲占17.7%。
为了研究GmWRKY58的进化情况,笔者构建了GmWRKY58的系统进化树(图2)。结果显示:GmWRKY58与大豆WRKY70(XP_003526799)序列一致性最高,为91%;其次为绿豆WRKY70(XP_014500012)、鹰嘴豆WRKY70(XP_004502820)、巨桉WRKY70 (XP_010067621)、毛果杨WRKY70 (XP_002319879)、胡杨WRKY70 (XP_011014496)、苹果WRKY70 (XP_008369379)、麻风树WRKY70 (XP_012091128)和可可WRKY70 (XP_007011367),序列一致性为43%~74%;而与野草莓亚种WRKY70(XP_004305079)、桑树WRKY70(XP_010093537)、甜瓜WRKY70(XP_008442314)、烟草WRKY70(XP_009586653)和芝麻WRKY70(XP_011085118)的一致性较低。

Pe.胡杨;Pt.毛果杨;Jc.麻疯树;Tc.可可;Gr.雷蒙德氏棉;Vv.葡萄;Eg.巨桉;Ca.鹰嘴豆;Vr.绿豆;Gm.大豆;Md.苹果;Nn.莲;Fv.野草莓亚种;Mn.桑树;Cm.甜瓜;Nt.烟草;Si.芝麻。
PeWRKY70.Populuseuphratica,XP_011014496;PtWRKY70.Populustrichocarpa,XP_002319879;JcWRKY70.Jatrophacurcas,XP_012091128;TcWRKY70.Theobromacacao,XP_007011367;GrWRKY70.Gossypiumraimondii,XP_012460911;VvWRKY70.Vitisvinifera,XP_002275401;EgWRKY70.Eucalyptusgrandis,XP_010067621;CaWRKY70.Cicerarietinum,XP_004502820;VrWRKY70.Vignaradiatavar.,XP_014500012;GmWRKY70.Glycinemax,XP_003526799;MdWRKY70.Malusdomestica,XP_008369379;NnWRKY70.Nelumbonucifera,XP_010252044;FvWRKY70.Fragariavescasubsp.,XP_004305079;MnWRKY70.Morusnotabilis,XP_010093537;CmWRKY70.Cucumismelo,XP_008442314;NtWRKY70.Nicotianatomentosiformis,XP_009586653;SiWRKY70.Sesamumindicum,XP_011085118.
图2大豆GmWRKY58与其他植物WRKY蛋白的系统演化分析
Fig.2PhylogeneticanalysisofthededucedaminoacidsequencebetweenGmWRKY58andotherWRKYproteins
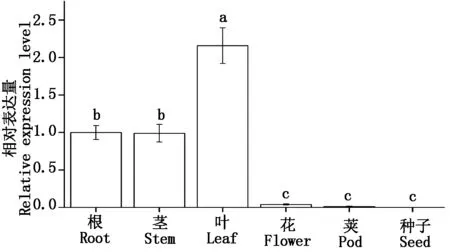
2.3 组织表达特性
GmWRKY58基因在根、茎、叶和花等器官中均有表达,在不同器官基因表达水平从高到低的顺序依次为:叶、根、茎、花、荚,在营养器官根、茎、叶中的表达量显著高于花和荚生殖器官中的表达量(图3)。其中,叶片中的表达量为根的2.16倍;在根和茎中的表达量相差不大,花和荚中的表达量较低。这可能是由于植物根茎叶在感知逆境信号和激素信号中起重要作用。
2.4 GmWRKY58 在非生物胁迫下的表达特性
用高盐、干旱、低温、SA、低N、低P、低K和缺Fe处理大豆幼苗,以qRT-PCR分析其表达动态(图4-6)。
结果表明,高盐胁迫能强烈诱导GmWRKY58表达,200 mmol/L的NaCl处理下(图4-A),GmWRKY58基因在根和茎中的表达呈先升高后下降的变化趋势,根和茎中上调表达至最高值分别出现在12,24 h,为相对表达量为0 h时的187.4,13.1倍;
不同小写字母表示在新复极差法分析中0.05水平上存在显著性差异。图4-6同。Bars superscripted by different lowercase letters are significantlydifferent at the 0.05 probability level.The same as Fig.4-6.
叶中表达呈下降趋势,在叶中的表达量则始终低于对照。2% PEG8000干旱处理后,在根、茎和叶中的诱导表达模式不尽相同,根中GmWRKY58表达量高于茎和叶。根和茎中最高表达量均出现在处理后48 h,分别是0 h时的15.1,18.4倍,表现为先升后降再升的趋势和上升趋势;叶中总体表现为先升后降的趋势,最高表达量出现在处理后12 h,是0 h时的10.0倍(图4-B)。低温(4 ℃)和水杨酸(SA)处理(图5)没有明显的上调或下调表达趋势,上调表达至最高值分别出现在处理后12 h 和24 h,相对表达量为0 h时的2.7,1.7倍。
低N、低P、低K和缺Fe处理后,GmWRKY58诱导表达模式存在差异。低N和缺Fe处理后,GmWRKY58基因在根中相对表达量高于叶片,最高表达量分别出现在处理后24,6 h,是0 h时的4.9,3.4倍(图6-A、D)。低P和低K处理后,GmWRKY58在叶中的相对表达量高于根,最高表达量均出现在叶中0 h,表现为先降后升的趋势(图6-B、C)。
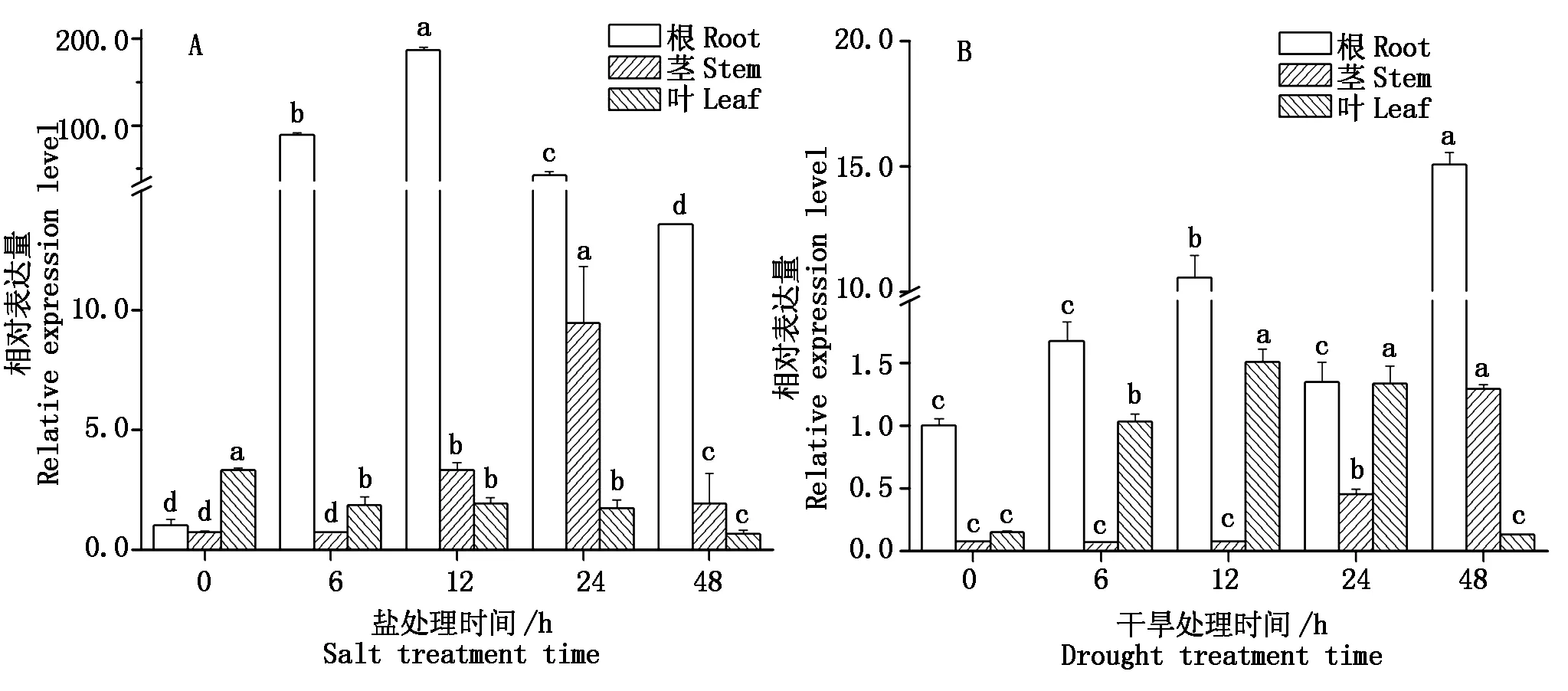
图4 盐胁迫(A)和干旱(B)处理条件下GmWRKY58基因的表达Fig.4 Expression of GmWRKY58 under salt stress (A) and drought (B) treatments
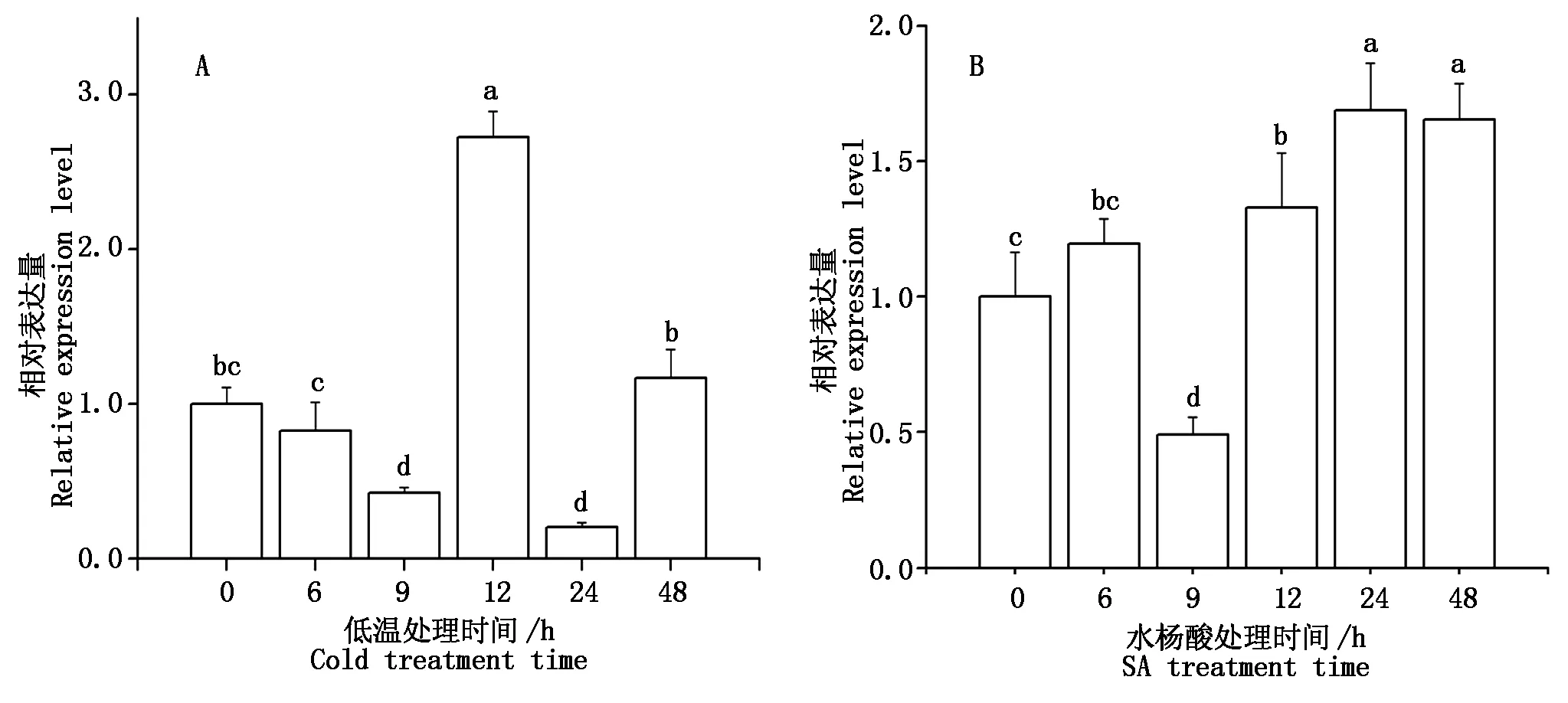
图5 低温4 ℃(A)和SA(B)诱导大豆叶片GmWRKY58表达 Fig.5 Expression of GmWRKY58 in leaf of soybean induced by cold (4 ℃,A) and SA (B) treatments
2.5 GmWRKY58 蛋白的亚细胞定位
通过PSORTII(http://www.genscript.com/wolf-psort.html) 网站对GmWRKY58蛋白进行亚细胞定位预测,结果定位于细胞核中。为了从试验上进一步证明其亚细胞定位,将瞬时表达载体35S∷GmWRKY58∷GFP转化到本氏烟叶片,温室培养24~72 h后,通过共聚焦激光扫描显微镜观察绿色荧光蛋白的信号。结果显示(图7),转pCambia1302-GFP 载体的绿色荧光分布在整个细胞膜、细胞核和细胞质中,转35S∷GmWRKY58∷GFP载体的绿色荧光分布在细胞核中,说明GmWRKY58定位在细胞核中,这与GmWRKY58基因序列上含有一个核定位信号(NLS)区域的特征相符,也与生物信息学预测结果相同,说明GmWRKY58是转录因子,定位在细胞核内调控下游相关基因的表达。
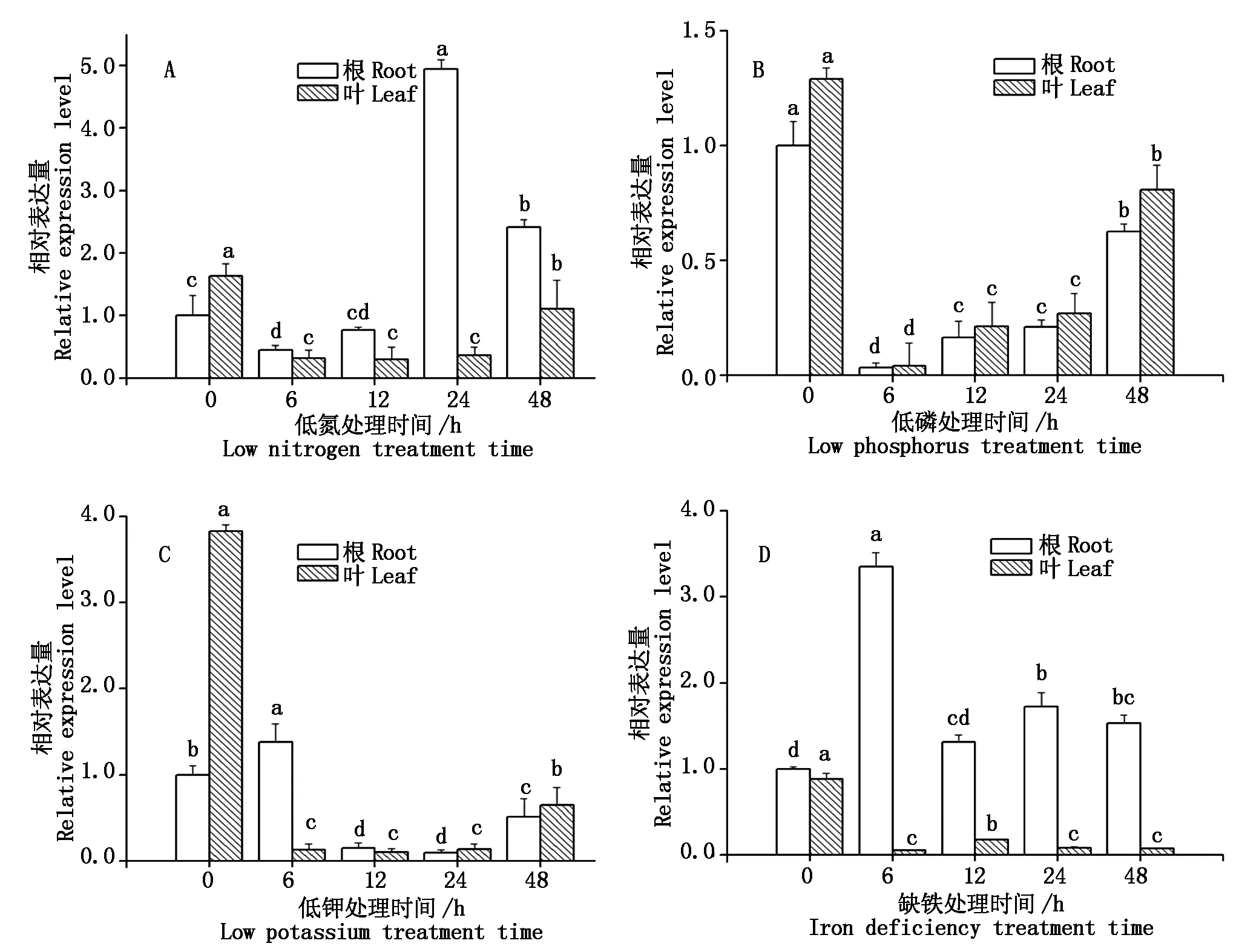
图6 低N(A)、低P(B)、低K(C)和缺Fe(D)诱导GmWRKY58表达 Fig.6 Expression of GmWRKY58 in soybean induce by low nitrogen (A),low phosphorus (B),low potassium (C) and iron deficiency (D) treatments
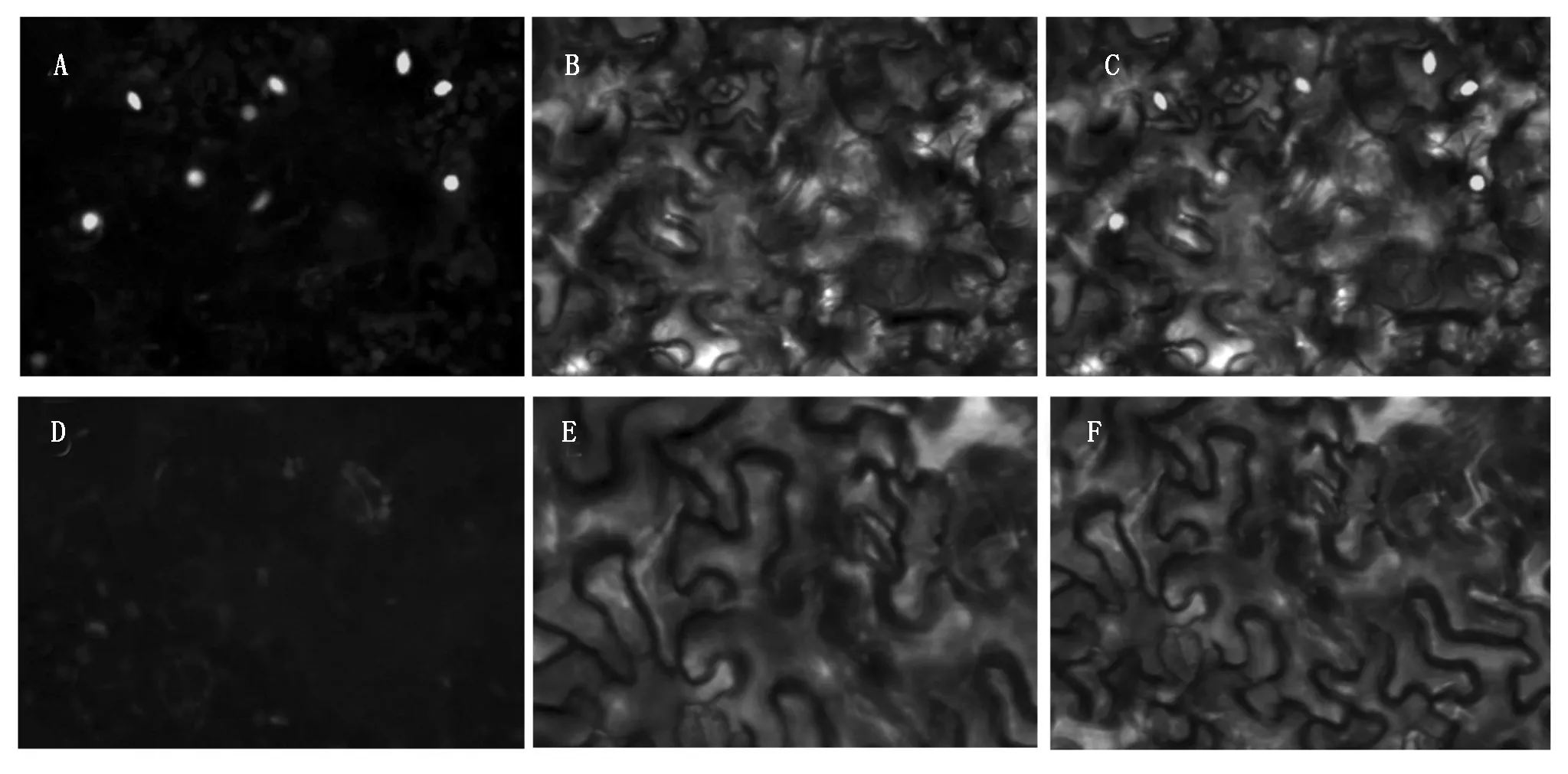
A、D.紫外激发的荧光信号;B、E.可见光的信号;C、 F. A和B叠加,D和E叠加。 A,D.UV-excited fluorescence signals;B,E.Signals under bright field;C,F.Merged image of A and B,D and E.
2.6 GmWRKY58的启动子分析
上述表达分析结果表明,转录因子GmWRKY58在非生物胁迫下存在一定功能,猜测其启动子序列上可能含有响应非生物胁迫的应答元件。在PlantCARE网站上预测GmWRKY58启动子的顺式作用元件,结果表明(表1),GmWRKY58基因的启动子序列中含有多个响应逆境和植物激素的顺式作用元件,例如:ACE类似序列、MBS核心序列、HSE核心序列、富含TC的重复序列、TGA盒和TATA盒等。这些调控元件暗示GmWRKY58在植物响应抗逆方面可能具有重要作用。
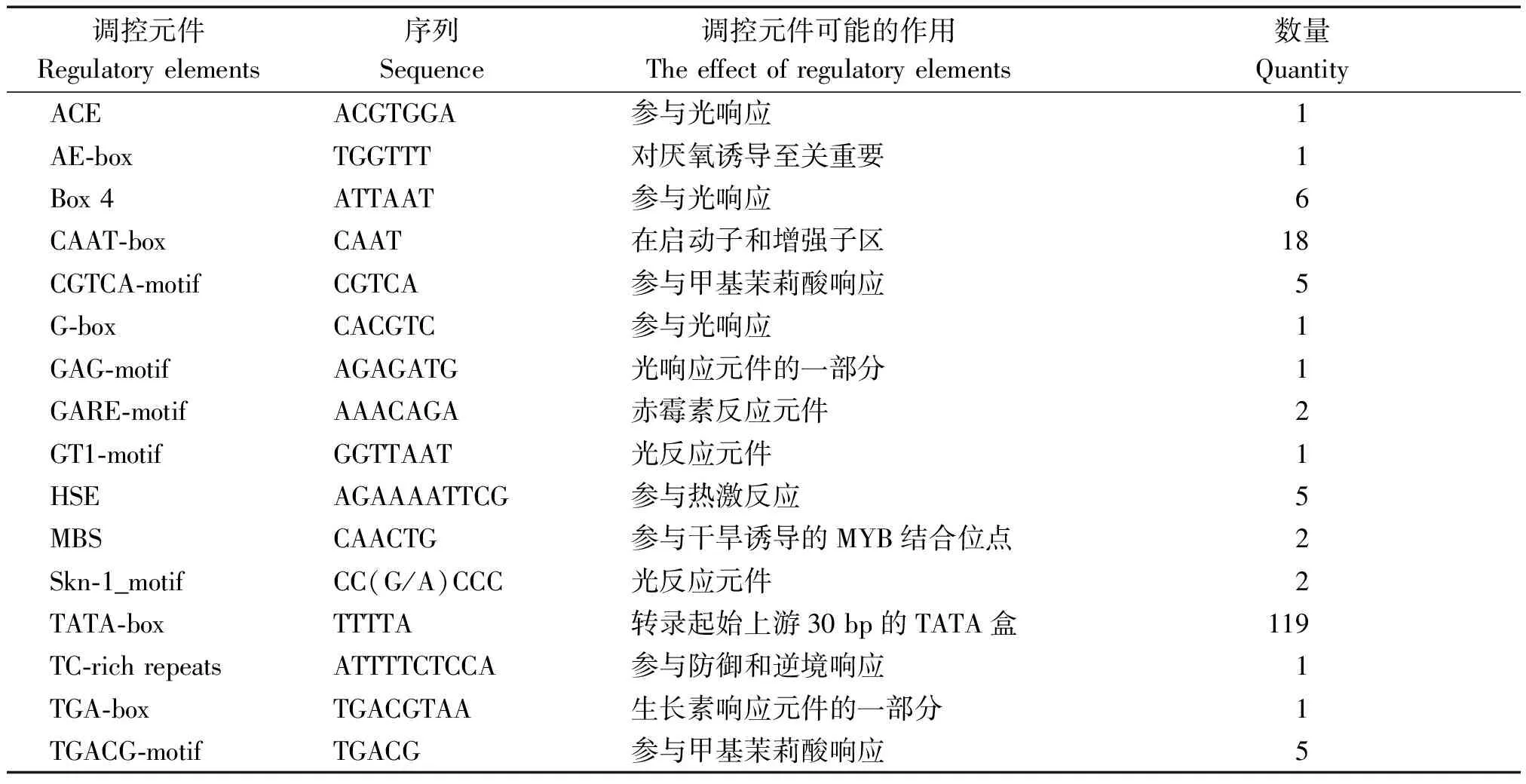
表1 GmWRKY58启动子上的顺式作用元件预测 Tab.1 The cis-acting elements in the promoter sequence of GmWRKY58
3 讨论与结论
转录因子调控类基因可以较快响应逆境胁迫[35]。本研究结果显示,大豆GmWRKY58基因受高盐、干旱、低N和缺Fe等多种逆境胁迫后在其根部强烈诱导表达,但表达模式不同,说明GmWRKY58基因在不同胁迫环境下可能具有不同的响应机制。根部组织对这几种胁迫更敏感,这可能与其最早受水分胁迫或渗透胁迫导致较快脱水有关。大豆GmWRKY75和GmWRKY6基因在低P、低N、低K和缺Fe胁迫下表达趋势基本一致,都呈先微弱下调再明显上调,但不同处理达到最高表达量的时间点不同[36],与本研究中GmWRKY58基因的表达模式存在着很大差异,这可能是由于它们基因结构和氨基酸序列的差异导致WRKY转录因子具有多种基因功能。
本研究中,GmWRKY58基因在大豆的根、茎、叶、花和荚中均有表达,根、茎、叶中的表达量显著高于花和荚,这与前人的研究结果一致[30],说明GmWRKY58对于大豆的营养与生殖器官发育都有一定的作用,但其发挥作用的大小、调控方式可能存在不同。
通过不同物种WRKY氨基酸序列同源性分析和 Blast 比对,结果表明GmWRKY58与大豆WRKY70之间序列一致性最高,其次为绿豆、鹰嘴豆、巨桉、毛果杨、胡杨、苹果、麻风树、可可,说明GmWRKY58在相同物种中保守性比在不同物种间保守性高;GmWRKY58基因在物种间进化过程中经受不同选择压力的选择。
蛋白质在细胞中的定位决定了其行使功能的部位。生物信息学预测GmWRKY58定位在细胞核中,本试验结果证实了这一预测,该结果与已报道的WRKY转录因子的亚细胞定位结果一致[30],同时也与GmWRKY58作为转录因子参与调控基因的表达相吻合。
WRKY通过结合启动子上保守的W box 顺式元件来调节目标基因的表达[37]。通过分析GmWRKY58基因上游1 500 bp的启动子片段,发现其含有多个与干旱、光响应、热激等逆境胁迫和生长素、赤霉素、茉莉酸等激素相关的诱导元件。GmWRKY58的启动子区域含有2个干旱应答的顺式元件MBS,推测该基因在大豆响应抗旱胁迫,调节干旱的抗性方面具有一定的功能,本试验也证实了这一点,如PEG处理下,大豆根中的GmWRKY58在短时间内即受到强烈的诱导,表达量提高近20倍。同时,在启动子区域还含有1个参与防御和逆境响应的顺式元件TC-rich repeats,当外界环境存在如高盐、低N和缺Fe时,通过这个诱导元件,与逆境相关转录因子可以激活GmWRKY58基因和蛋白质的表达,GmWRKY58转录因子又可以调控下游与逆境胁迫相关的功能基因表达,从而降低不良环境对植物造成的伤害。
大豆GmWRKY58基因开放阅读框长度为954 bp,编码317个氨基酸。GmWRKY58转录因子定位于细胞核中。GmWRKY58基因在根、茎、叶、花和荚等组织均有表达,种子中不表达,根茎叶中的表达量显著高于花和荚。该基因明显受高盐、干旱、低N和缺Fe胁迫诱导表达,说明GmWRKY58在大豆的抗盐、抗旱、低N和缺Fe等非生物胁迫响应中起到重要的作用。
参考文献:
[1] Choudhury S, Panda P, Sahoo L, et al. Reactive oxygen species signaling in plants under abiotic stress [J]. Plant Signaling & Behavior, 2013, 8(4):e23681.
[2] Nakashima K, Yamaguchi-Shinozaki K, Shinozaki K. The transcriptional regulatory network in the drought response and its crosstalk in abiotic stress responses including drought, cold, and heat [J]. Frontiers in Plant Science, 2014, 5:170.
[3] Jiang J, Ma S, Ye N, et al. WRKY transcription factors in plant responses to stresses [J]. Journal of Integrative Plant Biology, 2017, 59(2):86-101.
[4] Ishiguro S,Nakamura K. Characterization of a cDNA encoding a novel DNA-binding protein,SPF1,that recognizes SP8 sequences in the 5′upstream regions of genes coding for sporamin and β-amylase from sweet potato[J]. Molecular and General Genetics,1994,244(6):563-571.
[5] Rushton P J,Macdonald H,Huttly A K,et al. Members of a new family of DNA-binding proteins bind to a conserved cis-element in the promoters of α-Amy2 genes[J]. Plant Molecular Biology,1995,29(4):691-702.
[6] Rushton P J,Torres J T,Parniske M,et al. Interaction of elicitor-induced DNA-binding proteins with elicitor response elements in the promoters of parsley PR1 genes[J]. The EMBO Journal,1996,15(20):5690-5700.
[7] Abuqamar S,Chen X,Dhawan R,et al. Expression profiling and mutant analysis reveals complex regulatory networks involved inArabidopsisresponse to Botrytis infection[J]. The Plant Journal,2006,48(1):28-44.
[8] Dai X, Wang Y, Zhang W H.OsWRKY74, a WRKY transcription factor, modulates tolerance to phosphate starvation in rice [J]. Journal of Experimental Botany, 2015, 67(3): 947-960.
[9] Guan Y, Meng X, Khanna R, et al. Phosphorylation of a WRKY transcription factor by MAPKs is required for pollen development and function inArabidopsis[J]. PLoS Genetics, 2014, 10(5):e1004384.
[10] Zhou L, Wang N N, Gong S Y, et al.Overexpression of a cotton(Gossypiumhirsutum)WRKY gene,GhWRKY34, inArabidopsisenhances salt-tolerance of the transgenic plants [J]. Plant Physiology and Biochemistry, 2015, 96:311-320.
[11] Hui D,Iqbal J,Lehmann K,et al. Molecular interactions between the specialist herbivoreManducasexta(lepidoptera,sphingidae) and its natural hostNicotianaattenuata:V. microarray analysis and further characterization of large-scale changes in herbivore-induced mRNAs[J]. Plant Physiology,2003,131(4):1877-1893.
[12] Liu D, Leib K, Zhao P, et al. Phylogenetic analysis of barley WRKY proteins and characterization of HvWRKY1 and-2 as repressors of the pathogen-inducible geneHvGER4c[J]. Molecular Genetics and Genomics, 2014, 289(6): 1331-1345.
[13] Niu C F,Wei W,Zhou Q Y,et al. Wheat WRKY genes TaWRKY2 and TaWRKY19 regulate abiotic stress tolerance in transgenicArabidopsisplants[J]. Plant,Cell & Environment,2012,35(6):1156-1170.
[14] Chi Y, Yang Y, Li G, et al. Identification and characterization of a novel group of legume-specific, Golgi apparatus-localized WRKY and Exo70 proteins from soybean [J]. Journal of Experimental Botany, 2015,66(11): 3055-3070.
[15] Zhou Q Y,Tian A G,Zou H F,et al. Soybean WRKY-type transcription factor genes,GmWRKY13,GmWRKY21,andGmWRKY54,confer differential tolerance to abiotic stresses in transgenicArabidopsisplants[J]. Plant Biotechnology Journal,2008,6(5):486-503.
[16] Suttipanta N,Pattanaik S,Kulshrestha M A,et al. The transcription factor CrWRKY1 positively regulates the terpenoid indole alkaloid biosynthesis inCatharanthusroseus[J]. Plant Physiology,2011,157(4):2081-2093.
[17] Yu F,Huaxia Y,Lu W,et al. GhWRKY15,a member of the WRKY transcription factor family identified from cotton (GossypiumhirsutumL.),is involved in disease resistance and plant development[J]. BMC Plant Biology,2012,12:144.
[18] Jiang J, Ma S, Ye N, et al. WRKY transcription factors in plant responses to stresses [J]. Journal of Integrative Plant Biology, 2017, 59(2): 86-101.
[19] Ding Z J,Yan J Y,Li C X,et al. Transcription factor WRKY46 modulates the development ofArabidopsislateral roots in osmotic/salt stress conditions via regulation of ABA signaling and auxin homeostasis[J]. Plant Journal,2015,84(1):56-69.
[20] Jiang Y,Liang G,Yu D. Activated expression of WRKY57 confers drought tolerance inArabidopsis[J]. Molecular Plant,2012,5(6):1375-1388.
[21] Asai T,Tena G,Plotnikova J,et al. MAP kinase signalling cascade inArabidopsisinnate immunity[J]. Nature,2002,415(6875):977-983.
[22] 李立芹. 烟草WRKY12基因的分离、表达与多克隆抗体的制备[J]. 核农学报,2011,25(3):461-468.
[23] Jiang Y,Deyholos M K. Functional characterization ofArabidopsisNaCl-inducibleWRKY25 andWRKY33 transcription factors in abiotic stresses [J]. Plant Molecular Biology,2009,69(1-2):91-105.
[24] Li S J,Fu Q T,Chen L G,et al.ArabidopsisthalianaWRKY25,WRKY26,and WRKY33 coordinate induction of plant thermotolerance[J]. Planta,2011,233(6):1237-1252.
[25] Li S J,Fu Q T,Huang W D,et al. Functional analysis of anArabidopsistranscription factor WRKY25 in heat stress[J]. Plant Cell Reports,2009,28(4):683-693.
[26] Raineri J,Wang S H,Peleg Z,et al. The rice transcription factor OsWRKY47 is a positive regulator of the response to water deficit stress[J]. Plant Molecular Biology,2015,88(4/5):401-413.
[27] 钟贵买,伍林涛,王健美,等. 转录因子AtWRKY28亚细胞定位及在非生物胁迫下的表达分析[J]. 中国农业科技导报,2012,14(5):57-63.
[28] 张飞萃. 拟南芥WRKY28和WRKY42调控磷吸收和转运的机制研究[D]. 北京:中国农业大学,2015
[29] Xu Z L,Ali Z,Yi J X,et al. Over-expression of GmWRKY111 enhances NaCl tolerance of Salt-Sensitive genotype ofGlycinemax[J]. International Journal of Agriculture and Biology,2014,16(1):153-159.
[30] Yang Y,Chi Y J,Wang Z,et al. Functional analysis of structurally related soybean GmWRKY58 and GmWRKY76 in plant growth and development[J]. Journal of Experimental Botany,2016,67(15):4727-4742.
[31] Zhang G,Chen M,Li L,et al. Overexpression of the soybeanGmERF3 gene,an AP2/ERF type transcription factor for increased tolerances to salt,drought,and diseases in transgenic tobacco[J]. Journal of Experimental Botany,2009,60(13):3781-3796.
[32] 翟 莹,雷婷婷,闫 帆,等. 大豆逆境诱导基因GmPRP的克隆与表达[J]. 作物学报,2011,37(12):2152-2157.
[33] Livak K J,Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(T)(-Delta Delta C) method[J]. Methods,2001,25(4):402-408.
[34] Sparkes I A,Runions J,Kearns A,et al. Rapid,transient expression of fluorescent fusion proteins in tobacco plants and generation of stably transformed plants[J]. Nature Protocols,2006,1(4):2019-2025.
[35] Hirayama T,Shinozaki K. Research on plant abiotic stress responses in the post-genome era:past,present and future[J]. The Plant Journal :for Cell and Molecular Biology,2010,61(6):1041-1052.
[36] 徐 影. 大豆耐低磷相关转录因子GmWRKY75和GmWRKY6的克隆及功能分析[D]. 南京:南京农业大学,2014.
[37] Brand L H,Kirchler T,Hummel S,et al. DPI-ELISA:a fast and versatile method to specify the binding of plant transcription factors to DNAinvitro[J]. Plant Methods,2010,6(1):25.
