GSE74602芯片数据中直肠癌关键基因与治疗药物的生物信息学筛选
2018-05-07罗磊龚宝成刘福囝
罗磊,龚宝成,刘福囝
(中国医科大学附属第一医院 胃肠肿瘤外科,辽宁 沈阳 110001)
结直肠癌是世界癌症相关死亡的主要原因之一,其发病率在恶性肿瘤中排名第三[1]。结直肠癌的发生和发展是由多基因和细胞通路发生改变共同作用的结果[2]。当今,治疗结直肠癌的主要方式是手术和系统抗肿瘤治疗,目前尚不能精确地从分子机制方面分析结直肠癌的发病机制,因此了解结直肠癌细胞增殖、凋亡和侵袭的相关分子机制及筛选出潜在的治疗结直肠癌的小分子药物,对更有效的诊断结直肠癌及其治疗方案的制定尤为重要。
基因芯片分析提供了关于肿瘤进展中涉及的分子全面而详细的变化,从而更好地了解细胞癌变过程以及发现新的肿瘤标志物和治疗靶点[3]。目前仍然缺乏对临床有用的癌症诊断标记物,基因芯片分析之所以没有在临床上得到大量的应用,主要归结于下列几种原因:缺乏对比分析对每个已经获得的研究结果重叠度的比较;应用平台的类型、样品收集和处理以及数据分析的相关变异性;缺乏大规模对有价值的样本的分析;缺乏可靠的关于临床应用数据的理解[4-5]。
为了更加深入的研究结直肠癌的发病机制,本研究从基因表达大棚车(Gene Expression Omnibus,GEO)数据库中下载了GSE74602的基因表达芯片,然后利用R语言的Limma包筛选结直肠癌患者和正常的结直肠上皮组织之间的差异表达基因,并且对这些差异表达基因进行GO富集分析和KEGG通路分析,从而更好的研究与结直肠癌相关的分子机制。另外,利用STRING在线数据库构建差异表达基因之间的蛋白互作网络,并通过Cytoscape软件中的MCODE插件进行子网络模块分析,从而进一步发掘在结直肠癌发生过程中起着核心作用的基因,最后通过连通图筛选了前10个具有潜在治疗结直肠癌的小分子药物,为以后研究结直肠癌的分子机制和靶向药物提供重要方向以及依据。
1 材料与方法
1.1 基因芯片数据
从高通量表达数据库GEO(www.ncbi.nlm.nih.gov/geo)中下载结直肠癌的GSE74602基因表达谱数据芯片数据,该芯片是通过美国昂飞老鼠基因组芯片平台GPL1261进行注释的。GSE74602芯片包括30例结直肠患者样本和30例正常结直肠组织标本。
1.2 芯片数据的预处理
在芯片的预处理阶段,利用R语言中的Affy包对原始芯片数据之间进行标准化,去除一些不必要的影响因素。然后根据芯片平台注释信息将探针名字转换为基因名,对于多个探针对应1个基因的问题,采用这几个探针的平均值来表示,从而对应到该基因的表达量。
1.3 筛选差异表达基因
预处理芯片之后,利用R语言中的Limma包筛选癌症和正常组织之间的差异表达基因,采用t检验分析癌症和正常组织之间的表达,筛选出调整后P<0.05,而且|log2FC|>2的基因作为差异表达基因。然后对差异表达基因进行聚类分析,并且通过R语言中的gplots包绘制热图。
1.4 基因本体论(GO)富集分析与京都基因与基因组百科(KEGG)信号通路分析
DAVID在线数据库主要用来进行基因的注释、功能分类、信号通路分析以及基因之间编号的转换[6]。本研究使用的是功能注释,获得结直肠癌相关的差异表达基因之后,为了进一步分析结直肠癌的分子机制,将差异表达基因上传到DAVID在线数据库(https://david.ncifcrf.gov)中进行GO功能分析和KEGG信号通路分析,根据美国生物技术信息国立中心的重要功能分类,可以把GO分为分子功能(molecular function,MF)、生物学过程(biological process,BP)及细胞组分(cellular component,CC)3部分。最终得到差异表达基因及所参与的相关信号通路[7-8]。
1.5 蛋白互作网络构建和子网络模块分析
STRING数据库主要整合了来源不同的基因表达数据,如高通量测序数据,共表达数据以及实验相关数据等,构建一种特殊的根据基因之间的结合分数来评价蛋白间相关度的体系。本研究中,为了进一步探索与结直肠癌相关的核心基因以及作用机制,筛选到的差异表达基因上传到STRING在线数据库(http://www.string-db.org)进行蛋白间的相互作用网络分析,最终通过Cytoscape软件构建蛋白间作用网络,从而找出在结直肠癌发展过程中起到核心作用的基因(结合分数>0.4)。另外,为了更深入的研究在结直肠癌发展过程中起着更加精细的调控作用的核心基因,利用Cytoscape软件中的MCODE插件进行子网络模块分析,选出MCODE分数>3且基因数>4的子网络[9-10]。
1.6 连通图分析
连通图(The Connectivity Map,cMap,http://www.broad.mit.edu/cmap)是一个公共数据库,它是收集来自用小分子处理的培养的人类细胞,大约超过7 000多个的表达谱,以前已被应用于探索药物作用的机制以及确定新的潜在药物[11]。然后把蛋白互作网络图中的差异表达基因被映射到cMap数据库上,富集分数范围从-1到1,并使用富集评分的绝对值>0.7且P<0.05为标准。
2 结 果
2.1 差异表达基因的筛选
通过R语言中的Affy包将原始芯片数据的系统误差去除,从而得到标准化的基因表达数据。通过R语言中的Limma包对结直肠癌组织和正常组织标准化之后的数据进行差异表达分析,最终231个基因达到筛选标准(P<0.05且倍数变化的绝对值>2),其中有122个上调基因,109个下调基因(图1)。利用R语言中的pheatmap包绘制差异显著的前20个上调基因和前20下调基因的热图,直观的显示出差异表达基因(图2)。

图1 差异表达基因的火山图(红色表示上调基因,绿色表示下调基因,而黑色代表无差异表达基因)Figure 1 The volcano plot of differentially expressed genes (the red dots and green dots standing for the up-regulated genes and down-regulated genes respectively, and the black dots representing genes not differentially expressed)
2.2 差异基因功能和通路途径分析
为了进一步探究差异基因在结直肠癌发生发展过程中的作用,利用DAVID在线数据库进行功能富集分析以及信号通路分析,发现上调基因主要参与细胞中碳酸氢盐的运输,一碳代谢途径,调节细胞内的酸碱平衡等生物学过程;并且参与细胞外液、细胞间质以及细胞顶端质膜的组成;还可以促进钙离子通道以及转运蛋白酶激活(表1)。下调基因主要参与细胞核的有丝分裂、细胞增殖以及细胞之间的黏附等生物学过程;参与纺锤体、细胞核以及细胞液的构成;能够促进与蛋白激酶的结合,活化三磷酸腺苷及激活肽酶(表2)。
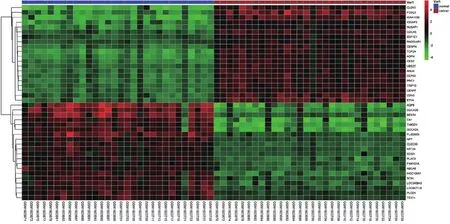
图2 热图中的40个差异表达基因(包括20个上调基因和20个下调基因;红色代表上调,绿色代表下调)Figure 2 Heat map of the top 40 differentially expressed genes(including 20 up-regulated genes and 20 downregulated genes; red color showing the up-regulated gene, and green color showing the down-regulated genes)
通过KEGG信号通路分析发现:上调基因主要参与细胞中矿物质的吸收,细胞中氮的代谢,近端小管对碳酸氢盐的重吸收作用,胆汁及胰液的分泌等信号通路。而下调差异表达基因主要参与细胞周期的形成,p53信号通路的传递等信号通路(表3)。
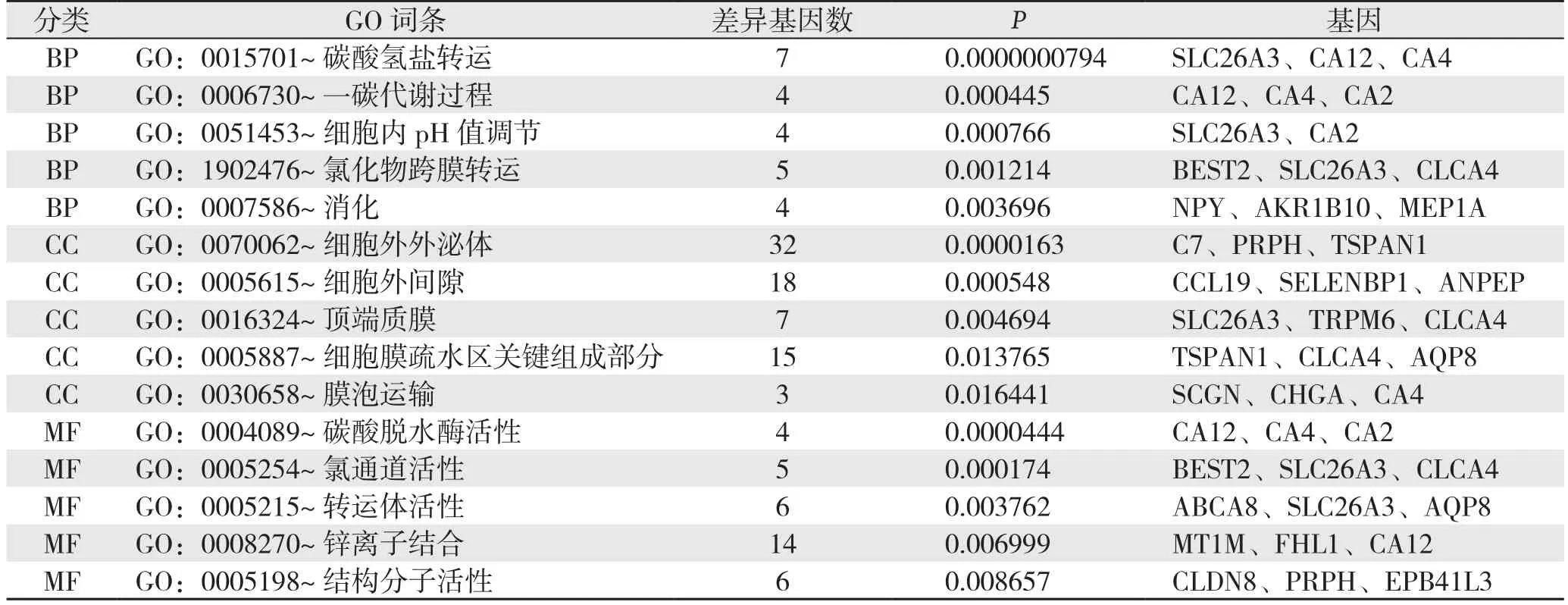
表1 上调基因的GO功能分析Table 1 GO enrichment analysis of the up-regulated genes
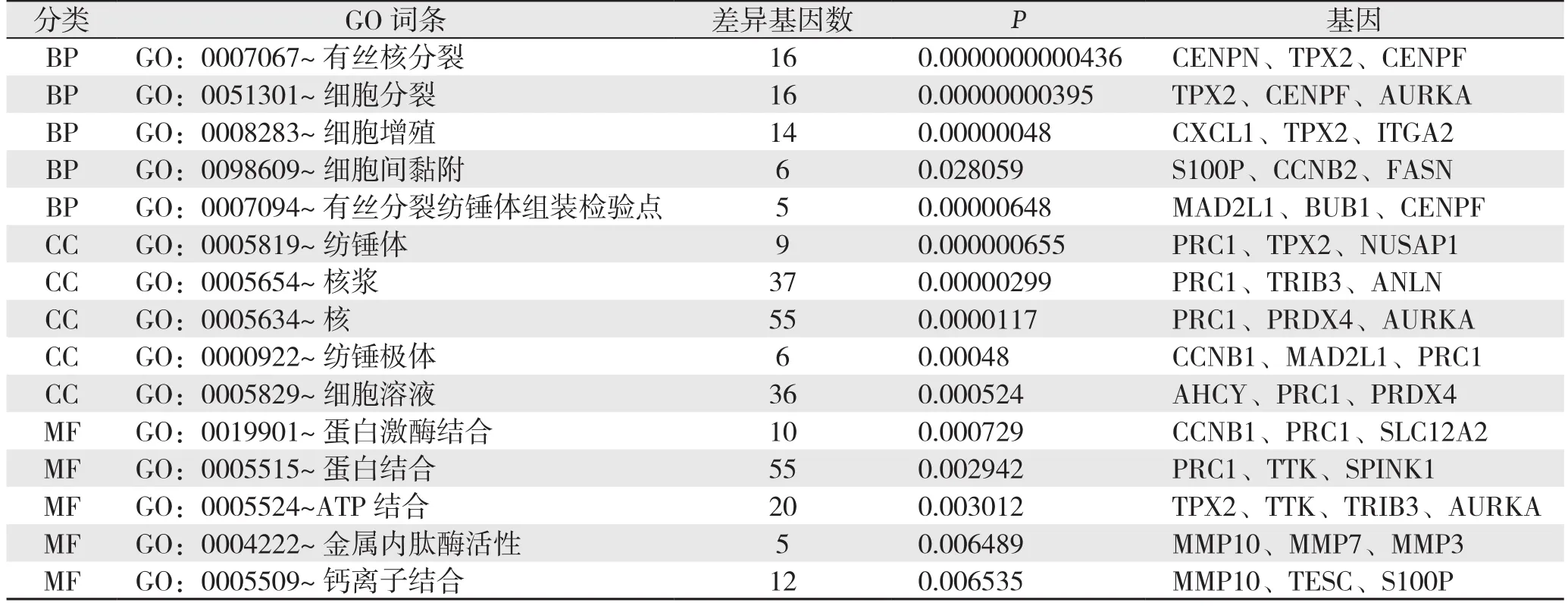
表2 下调基因的GO功能分析Table 2 GO enrichment analysis of the down-regulated genes

表3 差异表基因显著参与的KEGG信号通路Table 3 The KEGG pathway signi fi cantly enriched in differentially expressed genes
2.3 蛋白互作网络及子网络模块分析
为了进一步研究差异基因在结直肠癌中的作用机制,通过STRING数据库构建差异表达基因的蛋白间互作网络(图3),总共包含有159个节点和606条边。这里节点代表的是富集到STRING数据库中的差异表达基因,而边反映的是差异表达基因之间的相互作用。通过分析筛选了了前40个度值>10的基因(表4)。
另外结合子网络模块分析,发现了一些新的在结直肠癌发生的局部调控中起着重要作用的关键基因,如KIF20A、CENPF、NCAPG、PYY、IQGAP3,他们分别位于不同的子网络中(图4),这为以后研究结直肠癌的分子机制提供了更多的依据。
2.4 小分子药物筛选
基于cMap数据库映射的结果,总共筛选了前10个小分子药物,例如紫霉素、克洛索隆、纳多洛尔等(表5)。

图3 差异表达基因中的蛋白互作网络图(总共包含有159个节点和606条边;红色代表上调基因,绿色代表下调基因)Figure 3 Protein-protein interaction networks of the differential expressed genes (containing 159 nodes and 606 edges;red color showing the up-regulated gene, and green color showing the down-regulated genes)
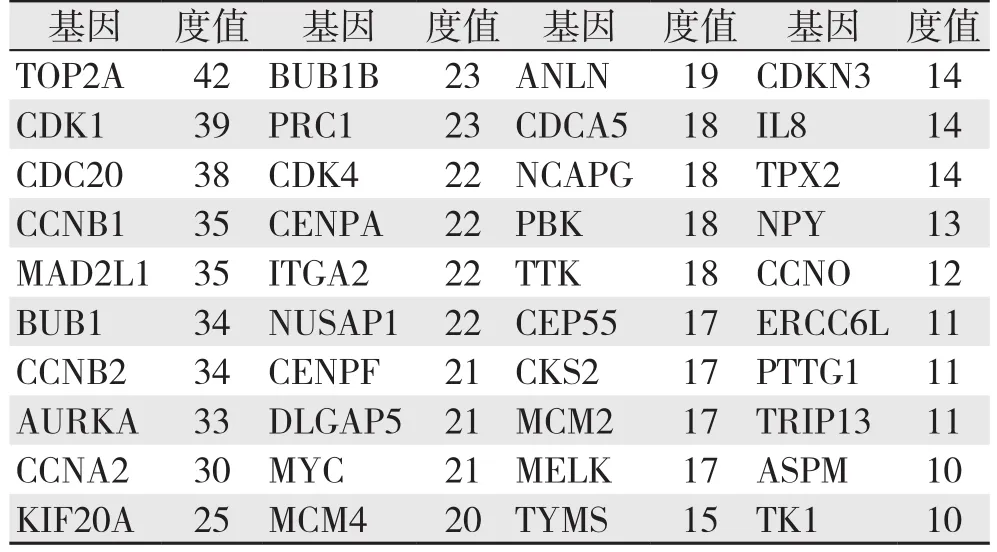
表4 筛选出度值大于10的40个基因Table 4 Forty screened genes with a degree greater than 10
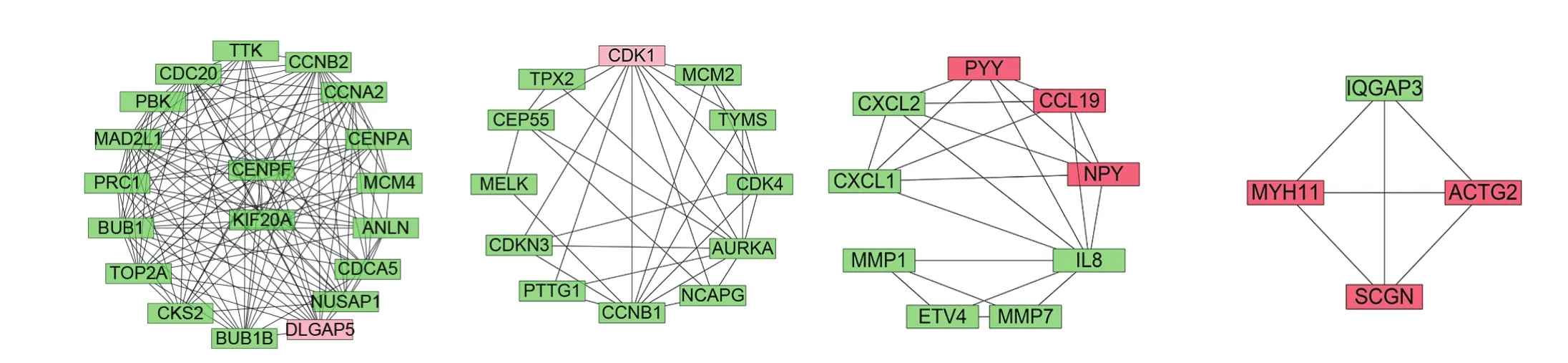
图4 蛋白互作网络图的子网络分析(包括4个模块)Figure 4 Sub-module analysis of the protein-protein interaction networks (including 4 modules)

表5 前10个具有潜在治疗结直肠癌的小分子药物Table 5 Top 10 small molecule drugs potentially against colorectal cancer
3 讨 论
结直肠癌作为世界上最常见的恶性肿瘤之一,主要是由于基因表达异常而引起各种遗传和表观遗传改变导致的结果[12]。本研究中,利用生物信息学方法对GSE74602的数据进行差异表达分析,得到231个显著差异表达基因,其中122个上调基因和1 0 9个下调基因。利用R语言中的pheatmap包绘制差异显著的前20个上调基因和前20下调基因的的热图,这样更能直观的显示出这些基因的表达差异结果。而且从该芯片通过筛选出度值(degree)大于10的40个基因,如表4所示。从表中可以看出度值位于前5的基因分别是,TOP2A、CDK1、CDC20、CCNB1、MAD2L1,值越大表明与恶性肿瘤的关系越密切。TOP2A是拓扑异构酶2α的简称,DNA复制和转录中的关键酶,并且能控制和改变DNA的拓扑功能状态,它与染色体浓缩、染色单体分离、DNA转录和复制期间发生的扭转应力的缓冲等过程有一定的关系,研究[13]表明它的高表达与结直肠癌的发生发展有关,可以作为肿瘤复发的标记物。研究[14]表明TOP2A的高表达在乳腺癌和前列腺癌中提示预后不良,Cao等[15]指出TOP2A的高表达可作为胃癌患者的潜在生物标志物,Zhou等[16]认为TOP2A的表达可能通过调节细胞周期和p53信号通路用来鉴定胰腺癌的进展和预后。CDK1,是细胞周期蛋白依赖性激酶1的缩写,Gao等[17]证明KPNA2可以通过增加CDK1的表达来促进肿瘤细胞增殖,并由此作为治疗肝癌干预的指标之一。在人类中,共有21个CDK基因,它们是细胞增殖,基因转录和mRNA加工的正调控因子,CDK4和CDK6在乳腺癌中被研究的最多,事实上,靶向CDK1或细胞周期蛋白B的药物已被证明能有效阻断肿瘤的生长和发展[18]。CDK1能够抑制细胞生长、增殖、迁移,延缓细胞周期和加速细胞凋亡,并通过联合下调基因细胞周期蛋白B、细胞周期蛋白D1和Bcl-2来抑制非小细胞肺癌的进展[19]。癌症基因组图谱转录组数据集揭示了人类结直肠癌中CDK1比正常结直肠要过度表达,CDK1是BRAFV600E结直肠癌中凋亡的新颖介质,其与MEK抑制剂的双重靶向结合可能是治疗结直肠癌有效的途径[20]。CDC20是细胞分裂周期20同系物的一种,它的过度表达以Bim依赖性方式促进前列腺癌细胞系对多西他赛的耐药性,将来很可能解决治疗前列腺癌对多西他赛的耐药性的问题[21]。多变量分析显示高CDC20表达是总生存的独立预测因子,CDC20上调基因与胃癌的侵袭性进展和不良预后相关,可将CDC20鉴定为预测胃癌患者临床预后分析的独立标记物[22]。CCNB1是细胞周期蛋白B1的简称,被认为是癌症疫苗的潜在靶标,因为它在许多恶性细胞中过表达,而在正常细胞中以几乎检测,像在包括乳腺癌,肺癌,结肠癌,前列腺癌和头颈部癌中的这几种人类实体瘤中都观察到过度表达[23]。MAD2L1基因反复参与细胞周期和有丝分裂的过程, 并发现MAD2L1高表达与肺癌预后不良有关,总之过度表达的MAD2L1有可能作为预测肺癌复发和生存的预后生物标志物[24]。
通过基因GO功能富集分析,发现这些差异表达基因主要参与细胞内的一碳代谢过程,细胞分裂、细胞增殖以及细胞间的黏附等作用。众多细胞生物学过程均与肿瘤的发生发展密切相关。细胞内一碳代谢途径一旦发生紊乱,会影响正常组织基因的表达,导致细胞坏死、变性,进而导致正常组织发生癌变[25]。癌症干细胞理论表明肿瘤细胞的发生是细胞的不对称分裂来维持的,细胞的不对称分裂又和分裂细胞的纺锤体形成密不可分[26]。细胞增殖和凋亡受细胞周期严密调控,一旦细胞周期发生紊乱将会诱导肿瘤细胞的发生细胞间的黏附。
从表2中可以看出,在生物学过程中,比较有意义的S100P、CCNB2、FASN这3个基因,它与结直肠癌有关的研究还不是很广泛。S100P蛋白具有C-末端赖氨酸残基,人类乳腺癌细胞中的S100P蛋白与患者生存率降低相关,它表达的值越高,表明在恶性肿瘤中的侵袭能力越强[27]。S100P高表达在各种恶性肿瘤中,并且在肿瘤生长、侵袭和转移中具有重要作用,并与结直肠癌的侵入和转移相关,然而在调控结直肠癌侵袭转移中的分子机制尚需进一步探讨[28]。CCNB2是细胞周期蛋白家族的一种,在G2/M周期转换的进展中起关键作用,高水平的CCNB2蛋白与肿瘤分化程度,肿瘤大小,淋巴结转移,远处转移和临床分期呈正相关,同时,CCNB2蛋白过表达是非小细胞肺癌患者总生存率的独立预后不良因素之一[29]。脂肪酸合成酶(FASN)是从头合成长链脂肪酸所需的关键酶,FASN通常在人类癌症中高度表达,而在大多数正常人类组织中通常不可检测或表现低表达,其表达与不良预后相关,可能由对药物或辐射的抗性介导有关,尽管FASN的表达在各种类型的癌症中,但其与非小细胞肺癌中的放射敏感性的关联尚不清楚,还需进一步研究[30]。上述3个基因虽然在人类恶性肿瘤中高度表达,代表其预后较差,但在结直肠癌中的高表达的分子机制尚不清楚,还需大量实验进一步证实。
KEGG信号通路分析发现这些差异基因主要参与细胞周期,以及p53信号传导通路。细胞周期是在DNA损伤之后影响细胞增值、生长以及细胞分裂的一个非常重要的调节因子,而且细胞周期检查点的缺失能够促进基因不稳定性的发生,从而导致细胞增值异常诱发癌症[31]。Han等[32]发现可以通过抑制转录因子sp1的表达促进结直肠癌细胞的凋亡以及细胞周期的停滞,从而抑制结直肠癌细胞的增殖。Zheng等[33]发现过表达细胞分裂周期蛋白Parafibromin能够抑制结直肠癌的分裂周期的进行,最终使结直肠癌细胞的生长受到抑制,并且为结直肠癌的基因治疗提供新的潜在的靶点。Chen等[34]发现过度表达的核糖体蛋白S15A能够通过p53信号通路诱导结直肠癌细胞的恶性转变,并且引起p53信号通路的错误调控。
为了进一步研究结直肠癌的分子机制,本研究对差异表达基因进行蛋白间互作网络分析以及子网络模块分析,发现了一些新的与结直肠癌发展有关的基因,如KIF20A、CENPF、NCAPG、PYY、IQGAP3,这些可能成为将来结直肠癌潜在的诊断、治疗的靶点和分子标志物。KIF20A是驱动蛋白超家族成员的一员,在细胞分裂中起着重要的作用[35],而且在黑色素瘤、膀胱癌中都能显著过表达。在胰腺癌细胞中敲除KIF20A能够明显抑制肿瘤细胞的增值、转移以及侵袭能力[36-37]。KIF20A在原发性肝癌中是显著过表达的,与肝癌患者的预后有着密切关系[38]。KIF20A的高表达与乳腺癌的恶化以及紫杉醇耐药有着密切关系[39]。但KIF20A在结直肠癌中的作用及机制我们还尚不清楚,需要进一步进行深入研究。
CENPF是着丝粒蛋白家族中的一员,在细胞有丝分裂过程中染色体的分离起着重要作用[40]。肝细胞肝癌中,CENPF对癌细胞的增殖没有影响,但是能够明显促进癌细胞的转移,而且CENPF的过表达与5氟类化疗药的耐药有着密切的关系[41]。CENPF的表达与染色体不稳定性,c-myc扩增以及端粒酶活性有密切联系,而且过度表达CENPF的乳腺癌患者预后较差[42]。因此,CENPF可能会成为一个新的潜在的基因治疗靶点。NCAPG是凝集素复合物I的一个亚基,在细胞减数分裂染色体分离过程中起重要作用[43],NCAPG不仅能够抑制高分化胶质瘤细胞的增殖,沉默NCAPG能够引起胶质瘤细胞停滞在G1期。iRNA-137能够通过抑制NCAPG的表达从而表现出抗肿瘤的活性[44]。另外,miRNA137在结直肠癌中表现出抑癌作用,因此,NCAPG在结直肠癌的发展过程中可能起着促进癌基因的作用。
PYY由36个氨基酸的肽组成,是胰腺多肽和神经肽Y构成的肽类家族的一员,也是远端肠道最丰富内分泌调节肽,低水平的PYY和肠道病变的恶性程度有着密切的关系[45]。而且PYY能够促进癌细胞的凋亡,从而抑制Barrets食管癌细胞的增殖[46-47]。另外,PPY及其合成类似物可能通过降低细胞内cAMP的水平,从而抑制乳腺癌的增长[48-49]。PYY不仅有缓解胰腺炎的作用,而且随着PYY浓度的升高以及暴露时间的延长,抑制胰腺癌生长的作用就越明显[50-51]。
IQGAP3是IQ模体GTP酶激活蛋白家族中最新被发现的一员,而且IOGAP3仅表达在大脑、肺、睾丸、小肠以及直肠中[52]。QGAP3有着与IQGAP1高达60%的相似结构,能够诱导细胞周期的发生,并且激活Ras依赖的ERK信号通路促进细胞的增殖,发挥癌基因的作用[53],另外,IQGAP3能够通过增强EGFR介导的ERK信号通路来促进肺癌细胞的生长、侵袭和转移[54],因此,IQGAP3在结直肠癌发展中也可能发挥着重要作用。
此外,本研究通过cMap筛选出了103个小分子药物,本文中列举了前10个小分子药物。紫霉素是富集分数绝对值最高的一种小分子药物,它是一种碱性多肽抗生素,是抗多药耐药结核病最有效的药物之一,它而且能抑制细菌蛋白质合成,并能抑制恶变细胞的增值,根据这一机制将来有可能成为治疗恶性肿瘤的小分子药物[55]。去甲骆驼蓬碱是一种β-咔啉生物碱,在当代生物医学研究和药物开发项目中表现出显着的重要性。它能改变含有细胞毒性的细胞结构和能量的特异性,进一步为抗肿瘤药物的研发提供了研究机制[56]。斑鸠霉素是一种具有强抗原生动物和细胞毒活性的抗生素。斑鸠霉素是一种先前的抗生素,已被证明可抑制巨噬细胞中氧化低密度脂蛋白的摄取,这样就间接的保护了人体免疫细胞,未来有可能作为抗肿瘤药物的制剂。上述经筛选的潜在小分子药物,像紫霉素、去甲骆驼蓬碱、斑鸠霉素等都有可能成为治疗结直肠癌的新型药物。
总而言之,本研究采用生物信息学方法筛选结直肠癌与正常结直肠组织之间的差异表达基因,并进行基因的GO功能富集分析以及KEGG信号通路分析,发现这些差异表达基因主要富集在癌症相关的细胞分裂、增殖、黏附等生物学过程,以及与癌症有关的细胞周期和p53信号通路,从而更好的了解结直肠癌发生的机制。而且本研究还发现一些新的在结直肠癌发展过程中起着核心作用的基因及潜在的治疗结直肠癌的小分子药物,如新型基因:KIF20A、CENPF、NCAPG、PYY和IQGAP3及紫霉素、去甲骆驼蓬碱、斑鸠霉素等小分子药物。为结直肠癌的诊断与治疗提供了潜在的标志物和靶点。这些都为理解结直肠癌发生过程中复杂的分子机制提供新的思路,也为临床进一步的对抗肿瘤药物的研究提供了大量依据,然而,考虑到本基础研究有一定的局限性,未来还需要进一步更深入的实验来验证本研究的结果。
[1]Torre LA, Bray F, Siegel RL, et al. Global cancer statistics,2012[J]. CA Cancer J Clin, 2015, 65(2):87–108. doi: 10.3322/caac.21262.
[2]Aran V, Victorino AP, Thuler LC, et al. Colorectal Cancer:Epidemiology, Disease Mechanisms and Interventions to Reduce Onset and Mortality[J]. Clin Colorectal Cancer, 2016, 15(3):195–203. doi: 10.1016/j.clcc.2016.02.008.
[3]Nannini M, Pantaleo MA, Maleddu A, et al. Gene expression profiling in colorectal cancer using microarray technologies:results and perspectives[J]. Cancer Treat Rev, 2009, 35(3):201–209. doi: 10.1016/j.ctrv.2008.10.006.
[4]Cardoso J, Boer J, Morreau H, et al. Expression and genomic profiling of colorectal cancer[J]. Biochim Biophys Acta, 2007,1775(1):103–137.
[5]Chan SK, Griffith OL, Tai IT, et al. Meta-analysis of colorectal cancer gene expression profiling studies identifies consistently reported candidate biomarkers[J]. Cancer Epidemiol Biomarkers Prev, 2008, 17(3):543–552. doi: 10.1158/1055–9965.EPI-07–2615.
[6]Huang da W, Sherman BT, Lempicki RA. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J]. Nat Protoc, 2009, 4(1):44–57. doi:10.1038/nprot.2008.211.
[7]Gene Ontology Consortium. The Gene Ontology (GO) project in 2006[J]. Nucleic Acids Res, 2006, 34(Database issue):D322–326.
[8]Ashburner M, Ball CA, Blake JA, et al. Gene ontology: tool for the unif i cation of biology. The Gene Ontology Consortium[J]. Nat Genet, 2000, 25(1):25–29.
[9]Saito R, Smoot ME, Ono K, et al. A travel guide to Cytoscape plugins[J]. Nat Methods, 2012, 9(11):1069–1076. doi: 10.1038/nmeth.2212.
[10]von Mering C, Huynen M, Jaeggi D, et al. STRING: a database of predicted functional associations between proteins[J]. Nucleic Acids Res, 2003, 31(1):258–261.
[11]Lamb J, Crawford ED, Peck D, et al. The Connectivity Map: using gene-expression signatures to connect small molecules, genes, and disease[J]. Science, 2006, 313(5795):1929–1935.
[12]Cuyle PJ, Prenen H. Current and future biomarkers in the treatment of colorectal cancer[J]. Acta Clin Belg, 2017,72(2):103–115. doi: 10.1080/17843286.2016.1262996.
[13]Tsavaris N, Lazaris A, Kosmas C, et al. Topoisomerase I and IIalpha protein expression in primary colorectal cancer and recurrences following 5-fluorouracil-based adjuvant chemotherapy[J]. Cancer Chemother Pharmacol, 2009, 64(2):391–398. doi: 10.1007/s00280–008–0886–4.
[14]Zheng H, Li X, Chen C, et al. Quantum dot-based immunof l uorescent imaging and quantitative detection of TOP2A and prognostic value in triple-negative breast cancer[J]. Int J Nanomedicine, 2016, 11:5519–5529.
[15]Cao Y, Zhang G, Wang P, et al. Clinical signif i cance of UGT1A1 polymorphism and expression of ERCC1, BRCA1, TYMS,RRM1, TUBB3, STMN1 and TOP2A in gastric cancer[J]. BMC Gastroenterol, 2017, 17(1):2. doi: 10.1186/s12876–016–0561-x.
[16]Zhou Z, Liu S, Zhang M, et al. Overexpression of Topoisomerase 2-Alpha Confers a Poor Prognosis in Pancreatic Adenocarcinoma Identified by Co-Expression Analysis[J]. Dig Dis Sci, 2017,62(10):2790–2800. doi: 10.1007/s10620–017–4718–4.
[17]Gao CL, Wang GW, Yang GQ, et al. Karyopherin subunit-alpha 2 expression accelerates cell cycle progression by upregulating CCNB2 and CDK1 in hepatocellular carcinoma[J]. Oncol Lett,2018, 15(3):2815–2820. doi: 10.3892/ol.2017.7691.
[18]Reese JM, Bruinsma ES, Monroe DG, et al. ERbeta inhibits cyclin dependent kinases 1 and 7 in triple negative breast cancer[J]. Oncotarget, 2017, 8(57):96506–96521. doi: 10.18632/oncotarget.21787.
[19]Pu S, Zhao Y, Zhou G, et al. Effect of CDK1 shRNA on proliferation, migration, cell cycle and apoptosis in non-small cell lung cancer[J]. J Cell Physiol, 2017, doi: 10.1002/jcp.26387.[Epub ahead of print]
[20]Zhang P, Kawakami H, Liu W, et al. Targeting CDK1 and MEK/ERK Overcomes Apoptotic Resistance in BRAF-Mutant Human Colorectal Cancer[J]. Mol Cancer Res, 2018, 16(3):378–389. doi:10.1158/1541–7786.MCR-17–0404.
[21]Guo W, Zhong K, Wei H, et al. Long non-coding RNA SPRY4-IT1 promotes cell proliferation and invasion by regulation of Cdc20 in pancreatic cancer cells[J]. PLoS One, 2018, 13(2):e0193483. doi:10.1371/journal.pone.0193483.
[22]Ding ZY, Wu HR, Zhang JM, et al. Expression characteristics of CDC20 in gastric cancer and its correlation with poor prognosis[J]. Int J Clin Exp Pathol, 2014, 7(2):722–727.
[23]Chevaleyre C, Benhamouda N, Favry E, et al. The Tumor Antigen Cyclin B1 Hosts Multiple CD4 T Cell Epitopes Differently Recognized by Pre-Existing Naive and Memory Cells in Both Healthy and Cancer Donors[J]. J Immunol, 2015, 195(4):1891–1901. doi: 10.4049/jimmunol.1402548.
[24]Shi YX, Zhu T, Zou T, et al. Prognostic and predictive values of CDK1 and MAD2L1 in lung adenocarcinoma[J]. Oncotarget,2016, 7(51):85235–85243. doi: 10.18632/oncotarget.13252.
[25]Myte R, Gylling B, Häggström J, et al. Untangling the role of one-carbon metabolism in colorectal cancer risk: a comprehensive Bayesian network analysis[J]. Sci Rep, 2017, 7:43434. doi:10.1038/srep43434.
[26]Powell AE, Shung CY, Saylor KW, et al. Lessons from development: A role for asymmetric stem cell division in cancer[J]. Stem Cell Res, 2010, 4(1):3–9. doi: 10.1016/j.scr.2009.09.005.
[27]Clarke CJ, Gross SR, Ismail TM et al. Activation of tissue plasminogen activator by metastasis-inducing S100P protein[J]. Biochem J, 2017, 474(19):3227–3240. doi: 10.1042/BCJ20170578.
[28]Zuo Z, Zhang P, Lin F, et al. Interplay between Trx-1 and S100P promotes colorectal cancer cell epithelial-mesenchymal transition by up-regulating S100A4 through AKT activation. J Cell Mol Med. 2018 Apr;22(4):2430–2441. doi: 10.1111/jcmm.13541.
[29]Qian X, Song X, He Y, et al. CCNB2 overexpression is a poor prognostic biomarker in Chinese NSCLC patients[J].Biomed Pharmacother, 2015, 74:222–227. doi: 10.1016/j.biopha.2015.08.004.
[30]Zhan N, Li B, Xu X, et al. Inhibition of FASN expression enhances radiosensitivity in human non-small cell lung cancer[J].Oncol Lett, 2018, 15(4):4578–4584. doi: 10.3892/ol.2018.7896.
[31]Malumbres M, Barbacid M. Cell cycle, CDKs and cancer: a changing paradigm[J]. Nat Rev Cancer, 2009, 9(3):153–166. doi:10.1038/nrc2602.
[32]Han D, Cho JH, Lee RH, et al. Antitumorigenic effect of atmospheric-pressure dielectric barrier discharge on human colorectal cancer cells via regulation of Sp1 transcription factor[J].Sci Rep, 2017, 7:43081. doi: 10.1038/srep43081.
[33]Zheng HC, Liu JJ, Li J, et al. The in vitro and vivo effects of nuclear and cytosolic paraf i bromin expression on the aggressive phenotypes of colorectal cancer cells: a search of potential gene therapy target[J]. Oncotarget, 2017, 8(14):23603–23612. doi:10.18632/oncotarget.15377.
[34]Chen J, Wei Y, Feng Q, et al. Ribosomal protein S15A promotes malignant transformation and predicts poor outcome in colorectal cancer through misregulation of p53 signaling pathway[J]. Int J Oncol, 2016, 48(4):1628–1638. doi: 10.3892/ijo.2016.3366.
[35]Fontijn RD, Goud B, Echard A, et al. The human kinesin-like protein RB6K is under tight cell cycle control and is essential for cytokinesis[J]. Mol Cell Biol, 2001, 21(8):2944–2955. doi:10.1128/MCB.21.8.2944–2955.2001
[36]Taniuchi K, Nakagawa H, Nakamura T, et al. Down-regulation of RAB6KIFL/KIF20A, a kinesin involved with membrane trafficking of discs large homologue 5, can attenuate growth of pancreatic cancer cell[J]. Cancer Res, 2005, 65(1):105–112.
[37]Stangel D, Erkan M, Buchholz M, et al. Kif20a inhibition reduces migration and invasion of pancreatic cancer cells[J]. J Surg Res,2015, 197(1):91–100. doi: 10.1016/j.jss.2015.03.070.
[38]Shi C, Huang D, Lu N, et al. Aberrantly activated Gli2-KIF20A axis is crucial for growth of hepatocellular carcinoma and predicts poor prognosis[J]. Oncotarget, 2016, 7(18):26206–26219. doi:10.18632/oncotarget.8441.
[39]Khongkow P, Gomes AR, Gong C, et al. Paclitaxel targets FOXM1 to regulate KIF20A in mitotic catastrophe and breast cancer paclitaxel resistance[J]. Oncogene, 2016, 35(8):990–1002.doi: 10.1038/onc.2015.152.
[40]Varis A, Salmela AL, Kallio MJ. Cenp-F (mitosin) is more than a mitotic marker[J]. Chromosoma, 2006, 115(4):288–295.
[41]Kim HE, Kim DG, Lee KJ, et al. Frequent amplification of CENPF, GMNN and CDK13 genes in hepatocellular carcinomas[J]. PLoS One, 2012, 7(8):e43223. doi: 10.1371/journal.pone.0043223.
[42]O'Brien SL, Fagan A, Fox EJ, et al. CENP-F expression is associated with poor prognosis and chromosomal instability in patients with primary breast cancer[J]. Int J Cancer 2007,120(7):1434–1443.
[43]Hirano T. Condensins: organizing and segregating the genome[J].Curr Biol, 2005, 15(7):R265–275.
[44]Liang ML, Hsieh TH, Ng KH, T et al. Downregulation of miR-137 and miR-6500–3p promotes cell proliferation in pediatric high-grade gliomas[J]. Oncotarget, 2016, 7(15):19723–19737. doi:10.18632/oncotarget.7736.
[45]Adrian TE, Ballantyne GH, Zucker KA, et al. Lack of peptide YY immunoreactivity in adenomatous colonic polyps: evidence in favor of an adenoma-carcinoma sequence[J]. J Surg Res, 1988,44(5):561–565.
[46]McFadden DW, Riggs DR, Jackson BJ, et al. Peptide YY inhibits the growth of Barrett's esophageal adenocarcinoma in vitro[J]. Am J Surg, 2004, 188(5):516–519.
[47]Tatemoto K. Isolation and characterization of peptide YY(PYY), a candidate gut hormone that inhibits pancreatic exocrine secretion[J]. Proc Natl Acad Sci U S A, 1982, 79(8):2514–2518.
[48]Grisé KR, Rongione AJ, Laird EC, et al. Peptide YY inhibits growth of human breast cancer in vitro and in vivo[J]. J Surg Res,1999, 82(2):151–155.
[49]Heisler T, Towf i gh S, Simon N, et al. Peptide YY and vitamin E inhibit hormone-sensitive and -insensitive breast cancer cells[J]. J Surg Res, 2000, 91(1):9–14.
[50]Liu CD, Kwan D, Simon N, et al. Synthetic peptide YY analog binds to a cell membrane receptor and delivers fl uorescent dye to pancreatic cancer cells[J]. J Gastrointest Surg, 2001, 5(2):147–152.
[51]Liu CD, Rongione AJ, Garvey L, et al. Adjuvant hormonal treatment with peptide YY or its analog decreases human pancreatic carcinoma growth[J]. Am J Surg, 1996, 171(1):192–196.
[52]Wang S, Watanabe T, Noritake J, et al. IQGAP3, a novel effector of Rac1 and Cdc42, regulates neurite outgrowth[J]. J Cell Sci,2007, 120(Pt 4):567–577.
[53]Nojima H, Adachi M, Matsui T, et al. IQGAP3 regulates cell proliferation through the Ras/ERK signalling cascade[J]. Nat Cell Biol, 2008, 10(8):971–978. doi: 10.1038/ncb1757.
[54]Yang Y, Zhao W, Xu QW, et al. IQGAP3 promotes EGFR-ERK signaling and the growth and metastasis of lung cancer cells[J].PLoS One, 2014,9(5):e97578. doi: 10.1371/journal.pone.0097578.
[55]Holm M, Borg A, Ehrenberg M, et al. Molecular mechanism of viomycin inhibition of peptide elongation in bacteria[J]. Proc Natl Acad Sci U S A, 2016, 113(4):978–983. doi: 10.1073/pnas.1517541113.
[56]Sarkar S, Bhadra K. Therapeutic role of harmalol targeting nucleic acids: Biophysical perspective and in vitro cytotoxicity[J]. Mini Rev Med Chem, 2017, doi: 10.2174/138955751866617121116483 0. [Epub ahead of print]
