荷花转录组测序及花青素苷合成相关基因表达分析
2018-04-25孟亚南刘召强史芳芳刘艺平孔德政
孟亚南 张 琳 刘召强 史芳芳 刘艺平 孔德政
(1. 河南农业大学林学院,河南 郑州 450002;2. 汝阳县第一高级中学,河南 洛阳 471200)
荷花 (Nelumbonucifera) 为睡莲科莲属多年生水生草本花卉,花大而美,多为红、粉、白、黄、复色等,具有很高的观赏价值[1]。而花色是观赏植物最重要的品质之一。观赏植物花色素形成、相关基因的筛选及呈色机理的研究,为花色改良和育种工作奠定理论和技术基础[2]。
目前,转录组测序技术已被广泛应用于植物相关基因组的研究[3-6]。针对不同观赏植物花色的研究也逐步从表型生理方向过渡到分子水平。如桂花 (Osmanthusfragrans)[7]、葡萄风信子 (Muscaribotryoides)[8]、凤丹 (Paeoniaostii)[9]、红花 (Carth-amustinctorius)[10]、切花菊 (Chrysanthemum×mori-folium)[11]等。然而,针对荷花花色形成相关分子机制缺乏深入研究,这在一定程度上限制了其花色的改良与创新。本实验采用转录组测序及RT-PCR技术,获得不同花色荷花品种的差异表达基因,筛选出与花青素苷生物合成相关关键基因,并验证了转录组数据的可靠性,为后期荷花花色改良提供参考依据。
1 材料与方法
1.1 实验材料
供试材料为荷花品种 ‘青玉’ 和 ‘白洋淀红莲’ 栽植于河南农业大学第3生活区试验基地。分别采集4个时期 (初蕾期、松蕾期、初花期、盛花期) 的荷花花瓣,用去离子水冲洗表面杂质,用锡箔纸包好放于液氮中速冻,并及时转移至-80 ℃超低温冰箱备用。
1.2 RNA的样品检测和文库构建
‘青玉’ 和 ‘白洋淀红莲’ 松蕾期花瓣样品,送往北京百迈克生物科技有限公司,采用PureLink Plant RNAReagent Kit试剂盒提取总RNA。采用Nanodrop检测RNA样品的浓度,样品检测合格后,进行cDNA文库的构建,通过PCR富集得到cDNA文库。采用Qubit 2.0和Agilent 2100检测RNA的浓度和完整度,用Q-PCR法准确定量文库的有效浓度,获得高质量的测序数据后用Illumina Hiseq2500进行高通量测序。
1.3 序列拼接和功能注释
用Trinity软件对转录组数据进行组装,组装成为Transcripts,将不同样品组装结果中多个可变剪接的Transcripts聚类到一个基因,得到unigene库。通过unigene在Mapped Reads上的分布,检验片段化的随机程度、基因数目的饱和情况。测序数据量可正向反映测序基因的数目,数据量越大,得到的基因数目越多。通过unigene核酸序列与数据库的BLAST比对,可得到unigene的蛋白功能注释信息,比对中选择参数E-value不大于10-5。
1.4 RNA提取及实时荧光定量PCR分析
选择与花青素苷合成的相关基因 (CHS基因、DFR基因、ANS基因、UF3GT基因) 进行RT-PCR验证分析。采用SK8661柱式植物总RNA抽提纯化试剂盒提取荷花品种 ‘青玉’ 和 ‘白洋淀红莲’ 花瓣的RNA。通过大连宝生物工程(大连)有限公司购买的PrimeScriptTMII1st Strand cDNA Synthesis Kit试剂盒方法合成cDNA。以荷花18S rRNA为内参基因,PCR引物序列运用primer premier 5.0设计,引物合成由北京Biomed公司完成,引物序列如表1所示[12]。按照Invitrogen公司的Power SYBR®Green PCR Master Mix试剂盒方法配制反应体系。

表1 荷花 ‘青玉’ 和 ‘白洋淀红莲’ RT-PCR所用引物序列Table 1 The primers of RT-PCR genes in lotus ′Qingyu′and ′Baiyangdianhonglian′
2 结果与分析
2.1 测序结果分析
2个品种荷花花瓣的转录组测序结果,共得到11.56 Gb的有效数据,其Q30碱基百分比均不小于87.44%,表明测序结果可靠,可用于后续的分析。对组装结果进行统计,得到序列重叠群 (Contig),转录本序列 (Transcript) 和单基因序列 (Unigene) 长度分布表,如表2所示。总共产生50 154条单基因序列和78 384条转录本序列,N50分别为1 412 bp和1 715 bp,组装完整性较高。其中长度区间位于200~300 bp的单基因序列数量最多,为16 788条,占33.47%。其次是长度区间位于300~400 bp和400~500 bp单基因序列数量较多。

表2 unigene组装结果统计Table 2 Statistics of unigene assembly result
2.2 功能分类
为获得unigene的功能分类信息,从NR蛋白分析、GO分析、COG分析、KEGG分析等几个方面进行注释分析。在NR蛋白分析中,共有32 194条 (64.19%) unigene在设定的E值范围内 (E-value ≤ 10-5),可以对比分析到NR蛋白数据库中。通过比对分析,同源蛋白质序列和相似功能蛋白质序列中具有数量最多是葡萄 (Vitisvinifera) (39.5%) 和可可树 (Theobromacacao) (10.6%)。其次,还有碧桃 (Amygdaluspersicavar.persicaf.duplex) (5.9%)、杨树 (Populussp.) (5.5%)、蓖麻 (Ricinuscommunis) (5.0%)。
在本实验的转录本中,能够注释到GO分类的unigene有23 753条 (47.36%),被分成了56个类别,被分成细胞组分 (16个)、分子功能 (16个)、生物学工程 (24个) 3类。有10 374条unigene可注释到COG数据库中,占总unigene的20.68%。其中有2 792条unigene注释到未知功能基因,占26.91%。其次数目较多的为重组和修复功能 (1 310条,12.63%),转录功能 (1 247条,12.02%),核糖体结构及生物学功能,信号转导机制,蛋白质翻译后修饰与转运、分子伴侣。
将unigene比对到KEGG数据库中,共有9 550条unigene参与了194个代谢通路,占总unigene的19.04%,分为五大代谢通路类型,分别是细胞过程、新陈代谢、生物系统、基因信息过程、环境信息过程,参与新陈代谢途径unigene的数量最多,其次为基因信息过程代谢途径。本研究集中花色形成相关因素富集分析,其中,共有40条unigene参与黄酮类化合物生物合成代谢通路,有34条unigene参与胡萝卜素生物合成代谢通路,有8条unigene参与黄酮和黄酮醇生物合成代谢通路,有92条unigene参与苯丙烷生物合成代谢通路。
2.3 差异表达基因分析
通过对样本 ‘青玉’ 花瓣和 ‘白洋淀’ 松蕾期的花瓣2个转录本文库进行差异基因的表达分析,共获得1 142条差异基因,包括587条上调基因,555条下调基因。在本研究中,以FDR < 0.01且FC ≥ 2为筛选标准。上调基因中差异较显著的有16条,下调基因中差异较显著的基因有13条。
2.3.1差异表达基因的功能注释和富集分析
对样本 ‘青玉’ 和 ‘白洋淀’ 的差异表达基因进行GO功能富集分析,能注释到的unigene有800条,分布在生物过程 (BP)、细胞组分 (CC)、分子功能 (MF) 三大类别上。其中,在细胞组分分类中,差异表达基因GO二级节点为:细胞成分、细胞、细胞器、生物膜、细胞器组分的分布数量最多;在分子功能分类中,差异表达基因GO二级节点分布数量较多的为催化活性、结合性;在生物学过程分类中,差异表达基因GO二级节点分布数量最多的为代谢过程、细胞过程、刺激应答、生物调控,具体结果见图1。
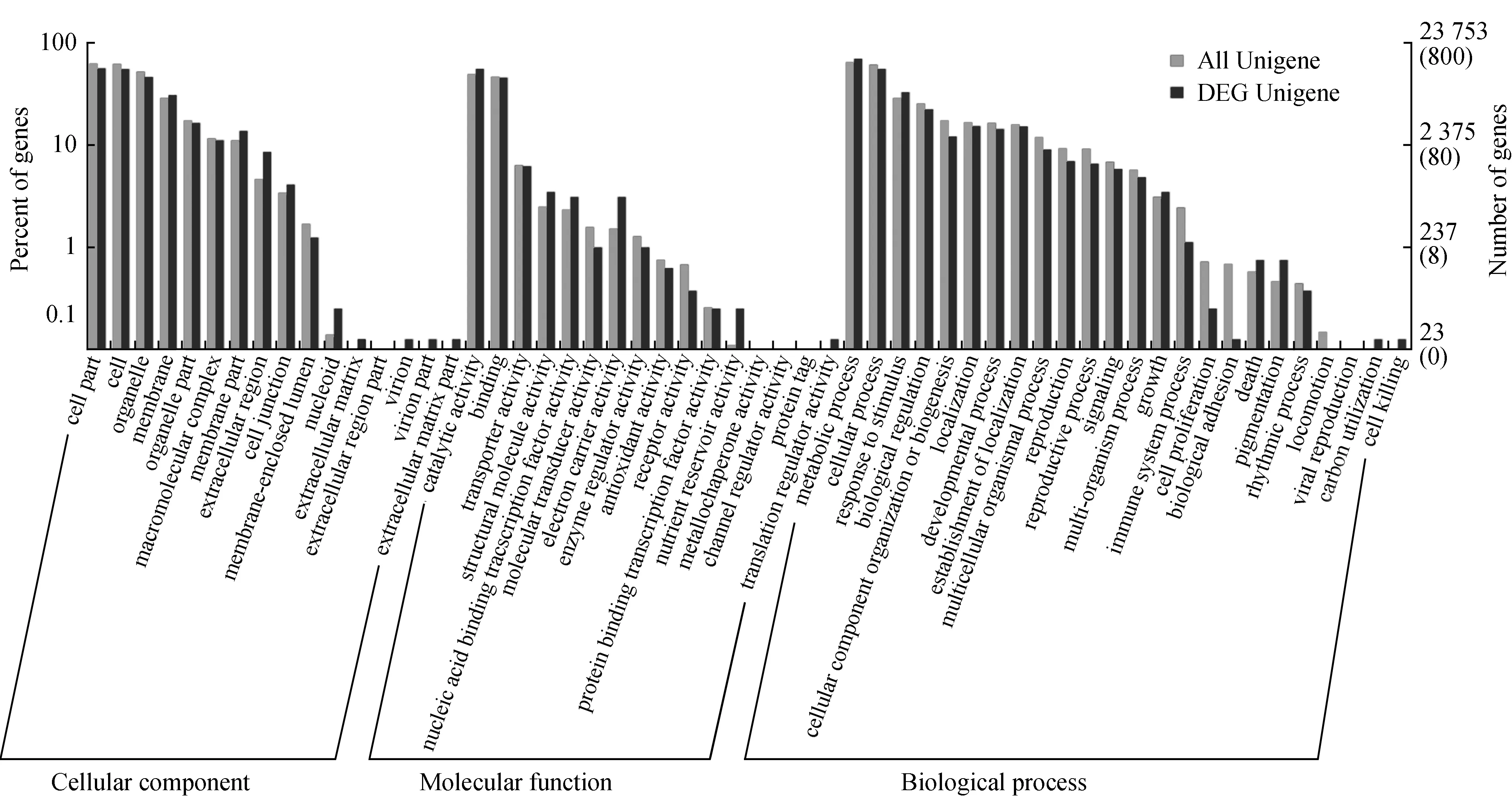
横坐标表示GO三大分类下的二级节点;纵坐标表示注释到该节点的基因数目及占所有基因数的百分比;右边纵坐标括号中代表DEG Unigene。
图1差异表达基因GO二级节点注释统计
Fig.1 GO branch classification of DEG genes
利用topGO软件,结合样品 ‘青玉’ 和 ‘白洋淀红莲’ 转录组的注释信息,对有GO功能的DEG gene进行分析,进行2组样品间的DEG gene的GO功能富集研究。以P-value ≤ 0.05为筛选显著GO term的标准。以CC为例,找出显著富集的GO term,并对显著富集的节点在GO体系中以有向无环图的形式进行直观展示。得知,topGO_CC term下显著富集到的功能分类中,显著性最高的GO term功能分类为叶绿体,KS值为2.50 × 10-9,注释到该term的基因数为3 547个,注释到该term的DEG gene数目为140个,注释到该term的DEG gene数目的期望值为113.99个。其次,在分子功能中显著性较高的GO term功能分类依次为光系统II (1.80 × 10-7)、光系统 (2.80 × 10-6)、叶绿体基质 (3.10 × 10-6)、叶绿体类囊体膜 (1.30 × 10-5)、内质网 (2.80 × 10-5)、细胞质 (5.50 × 10-5)、质外体 (6.30 × 10-5)、细胞壁 (7.60 × 10-5)、质体小球 (0.000 2)、高尔基体 (0.000 24) (表3)。
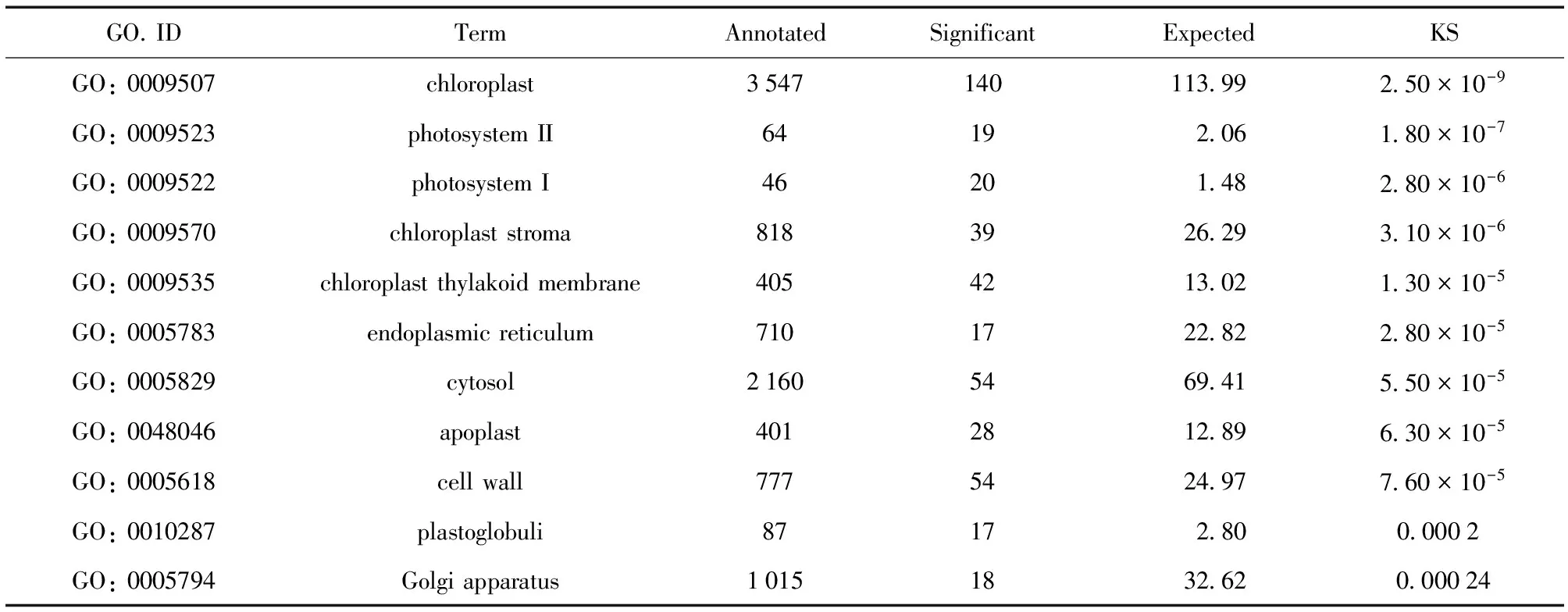
表3 差异表达基因topGO富集结果 (细胞组分)Table 3 TopGO enrichment results of DEG genes (cellular component)
注:GO.ID为GO节点编号;Term为GO节点名称;Annotated为所有基因注释到该功能的基因数;Significant为DEG注释到该功能的基因数;Expected为注释到该功能DEG数目的期望值;KS为富集节点的显著性统计,KS值越小,表明富集越显著。
由表4可知:topGO_BP term下显著富集到的功能分类中,镉的响应是最为显著富集的GO term,DEG gene注释到该功能的基因数为24个,期望值为22.35,KS值为2.50 × 10-9,其次较显著GO term的功能分类分别为盐胁迫效应 (8.00 × 10-8)、蛋白酶体核心 (4.30 × 10-6)、异戊烯二磷酸生物合成 (9.50 × 10-6)、过氧化氢反应 (2.10 × 10-5)、叶绿体组织 (2.30 × 10-5)、细胞质运输 (4.50 × 10-5)等。
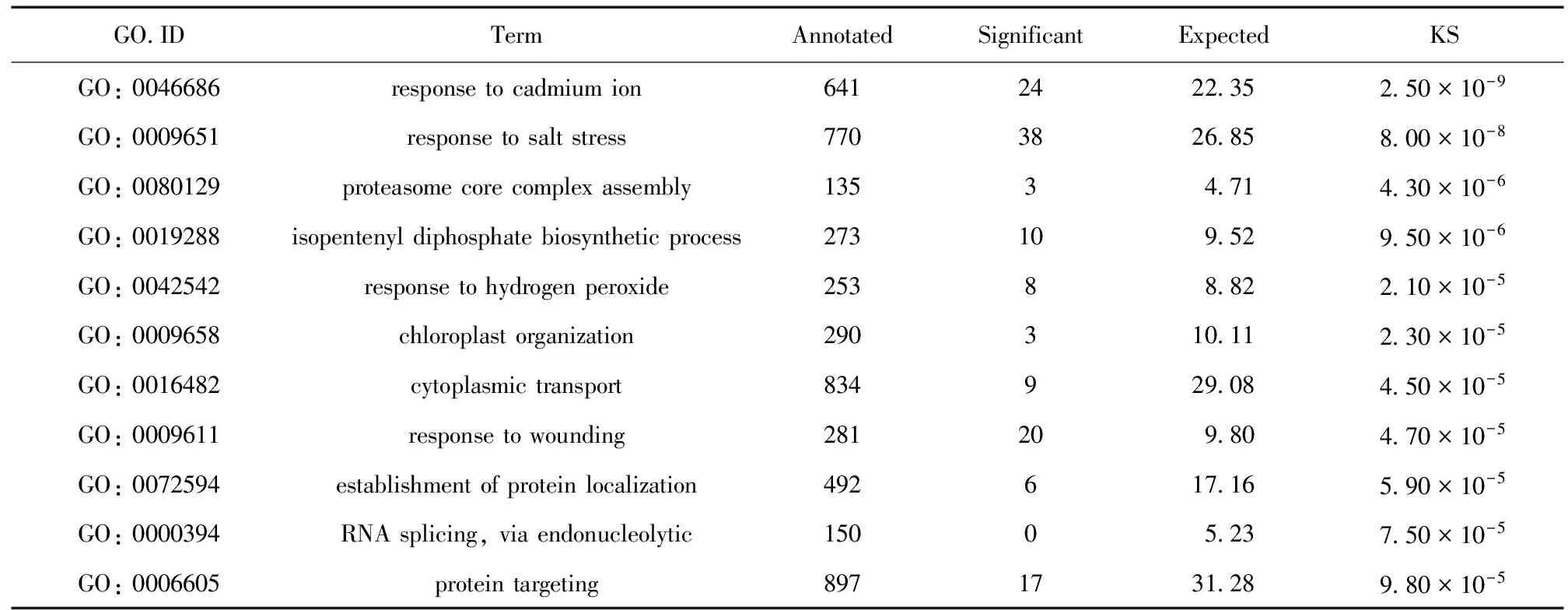
表4 差异表达基因topGO生物学过程富集结果Table 4 TopGO enrichment results of DEG genes (biological process)
topGO_MF (molecular function) term下显著富集到的功能分类中,显著性最高的GO term功能分类为叶绿体结合,KS值为2.20 × 10-6。注释到该term的基因数为40个,注释到该term的DEG gene数目为17个,注释到该term的DEG 数目的期望值为1.44。随后,在分子功能中显著性较高的GO term功能分类依次为丝氨酸内肽酶活性 (6.00 × 10-5)、谷胱甘肽转移酶活性 (0.000 11)、类固醇脱氢酶活性 (0.000 28)、烷烃加氧酶活性 (0.000 28)、铁离子结合 (0.001 11)、热休克蛋白结合 (0.001 13)、电子载体 (0.001 40)、木葡聚糖转移酶活性 (0.002 34)、锌离子结合 (0.002 49)、氧化还原酶活性 (0.003 03)、水通道活性 (0.003 28) (表5)。
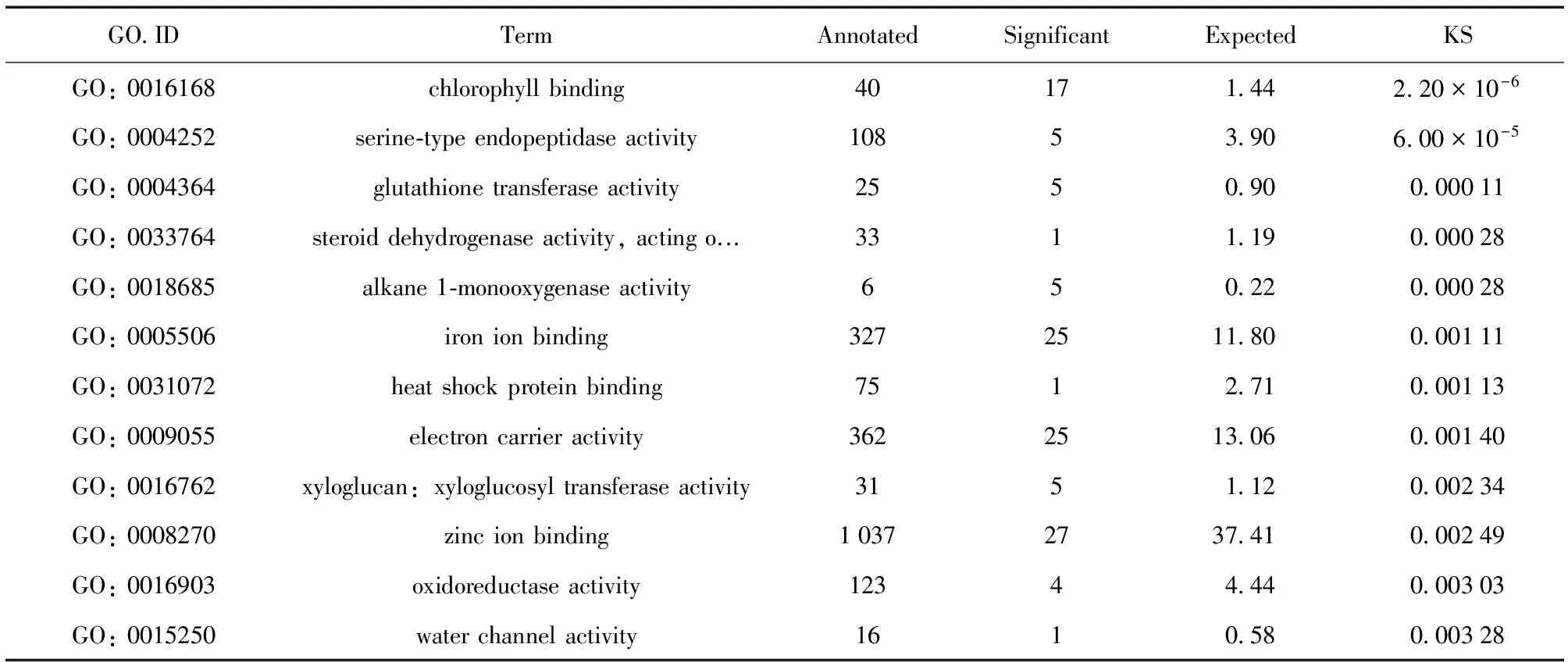
表5 差异表达基因topGO富集结果 (分子功能)Table 5 TopGO enrichment results of DEG genes (molecular function)
在 ‘青玉’ 和 ‘白洋淀红莲’ 的转录本中,能够注释到COG数据库的差异表达基因共有370个。分别为:基因功能预测、生物学功能、蛋白质修饰、转录、能量生产与节能。利用该数据库可以对基因产物进行直系同源分类,利用不同的功能分类,结合特定时间和条件下研究对象的分布,预测对应时期和对应环境下的某一类偏向,从而做出科学的筛选分析 (图2)。

图2差异表达基因COG注释分类统计
Fig.2 COG classification statistics of DEG genes
在 ‘青玉’ 和 ‘白洋淀红莲’ 的转录本中,能够注释到KEGG数据库的差异表达基因247个,共参与到76条pathway中。其中,归入unigene数目最多的5条KEGG途径分别为核糖体、光合作用、植物激素信号转导、淀粉与蔗糖代谢、柠檬酸循环。
本研究对差异表达基因的pathway进行了全面注释分析,可知在76条pathway中,黄酮类化合物生物合成4个、花青素苷合成1个、胡萝卜素生物合成1个、黄酮和黄酮醇生物合成1个、苯丙烷生物合成5个等5条代谢途径与花色素形成有密切的关系。通过所选择的5个与本研究相关的KEGG通路注释图,共找到相关类基因c35395.graph_c0、c27798.graph_c0、c34747.graph_c0、c37226.graph_c0、c40064.graph_c0、c41744.graph_c0、c32055.graph_c0、c37518.graph_c0等。
对差异表达基因进行KEGG代谢通路富集分析,结果显示,富集效果最为显著的代谢通路是丙氨酸,天门冬氨酸和谷氨酸代谢,其次是α-亚麻酸代谢、花青素苷生物合成、不饱和脂肪酸的生物合成、丁酸代谢、柠檬酸循环、黄酮类化合物生物合成、半乳糖代谢、糖酵解途径、氮代谢 (图3)。
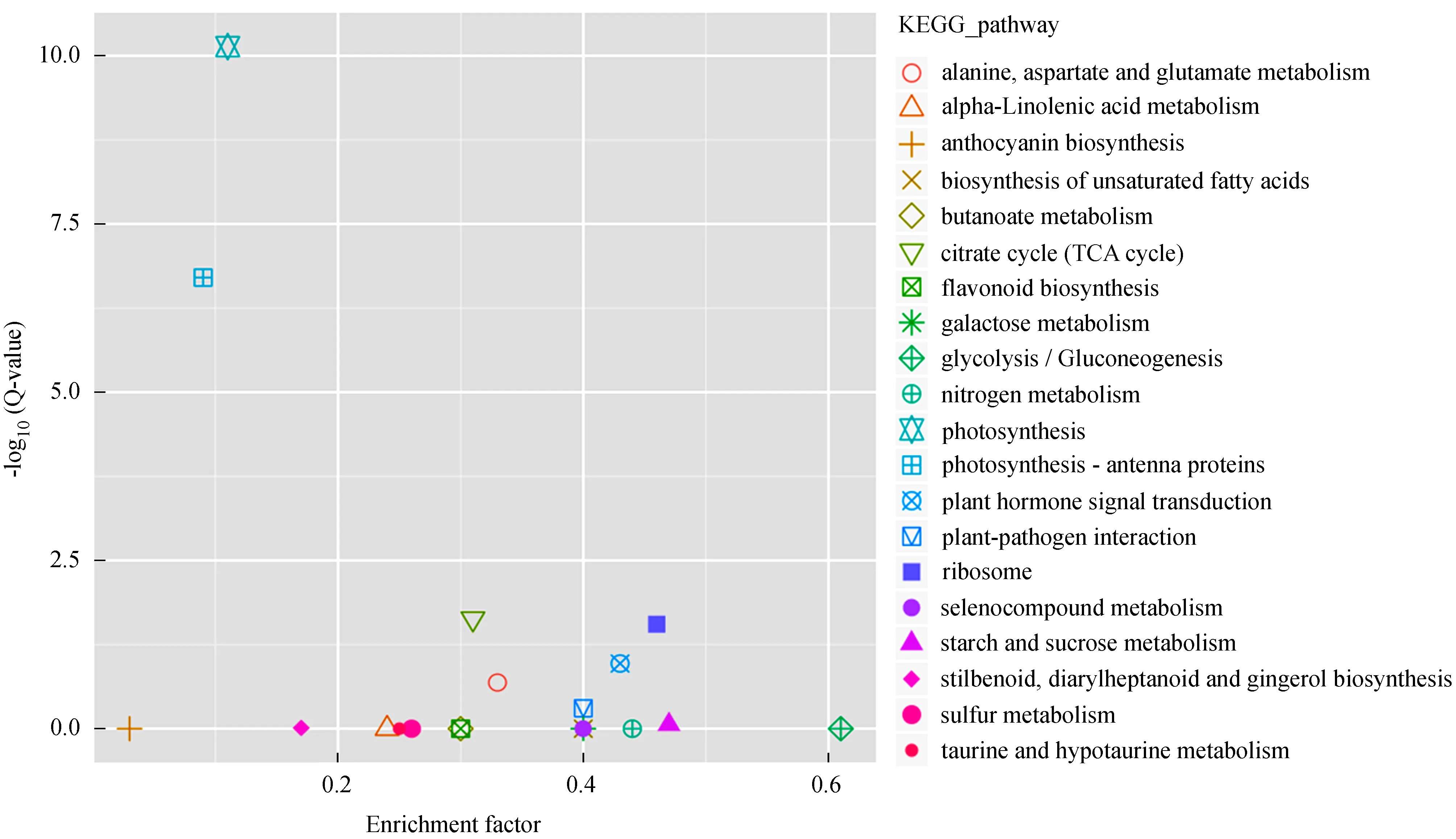
图3差异表达基因KEGG通路富集散点图
Fig.3 KEGG pathway enrichment scatter diagram of DEG genes
2.3.2荷花花色素苷相关基因筛选
通过对 ‘青玉’ 和 ‘白洋淀红莲’ 松蕾期花瓣的测序结果进行功能注释、功能分类及代谢途径分析,共找到与花青素苷代谢途径相关类差异表达基因如表6所示,其中ANS基因9个,分别是c37436.graph_c0、c47965.graph_c0、c41915.graph_c0、c47362.graph_c0、c35395.graph_c0、c50030.graph_c0、c37243. graph_c0、c38760.graph_c0、c1962-9.
graph_c0,包括6条基因呈现上调趋势,3条基因呈下调趋势,推测不同的ANS基因有不同的调节作用;CHS基因c42578.graph_c0,表达为上调;DFR基因c35275.graph_c0、c45153.graph_c0;UF3GT基因c35395.graph_c0等。其中有KEGG注释信息的差异表达基因有c35395.graph_c0、c41915.
graph_c0、c47362.graph_c0、c38760.gra-ph_c0,分别参与的KEGG pathway有ko00942 (花青素苷生物合成)、ko00561 (甘油酯代谢) 和ko00564 (甘油磷脂代谢)、ko00190 (氧化磷酸化)、ko00020 (柠檬酸循环)。
2.4 荷花花色苷相关基因的实时荧光定量PCR验证
以荷花18S rRNA作为内参基因,用相对定量的方法 (按2-ΔΔCt公式) 进行实时荧光定量PCR分析,并进行基因表达水平的比较,由图4~7:实时荧光定量PCR分析花青素苷相关基因c42578.graph_c0 (CHS基因)、c45153.graph_c0 (DFR基因)、c38760.graph_c0 (ANS基因)、c35395.graph_c0 (UF3GT基因) 的相对表达量,4个基因在2种类型花瓣不同时期中均有表达,表达量有显著差异。 ‘青玉’ 花瓣表达量总体明显低于 ‘白洋淀红莲’ 花瓣表达量。

表6 荷花 ‘青玉’ 和 ‘白洋淀红莲’ 转录组测序中花色相关差异基因的表达与功能Table 6 Expression and function of DEG genes related to flower color in transcriptome sequencing
注:Log2FC为表达量差异倍数的对数值。
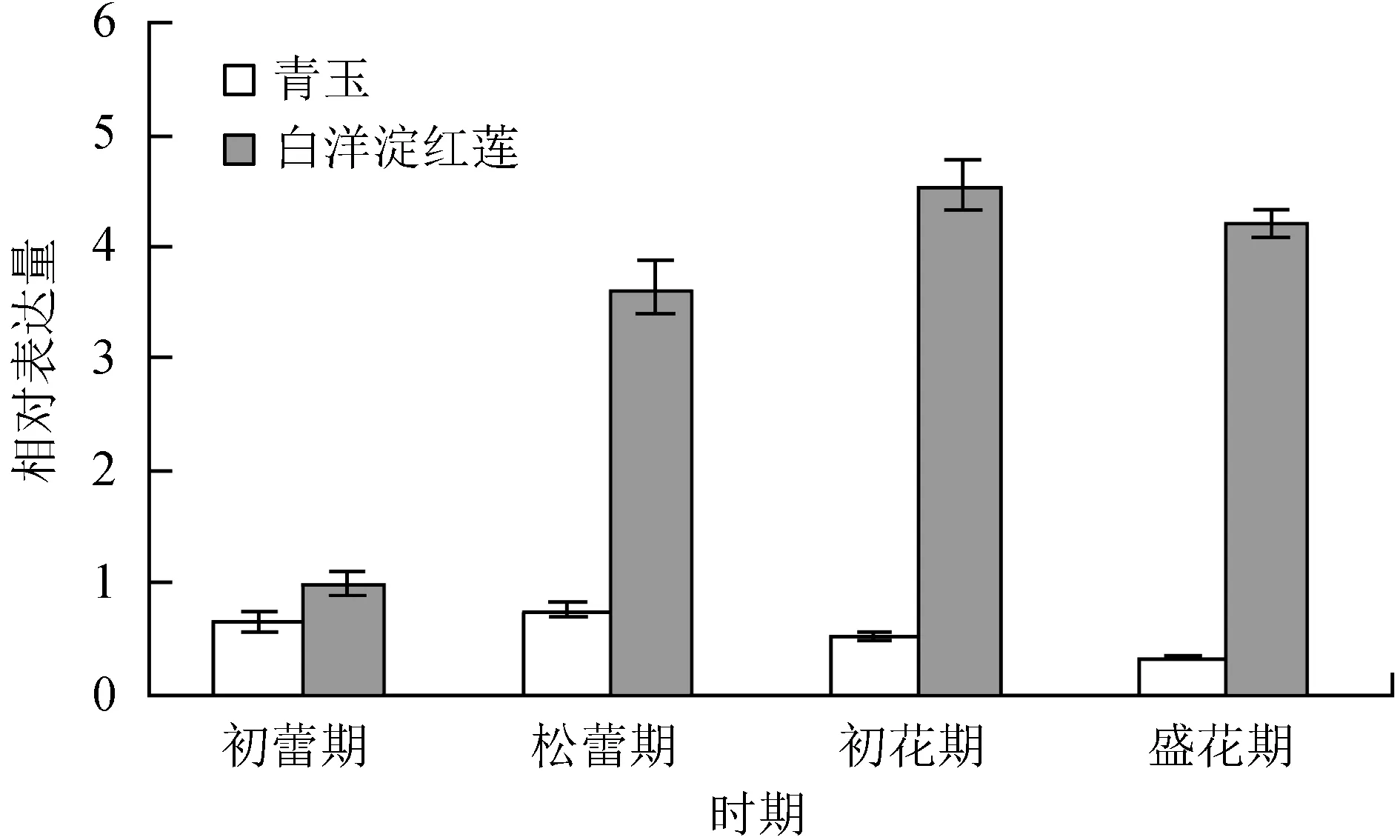
图4荷花CHS基因相对表达量
Fig.4 The relative expression ofCHSgene in lotus
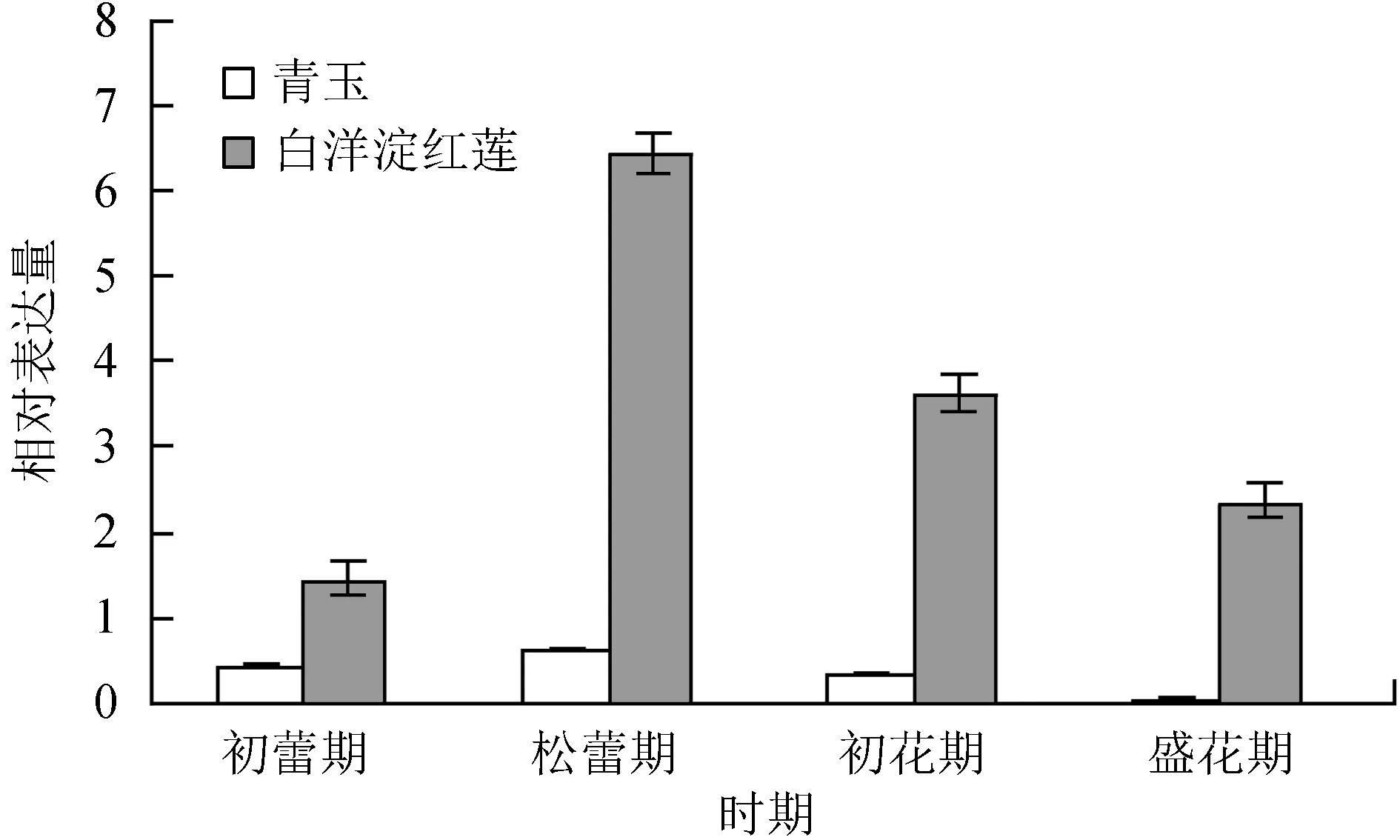
图5荷花DFR基因相对表达量
Fig.5 The relative expression ofDFRgene in lotus
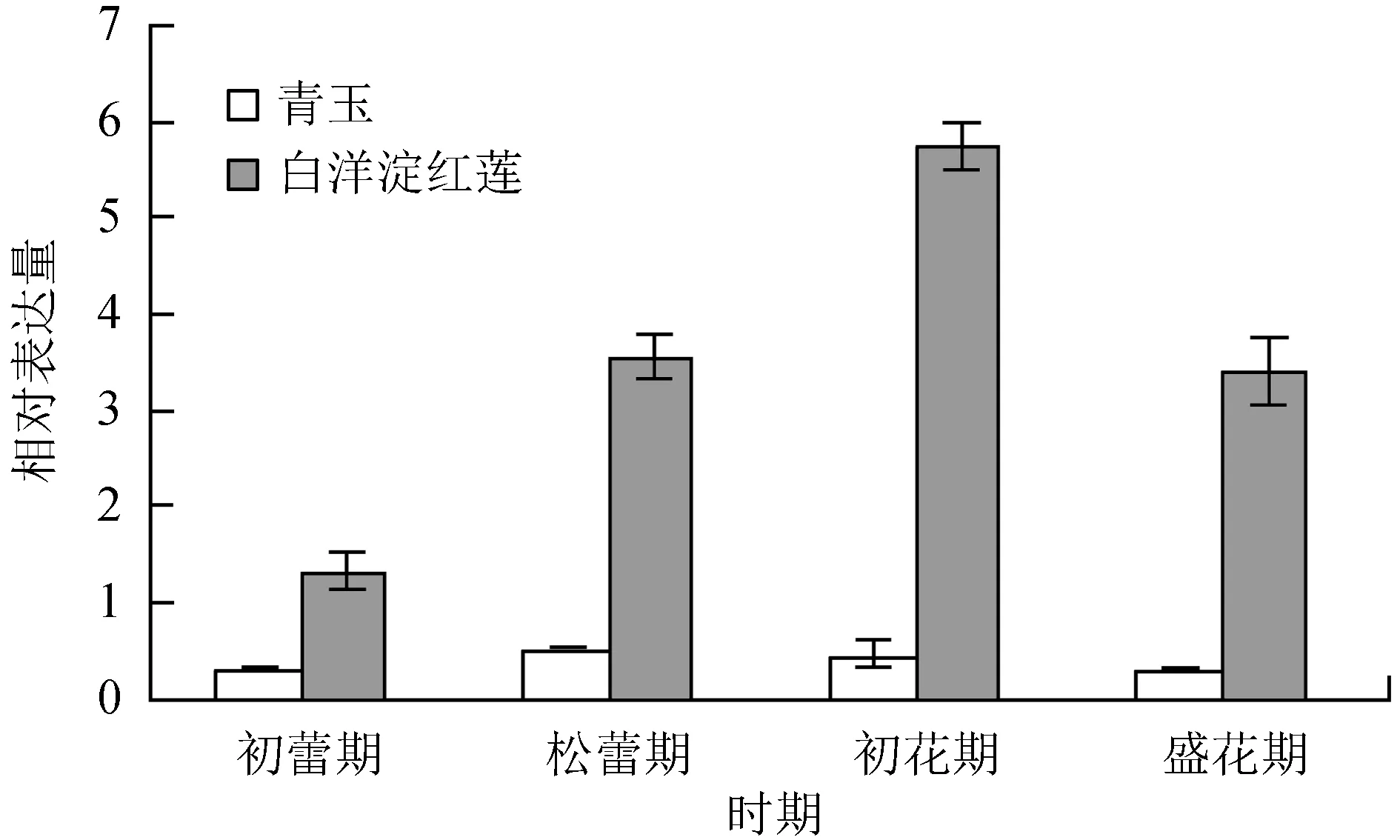
图6荷花ANS基因相对表达量
Fig.6 The relative expression ofANSgene in lotus

图7荷花UF3GT基因相对表达量
Fig.7 The relative expression ofUF3GTgene in lotus
分析得知,在 ‘青玉’ 花瓣中各个时期CHS基因表达量明显低于 ‘白洋淀红莲’,松蕾期表达量最高。在 ‘白洋淀红莲’ 花瓣中,初花期的表达量相对较高,在花朵开放过程中呈现上调趋势,在盛花期的表达量稍低于初花期。DFR基因在 ‘青玉’ 花瓣中表达量均较低,盛花期表达量仅为0.04,此基因在2个荷花品种中表达量存在显著差异。在 ‘白洋淀红莲’ 花瓣中,松蕾期的表达量相对较高,在花朵开放过程中呈现先上升后下降的趋势,在初蕾期到松蕾期上升效果明显。推测DFR基因可能是红色着色所必须的关键结构基因。ANS基因在 ‘青玉’ 花瓣各个时期中表达量明显低于CHS、DFR基因,推测ANS基因是后期花青素苷结构基因,在白色花中表达信号极弱。2个品种相比较而言,差异倍数较大,在松蕾期, ‘白洋淀红莲’ 基因表达量相较 ‘青玉’ 呈现上调趋势,与转录组测序数据一致。UF3GT基因在 ‘白洋淀红莲’ 花瓣中表达量较高,初花期为其表达量最高的时期。2个品种相比较而言,差异倍数较大,尤其是初花期与盛花期,差异倍数为131倍和127倍。在松蕾期,白洋淀红莲基因表达量相较青玉,呈现上调趋势,与转录组测序上调趋势一致,推测此基因表达量的高低,可能对花色的呈色起关键指导作用。
3 结论与讨论
通过Illumina Hiseq2500测序技术平台,共获得数据11.56 Gb clean data,共产生50 154条unigene。对样本 ‘青玉’ 花瓣和 ‘白洋淀红莲’ 松蕾期花瓣2个转录本文库进行差异基因的表达分析 (DEG分析),共获得1 142条差异表达基因,包括587条上调基因,555条下调基因。其中上调基因中差异较显著的有16条,下调基因中差异较显著的有13条。经过差异表达基因生物蛋白功能注释分析,在KEGG代谢途径中查找与花色相关的代谢途径,共找到与花色相关代谢通路5个,相关表达基因16个,并找到花青素苷相关基因4个,分别是CHS基因、DFR基因、ANS基因、UF3GT基因,对其进行实时荧光定量PCR表达验证,得知这4个基因在2个品种花瓣不同时期条件下,表达量存在显著差异,各个时期的表达量在 ‘白洋淀红莲’ 中比 ‘青玉’ 较高。在白色荷花品种 ‘青玉’ 花瓣中,各个时期花青素苷基因的表达量均较低,推测其花青素苷积累量可能较低。CHS基因、ANS基因、UF3GT基因在 ‘白洋淀红莲’ 中初花期的表达量最大,而在 ‘青玉’ 中松蕾期的表达量大。DFR基因在 ‘白洋淀红莲’ 中松蕾期的表达量最大。其中,在本实验中花青素苷合成晚期结构基因表达中,UF3GT基因在 ‘白洋淀红莲’ 花瓣中表达量远远大于 ‘青玉’ 花瓣。后期可以筛选不同花色的荷花品种,深入研究与花青素苷形成的相关基因,并分析出其关键基因。关于花青素苷的研究在其他植物中也有相关研究[13-16],与花色素苷相关的基因逐渐丰富,为后期花色的创新研究打下基础。在未来研究中,可进一步结合HPLC分析花青素苷的种类及含量。如邓娇等对108种不同花色的荷花花瓣类黄酮色素及着色机理进行研究,得知不同品种荷花花瓣中含有5种花青素,并对2个品种的荷花做蛋白组学分析,验证了相关基因的mRNA的转录水平和蛋白组水平的一致性[17]。另外,花青素的生物合成受结构基因和调节基因的共同控制,对花青素的代谢研究会有更加深刻的认识[18-20]。许传俊等人对不同花色、不同阶段的蝴蝶兰 (Phalaenopsisaphrodite) 进行研究,得知影响其花色素苷合成途径的10个结构基因和调节基因的表达量存在差异[21]。在本研究中,筛选出与荷花花瓣中花青素苷合成相关的4个结构基因,相关基因进行筛选与表达分析,后期也可以结合花青素生物合成的调节基因共同研究。深入挖掘影响花青素苷代谢途径的相关因素,为阐明荷花花色形成的机理提供理论依据,为荷花的花色改良分子育种奠定基础。
[参 考 文 献]
[1] 王其超, 张行言. 中国荷花品种图志 [M]. 北京: 中国林业出版社, 2005.
[2] 戴思兰, 洪艳. 基于花青素苷合成和呈色机理的观赏植物花色改良分子育种[J]. 中国农业科学, 2016, 49(3): 529-542.
[3] Vanburen R, Walters B, Ming R, et al. Analysis of expressed sequence tags and alternative splicing genes in sacred lotus (NelumbonuciferaGaertn) [J]. Plant Omics, 2013, 6(4): 311-317.
[4] Yang M, Xu L, Liu Y, et al. RNA-Seq Uncovers SNPs and alternative splicing events in Asian lotus (Nelumbonucifera) [J]. Plos One, 2015, 10(4): e0125702.
[5] Cai J, Liu X, Vanneste K, et al. The genome sequence of the orchidPhalaenopsisequestris[J]. Nature Genetics, 2015, 47(1): 65.
[6] Han X J, Wang Y D, Chen Y C, et al. Transcriptome sequencing and expression analysis of terpenoid biosynthesis genes inLitseacubeba[J]. Plos One, 2013, 8(10): e76890.
[7] 母洪娜, 孙陶泽, 杨秀莲, 等. 两个桂花品种花色色素相关基因的差异表达[J]. 南京林业大学学报 (自然科学版), 2015, 39(3): 183-186.
[8] Qian L, Liu Y, Qi Y, et al. Transcriptome sequencing and metabolite analysis reveals the role of delphinidin metabolism in flower colour in grape hyacinth[J]. Journal of Experimental Botany, 2014, 65(12): 3157.
[9] 刘洪峰. 凤丹不同颜色花瓣的色素及花色形成相关基因表达分析[D]. 北京: 北京林业大学, 2015.
[10] 刘秀明, 张宇, 姚娜, 等. 红花 (CarthamustinctoriusL.) 不同开花时期的转录组测序及黄酮合成相关基因的验证[J]. 中国细胞生物学学报, 2015, 37(9): 1207-1215.
[11] 韩科厅, 赵莉, 唐杏姣, 等. 菊花花青素苷合成关键基因表达与花色表型的关系[J]. 园艺学报, 2012, 39(3): 516-524.
[12] 刘艺平, 董姬秀, 张曼, 等. 荷花LEAFY基因的克隆及表达分析[J]. 植物生理学报, 2014, 50(2): 203-208.
[13] 朱满兰, 王亮生, 张会金, 等. 耐寒睡莲花瓣中花青素苷组成及其与花色的关系[J]. 植物学报, 2012, 47(5): 437-453.
[14] 李珍, 刘金兵, 刁卫平, 等. 辣椒花青素生物合成相关基因的表达分析研究[J]. 华北农学报, 2014, 29(4): 87-92.
[15] 钟淮钦, 黄敏玲, 吴建设, 等. 蝴蝶兰花青素苷合成途径关键酶基因的克隆与表达[J]. 福建农业学报, 2013(9): 854-858.
[16] 张彬, 尹美强, 温银元, 等. 羽衣甘蓝花青素合成途径结构基因的表达特性[J]. 山西农业科学, 2014, 42(4): 313-316.
[17] Deng J, Chen S, Yin X, et al. Systematic qualitative and quantitative assessment of anthocyanins, flavones and flavonols in the petals of 108 lotus (Nelumbonucifera) cultivars[J]. Food Chemistry, 2013, 139(1/4): 307-312.
[18] 杨鹏程, 周波, 李玉花. 植物花青素合成相关的bHLH转录因子[J]. 植物生理学报, 2012, 48(8): 747-758.
[19] 李莹, 高振蕊, 张驰, 等. 花青素合成途径中分子调控机制的研究进展[J]. 生态学杂志, 2015, 34(10): 2937-2942.
[20] 秦军, 潘腾飞, 郭志雄, 等. 多花水仙花色基因与遗传转化研究进展[J]. 福建农业科技, 2016, 47(4): 59-65.
[21] 许传俊, 黄珺梅, 黄雯, 等. 不同花色品种蝴蝶兰花色素苷含量分析及相关基因表达研究[J]. 华南师范大学学报 (自然科学版), 2015, 47(3): 93-99.
