绿豆环氧化物水解酶基因的克隆、分析及表达
2018-04-20邬敏辰叶慧华
姚 瑶, 胡 蝶, 邬敏辰, 叶慧华
(1.江南大学 药学院,江苏 无锡 214122;2.江南大学 生物工程学院,江苏 无锡 214122;3.江南大学 无锡医学院,江苏 无锡 214122)
手性环氧化物和邻二醇是一类合成手性化合物的重要砌块,被广泛应用于医药、农药、精细化工材料等的合成,如β-3肾上腺素受体激动剂、南部松小蠹诱剂和α-甜没药醇等的合成[1]。环氧化物水解酶 (epoxide hydrolase,EH,EC 3.3.2.3) 能选择性水解环氧化物生成相应的邻二醇[2],例如,其水解外消旋环氧苯乙烷((R,S)-SO) 可产生(R)-或(S)-苯基乙二醇(PED),该产物是液晶材料中不可缺少的手性添加剂,也是合成许多光学活性药物和农药的重要手性中间体。已发现的一些来源于动物、植物和微生物的EHs具有良好的对映选择性和区域选择性[3-4]。目前,对EH的研究主要集中在动力学拆分消旋体环氧化物制备手性环氧化物和邻二醇,但其生成1,2-二醇的对映体过量值(e.e.值)普遍较低,且最大理论产率为50%。如Aspergillus niger LCP 52 ANEH和Cupriavidus metallidurans-CH34 CMEH 动力学拆分外消旋(R,S)-SO 获得(R)-PED的 e.e.值分别为56%和51%[5-6]。
对映体会聚 (enantioconvergence)是指将2种构型的外消旋底物同时转化成一种构型的产物,发生对映体会聚的水解反应[7],其产物的最大理论产率为100%。许建和课题组从绿豆(Vigna radiata)中成功克隆出 2种 EHs基因 Vreh1[8]和 Vreh2[9],并实现其在大肠杆菌 (Escherichia coli)中的异源表达,获得的重组酶均能对映归一性水解对硝基苯基环氧乙烷生成 (R)-对硝基苯基乙二醇,当转化率为100%时,产物对映体过量e.e.值分别为70%和84.8%。本研究中通过对植物来源的EH进行基因多序列比对,设计2条简并引物,采用RT-PCR和作者所在研究室建立的侧翼未知DNA序列扩增技术THSO-PCR,克隆了一种新型绿豆EH的基因(Vreh3),并将其在 E.coli BL21(DE3) 中表达,利用该酶水解(R,S)-SO 制备(R)-PED。
1 材料与方法
1.1 菌株、质粒和培养基
E.coli JM109、BL21 (DE3) 和表达质粒 pET-28a(+)购自Novagen公司;克隆质粒pUCm-T购自上海Sangon公司;LB培养基:0.5 g/dL酵母提取物、1 g/dL蛋白胨和1 g/dL NaCl(固体培养基添加2 g/dL琼脂粉),pH 7.2。
1.2 主要试剂和仪器
绿豆,购自超市;UNIQ-10柱式Trizol总RNA抽提试剂盒和DNA胶回收试剂盒,购自上海Sangon公司;Ex Taq和rTaq DNA聚合酶、限制性内切酶、T4 DNA连接酶和RNA PCR Kit(AMV)Ver.3.0试剂盒,均购自大连 TaKaRa公司;(R,S)-SO 和(S)-PED,购自上海萨恩化学技术有限公司。气相色谱仪GC-2010,购自日本Shimadzu公司产品;手性气相色谱柱CYCLOSIL-B(30 m×0.25 mm×0.25 μm),购自美国Agilent科技公司产品。
1.3 绿豆总RNA及基因组DNA提取
选取饱满的绿豆种子置于培养皿中,加入少许去离子水覆盖培养皿底层,于37℃培养箱中孵育12 h,取发芽绿豆胚芽,使用UNIQ-10柱式Trizol总RNA抽提试剂盒提取绿豆总RNA。采用简化CTAB法[10]提取绿豆基因组DNA。
1.4 PCR引物的设计
将 Vignaradiata(ADP68585)、Glycinemax(NP_001238563) 和 Phaseolus vulgaris(XP_00714 7002)等21条来源于植物的EH氨基酸序列进行比对,在近N端和C端区域各选取一段保守序列(MHVAEKG) 和(AHFNNQ),分别对应其设计简并引物VrEH-F1和VrEH-R1,用于Vreh3基因部分cDNA序列的扩增;根据上述引物扩增获得的cDNA序列,在近其N端和C端非保守区分别设计特异下游引物VrEH-R2和特异上游引物VrEH-F2,用于Vreh3基因5′端DNA和3′端cDNA序列的扩增。根据获得的 Vreh3基因 3′端 cDNA和 5′端 DNA序列,设计上下游引物VrEH-F和VrEH-R,用于克隆Vreh3基因编码区DNA和cDNA序列。T-发卡结构序列HSO-T和引物HSO-F参照胡蝶[11]等所述方法合成。除试剂盒内引物,其余引物和HSO-T均由上海Sangon公司合成,见表1。
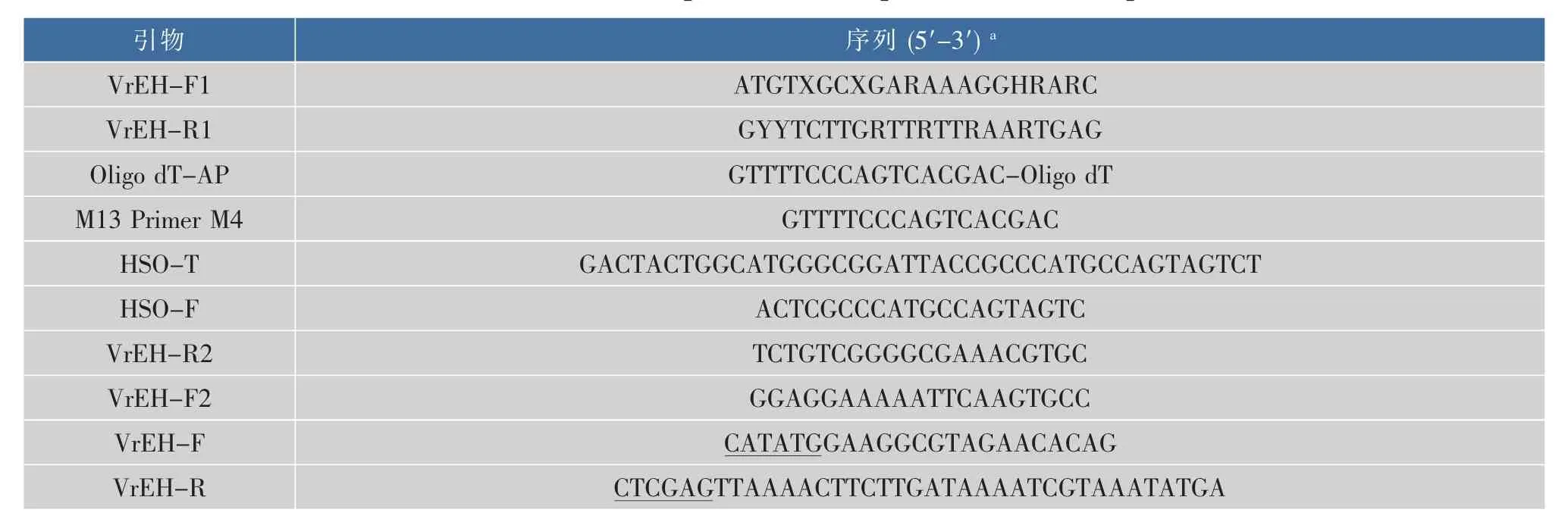
表1 PCR扩增引物和T-发卡结构序列Table 1 Primers and T-hairpin structure sequences for PCR amplification
1.5 Vreh3基因序列的扩增
1.5.1Vreh3基因3′端cDNA序列的扩增以绿豆总RNA为模板,Oligo dT-AP为引物,按照TaKaRa RNA PCR Kit(Ver.3.0)说明书进行反转录合成cDNA的第一链。以该链为模板,VrEH-F1和M13 Primer M4为引物进行第一轮PCR扩增。以第一轮PCR产物为模板,VrEH-F1和VrEH-R1为引物进行第二轮PCR扩增获得Vreh3基因部分cDNA片段,PCR扩增产物经琼脂糖凝胶电泳,割胶回收目的条带,连接pUCm-T载体,转化E.coli JM109,经蓝白斑筛选后,送上海Sangon测序。然后,以第一轮PCR产物为模板,VrEH-F2和M13 Primer M4为引物进行第二轮PCR扩增,获得部分cDNA片段。合并2次第二轮PCR扩增的cDNA片段获得Vreh3基因3′端cDNA序列。
1.5.2Vreh3基因5′端DNA序列的扩增参考胡蝶[11]等的 THSO-PCR技术扩增 Vreh3基因 5′端DNA。以HSO-T发夹结构与酶切修饰后绿豆基因组DNA的连接产物为模板,HSO-F与VrEH-R2为引物进行PCR扩增获得Vreh3基因5′端DNA序列。1.5.3Vreh3编码区DNA和cDNA序列的扩增以绿豆基因组DNA为模板,VrEH-R和VrEH-F为引物进行PCR扩增获得Vreh3基因编码区DNA序列。以逆转录合成cDNA第一链为模板,以VrEH-F和M13 Primer M4为引物进行第一轮PCR;以第一轮PCR产物为模板,VrEH-F和VrEH-R为引物进行第二轮PCR:94℃ 3 min,30个循环(94℃ 30 s,55 ℃ 30 s,72 ℃ 70 s),72 ℃ 10 min,获得 Vreh3基因编码区cDNA序列,PCR产物回收后与pUCm-T连接,转化E.coli JM109,测序正确的重组质粒命名为pUCm-T-Vreh3。
1.6 生物信息学分析
利 用 NCBI ORF Finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)分析 Vreh3开放阅读框;BDGP(http://www.fruitfly.org/index.html)预测转录起始 位 点 ;PLACE (http://www.dna.affrc.go.jp/PLACE/signalscan.html)预测顺式调控作用元件;ProtParam(http://web.expasy.org/protparam/) 工具分析 VrEH3理化性质;DNAMAN 6.0和ClustalW2软件用于蛋白质一级结构的分析。
1.7 Vreh3在 E.coli BL21(DE3)中的表达
以cDNA第一链为模板,以VrEH-F和M13 Primer M4为引物进行第一轮PCR;以该轮PCR产物为模板,VrEH-F和VrEH-R为引物进行第二轮PCR获得Vreh3基因编码区cDNA序列,产物回收后与pUCm-T连接,转化E.coli JM109,测序正确的重组质粒命名为pUCm-T-Vreh3。
用Nde I和Xho I双酶切pUCm-T-Vreh3,割胶回收目的基因Vreh3,与经同样双酶切的pET-28a(+) 连接,获得重组表达质粒 pET-28a(+)-Vreh3,转化E.coli BL21(DE3),获得工程菌命名为E.coli BL21/pET-28a(+)-Vreh3。在 IPTG终浓度 0.15 mmol/L,16℃诱导表达重组VrEH3。100 mL诱导发酵液经8 000 r/min离心收集菌体,用5 mL磷酸钾缓冲液(100 mmol/L,pH 7.0) 悬浮,用于 SDS-PAGE分析及活性测定。不含目的基因的pET-28a(+)转化E.coli BL21(DE3)作为空白对照,命名为E.coli/pET-28a(+)。
1.8 气相色谱分析及计算
样品分析采用气相色谱仪GC-2010、手性气相色谱柱CYCLOSIL-B和氢火焰离子化检测器。进样口和检测器温度均为250℃;初始柱温100℃,以5℃/min升温至210℃;载气为氮气,流速3.0 mL/min,分流比 1∶50。在此检测条件下,正己醇、(R)-SO、(S)-SO、(S)-PED 和 (R)-PED 的保留时间分别为3.738、6.368、6.492、17.437 min 和 17.574 min。 (R)-PED 摩尔产率 =(S/RS0) × 100%,e.e.=[(S-R)/(S+R)]× 100%; 其中:S和 R 分别代表 (S)-和 (R)-PED的最终摩尔浓度,RS0代表(R,S)-SO的初始摩尔浓度。
1.9 EH活性的测定
在 2 mL EP管中加入 800 μL菌液和 150 μL磷酸钾缓冲液 (100 mmol/L,pH 7.0),25 ℃预热 5 min;加入 50 μL(R,S)-SO(200 mmol/L),25 ℃反应3.5 h后8 000 r/min离心2 min, 取100 μL上清液于1 mL乙酸乙酯 (含1 mmol/L的正己醇作为内标),激烈震荡,8 000 r/min离心2 min,吸取上层有机相,经无水硫酸镁干燥,过0.22 μm有机膜,按1.8方法进行气相色谱分析。
2 结果与分析
2.1 Vreh3基因序列的扩增
2.1.1Vreh3基因3′端cDNA序列的扩增据1.5所述,以VrEH-F1和M13 Primer M4为引物进行PCR 获得约 1 800 bp的条带(图 1(a) 泳道 1);以VrEH-F1和VrEH-R1为引物,第二轮PCR产物获得约 850 bp条带(图 1(a)泳道 2);以 VrEH-F2和M13 Primer M4为引物,第二轮PCR产物获得约400 bp、500 bp 2 条清晰的条带(图 1(a) 泳道 3),测序结果显示约500 bp处条带为Vreh3基因序列。合并2次第二轮PCR产物获得的序列,获得的Vreh3基因3′端cDNA序列长1 080 bp。
2.1.2Vreh3基因5′端DNA序列的扩增不同限制性内切酶酶切基因组构建了3个PCR模板库,以HSO-F和VrEH-R2为引物分别进行PCR扩增,获得约 2 100 bp、220 bp 和 220 bp 的条带(图 1(b))。测序结果显示3个条带均为Vreh3基因5′端DNA序列。
2.1.3Vreh3基因编码区DNA和cDNA序列的扩增用DNAMAM软件分析3′端cDNA序列确定终止子为TAA,设计特异性下游引物VrEH-R;用BestORF分析5′端DNA序列确定起始密码子ATG的位置,设计特异性上游引物VrEH-F。以绿豆总RNA逆转录合成cDNA第一链为模板,VrEH-F和VrEH-R为引物,经二轮PCR扩增获得Vreh3基因的编码区cDNA长约960 bp(图1(c)泳道1)。以绿豆基因组DNA为模板,VrEH-F和VrEH-R为引物,经PCR扩增获得Vreh3基因的编码区DNA长约1 200 bp的序列(图1(c)泳道2)。测序获得编码区cDNA序列为 957 bp(GenBank登录号为KR013755),编码区DNA序列为1 178 bp。
2.2 Vreh3的核酸序列分析
用DNAMAN合并扩增获得Vreh3序列,编码区DNA序列包含2个142 bp和79 bp的内含子和957 bp的开放阅读框,编码318个氨基酸。利用BDGP预测,位于起始密码子ATG上游的94 bp处的鸟嘌呤(G)为转录起始位点;利用PLACE分析在转录起始位点上游22 bp处发现真核生物中典型的TATA-box,在终止密码子下游360 bp处发现AATAATA的多聚腺苷酸加尾信号,分析结果表明Vreh3的5′-非翻译区和 3′非编码区长分别为 94 bp 和 446 bp(图 2)。
2.3 VrEH3氨基酸序列分析
利用ProtParam分析VrEH3的理论相对分子质量和等电点分别为36.2×103和5.59。通过将VrEH3与不同来源的5种EH进行氨基酸序列比对,发现VrEH3 与来源 于 Glycine max(NP_001238563)、Glycine max(NP_001240943)、Vigna radiata(ADP685 85,VrEH1)[8]、Phaseolus vulgaris (XP_007147002)和Solanum lycopersicum (XP_004230258)的EH氨基酸序列同源性分别为80.8%、73.9%、72.4%、71.6%和66.0%(图3)。根据文献报道分析,VrEH3保守的Asp101、Asp262和 His297组成催化三联体,Tyr150和Tyr232为质子供体。
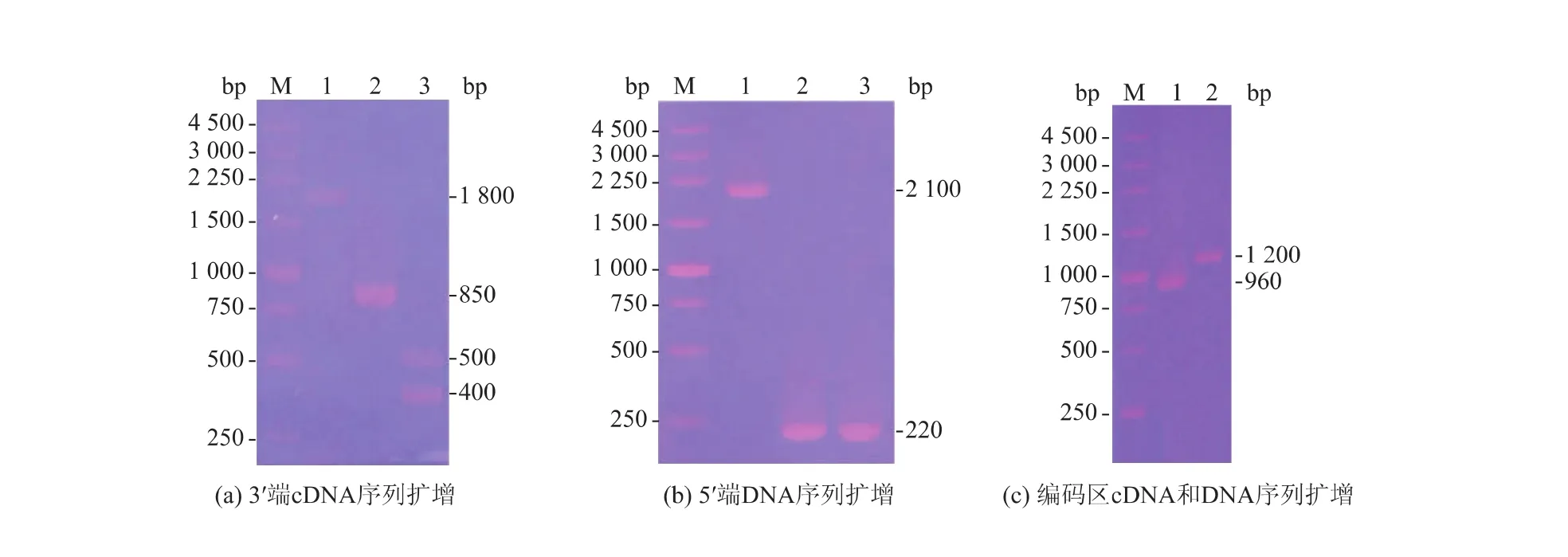
图1 Vreh3序列扩增的PCR产物Fig.1 PCR products of the amplification of Vreh3

图2 Vreh3的核酸序列分析Fig.2 Analysis of nucleotide sequence of Vreh3
2.4 Vreh3在E.coli中的表达
构建的重组菌VrEH3按1.7方法进行诱导表达。SDS-PAGE分析结果显示,VrEH3全细胞在约30.1×103处有特异性目的条带(图 4,泳道 2),小于VrEH1(约 41.0×103)[8]和 VrEH2(约 36.0×103)[9]的相对分子量;E.coli/pET-28a(+)在该处无特异性条带(图 4泳道 1),表明 VrEH3成功在 E.coli BL21(DE3)中表达。
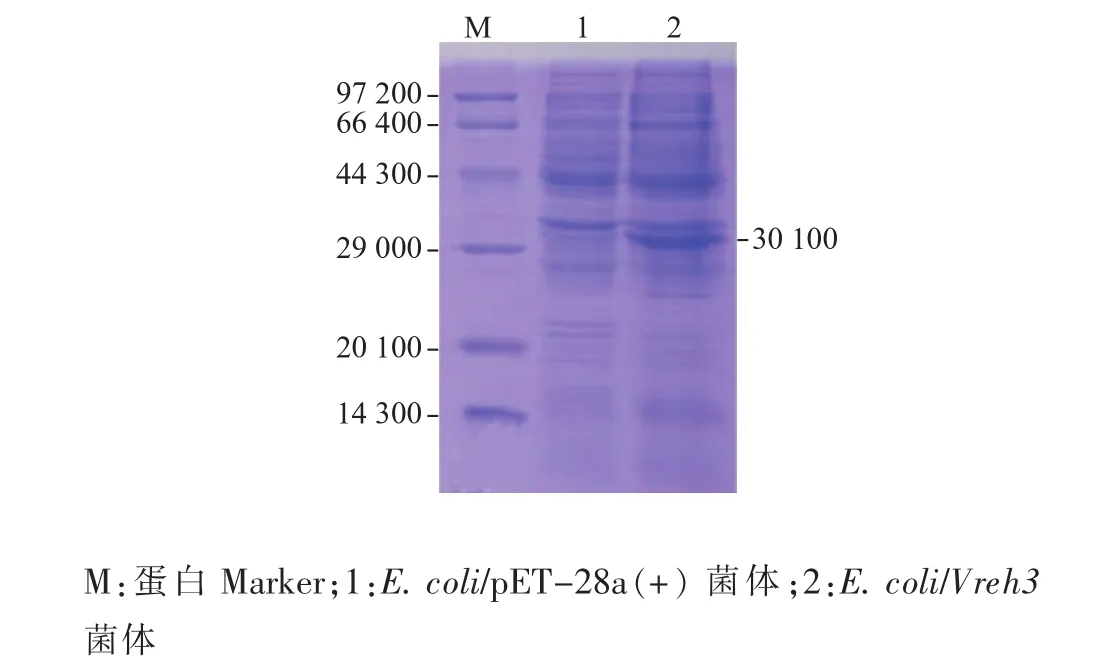
图4 重组大肠杆菌表达产物的SDS-PAGE分析Fig.4 SDS-PAGE analysis of the expressed products by recombinant E.coli
2.5 VrEH3 对映归一性水解(R,S)-SO
按1.9所述方法,诱导后的工程菌E.coli/Vreh3 对10 mmol/L(R,S)-SO 进行水解反应,反应进程如图5。结果表明,VrEH3优先水解(S)-SO,而对(R)-SO 反应较慢。 在 0~2 h内,(S)-SO 的摩尔浓度迅速降低,(R)-SO的e.e.值迅速上升,同时产物e.e.值一直保持在95%以上。当反应3.5 h时,(S)-SO 全部水解,(R)-SO 的 e.e.值>99%,得率为20.6%;产物(R)-PED 的 e.e.值为 94.7%,得率为79.4%,高于绿豆粉水解(R,S)-SO 生成(R)-PED的最大 e.e.值(82.7%) 和最高得率(48.2%)[12]。目前,大部分报道的重组表达EHs,如A.niger ANEH[5],Sphingomonassp.HXN-200SpEH[13]和Agrobacterium radiobacter AD1[14]EchA等均具有较高的对映选择性,通过酶动力学拆分的制备(R)-或(S)-SO,其 e.e.值>99%,得率<50%,但(R)-或(S)-PED的e.e.值并不高。少部分EHs,如Caulobacte crescentus CcEH[15],Solanum tuberosum StEH[16]和 绿豆VrEH1[8]和VrEH2[9]等具有对映体归一性,其中CcEH 和 StEH 归一性水解(R,S)-SO 生成(R)-PED的e.e.值分别为90%和86%,均小于VrEH3获得(R)-PED 的 e.e.值(94.7%)。 该结果说明 VrEH3 不仅具有较好的对映选择性,且具有良好的对映体归一性(如图6),有着广阔的应用前景。
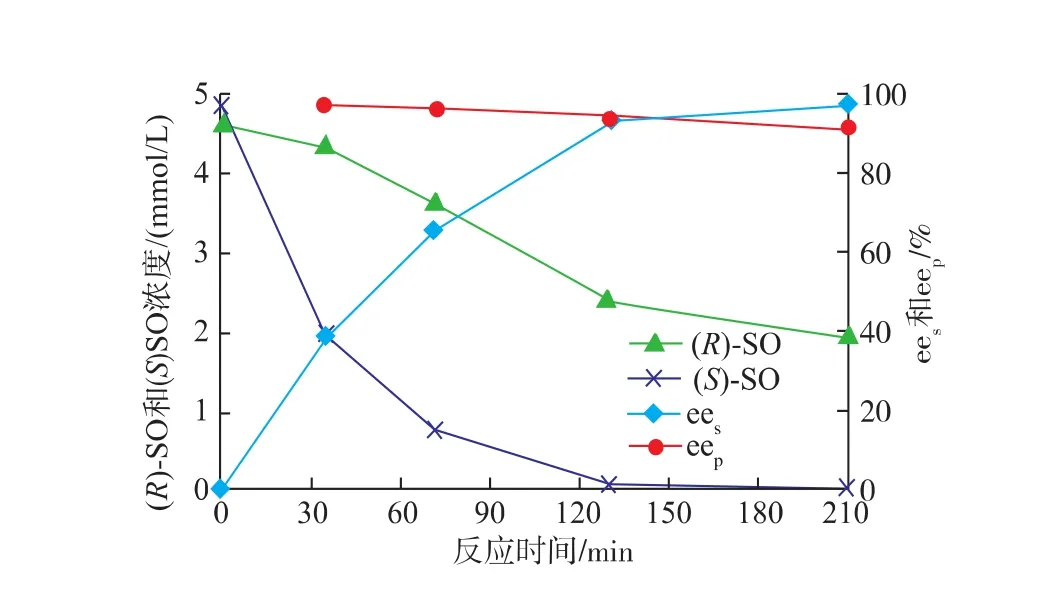
图 5 对映归一性水解(R,S)-SO的时间进程Fig.5 Time course of enantioconvergent hydrolysis of(R,S)-SO
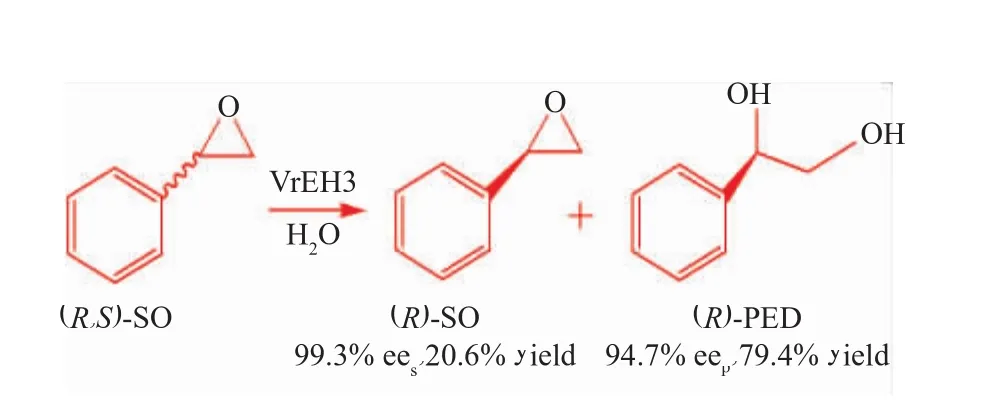
图6 VrEH3催化水解(R,S)-SO的反应Fig.6 Catalytic hydrolysis reaction of (R,S)-SO by VrEH3
3 结 语
本研究运用不同PCR技术,从绿豆中获得了一种新型环氧化物水解酶基因Vreh3,并成功实现了在E.coli中的异源表达。初步测定重组VrEH3活性,发现VrEH3对映归一性水解(R,S)-SO可同时获得高对映体纯的(R)-SO(e.e.值>99%) 和(R)-PED(e.e.值为94.7%)。VrEH3可作为一个良好的生物催化剂,对于制备高纯度的手性环氧化物或邻二醇具有巨大的潜在应用价值,其催化机理及应用需进一步研究。
参考文献:
[1]SOLARES L F,MATEO C.Improvement of the epoxide hydrolase properties for the enantioselective hydrolysis of epoxides[J].Current Organic Chemistry,2013,17(7):744-755.
[2]李从发.新型环氧化物水解酶筛选及其在手性合成中的应用[D].济南:山东大学,2003.
[3]PENG Huasong,ZONG Minhua,LOU Wenyong,et al.The application of microbial epoxide hydrolase in organic synthesis[J].Microbiology,2003,30(2):92-97.(in Chinese)
[4]XU W,XU J H,PAN J,et al.Enantioconvergent hydrolysis of styrene epoxides by newly discovered epoxide hydrolases in mung bean[J].Organic Letters,2006,8(8):1737-1740.
[5]KUMAR R,WANI S I,CHAUHAN N S,et al.Cloning and characterization of an epoxide hydrolase from Cupriavidus metallidurans-CH34[J].Protein Expression and Purification,2011,79(1):49-59.
[6]PEDRAGOSAMOREAU S,ARCHELAS A,FURSTOSS R.Microbiological transformations.28.Enantiocomplementary epoxide hydrolyzes as a preparative access to both enantiomers of styrene oxide.Journal of Organic Chemistry,1994,58(7):5533-5536.
[7]JU Xin,PAN Jiang,XU Jianhe.Enantioconvergent hydrolysis of p-nitrostyrene oxide catalyzed by Mung bean epoxide hydrolase[J].Chinese Journal of Catalysis,2008,29(8):696-700.(in Chinese)
[8]ZHU Q Q,HE W H,KONG X D,et al.Heterologous overexpression of Vigna radiata epoxide hydrolase in Escherichia coli and its catalytic performance in enantioconvergent hydrolysis of p-nitrostyrene oxide into (R)-p-nitrophenyl glycol[J].Applied Microbiology and Biotechnology,2014,98(1):207-218.
[9]YAN W W,XU D K,QING Q Z,et al.Chemoenzymatic enantioconvergent hydrolysis of p-nitrostyrene oxide into (R)-p-nitrophenyl glycol by a newly cloned epoxide hydrolase VrEH2 from Vigna radiata[J].Catalysis Communications,2015,58:16-20.
[10]ZHANG Xiaoxiang,WANG Ling,SHOU Lulu.A rapid modified CTAB method of extracting genomic DNA from wheat leaf[J].Chinese Agricultural Science Bulletin,2012,28(36):46-49.(in Chinese)
[11]HU Die,ZHU Lijuan,WU Minchen,et al.Cloning and bioinformatics analysis of epoxide hydrolase gene from Aspergillus usamii[J].Journal of Food Science and Biotechnology,2013,32(12):1244-1252.(in Chinese)
[12]CHEN W J,LOU W Y,ZONG M H.Efficient asymmetric hydrolysis of styrene oxide catalyzed by mung bean epoxide hydrolases in ionic liquid-based biphasic systems[J].Bioresource Technology,2012,115(115):58-62.
[13]WU S,LI A,CHIN Y S,et al.Enantioselective hydrolysis of racemic and meso-epoxide with recombinant Escherichia coli expressing epoxide hydrolase from Sphingomonas sp.HXN-200:preparation of epoxides and vicinal diols in high ee and high concentration[J].ACS Catalysis,2013,3(4):752-759.
[14]LOO B V,KINGMA J,HEYMAN G,et al.Improved enantioselective conversion of styrene epoxides and meso-epoxides through epoxide hydrolases with a mutated nucleophile-flanking residue[J].Enzyme and Microbial Technology,2009,44(3):145-153.
[15]HWANG S,CHOI C Y,LEE Y E.Enantioconvergent bioconversion of p-chlorostyrene oxide to (R)-p-chlorophenyl-1,2-ethandiol by the bacterial epoxide hydrolase of Caulobacter crescentus[J].Biotechnology Letters,2008,30(7):1219-1225.
[16]MONTERDE M I,LOMBARD M,ARCHELAS A,et al.Enzymatic transformations.Part 58:Enantioconvergent biohydrolysis of styrene oxide derivatives catalyzed by the Solanum tuberosum epoxide hydrolase[J].Tetrahedron:Asymmetry,2004,15(18):2801-2805.
