黄酒麦曲微生物总DNA提取方法比较
2018-04-20薛景波刘双平
薛景波 , 毛 健 *, 刘双平
(1.粮食发酵工艺与技术国家工程实验室,江南大学,江苏 无锡214122;2.江南大学 食品学院,江苏 无锡214122)
“以麦制曲,用曲酿酒”是中国黄酒的特色,黄酒麦曲是以轧碎的小麦为原料,经过加水拌曲、压块成型并堆放在一定的温度和湿度条件下,富集培养酿酒有益微生物制得的糖化发酵剂[1]。麦曲在黄酒制作中不仅提供各种酶系,同时还是黄酒酿制的主要微生物来源,这些微生物在黄酒制作过程中共同作用,最终形成了黄酒独特的风格,因此黄酒麦曲有着“酒之骨”的美誉[2-4]。
黄酒麦曲中微生物的研究一直是麦曲的研究重点,传统的分离培养不仅工作量大,耗时耗力,而且无法全面的了解微生物的组成,利用分子手段研究麦曲则能够避免传统方法的不便。利用分子手段分析一定环境下微生物群落结构时,基因组DNA提取的质量对后续的分析有着很大的影响[5],高质量的提取麦曲中的总DNA对全面的剖析麦曲微生物群落结构有着重要作用。
麦曲的制作在自然开放的环境中完成,且制曲时间较长(3个月左右),微生物来源广泛(制曲用水、小麦、空气微生物等)[6],造就了麦曲复杂的微生物体系。汪建国[7]在相关研究中将麦曲制作过程中微生物的生长变化分为孢子发芽期、生长繁殖期和产酶成熟期3个阶段,之后麦曲还要经过长时间的放置过程,随着水分含量的降低,麦曲中的大量霉菌、细菌产生成孢子、芽孢,而且小麦中丰富的蛋白质、淀粉等杂质易混入提取过程,这些都为基因组的提取带来困难。所以麦曲是一种含有真菌、细菌的营养体、孢子和芽孢的复杂体系,在进行群落结构解析时需要同时获得他们的基因组,然而目前少有专门针对黄酒麦曲中宏基因组提取法方法的报道,为此本研究通过综合比较现有方法优化出一种高质量提取麦曲中总DNA方法。
1 材料与方法
1.1 材料
1.1.1麦曲样品麦曲样品取自浙江古越龙山绍
兴酒股份有限公司,取样后分装于7℃保存。
1.2 麦曲总DNA提取及纯化方法
1.2.1麦曲样品预处理取5 g麦曲样品加15 mL ddH2O置于50 mL的离心管中,加入适量玻璃珠,充分振荡5 min;4℃条件于KQ700E超声波清洗器中超声振荡5 min;200g离心5 min,取上清液10000 g离心10 min;收集沉淀,加2 mL ddH2O混悬均匀转移至2 mL EP管;10 000 g离心10 min,得菌沉淀。1.2.2麦曲总DNA提取方法方法一:SDS法,参考Zhou等[8]报道方法,具体步骤如下:
菌沉淀加入0.5 mL DNA抽提液 (100 mmol/L Tris-HCl,pH 8.0,100 mmol/L EDTA,pH 8.0,100 mmol/L Na3PO4,1.5 mol/L NaCl)混悬,液氮条件下充分研磨菌体,之后加入 10 μL 溶菌酶(50 mg/mL),37℃条件下放置30 min;加入125 μL质量分数10%SDS,立即加入 5 μL 蛋白酶 K(20 mg/mL),混均后65℃水浴2 h(每隔10 min上下颠倒混均样品);6 000 g离心 10 min,取上清液。
方法二:氯化苄法,参照文献[9]进行操作。
方法三:CTAB法,参考文献[10]略有改动,具体具体方法如下:
菌沉淀加入0.5 mL DNA抽提液混悬,液氮条件下充分研磨菌体,之后加入700 μL CTAB提取缓冲液 (质量分数 2%CTAB,1.4 mol/L NaCl,1 mol/L Tris-HCl,0.5 mol/L EDTA),其余操作与方法一同。
方法四:超声波法,参考文献[11]进行操作。
方法五:Soil DNA kit提取法,菌体加入0.5 mL ddH2O混悬后,后续提取操作参照E.Z.N.A.Soil DNA Kit(美国OMEGA公司)说明书。
方法六:SDS高盐法,DNA抽提液中NaCl浓度提高为2.5 mol/L,其余操作与方法一同。
方法七:SDS-CTAB法,SDS法65℃水浴处理样品1 h后加入700 μL CTAB缓冲液,混均后再进行65℃水浴1 h,其余操作与方法一同。
1.2.3DNA纯化方法提取的DNA除了方法5以外,其他方法参考文献[12]对DNA进行纯化操作。
1.3 麦曲总DNA质量检测
1.3.1电泳检测用0.8 g/dL琼脂糖对所得麦曲总DNA进行电泳实验,跑胶完成后用伯乐DC XR+凝胶成像仪进行拍照观察。
1.3.2纯度检测不同方法处理得到的样品用紫外分光光度计于波长260、280 nm及230 nm处测定吸光值 A260、A280及 A230,计算比值 A260/A280及 A260/A230检测样品纯度。
1.3.3PCR检测将得到的麦曲总DNA进行TouchPCR扩增,对其中真菌的18S rDNA区进行扩增,对细菌的16S rDNA区进行扩增。真菌采用张霞等[13]在相关研究中所用通用引物,上游引物序列为NS1:5’-GTAGTCATATGCTTGTCTC-3’, 下游引物序 列 为 NS8:5’-TCCGCAGGTTCACCTACGCGA-3’,扩增片段大小为1 700 bp左右;细菌采用Delong[14]在相关研究中所用通用引物,上游引物序列 为 27f:5’-AGAGTTTGATCCTGGCTCAC-3’,下游 引 物 序 列 为 1492r:5’-TACGGCTACCTTGTTAC GACTT-3’,扩增片段大小为1 500 bp左右。
真菌 PCR 反应体系如下 (20 μL):Taq PCR master mix 10 μL,上下引物各 0.2 μL,模板 1 μL,补无菌水至20 μL。相应PCR反应条件为:95℃预变性 5 min;94℃变性 30 s,60℃退火 30 s,72℃延伸 2 min(Touch down PCR,10个循环,每个循环降低 1℃);94℃变性 30 s,58℃退火 30 s,72℃延伸2 min(20个循环);72℃终延伸7 min。细菌PCR反应体系如下(20 μL):Taq PCR master mix 10 μL,上下引物各 0.4 μL,模板 1 μL,补无菌水至 20 μL。相应PCR反应条件为:95℃预变性5 min;94℃变性30 s,57 ℃退火 30 s,72 ℃延伸 2 min (Touch down PCR,10个循环,每个循环降低1℃);94℃变性30 s,55 ℃退火 30 s,72 ℃延伸 2 min (20个循环);72℃终延伸7 min。PCR产物用0.8 g/dL琼脂糖凝胶电泳检测。
1.3.4Real-time PCR检测采用伯乐CFX Connect实时定量PCR仪对总DNA进行Real-time PCR扩增。为比较不同处理方法得到的总DNA样品中真菌和细菌相对模板数的大小,本实验以SDS法提取得到的总DNA样品作为参考制作稀释曲线,稀释倍数分别为 10、50、100、200、500、1 000,细菌Real-time PCR采 用 引 物 Eub338F:5’ -ACTCCTACGGGAGGCAGCAG-3’ 及 Eub518R:5’-ATTACCGCGGCTGCTGG-3’进行扩增[15],反应体系参考文献[15]进行操作;真菌Real-time PCR采用引物 ITS1F:5’ -CTTGGTCATTTAGAGGAAGTAA-3’及 ITS2:5’-GCTGCGTTCTTCATCGATGC-3’进行扩增[16],参反应体系考文献[16]进行操作。
2 结果与分析
2.1 不同提取方法电泳结果比较
7种不同方法提取麦曲总DNA样品以0.8 g/dL琼脂糖进行电泳得到的图谱如图1所示。不同方法得到的DNA片段唯一,片段较为完整,没有杂带,大小15 Kbp左右,但不同方法得到的条带在上样量相同的条件下亮度不同,其中SDS法、CTAB法、SDS高盐法及SDS-CTAB法得到的条带较亮且清晰。
2.2 不同提取方法纯度比较
DNA样品的纯净程度影响着后续PCR的结果,由表1可以看出,不同处理方法得到的DNA样品在纯度的得率上面有着较大的差别。Soil DNA kit提取法得到的总DNA样品的A260/A280及A260/A230值都偏小,DNA质量浓度较低,说明此种方法提取的总DNA有着蛋白、多糖及各种盐离子的污染,并且提取效果不佳[17];SDS法提取得到的总DNA样品质量浓度较高,但 A260/A280<1.7,A260/A230<2.0,说明存在着蛋白、多糖、盐离子等的污染;氯化苄法、超声波法及SDS高盐法提取得到的总DNA样品A260/A280值在1.8左右,A260/A230<2.0,说明样品中蛋白去除较为彻底,但存在着多糖等的污染;CTAB法及SDSCTAB法提取得到的总DNA样品A260/A280及A260/A230值相对较为合适,得到的DNA质量浓度较高。

图1 7种不同方法提取麦曲的总DNA凝胶电泳分析Fig.1 Electrophoresis analysis of total DNA isolated from Wheat Qu samples using 7 different methods

表1 7种不同方法提取的总DNA产量及纯度Table 1 Yield and purity of total DNA extracted by 7different methods
2.3 不同提取方法PCR结果比较
分别利用细菌引物 27f、1492r及真菌引物NS1、NS8对细菌16S全序列及真菌的18S全序列进行PCR扩增,扩增结果进行0.8%琼脂糖凝胶电泳得到谱图如图2、图3所示。

图2 7种不同方法提取的麦曲总DNA进行细菌PCR扩增电泳图Fig.2 Agarose gel ectrophoresis of PCR products of bacteria in samplesextracted by 7 different methods

图3 7种不同方法提取的麦曲总DNA进行真菌PCR扩增电泳图Fig.3 Agarose gel ectrophoresis of PCR products of fungi in samples extracted by 7 different methods
从图中可以看出,细菌和真菌相应片段扩增后电泳得到的条带单一,无杂带,说明各自所选通用引物合适,同时细菌扩增条带大小为1 500 bp左右,真菌扩增条带大小为1 700 bp左右,与相关文献相符[13-14]。7种方法得到的产物进行细菌PCR扩增后都能得到清晰明亮的条带,但进行真菌PCR得到的结果有着较大的差别,其中超声波法提取得到的样品在真菌PCR时没有得到相应条带,同时氯化苄法及Soil DNA kit法提取得到的样品进行真菌PCR得到的条带较弱。SDS法、CTAB法、SDS高盐法及SDS-CTAB法在细菌和真菌PCR结果中都有着亮而清晰的条带,说明这4种方法对真菌和细菌的提取效果相对于其他3中方法较好。
2.4 不同提取方法Real-time PCR结果比较
对SDS法提取的总DNA进行梯度稀释,稀释倍数分别为 10、50、100、200、500、1 000, 进行细菌和真菌Real-time PCR绘制熔解曲线和稀释曲线如图4、图5所示。
由图4、图5可知,以SDS提取的总DNA进行细菌和真菌Real-time PCR所用引物特异性较好,细菌Real-time PCR稀释曲线为Y=-3.337 9X+27.835,R2=0.999,扩增效率为99.3%;真菌Realtime PCR稀释曲线为 Y=-3.172 3X+30.804,R2=0.998,扩增效率为106.6%,均符合后续分析需求。
将不同提取方法所得样品稀释10倍后进行细菌和真菌Real-time PCR测定Ct值比较相对模板量如表2和表3所示。

图4 SDS法提取总DNA进行细菌Real-time PCR得到的熔解曲线和稀释曲线Fig.4 Melting curveanddilution curve constructed by bacteria Real-time PCR using DNA templates obtained by SDS method

图5 SDS法提取总DNA进行真菌Real-time PCR得到的熔解曲线Fig.5 Melting curve and dilution constructed by fungi Real-time PCR using DNA templates obtained by SDS method
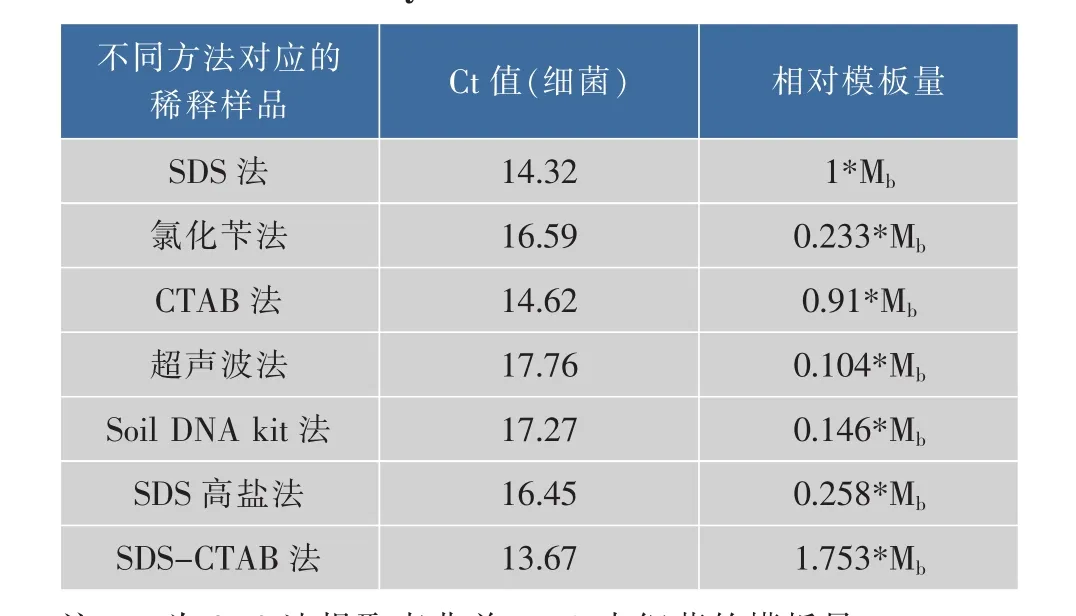
表2 不同提取方法所得样品中细菌相对模板量Table 2 Relative bacteria template amount in the samples extracted by 7 different methods

表3 不同提取方法所得样品中真菌相对模板量Table 3 Relative fungi template amount in the samples extracted by 7 different methods
从表2和表3可以看出,SDS法、CTAB法、SDS高盐法及SDS-CTAB法提取得到的总DNA中在细菌和真菌模板量上都处于较高水平,虽然超声波法提取的总DAN在细菌PCR时条带较亮且清晰,但进行实时定量PCR时得到细菌模板量最少,可能是PCR条件的不同及引物不同的原因,其中Soil DNA kit法提取得到的真菌模板量相对最少,这与真菌PCR的结果相符,综合不同样品真菌及细菌Realtime PCR结果,SDS-CTAB法提取得到的样品不论在真菌和细菌的模板数都相对最多,说明两种方法联用更能有效的提取麦曲中的总DNA。
3 讨 论
黄酒麦曲的制作在开放的自然环境中完成,微生物种类多且体系杂,作为黄酒制作的主要微生物来源,高质量的提取麦曲中微生物的总DNA对于分析麦曲微生物组成从而为提高黄酒品质奠定基础有着重要作用。
在提取DNA时,菌体细胞的裂解是提取的关键步骤,菌体裂解方法一般有以下3种:物理法、化学法、酶解法[18]。本文研究比较的7种方法对3种裂解方法都有涉及,其中溶菌酶是专门作用于微生物细胞壁的水解酶,对于破坏革兰氏阳性菌的细胞壁有着重要作用。SDS是一种亲水性的表面活性剂,能够溶解细胞膜上面的脂类和蛋白质,从而破坏细胞膜,并且能够解离细胞中的核蛋白,与其结合形成沉淀。蛋白酶K能够很好的水解与DNA结合的蛋白,在SDS的溶液里面更能够很好的发挥作用。CTAB是一种阳离子表面活性剂,在高离子强度的溶液中(>0.7 mol/L NaCl),CTAB与蛋白质和多聚糖形成复合物,只是不能沉淀核酸,通过有机溶剂抽提,去除蛋白质、多糖、酚类等杂质后加入乙醇沉淀即可使核酸分离出来[19]。
本文中研究了SDS法和超声波法比较了超声波破碎和液氮研磨对菌体细胞壁及真菌孢子壁的破碎效果,通过提取率、PCR验证及Real-time PCR分析说明超声波法对于细菌具有一定的提取效果,但对于真菌的提取效果并不显著,可能实验所用的超声波强度不能产生对真菌尤其是以孢子形式存在的真菌细胞壁很好的破壁效果,从而阻碍了后续总DNA的提取,且提取得到的样品有较为严重的多糖及其他小分子的污染。土壤与麦曲都是真菌、细菌混杂的微生物体系,有相关文献以Soil DNA kit对麦曲中总DNA进行提取[20],本实验对其提取效果进行比较发现Soil DNA kit对麦曲总DNA的提取效率较低,其提取纯度只有11.5 ng/μL,在7种提取方法中显著偏低,相对于超声波法,Soil DNA kit法提取对真菌和细菌都有一定的效果,但真菌的提取效率也处于较低水平。张中华等[21]以氯化苄法对麦曲进行了总DNA的提取,本实验参考其做法并对提取产物进行检测,纯度检测表明氯化苄法能够很好的除去提取中的蛋白质,但对多糖的清除效果一般,通过提取率检测、PCR验证及Real-time PCR分析表明氯化苄法对麦曲中真菌和细菌都有一定提取效果,这与文献报道相一致。SDS法对麦曲总DNA的提取有着较好的效果,说明SDS适用于各种较为复杂的体系,这与Zhou[8]的研究相符,但由于麦曲以轧碎的小麦为原料,小麦中的淀粉进入提取体系,在DNA提取过程中经过65℃水浴操作,淀粉糊化产生大量多糖干扰提取效果,使得SDS法提取的样品有着较多的多糖污染,说明SDS法对含有多糖的体系除去多糖效果较差。CTAB法对于含有多糖等杂质的提取环境有着很好的提取效果,本文中对CTAB法提取麦曲总DNA效果进行考察表明,相对于SDS法,CTAB法得到的样品澄清透亮,从纯度分析可知,CTAB法大幅度的减少了多糖对样品的污染,且蛋白质污染也较少,提取率较高,对于麦曲是一种较好的总DNA提取方法。相关文献指出高盐条件下提高了多糖在乙醇溶液中的溶解度,从而达到除去多糖的效果[22],本实验中比较了SDS法以及SDS高盐法提取效果,结果发现提高了提取体系的盐浓度后多糖的污染并没有减少,反而更加严重,这与文献结果不相同,但该方法总DNA提取率依然处于较高水平。SDS-CTAB法结合了SDS法和CTAB法的优势,结果表明,SDS-CTAB不仅增强了对样品的处理程度,使得到的总DNA在质量浓度和模板数都有显著提高,同时很大程度上减少了多糖对样品的污染,但是2种方法联合并没有单独用CTAB法更具去除多糖的效果,可能是由于65℃水浴1 h时更多的淀粉糊化溶解在体系形成更大量的多糖。通过对纯度检测、PCR验证以及Real-time PCR分析都表明,SDS-CTAB法对于麦曲体系的提取综合效果最好,是最为合适的提取方法。
4 结 语
本研究中通过对7种不同提取方法作用于麦曲效果进行比较表明,SDS-CTAB法联用能够对麦曲起到最佳的提取效果,最终得到总DNA样品中DNA质量浓度达到149.6 ng/μL,蛋白质、多糖等污染较少,PCR扩增后电泳得到条带单一明亮,Realtime PCR结果表明相对于SDS法,细菌和真菌模板数分别达到其2.343倍和1.753倍,是一种适合于黄酒麦曲总DNA提取的优良方法。
参考文献:
[1]周家骐.黄酒生产工艺[M].第2版.北京:中国轻工业出版社,1996:59-118.
[2]CAO Yu,LU Jian,FANG Hua,et al.Fungal diversity of Wheat Qu of Shaoxing Rice Wine[J].Food Science,2008,29(3):277-282.(in Chinese)
[3]陈亮亮.黄酒麦曲制曲工艺的优化研究[D].无锡:江南大学,2013.
[4]余培斌.改善绍兴黄酒麦曲品质的初步研究[D].无锡:江南大学,2013.
[5]ZHANG D,LI W,ZHANG S,et al.Evaluation of the impact of DNA extraction methods on BAC bacterial community compositionmeasuredbydenaturinggradientgelelectrophoresis[J].Lettersin Applied Micrology,2011,53(1):44-49.(inChinese)
[6]CAO Yu,CHEN Jianyao,XIE Guangfa,et al.Study on the factors of fungal community formation during the fermentation course of Wheat Qu[J].Journal of Food Science and Biotechnology,2008,27(5):95-101.(in Chinese)
[7]WANG Jianguo.Function of traditional koji in rice wine and its characteristics[J].Chinese Brewing,2004,(10):29-31.(in Chinese)
[8]ZHOU J Z,BRUNS M A,TIEDJE J M.DNA recovery from soils of diverse composition[J].Appliedand Environmental Microbiology,1996,62(2):316-322.
[9]ZHU Heng,QU Feng,ZHU Lihuang.Isolation of genomic DNAs from fungi using benzyl chloride[J].Acta Mycologica Sinica,1994,13(1):34-40.(in Chinese)
[10]SOARES S,AMARAL J S,OLIVEIRA M B P P,et al.Improving DNA isolation from honey for the botanical origin identification[J].Food Control,2015,48:130-136.
[11]LI Peng,BI Xuejun,RU Shaoguo.On the influence of DNA extraction from activated sludge for denaturing gradient gel electrophoresis on microbial diversity analysis[J].Journal of Safety and Environment,2007,7(2):53-57.(in Chinese)
[12]NI Zhengfei,XU Wei,DOU Wenfang,et al.Comparison of total microbial DNA extraction methods from solid-culture of Zhenjiang Vinegar[J].Acta Microbiologica Sicina,2010,50(1):119-125.(in Chinese)
[13]ZHANG Xia,WU Zhifang,ZHANG Shengchao,et al.Phylogenetic analysis of 18S rDNA sequence of mold from Luzhou-flavor Daqu[J].Chin J Appl Environ Biol,2011,17(3):334-337.(in Chinese)
[14]DELONG E F.Archaeain coastalmarine environments[J].Proceedingsofthe National Academyof Sciencesofthe United Statesof America,1992,89(12):5685-5689.
[15]GUO X,XIA X,TANG R,et al.Real-time PCR quantification of the predominant bacterial divisions in the distal gut of Meishan and Landrace pigs[J].Anaerobe,2008,14(4):224-228.
[16]NICOLAISEN M,JUSTESEN A F,KNORR K,et al.Fungal communities in wheat grain show significant co-existence patterns among species[J].Fungal Ecology,2014,11:145-153.
[17]CAO Nannan,PING Baohong,SI TU bo,et al.Detection and identification of aspergillus with panfungal primers and highresolution melting curve analysis[J].J Trop Med,2012,12(5):589-592.(in Chinese)
[18]倪峥飞.镇江香醋固态发酵过程中酿造微生物强化及醋醅总DNA提取方法的初步研究[D].无锡:江南大学,2009.
[19]YANG Mohua,LI Zhihui,ZHANG Donglin,et al.DNA isolation from pinus massoniana needles[J].Journal of Central South University of Forestry&Technology,2008,28(3):39-44.(in Chinese)
[20]YE Guangbin,LI Danyu,YANG Yuehaun,et al.Study on the succession law of fungal community during the fermentation and storage process of Luzhou-flavor Daqu by PCR-DGGE[J].Journal of Sichuan University of Science & Engineering(Natural Science Edition),2013,26(4):5-9.(in Chinese)
[21]张中华.绍兴黄酒麦曲中微生物群落结构的研究[D].无锡:江南大学,2012.
[22]WULFF E G,TORRES S,GONZALES VigilE.Protocol for DNA extraction from potato tubers[J].Plant Mol Bio Rep,2002,20:187.
