猪MYNN基因可变剪接体的克隆及表达特性研究
2018-04-04郭晓红高鹏飞曹果清成志敏张宁芳乐宝玉刘剑锋刘小军李步高
郭晓红,李 萌,高鹏飞,曹果清,成志敏,张宁芳,乐宝玉,刘剑锋,刘小军,李步高*
(1. 山西农业大学动物科技学院,太谷 030801; 2. 中国农业大学动物科技学院 农业部动物遗传育种与繁殖国家重点实验室,北京 100193; 3. 河南农业大学牧医工程学院,郑州 450002)
BTB(Broad complex, tramtrack, bricabrac)蛋白家族在转录的抑制和激活、染色质重塑以及细胞骨架的构建上发挥重要作用[1]。BTB蛋白既是转录激活剂,也是转录抑制剂[2-5],可通过在核小体上的旋转和重建来激活转录[6],还可通过招募转录辅阻遏物[7-9]抑制转录因子的活性,进一步抑制相关基因的转录活性[10-12]。锌指蛋白(Zinc finger protein, ZF)为常见的一类转录调控蛋白,主要通过与靶基因特异性序列结合,以及与自身或其他锌指蛋白的结合,来调节靶基因的表达,使之适应生物体发育、分化和成熟等不同阶段个体的需要[13]。研究发现,大约有5%~10%的C2H2型锌指蛋白的N端含有BTB结构域[14],这便形成一类新的转录因子-BTB/POZ和锌指蛋白 (BTB/POZ-ZF)家族。该家族通常与其他转录因子或者辅因子共同作用形成转录复合体从而激活或者抑制转录。Myoneurin(MYNN)是BTB/POZ-ZF家族的一员,其蛋白包括1个BTB/POZ结构域和8个串联重复的C2H2类型的锌指结构[15-16],在基因的表达调控上具有重要作用。
MYNN已在小鼠的胚胎细胞中被鉴定出来,并且发现与人的MYNN序列具有较高的同源性[17]。前人研究发现,MYNN在哺乳动物各个组织中均有表达[18-20]。通过分析MYNN的整个结构并检测其在人肌肉组织中的表达规律,发现MYNN可以在人的肌肉组织中调控某些基因的表达[16]。研究发现,若将神经切断或细胞核突触外的轴突切断,MYNN的表达会出现异常,说明MYNN是介导突触特异基因表达的候选基因[17]。另外还有研究认为,MYNN是潜在的促癌因子[21],因此,其生物学功能及与肿瘤的发生发展关系值得进一步剖析。
目前,国内外关于MYNN基因的研究主要集中在人和鼠上,关于猪MYNN的研究尚未见报道。本研究利用RT-PCR等技术对猪MYNN基因的全长CDS区进行克隆,并对其编码蛋白的结构与功能进行预测,采用实时荧光定量PCR(Quantitative real-time PCR,qPCR)技术对马身猪各组织表达谱及特定组织的发育性表达规律进行研究,从而为猪MYNN基因生物学功能的深入研究提供一定的理论依据。
1 材料与方法
1.1 试验动物和样品采集
本试验选取的试验动物为山西省地方猪种马身猪12头,由山西大同市种猪场提供。分别在1、90、180日龄3个时间点屠宰采样,每个时间点各4头(公、母各半,公猪断奶时去势),屠宰后,取其心、肝、脾、肺、肾、小脑、小肠、胰、胃、背最长肌及脂肪(无1日龄的脂肪组织样品)组织样品,迅速置于液氮中,后转至-80 ℃冰箱保存备用。
1.2 主要试剂
RNAiso Plus regent(TaKaRa, 大连);PrimeScriptTMRT reagent Kit with gDNA Eraser(perfect Real Time)(TaKaRa, 大连);2×TaqPCR MasterMix(天根生化科技有限公司,北京);Gel DNA Extraction Kit(TaKaRa, 大连); Trans-5α感受态细胞(全式金生物科技有限公司,北京); pMDTM19-T Vector(TaKaRa, 大连);SYBR Premix ExTaqⅡ (Tli RNaseH Plus)(TaKaRa,大连)。
1.3 试验方法
1.3.1总RNA提取和cDNA合成按照RNAiso Plus regent试剂盒说明书提取不同组织的总RNA,用ND-1000微量核酸蛋白测定仪及1%琼脂糖凝胶电泳测定总RNA的浓度和纯度。按照PrimeScriptTMRT reagent Kit with gDNA Eraser(perfect Real Time)反转录试剂盒操作说明反转录合成cDNA。合成cDNA采用两步法,第一步反应体系:gDNA Eraser 1 μL,gDNA Eraser buffer 2 μL,RNA 500 ng,加RNase-free水至10 μL。反应程序:42 ℃ 2 min;4 ℃ 5 min。第二步反应体系:5×primer 4 μL,mixⅠ1 μL,RTmix 1 μL,加RNase-free水至20 μL。反应条件:37 ℃ 15 min;85 ℃ 5 s;4 ℃ 5 min。合成的cDNA置于4 ℃冰箱保存备用。
1.3.2引物的设计与合成根据GenBank序列(登录号:NC_010455.5),用NCBI Primer-BLAST(https://blast.ncbi.nlm.ni,h.gov/Blast.cgi)和Oligo 7软件设计克隆MYNN基因全长CDS区的引物P1以及扩增MYNN两个转录本(MYNN-1和MYNN-2)的特异性引物P2和P3。所有引物由生工生物工程(上海)股份有限公司合成,引物位置如图1,各引物详细信息见表1。图1中两个F3是因为F3的位置为外显子5的末端及外显子7的前端。F3的序列前面部分与外显子5的3′末端相同,后半部分与外显子7的5′端的开始序列相同,所以标注两个F3,其实是一个,即P3的上游引物。
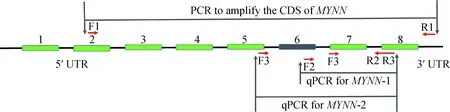
F. 上游引物;R. 下游引物F. Forward primer; R. Reverse primer图1 引物在猪MYNN基因上的位置信息Fig.1 Positional information of primers in pig MYNN
1.3.3目的基因的扩增及克隆以所有组织的cDNA混合物为模板,扩增猪MYNN基因全长CDS区,PCR反应体系为20 μL:cDNA pool 50 ng,上下游引物(P1)各0.5 μmol·L-1,2×TaqPCR MasterMix 10 μL,加 ddH2O至20 μL。反应程序:94 ℃预变性5 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸40 s,35个循环;72 ℃延伸2 min。反应产物用2%的琼脂糖凝胶电泳进行检测,用Gel DNA Extraction Kit对目的片段进行回收。
表1猪MYNN基因的PCR引物
Table1PCRprimersamplifyingpigMYNN

引物名称Primername引物序列(5'→3')Primersequence片段长度/bpProductlength注释NoteP1F1:AGAACAAGGGTAAAATTCGTTTGTGR1:TGCAGCATCAGGTGCTTTTA1928PCR扩增MYNN基因CDS区P2F2:CCTCAGGAGAGCTCAACAAACAR2:TGGACTCTTTTTCACTCAAGGGAT186qPCR扩增MYNN-1P3F3:CACTCATTCTCGAAAACATACAGGAR3:TGGACTCTTTTTCACTCAAGGGAT170qPCR扩增MYNN-218SrRNAF:CCCACGGAATCGAGAAAGAGR:TTGACGGAAGGGCACCA132qPCR扩增18SrRNA
将回收的目的片段与pMDTM19-T Vector 4 ℃过夜连接;采用热激法将连接产物转化至Trans-5α感受态细胞,用Amp抗性固体培养基进行蓝白斑筛选,37 ℃避光培养12 h;选取阳性白色单克隆菌在含Amp的LB液体培养基中扩大培养,之后对菌液进行PCR鉴定,筛选阳性菌液进行测序(北京六合华大科技股份有限公司)。
1.3.4生物信息学分析测序结果采用DNASTAR软件进行序列比对;运用NCBI上的ORF Finder程序对猪MYNN基因的开放性阅读框进行分析(https://www.ncbi.nlm.nih.gov/orffinder/);应用ProtParam软件对猪MYNN的理化性质进行预测(http://web.expasy.org/protparam/);采用NCBI在线软件进行蛋白保守结构域预测(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi);用SOPMA软件对猪MYNN蛋白质的二级结构进行预测(https://www.predictprotein.org/);用ProtFun 2.2软件对MYNN功能进行预测(http://www.cbs.dtu.dk/services/ProtFun/);用BLAST软件对猪、人、东北虎、马、羊驼、驴、家猫、家犬、北极熊、山羊的MYNN蛋白氨基酸序列进行同源性比较(https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastp&PAGE_TYPE=BlastSearch&LINK_LOC=blasthome);用MEGA4软件对各物种进行进化树的构建。
1.3.5MYNN基因的时空表达特性研究采用qPCR技术及引物P2和P3对马身猪各组织MYNN两个转录本的表达规律进行研究。qPCR反应体系为20 μL:2×SYBR Premix ExTaqII 10 μL,上、下游引物(10 μmol·L-1)各0.8 μL,50×ROX Reference Dye II 0.4 μL,cDNA 50 ng,RNAase Free ddH2O补至20 μL。参照SYBR Premix ExTaqⅡ (Tli RNaseH Plus)说明书确定反应程序:95 ℃预变性30 s;95 ℃变性20 s,64 ℃退火延伸34 s,45个循环;95 ℃ 15 s,60 ℃ 1 min,95 ℃ 30 s。每个样本重复3次,反应产物用1%的琼脂糖凝胶电泳进行检测。
1.3.6数据处理采用2-ΔΔCt法对相对定量结果进行处理,结果用“平均值±标准误”表示[22]。利用SPSS 22.0软件单因素方差分析对MYNN基因的表达差异显著性进行判断,P<0.05表示差异显著,P<0.01表示差异极显著,P>0.05表示差异不显著。
2 结 果
2.1 猪MYNN基因可变剪接体的克隆及生物信息学分析
用1%的琼脂糖凝胶电泳检测提取的总RNA,发现总RNA存在28S、18S、5S完整的3条带(图略),且A260 nm/A280 nm的比值为1.8~2.0,表明其RNA完整性好,且无蛋白质与DNA污染,测得的浓度适宜,可进一步用于RT-PCR分析。
2.1.1猪MYNN基因可变剪接体的克隆及序列分析引物P1的扩增结果如图2所示,产物为清晰的两条带,大小分别为2 012和1 928 bp,推测其为MYNN的两个不同的转录本。经克隆测序得到猪MYNN基因的全长CDS区及部分非编码区。
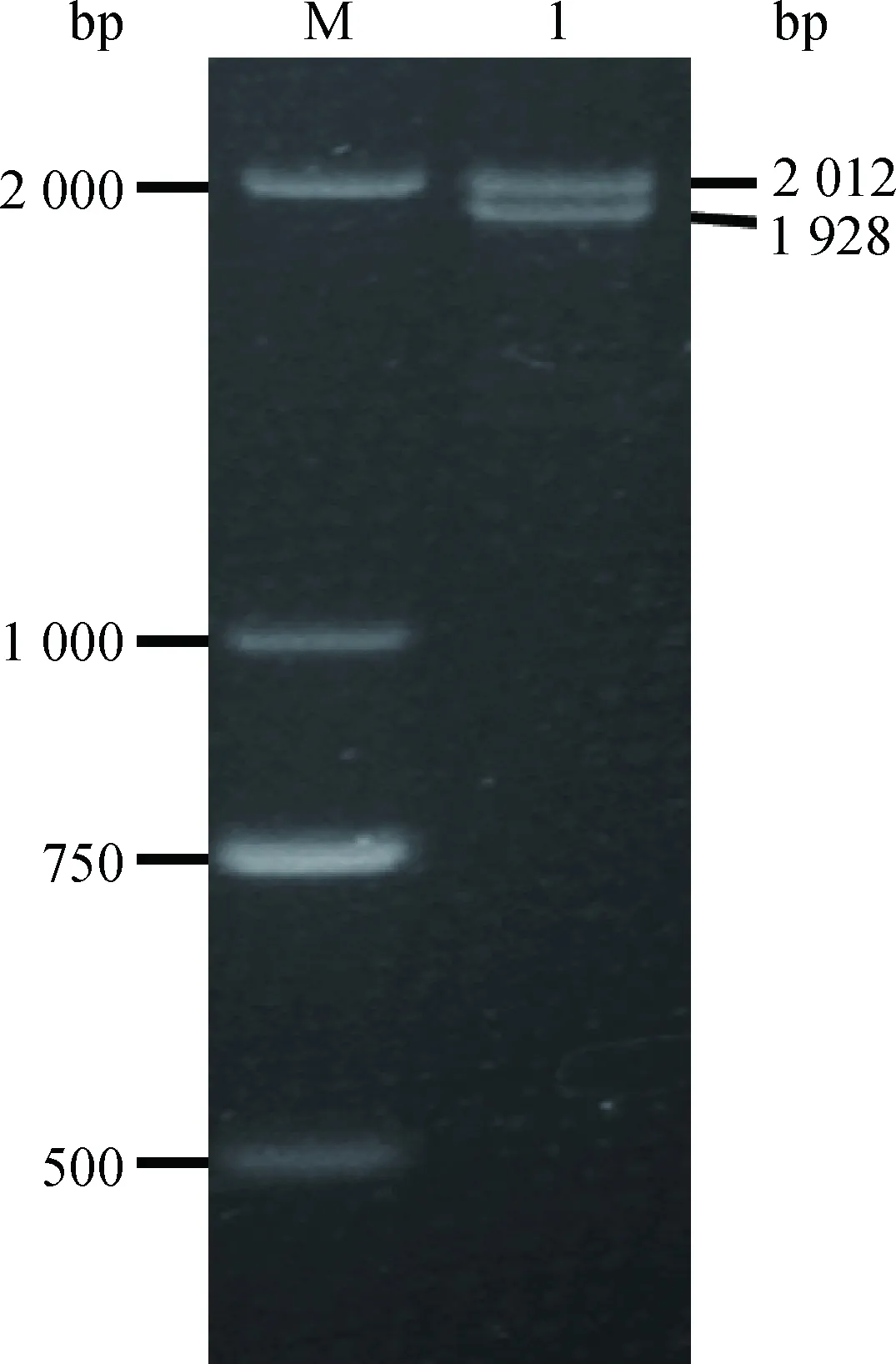
M. DNA 相对分子质量标准;1. 引物P1的扩增产物M. DL2000 DNA marker; 1. Amplicons of P1图2 引物P1扩增产物的电泳图Fig.2 Agarose gel electrophoresis of RT-PCR amplicons of P1
经过序列比对分析发现,MYNN基因在猪上存在两个转录本,分别命名为MYNN-1和MYNN-2。运用NCBI上的ORF Finder对猪MYNN基因序列分析发现,MYNN-1含有一个长1 830 bp的ORF,其起始密码子为ATG,位于34 bp处,终止密码子为TGA,位于1 863 bp处;MYNN-2含有一个长1 746 bp的ORF,其起始密码子为ATG,位于34 bp处,终止密码子为TGA,位于1 779 bp处。由此可得出,MYNN-1的CDS区全长1 830 bp,编码609个氨基酸;MYNN-2的CDS区全长1 746 bp,编码581个氨基酸。与MYNN-1相比,MYNN-2少了84 bp,缺失第6外显子,并且发现第6外显子的剪切符合GT-AG法则,可变剪切的类型为外显子跳跃(图3)。将两个转录本的全长CDS区序列及部分UTR序列提交至GenBank:MYNN-1的登录号: KY470829,MYNN-2的登录号: KY670835。

“∣”表示相同的核苷酸;“-”表示缺失的核苷酸“∣” indicate the same nucleotides; “-” indicate the deleted nucleotides图3 MYNN-1和MYNN-2的部分核苷酸序列Fig.3 Partial nucleotide sequences of MYNN-1 and MYNN-2
2.1.2猪MYNN理化性质分析用ProtParam软件预测了猪MYNN的理化性质。MYNN-1由609个氨基酸组成,其分子式为C3009H4787N859O928S32,分子量为68 871.23 u,理论等电点PI为8.77,说明MYNN-1为碱性氨基酸;Ser(9.5%)、Lys(9.4%)和Leu(8.7%)出现的频率较高;在氨基酸组成中,极性氨基酸占33.83%,疏水性氨基酸占27.59%,带电荷氨基酸占24.63%,其中包括13.46%的酸性氨基酸和11.17%的碱性氨基酸;消光系数(mol·L-1·cm-1γ=280 nm)为35 310,预测其在哺乳动物红细胞的半衰期为30 h,其不稳定系数为39.76,因此属于稳定蛋白质;疏水性指数为72.18,平均亲水性为-0.603,属于可溶性蛋白质。MYNN-2由581个氨基酸组成,其分子式为C2873H4578N820O889S30,分子量为65 792.71 u,理论等电点PI为8.71,说明MYNN-2为碱性氨基酸;Ser(9.3%)、Lys(9.3%)和Leu(9.0%) 出现的频率较高,在氨基酸组成中,极性氨基酸占33.91%,疏水性氨基酸占27.88%,带电荷氨基酸占24.78%,其中包括13.42%的酸性氨基酸和11.36%的碱性氨基酸;消光系数(mol·L-1·cm-1γ=280 nm)为35 310,预测其在哺乳动物红细胞的半衰期为30 h,其不稳定系数为40.83,因此属于不稳定蛋白质;疏水性指数为72.98,平均亲水性为-0.609,属于可溶性蛋白质。
2.1.3猪MYNN保守结构域的预测利用NCBI在线软件进行蛋白保守结构域预测:MYNN-1的N端含有一个BTB结构域,C端含有8个串联重复C2H2类型的锌指结构(图4,左侧为N端BTB结构域,右侧C端为锌指蛋白结构域)。MYNN-2的N端同样含有一个BTB结构域,不同的是,其C端相对MYNN-1少了一个锌指蛋白结构域(图4)。
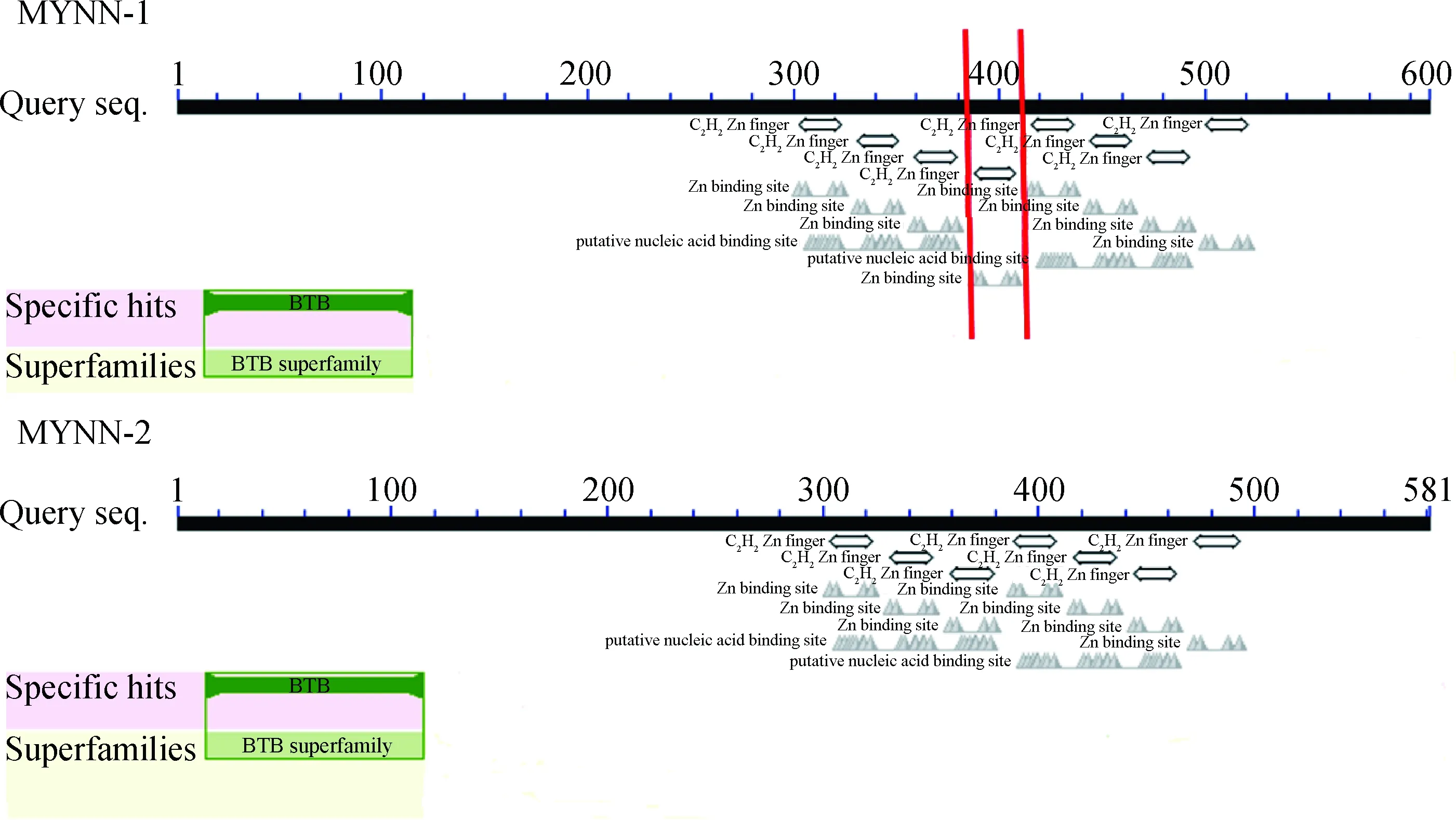
双竖线标出的是MYNN-1与MYNN-2结构域预测不同的地方Double vertical line indicate the difference of predicted domains between MYNN-1 and MYNN-2图4 猪MYNN蛋白结构与分析Fig.4 Conservative domains analysis of pig MYNN protein
2.1.4猪MYNN二级结构预测用SOPMA软件对猪MYNN蛋白质的二级结构进行预测,结果显示, MYNN-1的二级结构主要由α-螺旋(36.32%)、延伸(19.97%)、β-折叠(8.78%)和无规则卷曲(34.94%)组成,MYNN-2的二级结构中α-螺旋、延伸、β-折叠及无规则卷曲的比例分别为35.63%、20.53%、10.02%及33.83%。
2.1.5猪MYNN功能预测用ProtFun 2.2软件对MYNN功能进行了预测,结果表明,该蛋白在运输和结合、嘌呤和嘧啶、辅因子及翻译的生物合成中发挥功能的可能性分别为0.773、0.331、0.210及0.071,作为电压激活离子通道、转录因子、信号转导分子、离子通道、阳离子通道及转录调控因子发挥作用的可能性分别为0.279、0.219、0.205、0.169、0.146、及0.111(表2)。由此推测,MYNN可能在运输和结合、嘌呤和嘧啶、辅因子及翻译的生物合成中发挥转录调控及信号转导等重要作用。
2.1.6MYNN氨基酸序列进化树的构建用BLAST与MEGA4软件对猪、东北虎、马、山羊、羊驼、驴、家猫、家犬、北极熊、人的氨基酸序列进行同源性的比较以及进化树的构建(图5)。结果表明,猪MYNN的氨基端序列与北极熊、山羊、马、家犬等在系统进化树中的遗传距离较近,且同源性在95%以上。MYNN氨基酸序列进化树与生物进化物种树基本一致,符合物种进化的规律,说明MYNN基因的编码区在物种间具有很高的保守性。
表2猪MYNN功能分析
Table2AnalysisofMYNNfunction

功能分类Functionalcategory概率Probability基因本体分类GeneOntologycategory概率Probability氨基酸生物合成Aminoacidbiosynthesis0.011信号转导分子Signaltransducer0.205辅因子的生物合成Biosynthesisofcofactors0.210受体Receptor0.007细胞被膜Cellenvelope0.033转运蛋白Transporter0.025细胞加工Cellularprocesses0.030离子通道Ionchannel0.169中央中间代谢Centralintermediarymetabo-lism0.048电压激活离子通道Voltage-gatedionchannel0.279能量代谢Energymetabolism0.035阳离子通道Cationchannel0.146脂肪酸代谢Fattyacidmetabolism0.017转录因子Transcriptionfactor0.219嘌呤和嘧啶Purinesandpyrimidines0.331转录调控因子Transcriptionregulationfactor0.111监管职能Regulatoryfunctions0.034应激反应Stressresponse0.073复制和转录Replicationandtranscription0.020免疫反应Immuneresponse0.011翻译Translation 0.071生长因子Growthfactor0.005运输和结合Transportandbinding0.773金属离子转运Metaliontransport0.018

图5 MYNN 氨基酸序列的系统进化树Fig.5 Phylogenetic tree of amino acid sequences of MYNN
2.2 猪MYNN基因的时空表达特性研究
2.2.1MYNN基因各转录本在猪不同组织的表达谱分析由图6可以看出,MYNN-1和MYNN-2在马身猪各组织中均有表达,且组织间表达差异显著(P<0.05);MYNN-1和MYNN-2在马身猪的胃中表达量最高,其次为胰、小肠、肺、小脑、肝、肾,在脂肪组织中的表达量最低;相同组织中,除了肾组织,MYNN-1的表达量均显著或极显著高于MYNN-2(P<0.05或P<0.01)。
2.2.2MYNN基因各转录本在猪胃和背最长肌中的发育性表达特征分析MYNN-1和MYNN-2在马身猪胃组织的表达量随着日龄的增加呈下降趋势(图7);在背最长肌中的表达量随着日龄的增加呈先上升后下降的趋势(图8);在胃和背最长肌几乎所有时期中,MYNN-1的表达量均极显著高于MYNN-2(P<0.01)(图7、图8)。

*、**分别表示同一组织不同转录本间表达差异显著或极显著(P<0.05,P<0.01)。不同字母表示同一转录本在不同组织间表达差异显著(P<0.05),下图同*, ** indicate the significant or extremely significant expression difference in the same tissue between the two transcripts(P<0.05, P<0.01). The different letters indicate the significant difference of the same transcript expression among different tissues(P<0.05). The same as below图6 MYNN-1和MYNN-2在马身猪各组织中的表达Fig.6 The relative expression of MYNN-1 and MYNN-2 in different tissues of Mashen pig

图7 MYNN-1和MYNN-2 mRNA在胃组织的表达Fig.7 The relative expression of MYNN-1 and MYNN-2 mRNA in stomach

图8 MYNN-1和MYNN-2 mRNA在背最长肌组织的表达Fig.8 The relative expression of MYNN-1 and MYNN-2 mRNA in longissimus dorsi
3 讨 论
可变剪接是由pre-mRNA通过不同的剪接方式产生的,最终会导致蛋白质结构和功能的多样性[23]。可变剪接在机体中广泛存在,并且与蛋白质功能的改变密切相关[24]。同一基因的各个转录本的表达程度大小不同,表达量最高的被称为主要的亚型形式,而那些相对表达少的转录本被称为次要亚型[25]。本试验成功克隆出猪MYNN基因的两个转录本,MYNN-1的CDS区全长1 830 bp,编码609个氨基酸,MYNN-2的CDS区全长1 746 bp,编码581个氨基酸。在马身猪90日龄的各个组织,以及胃和背最长肌几乎所有的时期,MYNN-1 mRNA的表达量显著或极显著高于MYNN-2(P<0.05,P<0.01),因此,在猪上MYNN-1是主要的亚型。
TF IIIA是典型的C2H2类型的锌指蛋白,其虽含有9个锌指结构,但并非每个锌指都可以与DNA结合,它主要依靠锌指1~3与DNA结合。锌指7~9与DNA结合的能力显著低于锌指1~3[26-27],且锌指4~6不能与DNA结合,它会形成一个开放性的延展结构,来促进锌指1~3、7~9与DNA的结合[28-29]。功能结构域预测发现,MYNN-1比MYNN-2多了一个C2H2类型的锌指结构,该锌指结构有Zn的结合位点,但不能结合核酸。MYNN-1比MYNN-2多出来的锌指结构虽然不能结合DNA,但有可能会促进其他锌指结构与DNA的结合,使MYNN-1与DNA结合的能力更强,从而发挥主要作用。
前人研究发现,蛋白质的半衰期和其稳定性有密切关系,半衰期长的蛋白质一般具有较高的稳定性[30]。本研究发现,猪MYNN-1与MYNN-2同样具有较长的半衰期,但前者属于稳定蛋白,后者却为不稳定蛋白质,推测该特性与其特定的功能有关。通过对猪MYNN功能的预测,其可能在运输和结合、嘌呤和嘧啶及辅因子的生物合成中发挥转录调控及信号转导等重要作用。对猪MYNN基因序列与氨基酸序列的比对分析发现,猪MYNN基因的长度与其所编码氨基酸数和其他物种基本一致,与个别物种略有差异。分析与其他物种的同源性发现,猪与人、羊驼、家犬等9个物种的亲缘关系较近,其同源性为95%~100%。该结果说明MYNN基因的编码区在生物进化过程中具有很强的保守性。
基因表达的差异与生物学功能密切相关,所以研究MYNN基因的表达规律对于探究其功能是十分必要的。前人研究发现,MYNN在哺乳动物各个组织中均有表达[18-20]。P.M.Alliel等[15]通过Northern blot分析发现,MYNN在人的各组织中均有表达,且肌肉组织中的表达量显著高于睾丸、卵巢及胚胎等其他组织。据报道,MYNN在小鼠的小脑、骨骼、肌肉、睾丸、心、脑及肝中都有表达[31]。本研究发现,MYNN-1和MYNN-2在马身猪的心、肝、脾、肺、肾、小脑、小肠、胃、胰、肌肉和脂肪中都有表达,且各组织间表达差异显著(P<0.05),与前人的研究结果基本一致。MYNN基因的这种广谱型表达特征与其作为转录因子来调节基因转录的功能相符。进一步分析发现,MYNN-1和MYNN-2在马身猪的胃、胰、小肠中高表达,胃、胰和小肠是主要的消化器官,据此推测,MYNN与马身猪的消化吸收作用密切相关。
由此,本研究进一步对MYNN-1与MYNN-2在马身猪胃组织中的发育性表达规律进行研究,结果显示,随着日龄的增加,MYNN-1与MYNN-2的表达量均逐渐降低。仔猪出生时的胃肠道发育不完全,其重量和容积均相对较小,且消化酶的分泌量不足,消化吸收功能也不完善,例如胃蛋白酶在仔猪出生时是以酶原状态存在的,其为不可消化蛋白质,特别是植物性蛋白。初生1 d仔猪的胃重仅为4~8 g,只能容纳40 mL左右的乳汁。仔猪胃肠道在整个哺乳期会快速生长发育,胃蛋白酶在35~40日龄便开始具有消化功能,75日龄时消化机能逐渐完善,180日龄时,胃肠道基本不再发育。由此推测,MYNN基因可能通过调控某些基因的表达来促进胃肠道的发育。
研究推测MYNN基因在肌肉发育中有重要作用。因此,为了更深入地研究MYNN在肌肉中的功能,本试验分析了MYNN基因在马身猪背最长肌中的发育性表达规律,结果发现,随着日龄的增加,MYNN-1与MYNN-2的表达量呈现先上升后下降的趋势。骨骼肌的生长发育受到一系列生肌决定因子在时间和空间上的精密调控。本课题组前期研究的MyoD及MyoG基因均属生肌调控因子家族,该家族基因参与肌纤维的形成,是影响肌肉生长和肉品质的候选基因[31]。在初生1~180日龄,MyoD呈现先上升后下降的趋势,MyoG的表达量在1和180日龄均显著低于90日龄,90日龄时达到最高值[31]。MYNN-1与MYNN-2的发育性表达规律与这两个生肌决定因子基本相同,这说明MYNN-1与MYNN-2很可能在骨骼肌的生长发育调节过程中起到一定的作用,但其具体机制还需要进一步的研究。
4 结 论
本试验获得了猪MYNN基因的两个新转录本,分别命名为MYNN-1(GenBank登录号:KY470829)和MYNN-2(GenBank登录号:KY670835),MYNN-1的CDS区全长1 830 bp,编码609个氨基酸,MYNN-2的CDS区全长1 746 bp,编码581个氨基酸。与MYNN-1 相比,MYNN-2少84 bp,缺失第6外显子,且MYNN-2比MYNN-1少一个C2H2类型的锌指蛋白结构域。分析马身猪MYNN-1和MYNN-2在各组织表达谱及在胃和背最长肌组织中发育性表达规律发现,MYNN-1是主要的亚型,且MYNN在马身猪的消化吸收及骨骼肌的生长发育过程中具有重要作用。
参考文献(References):
[1]ARAVIND L, KOONIN E V. Fold prediction and evolutionary analysis of the POZ domain: structural and evolutionary relationship with the potassium channel tetramerization domain[J].JMolBiol, 1999, 285(4): 1353-1361.
[2]LI X M, PENG H Z, SCHULTZ D C, et al. Structure-function studies of the BTB/POZ transcriptional repression domain from the promyelocytic leukemia zinc finger oncoprotein[J].CancerRes, 1999, 59(20): 5275-5282.
[3]TAKENAGA M, HATANO M, TAKAMORI M, et al. Bcl6-dependent transcriptional repression by BAZF[J].BiochemBiophysResCommon, 2003, 303(2): 600-608.
[4]PHAN R T, DALLA-FAVERA R. The BCL6 proto-oncogene suppresses p53 expression in germinal-centre B cells[J].Nature, 2004, 432(7017): 635-639.
[5]KELLY K F, OTCHERE A A, GRAHAM M, et al. Nuclear import of the BTB/POZ transcriptional regulator Kaiso[J].JCellSci, 2004, 117(25): 6143-6152.
[6]HUYNH K D, BARDWELL V J. The BCL-6 POZ domain and other POZ domains interact with the co-repressors N-CoR and SMRT[J].Oncogene, 1998, 17(19): 2473-2484.
[7]DAVID G, ALLAND L, HONG S H, et al. Histone deacetylase associated with mSin3A mediates repression by the acute promyelocytic leukemia-associated PLZF protein[J].Oncogene, 1998, 16(19): 2549-2556.
[8]MELNICK A, CARLILE G, AHMAD K F, et al. Critical residues within the BTB domain of PLZF and Bcl-6 modulate interaction with corepressors[J].MolCellBiol, 2002, 22(6): 1804-1818.
[9]AHMAD K F, MELNICK A, LAX S, et al. Mechanism of SMRT corepressor recruitment by the BCL6 BTB domain[J].MolCell, 2003, 12(6): 1551-1564.
[10]LI X C, ZHANG D, HANNINK M, et al. Crystal structure of the Kelch domain of human Keap1[J].JBiolChem, 2004, 279(52): 54750-54758.
[11]LEDEE D R, GAO C Y, SETH R, et al. A specific interaction between muskelin and the cyclin-dependent kinase 5 activator p39 promotes peripheral localization of muskelin[J].JBiolChem, 2005, 280(22): 21376-21383.
[12]PRAG S, COLLETT G D M, ADAMS J C. Molecular analysis of muskelin identifies a conserved discoidin-like domain that contributes to protein self-association[J].BiochemJ, 2004, 381(2): 547-559.
[13]周畅, 李麓芸. C2H2型锌指蛋白的研究进展[J]. 生命科学研究, 2004, 8(3): 215-220.
ZHOU C, LI L Y. Advances in C2H2zinc finger proteins[J].LifeScienceResearch, 2004, 8(3): 215-220. (in Chinese)
[14]VENTER J C, ADAMS M D, MYERS E W, et al. The sequence of the human genome[J].Science, 2001, 291(5507): 1304-1351.
[15]ALLIEL P M, SEDDIQI N, GOUDOU D, et al. Myoneurin, a novel member of the BTB/POZ-Zinc Finger family highly expressed in human muscle[J].BiochemBiophysResCommon, 2000, 273(1): 385-391.
[16]CIFUENTES-DIAZ C, BITOUN M, GOUDOU D, et al. Neuromuscular expression of the BTB/POZ and zinc finger protein myoneurin[J].MuscleNerve, 2004, 29(1): 59-65.
[18]MITCHELMORE C, KJAERULFF K M, PEDERSEN H C, et al. Characterization of two novel nuclear BTB/POZ domain zinc finger isoforms. Association with differentiation of hippocampal neurons, cerebellar granule cells, and macroglia[J].JBiolChem, 2002, 277(9): 7598-7609.
[19]SAKASHITA C, FUKUDA T, OKABE S, et al. Cloning and characterization of the humanBAZFgene, a homologue of theBCL6 oncogene[J].BiochemBiophysResCommon, 2002, 291(3): 567-573.
[20]TRAPPE R, BUDDENBERG P, UEDELHOVEN J, et al. The murine BTB/POZ zinc finger geneZnf131: predominant expression in the developing central nervous system, in adult brain, testis, and thymus[J].BiochemBiophysResCommon, 2002, 296(2): 319-327.
[21]孟莹莹, 许兴智, 廖蓟. 转录因子ZBTB家族与肿瘤[J]. 生命科学, 2015, 27(4): 477-485.
MENG Y Y, XU X Z, LIAO J. ZBTB transcription factors and tumorigenesis[J].ChineseBulletionofLifeSciences, 2015, 27(4): 477-485. (in Chinese)
[22]RAMAKERS C, RUIJTER J M, DEPREZ R H L, et al. Assumption-free analysis of quantitative real-time polymerase chain reaction (PCR) data[J].NeurosciLett, 2003, 339(1): 62-66.
[23]张天,李祥龙,周荣艳,等. 不同毛色山羊皮肤组织Agouti基因剪接体类型研究[J]. 畜牧兽医学报, 2015, 46(11): 1934-1943.
ZHANG T,LI X L,ZHOU R Y,et al. Study onAgoutispliceosome types in goat skin with different coat color[J].ActaVeterinariaetZootechnicaSinica, 2015, 46(11): 1934-1943.(in Chinese)
[24]MODREK B, LEE C. A genomic view of alternative splicing[J].NatGenet, 2002, 30(1): 13-19.
[25]FENG H J, QIN Z Y, ZHANG X G. Opportunities and methods for studying alternative splicing in cancer with RNA-Seq[J].CancerLett, 2013, 340(2): 179-191.
[26]LIAO X B, CLEMENS K R, TENNANT L, et al. Specific interaction of the first three zinc fingers of TFIIIA with the internal control region of theXenopus5 S RNA gene[J].JMolBiol, 1992, 223(4): 857-871.
[27]BRAYER K J, SEGAL D J. Keep your fingers off my DNA: protein-protein interactions mediated by C2H2zinc finger domains[J].CellBiochemBiophys, 2008, 50(3): 111-131.
[28]NOLTE R T, CONLIN R M, HARRISON S C, et al. Differing roles for zinc fingers in DNA recognition: structure of a six-finger transcription factor IIIA complex[J].ProcNatlAcadSciUSA, 1998, 95(6): 2938-2943.
[29]NEELY L, TRAUGER J W, BAIRD E E, et al. Importance of minor groove binding zinc fingers within the transcription factor IIIA-DNA complex[J].JMolBiol, 1997, 274(4): 439-445.
[30]贾浩, 张小白, 宋晓峰. 人类胞内蛋白半衰期与其亚细胞定位的相关性研究[J]. 计算机与应用化学, 2011, 28(4): 411-414.
JIA H, ZHANG X B, SONG X F. Relationship between intracellular protein half-life and subcellular localization in human cells[J].ComputersandAppliedChemistry, 2011, 28(4): 411-414. (in Chinese)
[31]牛姣艳.Pax7、MyoD和MyoG基因在猪背最长肌中的发育性表达研究[D]. 太谷: 山西农业大学, 2015.
NIU J Y. Study on the developmental expression ofPax7,MyoDandMyoGgenes inlongissimusdorsiof pigs[D]. Taigu: Shanxi Agricultural University, 2015. (in Chinese)
