circRNA研究进展及其法医学应用展望
2018-03-30涂春艳金恺迪邵诚臣刘宝年张雅琪谢建辉沈忆文
涂春艳,金恺迪,邵诚臣,刘宝年,张雅琪,谢建辉,沈忆文
(复旦大学基础医学院法医系,上海 200032)
环状 RNA(circular RNA,circRNA)[1]呈环状,是一类新发现的特殊RNA分子,由前体RNA转录后反向剪接产生,参与基因的转录和转录后调控,在生物体内发挥着重要的作用。二代测序及分析技术的快速发展,使我们认识到circRNA在生物体内广泛表达,可能发挥着重要的功能。circRNA稳定,具有组织表达特异性,是分子生物学领域新的研究热点。近年来,circRNA作为分子生物标记用于临床疾病机制研究以及疾病诊断的研究报道较多,现对circRNA作为生物标记用于临床研究的现状进行综述,并对其帮助解决某些法医学难题的应用进行展望,希望为circRNA在法医学领域的应用提供参考。
1 circRNA的一般情况
1.1 circRNA的发现历程
circRNA最初发现于植物病毒体内,随后相继在肝炎病毒及其他动物病毒中发现[2],因而曾经被认为是病毒中特有的RNA[3]。1979年,HSU等[4]用电子显微镜在真核细胞中发现了circRNA;1993年,CAPEL等[5]在小鼠精子决定基因SRY中发现环状转录;2012年,SALZMAN等[6]通过RNA-Seq方法首次报道了人大量基因表达circRNA。至此,高通量测序技术证实了circRNA表达。随后,大量的circRNA分子被相继发现,如JECK等[7]在人类成纤维细胞中检测出了高达 25 000多种的 circRNA;而 MEMCZAK等[8]通过RNA-seq测序结合人白细胞数据库鉴定出1 950种人类circRNA、1903种小鼠circRNA(其中81种与人类circRNA相同)和724种线虫circRNA。此后,关于circRNA的研究报道不断丰富了人们对于circRNA的认识[9-13]。
1.2 circRNA的形成机制、生物学特性和功能
circRNA呈闭合环状;大部分属于非编码RNA,主要由外显子衍生而来,且由一个或多个外显子反向剪接形成(图1A);同一基因位点可通过选择性环化产生多种circRNA(图1B);环化过程与mRNA剪接过程彼此竞争;缺少5′末端和3′polyA尾。因此,circRNA不易被RNA核酸外切酶等核酸酶降解,具有相对稳定性[14]。circRNA广泛存在于人体细胞中,JECK等[7]通过高通量测序在人的成纤维细胞中发现的两千多种circRNA中,部分circRNA的表达量是线性mRNA的10倍。circRNA相对保守,在古生菌与真核细胞中均有表达。此外,EBERT等[15]的研究表明:部分circRNA分子含miRNA应答元件,可充当竞争性内源RNA,与miRNA结合,在细胞中起到miRNA海绵的作用,进而解除miRNA对靶基因的抑制作用,上调靶基因的表达水平,从而帮助阐明某些疾病的发病机制;circRNA有较高的组织表达特异性[6],在不同组织器官中差异表达,可以根据circRNA表达变化帮助诊断相应的组织器官疾病。然而,由于传统分子检测技术的局限性[16],不能探查到circRNA分子。新一代测序技术和分子生物学的发展使circRNA重新走进科研工作者的视野,并且成为分子生物标记的研究热点。
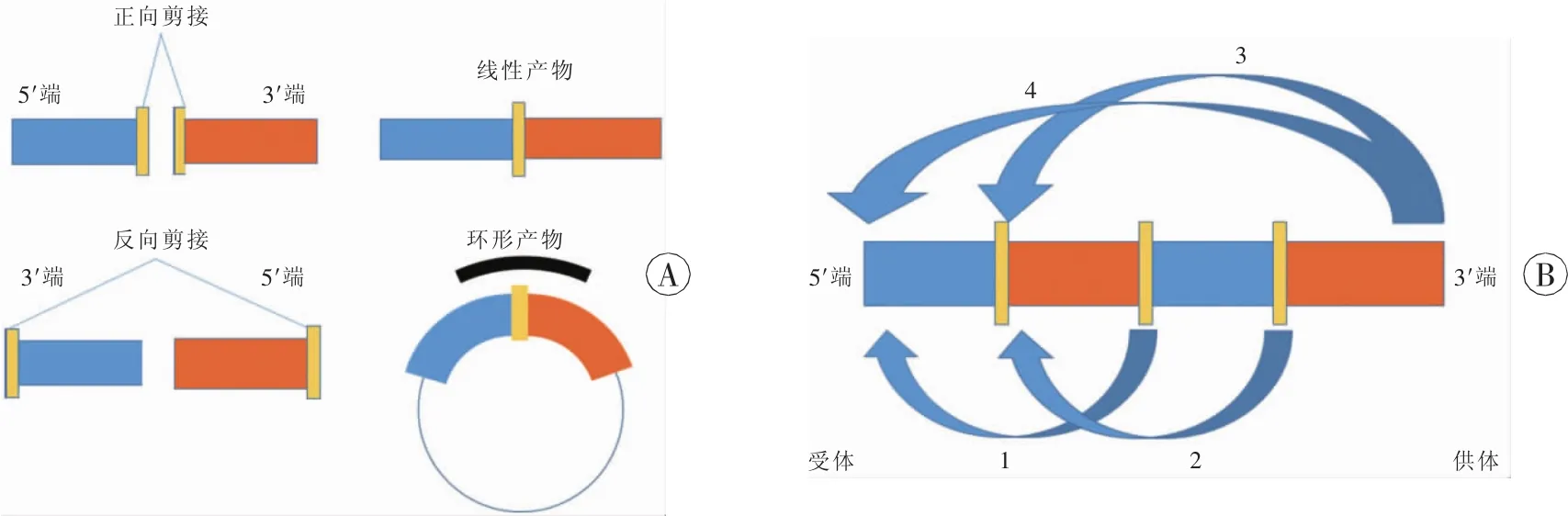
图1 circRNA形成方式
1.3 circRNA的预测及分析
自从人们意识到circRNA的重要性以来,circRNA的预测分析技术不断发展,现对其中重要并且常用的分析工具和方法进行介绍。
1.3.1 RNA-Seq技术
最初的转录组研究主要以基因芯片微阵列技术为基础,由于基因芯片技术的检测范围取决于芯片上的探针信息,所以只能检测已知序列的特征,缺少发现新基因的能力。小RNA由于序列较短,同源性较高,单纯利用基因芯片技术检测非常困难。RNA-Seq测序技术由于通量高、可重复性好、检测范围宽、定量准等优点,可以很好地弥补基因芯片技术在这方面的不足。RNA-Seq测序在过去十年中快速发展,促使关于生物的功能基因组研究日益兴起[17],人们利用高通量测序技术研究了从简单模式生物(如酵母、拟南芥、水稻等)到人等一些高等物种的基因组中DNA修饰和RNA的定性定量变化等动态的基因组位点的特性,成为发现识别小RNA分子的革命性技术。
1.3.2 常用的circRNA分析工具和数据库
目前对circRNA分子的研究还不太成熟,建立有价值的circRNA数据库可为后续研究提供一定的参考,推动相关研究[18]。目前,较常用的circRNA分析工具很多,CIRCexplorer[19](http://yanglab.github.io/CIRC explorer)是其中一种circRNA检测工具,通过过滤出Tophat(一个将RNA-Seq数据进行快速剪接映射的程序)无法比对上的核酸序列,再把这些序列用Tophat-Fusion比对到基因组上。那些用Tophat-Fusion比对到基因组上的非线性候选位置的序列就是潜在的反向剪接位点。这些序列会在有基因注释的帮助下,确定更加精确的供体、受体位置,最后,对circRNA进行注释。其优点是具有高度的准确性和敏感性,但是分析速度慢。
circRNA 常用数据库主要有:(1)circBase[20](http://www.circbase.org),由GLAZAR等开发的一个数据库,收集并合并了大量包括人、小鼠、腔棘鱼、线虫、果蝇等成套的circRNA相关数据资料,能够浏览访问和下载相关数据资料。该数据库能够鉴定RNA测序结果中目前已知的circRNA,可以通过互联网免费访问网站数据库,但该数据库更新不够及时,所收录的基因种属种类不够多、组织来源不够全面。(2)Circ2Traits[21](http://gyanxet-beta.com/circdb),是一个收集与疾病或性状相关的circRNA数据库。该数据库识别与miRNA相关的疾病和circRNA的相互作用,然后计算circRNA与疾病相关的可能性。针对特定疾病,该数据库可以构建预测的miRNA与蛋白编码基因、非编码基因以及circRNA基因的相互作用的网络图。此外,数据库还能比对出疾病相关的单核苷酸多态性(single nucleotide polymorphism,SNP)在 circRNA 上的位置,然后通过Ago分析两者之间的相互作用。(3)deepBase[22](http://rna.sysu.edu.cn/deepBase/),是一个研究非编码RNA的综合数据库,同时收录小RNA、长非编码RNA以及circRNA等非编码RNA的数据,旨在研究深度测序发现的各种非编码RNA的表达形式与功能,包含了大约15万的circRNA基因(人、鼠、果蝇、线虫等),构建了最全面的circRNA表达图谱。
2 circRNA参与的生物学功能
2.1 circRNA在生物生长发育过程中的变化规律
circRNA具有组织表达特异性和发育阶段的差异表达。circ-FoxO3是由转录子转录而来,在老年病人心脏样本中高表达。DU等[23]的研究表明,circ-FoxO3作用于抗衰老因子 tID-1、E2F1、FAK和 HIF1a,抑制后者作用。GRUNER等[24]发现,circRNA在老龄化的小鼠大脑中富集,而所对应的心脏中表达的circRNA没有发现明显的富集效应,说明circRNA在脑中的富集行为可能在生长发育过程中具有特定的功能。DONG等[25]发现伴随着进化进程,短散在核重复序列(short interspersed element,SINE)的数目明显增多,互补配对的能力也逐渐提高,与此对应的是circRNA的种类和表达量均有提高,说明circRNA与进化过程有关。LI等[26]利用芯片技术筛选出与大鼠肝再生过程密切相关的6种关键circRNA分子:circ432、circ2077、circ1366和 circ15分别对应调控 MAPK14、FN1、TNFRSF21和GOT1的表达,circ137与circ2270通过调控miR-127影响肝再生过程。
2.2 circRNA参与疾病发生的机制
以下研究表明,circRNA主要通过调节miRNA(即miRNA海绵作用)调控疾病的发生发展过程。LI等[27]对与神经系统疾病有关的circRNA研究发现,ciRS-7和circSry作为miRNA海绵在阿尔茨海默病 (Alzheimer’s disease,AD)、帕金森病(Parkinson disease,PD)等神经退行性疾病或脑部肿瘤中致病;circMbl与强直性肌营养不良症发病有关;cANRIL与脑血管疾病相关;hsa-circRNA 2149、has-circRNA 100783及circSry在神经系统免疫性疾病中发挥作用等。BONIZZATO等[28]介绍了经常导致血液系统肿瘤的染色体易位突变引入的融合性circRNA (f-circRNAs)在血液系统肿瘤中的作用,circRNA表现出的组织和细胞类型特异性特征与所对应的基因与细胞增殖、凋亡等信号通路关系密切。ZHU等[29]通过基因敲除发现,circBANP在结肠癌中高表达,敲除circBANP可明显影响结肠癌细胞的增殖。TANG等[30]描述了一个circRNA-000203调控心肌纤维化的信号通路:miR-26b-5p通过结合Col1a2和CTGF的3′UTR区,阻止后者的基因表达,CircRNA_000203结合miR-26b-5p,促进了心肌纤维化作用有关基因的表达。WENG等[31]研究发现,结肠直肠癌中ciRS-7明显高表达,且与患者的预后相关,表达高的患者预后较差,在HCT116和HT29细胞中可阻止miR-7的抑癌作用,并激活EGFR和RAF等癌基因的功能,因此认为,ciRS-7可作为结直肠癌的标志物和治疗靶标。
2.3 circRNA作为疾病诊断标记的研究
circRNA所具有的稳定性、组织表达特异性以及强大的疾病调控作用引起了临床科研工作者对其作为某些临床诊断困难疾病的诊断标记的关注。高通量测序技术结合强大的计算机分析技术以及实时荧光定量技术为circRNA应用于疾病生物学标记的研究提供了有力的技术支持。2016年,KULCHESKI等[32]对circRNA作为疾病诊断标记进行了可行性分析,认为circRNA作为miRNA海绵功能分子与关节炎、糖尿病、神经系统疾病及某些类型的肿瘤均有密切关系,其相对更高的稳定性及组织和疾病相关的差异性表达特性,可能成为相关疾病稳定且特异的诊断标记。同年,VAUSORT等[33]发现了一种心肌梗死特征性的circRNA,在外周血中检测该circRNA可以预测左心室的功能。NAIR等[34]研究发现,circRNA与乳腺癌分型有关,不同类型的乳腺癌特征性circRNA的差别较大,可能辅助诊断乳腺癌类型。随后,DONG等[35]的研究有了巨大突破,该研究团队通过芯片技术在精液、血浆中发现大量全新的circRNA,其中10792种为首次发现和报道,这开辟了新的circRNA研究方向,也为无创诊断提供了新的探索方向。CHEN等[36]在胃癌的研究中发现,circPVT1可作为胃癌预后指标,并独立于肿瘤大小、分期等指标,如果肿瘤分期结合circPVT1表达情况一起作为预后分析的指标将更有价值。2017年,CHEN等[37]的研究表明,组织和血浆中hsa_circ_0000190表达量的统计学分析诊断可信度分别可达到0.75和0.60,而将两者联合后可信度可达到0.775。
3 circRNA在法医学中的应用展望
死亡原因分析和死亡时间判断是法医病理学研究的两大主要问题,而个体识别是法医物证学研究的主要内容之一。由于法医学的研究对象主要是尸体,而机体在死后会发生各种代谢变化,这对法医寻求可靠的分子标记以判断死因、推断死亡时间以及个体识别是一大挑战。circRNA具有较一般核酸分子稳定的特性,应该引起法医学工作者对circRNA分子研究的重视,寻求应用circRNA解决法医学相关问题。
3.1 circRNA在法医学晚期死亡时间推断中的应用
死亡时间推断是法医学鉴定中需要解决的重要问题之一。寻找灵敏、特异的检测指标是死亡时间特别是晚期死亡时间推断的新思路[38-40]。核酸存在于细胞内,所处环境相对稳定,张志宏等[41]的研究发现,核酸等体内有机物质的降解随着死亡时间的延长呈现一定的规律性,因此利用分子生物学技术研究核酸降解规律用于死亡时间推断成为近年来死亡时间推断研究的热点。mRNA虽然没有DNA稳定,但可以很好地弥补检材缺少细胞核而检不出DNA的问题。吕叶辉等[42-43]研究mRNA的降解规律和死亡时间的关系,利用实时荧光定量PCR技术对死后不同时间的靶基因进行定量,尝试建立死亡时间模型以期望将该方法用于法医学死亡时间推断实践中。但该研究发现,一般核酸分子在机体死后其含量的变化容易受环境温度、淤血程度、细菌繁殖、疾病等的影响,这些影响会对推断造成一定的偏差。circRNA由于自身结构呈环状,相对稳定,较少受这些因素的影响,并且,这种稳定性会使circRNA降解更缓慢[44]。因此,circRNA有望成为法医学死亡时间推断的可靠分子生物学标记。
3.2 circRNA应用于法医学疑难死因分析中的展望
一般情况下,致命性损伤、重要器官的严重病变或者衰老等导致的死亡,通过全面系统的尸体检验以及毒(药)物检验等即能明确死因。但是,对于某些缺乏明显病理学改变的疾病,如糖尿病等内分泌系统疾病导致的死亡,特别是猝死,死因分析就较为困难,此类案件往往由于缺乏明确的临床检查资料以及法医病理学证据而引起纠纷。分子生物学标记诊断是缺乏特征性病理学改变或者病理学证据不明确时的重要辅助诊断工具,这启发法医工作者,进行特殊死因分析时,可探索运用表达特异且相对稳定的circRNA分子作为死因判断的辅助证据。2017年,ZHAO等[45]的研究表明,检验外周血中的hsa-circ-0054633可以帮助诊断糖尿病,该研究提示circRNA有助于法医学诊断。心脏性猝死一直是法医病理学研究的热点和难点,因为此类死亡较常见,且常常缺乏明显的、致命的病理学改变。ZHAO等[46]利用芯片法筛选到了22种变化明显的circRNA,挑选了5种circRNA进行验证实验,结果表明,hsa-circ-0124644可以作为冠状动脉疾病诊断的标志物。法医学工作者可以利用此类circRNA作为生物标记,帮助明确部分疑难案件的死因。
3.3 circRNA应用于生物检材来源鉴别的研究
法医学实践中,有时会遇到不明来源的组织或者疑似体液的生物检材,一般情况下,器官和组织的来源可以通过观察大体标本或者组织切片进行判断。相比之下,体液鉴别较为困难,如果获得的组织较少或者腐败严重,组织来源就更难以判断[47]。因此,法医学工作者需要探索更灵敏特异的方法用于鉴别生物检材的来源。传统的生物化学和免疫分析法鉴别组织来源简便快捷,但会破坏检材且特异性较低。DNA没有组织特异性,mRNA虽然灵敏性和特异性高,但相对不稳定,不适用于陈旧或腐败样本[48]。由于circRNA的稳定性和组织表达特异性,可以用于不同生物检材的来源鉴定[49],例如,Y染色体性别决定区特异表达的circRNA可以用于性别鉴别[50]。因此,circRNA对于生物检材来源鉴别,特别是对于陈旧的或降解生物检材检测可能具有极大的应用前景。
[1]HANSEN T B,JENSEN T I,CLAUSEN B H, et al.Natural RNA circles function as efficient microRNA sponges[J].Nature,2013,495(7441):384-388.
[2]SANGER H L,KLOTZ G,RIESNER D, et al.Viroids are single-stranded covalently closed circular RNA molecules existing as highly base-paired rod-like structures[J].Proc Natl Acad Sci USA,1976,73(11):3852-3856.
[3]KOS A,DIJKEMA R,ARNBERG A C, et al.The hepatitis delta (δ) virus possesses a circular RNA[J].Nature,1986,323(6088):558-560.
[4]HSU M T,COCA-PRADOS M.Electron microscopic evidence for the circular form of RNA in the cytoplasm of eukaryotic cells[J].Nature,1979,280(5720):339-340.
[5]CAPEL B, SWAIN A, NICOLIS S, et al.Circular transcripts of the testis-determining gene Sry in adult mouse testis[J].Cell,1993,73(5):1019-1030.
[6]SALZMAN J, GAWAD C, WANG P L, et al.Circular RNAs are the predominant transcript isoform from hundreds of human genes in diverse cell types[J].PLoS One,2012,7(2):e30733.
[7]JECK W R,SORRENTINO J A,WANG K,et al.Circular RNAs are abundant, conserved, and associated with ALU repeats[J].RNA,2013,19(2):141-157.
[8]MEMCZAK S,JENS M,ELEFSINIOTI A,et al.Circular RNAs are a large class of animal RNAs with regulatory potency[J].Nature,2013,495(7441):333-338.
[9]李景林,郑汪洋,李鑫恒,等.环状RNA在肿瘤中调控作用的研究进展[J].中华病理学杂志,2017,46(8):578-581.
[10]MENG S,ZHOU H,FENG Z,et al.CircRNA:functions and properties of a novel potential biomarker for cancer[J].Mol Cancer,2017,16(1):94.
[11]LASDA E, PARKER R.Circular RNAs: diversity of form and function[J].RNA,2014,20(12):1829-1842.
[12]Du WW,ZHANG C,YANG W,et al.Identifying and characterizing circRNA-protein interaction[J].Theranostics,2017,7(17):4183-4191.
[13]HUANG C,SHAN G.What happens at or after transcription:insights into circRNA biogenesis and function[J].Transcription,2015,6(4):61-64.
[14]JECK W R,SHARPLESS N E.Detecting and characterizing circular RNAs[J].Nat Biotechnol,2014,32(5):453-461.
[15]EBERT M S, SHARP P A.MicroRNA sponges:progress and possibilities[J].RNA,2010,16(11):2043-2050.
[16]XU S, XIAO S, QIU C, et al.Transcriptome-wide identification and functional investigation of circular RNA in the teleost large yellow croaker(Larimichthys crocea)[J].Mar Genomics,2017,32:71-78.
[17]滕晓坤,肖华胜.基因芯片与高通量DNA测序技术前景分析[J].中国科学(C 辑:生命科学),2008,38(10):891-899.
[18]MENG X,LI X,ZHANG P,et al.Circular RNA: an emerging key player in RNA world[J].Brief Bioinform,2016:w45.
[19]DUDEKULA D B,PANDA A C,GRAMMATIKAKIS I, et al.CircInteractome: a web tool for exploring circular RNAs and their interacting proteins and microRNAs[J].RNA Biol,2016,13(1):34-42.
[20]GLAŽAR P, PAPAVASILEIOU P, RAJEWSKY N.circBase: a database for circular RNAs[J].RNA,2014,20(11):1666-1670.
[21]GHOSAL S, DAS S, SEN R, et al.Circ2Traits: a comprehensive database for circular RNA potentially associated with disease and traits[J].Front Genet,2013,4:283.
[22]ZHENG L L, LI J H,WU J,et al.deepBase v2.0:identification, expression, evolution and function of small RNAs,LncRNAs and circular RNAs from deepsequencing data[J].Nucleic Acids Res,2016,44(D1):D196-D202.
[23]DU W W,YANG W,CHEN Y,et al.Foxo3 circular RNA promotes cardiac senescence by modulating multiple factors associated with stress and senescence responses[J].Eur Heart J,2017,38(18):1402-1412.
[24]GRUNER H,CORTES-LOPEZ M,COOPER D A,et al.CircRNA accumulation in the aging mouse brain[J].Sci Rep,2016,6:38907.
[25]DONG R,MA X K,CHEN L L,et al.Increased complexity of circRNA expression during species evolution[J].RNA Biol,2017,14(8):1064-1074.
[26]LI L, GUO J, CHEN Y, et al.Comprehensive circRNA expression profile and selection of key circRNAs during priming phase of rat liver regeneration[J].BMC Genomics,2017,18(1):80.
[27]LI T R, JIA Y J, WANG Q, et al.Circular RNA:a new star in neurological diseases[J].Int J Neurosci,2017,127(8):726-734.
[28]BONIZZATO A,GAFFO E,TE K G,et al.CircRNAs in hematopoiesis and hematological malignancies[J].Blood Cancer J,2016,6(10):e483.
[29]ZHU M,XU Y,CHEN Y,et al.Circular BANP,an upregulated circular RNA that modulates cell proliferation in colorectal cancer[J].Biomed Pharmacother,2017,88:138-144.
[30]TANG C M,ZHANG M,HUANG L,et al.CircRNA_000203 enhances the expression of fibrosis-associated genes by derepressing targets of miR-26b-5p,Col1a2 and CTGF, in cardiac fibroblasts[J].Sci Rep,2017,7:40342.
[31]WENG W,WEI Q,TODEN S,et al.Circular RNA ciRS-7-A promising prognostic biomarker and a potential therapeutic target in colorectal cancer[J].Clin Cancer Res,2017,23(14):3918-3928.
[32]KULCHESKI F R, CHRISTOFF A P, MARGIS R.Circular RNAs are miRNA sponges and can be used as a new class of biomarker[J].J Biotechnol,2016,238:42-51.
[33]VAUSORT M,SALGADO-SOMOZA A,ZHANG L,et al.Myocardial infarction-associated circular RNA predicting left ventricular dysfunction[J].J Am Coll Cardiol,2016,68(11):1247-1248.
[34]NAIR A A,NIU N,TANG X,et al.Circular RNAs and their associations with breast cancer subtypes[J].Oncotarget,2016,7(49):80967-80979.
[35]DONG W W,LI H M,QING X R,et al.Identification and characterization of human testis derived circular RNAs and their existence in seminal plasma[J].Sci Rep,2016,6:39080.
[36]CHEN J,LI Y,ZHENG Q,et al.Circular RNA profile identifies circPVT1 as a proliferative factor and prognostic marker in gastric cancer[J].Cancer Lett,2017,388:208-219.
[37]CHEN S,LI T,ZHAO Q,et al.Using circular RNA hsa_circ_0000190 as a new biomarker in the diagnosis of gastric cancer[J].Clin Chim Acta,2017,466:167-171.
[38]LV Y, MA K, ZHANG H, et al.A time course study demonstrating mRNA,microRNA,18S rRNA,and U6 snRNA changes to estimate PMI in deceased rat’s spleen[J].J Forensic Sci,2014,59(5):1286-1294.
[39]LI W,MA K,LV Y,et al.Postmortem interval determination using 18S-rRNA and microRNA[J].Sci Justice,2014,54(4):307-310.
[40]ZHANG H, ZHANG P, MA K, et al.The selection of endogenous genes in human postmortem tissues[J].Sci Justice,2013,53(2):115-120.
[41]张志宏,袁雅洁.核酸与死亡时间的相关性及其检测方法[J].中国司法鉴定,2013(1):95-97.
[42]吕叶辉,李志宏,托娅,等.不同温度下大鼠脑组织RNA降解与早期 PMI的相关性[J].法医学杂志,2016,32(3):165-170.
[43]吕叶辉,马开军,潘晖,等.不同死因大鼠心肌多种RNA表达量与死亡时间的关系[C].北京:全国第九次法医学术交流会,2013.
[44]SCHMIDT C A, NOTO J J, FILONOV G S, et al.A method for expressing and imaging abundant,stable,circular RNAs in vivo using tRNA splicing[J].Methods Enzymol,2016,572:215-236.
[45]ZHAO Z,LI X,JIAN D,et al.Hsa_circ_0054633 in peripheral blood can be used as a diagnostic biomarker of pre-diabetes and type 2 diabetes mellitus[J].Acta Diabetol,2017,54(3):237-245.
[46]ZHAO Z, LI X, GAO C, et al.Peripheral blood circular RNA hsa_circ_0124644 can be used as a diagnostic biomarker of coronary artery disease[J].Sci Rep,2017,7:39918.
[47]李岩.法医相关体液鉴定技术的研究进展[J].科技展望,2015(11):106-107.
[48]马丽莉,马红杜,黄代新.人体组织/体液来源的法医学鉴定[J].中国法医学杂志,2013,28(4):291-294.
[49]张雅琪,邵诚臣,李成涛,等.circRNA的研究进展及其法医学意义[J].法医学杂志,2016,32(2):131-133.
[50]CAO Q P, GAUDETTE M F, ROBINSON D H,et al.Expression of the mouse testis-determining gene Sry in male preimplantation embryos[J].Mol Reprod Dev,1995,40(2):196-204.
