药用美洲大蠊全基因组测序分析
2018-03-26晋家正李午佼牟必琴沈咏梅耿福能岳碧松范振鑫
晋家正, 李午佼, 牟必琴, 沈咏梅, 耿福能, 岳碧松, 范振鑫, *
1.生物资源与生态环境教育部重点实验室,四川大学生命科学学院,成都610065; 2.药用美洲大蠊四川省重点实验室,四川西昌615000; 3.四川省药用动物工程技术研究中心,四川西昌615000)
美洲大蠊Periplanetaamericana属昆虫纲Insecta蜚蠊目Blattaria蜚蠊科Blattidae大蠊属Periplaneta,是人们熟知的卫生害虫,同时也是重要的药用昆虫。蜚蠊入药早在《神农本草经》中就有记载,中医学认为蟑螂具有活血散瘀、解毒消疳和利尿消肿的功效,还被广泛应用于治疗疔疮、肿毒及虫蛇咬伤等方面。四川好医生攀西药业有限责任公司在四川省西昌市建立了药用美洲大蠊人工养殖基地,实现了规模化和标准化饲养,成为国内首家昆虫类药材——美洲大蠊GAP认证养殖基地,用美洲大蠊制成的康复新液等多种药物临床应用效果良好,特别是在治疗人腔道溃疡损伤和促创面修复等方面效果显著。近年来,随着对美洲大蠊及其提取物研究的不断深入,其抗肿瘤、抗氧化、增强免疫及其对糖尿病和心血管疾病的效果也受到关注(肖小芹等,2007;马俊等,2015;谭巧云等,2016;夏超等, 2016;陈佳松等,2017;李娇等,2017;张蕊等,2017)。
有关美洲大蠊的研究,国外主要集中在美洲大蠊生物学、生态学习性、抗药性及防治、生理生化及过敏原等方面(Ahmedetal.,2010;Kimetal.,2016;Tahiretal.,2017)。近年来,国内的研究主要集中在化学成分分析、临床应用和药用价值评估等方面(郭美仙等,2017;Yunetal.,2017;Zhaoetal.,2017)。随着测序技术和基因组分析方法的飞速发展,大量动物、植物和微生物的基因组测序陆续完成,取得了丰硕的研究成果。到目前为止,还没有美洲大蠊全基因组测序的研究报道。本文以人工繁殖的药用美洲大蠊为材料,完成了全基因组测序、组装和注释,对进一步从基因组水平阐释美洲大蠊的遗传进化、生理生化、环境适应及药物研发等具有重要意义。
1 材料与方法
1.1 美洲大蠊样品及DNA提取
测序用美洲大蠊样品由四川好医生攀西药业有限责任公司在四川省西昌市的药用美洲大蠊规范化封闭式人工养殖基地提供。在去除3只美洲大蠊雄性成虫的翅膀、附肢和消化道并进行表面消毒后,直接送杭州和壹基因科技有限公司建库测序。
1.2 文库构建和测序
美洲大蠊DNA提取、文库构建和测序均由杭州和壹基因科技有限公司完成。共构建了37个文库,其中包括长片段文库24个,插入片段大小分别为2 kb、5 kb、10 kb和20 kb;短片段文库13个,插入片段大小分别为250 bp、500 bp和800 bp。采用二代测序平台的Illumina HiSeq 2000和三代测序平台的PacBio SMRT进行高通量测序,原始测序数据经过滤后用于基因组组装与质量评估。
1.3 基因组组装与质量评估
利用Jellyfish(Marçais & Kingsford,2011)进行k-mer统计分析,对基因组大小、杂合度和重复序列进行评估。分别使用DISCOVAR(Weisenfeldetal.,2014)和BESST(Sahlinetal.,2014)构建Contig序列和scaffold序列,并通过Soap Gapcloser(Luoetal.,2012)补缺得到更长的scaffold片段,最后利用三代测序数据补洞和延伸,完成基因组组装。利用BUSCO(Simãoetal.,2015)进行单拷贝基因完整性分析,并使用bowtie2(Langmead & Salzberg,2012)将所有小片段序列比对回组装好的基因组,以检查基因区是否完整。最后,进行GC深度分析,检查组装好的基因组中是否存在污染序列。
1.4 基因结构与功能注释
基因注释采用Denovo结构注释、同源结构注释和基于转录本预测相结合的方法。Denovo结构注释是在屏蔽重复序列之后,用内华达古白蚁Zootermopsisnevadensis和德国小蠊Blattellagermanica基因组信息训练Augustus(Stankeetal.,2008)和SNAP (Korf,2004)的预测模型;同源结构注释通过下载7种昆虫的蛋白质序列文件,利用TBLASTN比对到美洲大蠊基因组,最后利用GeneWise(Birneyetal.,2004)对可能的基因区域进行结构预测;基于转录本预测是将美洲大蠊转录组数据通过Blast比对回基因组,然后利用PASA(Haasetal.,2003)对基因进行预测。最后利用EVidenceModeler(Haasetal.,2008)和Apollo(Lewisetal.,2002)进行整合。用Blastp将翻译的蛋白质序列与3个非冗余蛋白质数据库(Swiss-Prot,KOG和NCBI NR)进行比对,同时使用InterProScan(Hunteretal.,2008)完成美洲大蠊基因的功能注释。
2 结果
2.1 文库构建和测序
本研究共获得小片段文库测序850 Gb和大片段文库测序2.1 Tb的原始数据,经质量控制删除后得到1.4 Tb的二代数据,覆盖深度大约446×(表1);同时还获得约10×的三代数据,用于scaffold序列延伸(表2)。
2.2 基因组组装与质量评估
当k=17时的k-mer分布如图1。双峰分布表明美洲大蠊基因组的杂合程度较高,所有的k-mer总数为173 392 497 379,峰值为54,计算得到美洲大蠊基因组的大小约为3.2 Gb,杂合率约为0.635%,属于复杂基因组。

表1 美洲大蠊Illumina HiSeq 2000平台测序Table 1 Genomic sequencing of Periplaneta americana by Illumina HiSeq 2000 platform

表2 美洲大蠊PacBio SMRT平台测序Table 2 Genomic sequencing of Periplanetaamericana by PacBio SMRT

图1 美洲大蠊基因组17 bp-mer分布图Fig. 1 17 bp-mer distribution curve of Periplaneta americana genome
横坐标为17 bp-mer深度, 纵坐标为该深度下的k-mer频数所占比例
X-axis: 17 bp-mer depth, Y-axis: proportion of k-mer frequency
通过Denovo组装,基因组大小为3.26 Gb,与预测基因组大小基本符合。组装的Contig N50为28.2 kb,scaffold N50为315 kb(表3)。通过BUSCO评估显示,单拷贝基因完整性为88.1%,小片段文库比对率平均为99.8%,表明基因组的基因完整性较高(表4)。基因组GC含量为0.346,GC深度分析表明,美洲大蠊基因组GC含量分布在一个合理的范围,没有明显的污染差异区域(图2)。

表3 美洲大蠊基因组组装Table 3 Statistics of Periplaneta americana genome assembly
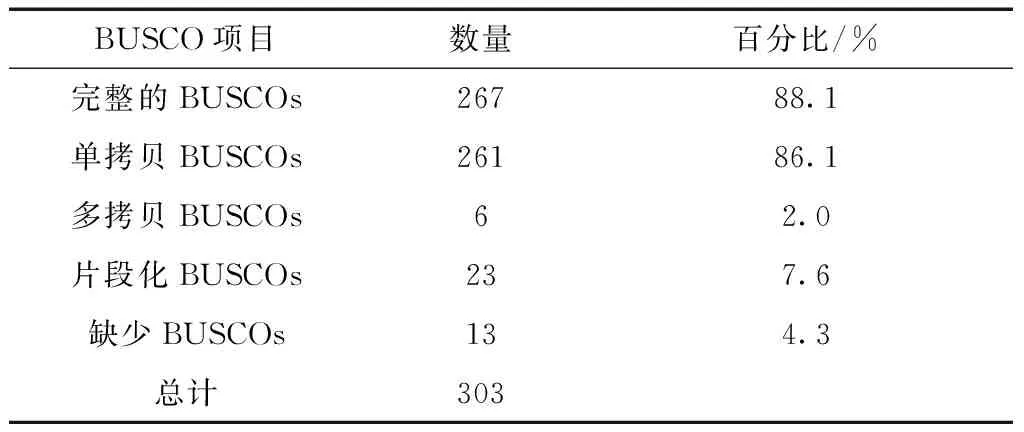
表4 BUSCO基因完整性评估Table 4 Genome integrality based on BUSCO
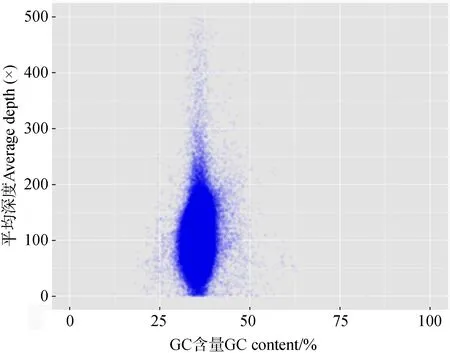
图2 美洲大蠊基因组GC含量深度分布Fig. 2 Correlation between GC content and sequencing depth in the genome of Periplaneta americana
2.3 基因结构与功能注释
在美洲大蠊基因组重复序列注释中,共注释到1 993.9 Mb的重复序列。其中,DNA转座子最多,达到517 Mb,占基因组大小的16.18%,其次是LINE转座子,达到436 Mb,SINE转座子和LTR转座子所占基因组的比例都比较小,只有1.8%左右。
在Denove预测中使用的3个软件预测到的基因数差异较大(表5),同源预测得到3.5万个基因,而基于转录本预测到约23万个基因。利用EVidenceModeler整合所有的预测结果,在去除仅有
Denovo证据支持的基因后,最终得到14 568个可信度较高的基因序列。
在预测的14 568个基因中,有13 464个基因能由4个数据库(NR库、KOG库、Interpro库和Swiss-Prot库)获得功能注释(图3),注释率达92.4%,能被4个数据库同时注释到的基因数为10 773个,占功能注释基因总数的80.0%。

图3 美洲大蠊基因功能注释统计Fig. 3 Gene function annotations of Periplaneta americana
3 讨论
随着基因组学的飞速发展,完成基因组测序的动物越来越多,组装的质量也越来越高。已经报道的昆虫基因组大小变化很大,有的只有几百兆,东亚飞蝗Locustamigratoria的基因组达6.5 Gb(Wangetal.,2014),而本研究的美洲大蠊基因组为3.26 Gb。基因组的复杂程度也各不相同。美洲大蠊的基因组重复序列含量高、杂合度高,属于复杂基因组,给测序和组装带来极大困难。尽管大大提高了文库数量和测序量,并辅以三代测序方法,但美洲大

表5 3种方法预测的美洲大蠊基因数Table 5 Predicted protein-coding genes of Periplaneta americana using 3 methods
蠊基因组的组装质量还是不能与脊椎动物及其他简单基因组相比。但与近年来完成的几种昆虫基因组相比,美洲大蠊基因组的组装质量较高(表6),能够满足进一步分析的要求。
目前公布的昆虫基因组中,蜚蠊目昆虫只有3种,即德国小蠊和2种白蚁:内华达古白蚁和Macrotermesnatalensis。美洲大蠊是第一个完成基因组测序的大蠊属昆虫,对于进一步研究大蠊属昆虫的起源进化及药用资源开发利用具有重要意义。

表6 7种昆虫的基因组测序和组装结果比较Table 6 Comparison of 7 insect genome sequences
注Notes: 1. Consortiumetal., 2012, 2. Keelingetal., 2013, 3. Geoffreyetal., 2014, 4. Terraponetal., 2014, 5. Wangetal., 2014, 6. Chenetal., 2015
致谢:感谢药用美洲大蠊四川省重点实验室提供美洲大蠊样品,四川好医生攀西药业有限责任公司提供经费赞助。
陈佳松, 陈峰, 彭锐, 等. 2017. 基于转录组测序分析美洲大蠊提取物促进小鼠创面愈合的分子机制[J]. 四川动物, 36(4): 398-403.
郭美仙, 刘晓波, 刘光明, 等. 2017. 美洲大蠊提取物对H22腹水瘤小鼠肿瘤微环境的影响[J]. 大理学院学报,2(10): 5-9.
李娇, 郭美仙, 张冰清, 等. 2017. 美洲大蠊提取物CⅡ-3对肝癌H22荷瘤小鼠作用研究[J]. 中华肿瘤防治杂志,24(11): 739-744.
马俊, 曾远生, 刘炜. 2015. 美洲大蠊提取物对力竭运动大鼠心血管氧化损伤的保护作用[J]. 中国应用生理学杂志, 31(5): 404-406.
谭巧云, 满红霞, 那凯歌, 等. 2016. 美洲大蠊提取物对口腔溃疡大鼠模型的作用[J]. 中国临床药理学杂志, 32(11): 1014-1016.
夏超, 王佳佳, 李芳群, 等. 2016. 美洲大蠊水提取物对免疫性肝纤维化大鼠的保护作用[J]. 安徽医科大学学报, 51(2): 199-204.
肖小芹, 汪世平, 徐绍锐, 等. 2007. 美洲大蠊提取物抗炎, 镇痛作用的实验研究[J]. 中国病原生物学杂志, 2(2): 140-143.
张蕊, 袁发璐, 李婷, 等. 2017. 美洲大蠊提取物对人肝癌HepG2细胞的作用机制研究[J]. 中国现代医学杂志, 27(12): 1-8.
Ahmed A, Minha K, Namood-e-Sahar,etal. 2010.Insilicoidentification of potential American cockroach (Periplanetaamericana) allergens[J]. Iranian Journal of Public Health, 39(3): 109-115.
Birney E, Clamp M, Durbin R. 2004. GeneWise and genomewise[J]. Genome Research, 14(5): 988-995.
Chen XG, Jiang XT, Gu JB,etal. 2015. Genome sequence of the Asian tiger mosquito,Aedesalbopictus, reveals insights into its biology, genetics, and evolution[J]. Proceedings of the National Academy of Sciences of the United States of America, 112(44): E5907. DOI:10.1073/pnas.1516410112.
Consortium THG, Kanchon KD, James RW,etal. 2012. Butterfly genome reveals promiscuous exchange of mimicry adaptations among species[J]. Nature, 487(7405): 94-98.
Geoffrey MA, Patrick PA, Joanna EA,etal. 2014. Genome sequence of the tsetse fly (Glossinamorsitans): vector of African trypanosomiasis[J]. Science, 344(6182): 380-386.
Haas BJ, Delcher AL, Mount SM,etal. 2003. Improving the arabidopsis genome annotation using maximal transcript a lignment assemblies[J]. Nucleic Acids Research, 31(19): 5654-5666.
Haas BJ, Salzberg SL, Zhu W,etal. 2008. Automated eukaryotic gene structure annotation using EVidenceModeler and the program to assemble spliced alignments[J]. Genome Biolology, 9(1): R7.
Hunter S, Apweiler R, Attwood TK,etal. 2008. InterPro: the integrative protein signature database[J]. Nucleic Acids Research, 37(suppl_1): D211-D215.
Keeling CI, Yuen MM, Liao NY,etal. 2013. Draft genome of the mountain pine beetle,DendroctonusponderosaeHopkins, a major forest pest[J]. Genome Biology, 14(3): R27.
Kim IW, Lee JH, Subramaniyam S,etal. 2016.Denovotranscriptome analysis and detection of antimicrobial peptides of the American cockroachPeriplanetaamericana(Linnaeus)[J]. PLoS ONE, 11(5): e0155304. DOI:10.1371/journal.pone.0155304.
Korf I. 2004. Gene finding in novel genomes[J]. BMC Bioinformatics, 5(1): 59.
Langmead B, Salzberg SL. 2012. Fast gapped-read alignment with Bowtie 2[J]. Nature Methods, 9(4): 357-359.
Lewis SE, Searle S, Harris N,etal. 2002. Apollo: a sequence annotation editor[J]. Genome Biolology, 3(12): research0082.1-82.14.
Luo R, Liu B, Xie Y,etal. 2012. SOAPdenovo2: an empirically improved memory-efficient short-readdenovoassembler[J]. Giga Science, 1(1): 18.
Marçais G, Kingsford C. 2011. A fast, lock-free approach for efficient parallel counting of occurrences of k-mers[J]. Bioinformatics, 27(6): 764-770.
Sahlin K, Vezzi F, Nystedt B,etal. 2014. BESST-efficient scaffolding of large fragmented assemblies[J]. BMC Bioinformatics, 15(1): 281.
Simão FA, Waterhouse RM, Ioannidis P,etal. 2015. BUSCO: assessing genome assembly and annotation completeness with single-copy orthologs[J]. Bioinformatics, 31(19): 3210-3212.
Stanke M, Diekhans M, Baertsch R,etal. 2008. Using native and syntenically mapped cDNA alignments to improvedenovogene finding[J]. Bioinformatics, 24(5): 637-644.
Tahir HM, Mustafa R, Khan AA,etal. 2017. Toxicity and resistance of American cockroach,PeriplanetaamericanaL.(Blattodea: Blattidae) against malathion[J]. African Entomology, 25(2): 361-366.
Terrapon N, Li C, Robertson HM,etal. 2014. Molecular traces of alternative social organization in a termite genome[J]. Nature Communications, 5(6183): 3636.
Wang X, Fang X, Yang P,etal. 2014. The locust genome provides insight into swarm formation and long-distance flight[J]. Nature Communications, 5(5): 2957.
Weisenfeld NI, Yin S, Sharpe T,etal. 2014. Comprehensive variation discovery in single human genomes[J]. Nature Genetic, 46(12): 1350-1355.
Yun JE, Hwang JS, Lee DG. 2017. The antifungal activity of the peptide, periplanetasin-2, derived from American cockroachPeriplanetaamericana[J]. Biochemical Journal, 474(17): 3027-3043.
Zhao Y, Yang A, Tu P,etal. 2017. Anti-tumor effects of the American cockroach,Periplanetaamericana[J]. Chinese Medicine, 12(1): 26. DOI: 10.1186/s13020-017-0149-6.
