单增李斯特菌食品分离株LM201的双组分信号转导系统的生物信息学分析
2018-03-21郑金水
张 娜, 欧 娅, 郑金水, 刘 梅,,3*
(1.华中农业大学动物医学院 兽医微生物学与免疫学实验室,湖北 武汉 430070; 2.华中农业大学 农业微生物学国家重点实验室,湖北 武汉 430070; 3.华中农业大学 预防兽医学湖北省重点实验室,湖北 武汉 430070)
单增李斯特菌(Listeriamonocytogenges,Lm)是导致人兽共患病—李斯特菌病(listeriosis)的食源性病原菌。Lm的易感者为免疫缺陷和免疫力低下者,其侵入人和动物机体导致胃肠炎、脑膜炎、脑炎、败血症、流产和死胎等病症。李斯特菌病虽发病率不高,但病死亡率可达20%~30%[1]。近几年,我国不断有人和动物感染李斯特菌病的报道[2],而且报告病例涉及我国大部分省市,严重威胁着人们的健康和畜牧业发展。因此,对Lm毒力调控方面的研究工作不容忽视。Lm于外界环境中时是腐生菌,侵入宿主机体内则变为致病菌,其转变机制主要是Lm中有一个转录调控因子—PrfA,它像一个开关,当Lm处于外界环境时,PrfA处于低活性状态,受其调控的相关毒力基因表达水平低;当Lm侵入宿主细胞后,PrfA被激活,进而诱导InlA、InlB、LLO、PlcA、PlcB、Hpt和ActA等毒力因子基因的表达,使得Lm能在宿主细胞中繁殖及在细胞间传播[3]。转录调控因子活性的激活,需要细菌首先感应环境变化,然后将信号传递至菌体内,从而做出相应的调控反应,这个过程涉及到细菌的信号转导系统,其中双组分信号转导系统(Two-component signal transduction systems,TCSs)在细菌的信号传导过程中起至关重要的作用[4]。TCSs参与调控细菌的生长代谢、生物被膜形成以及病原菌的毒力等。因此,研究Lm的TCSs是探究Lm致病机制的重要方面[5]。TCSs通过调控蛋白质磷酸化来实现信号转导。典型的TCS由组氨酸蛋白激酶(Histidine kinase,HK)和应答调节蛋白(Response regulator,RR)组成,HK和RR通常是由基因组上同一个操纵子的两个邻近基因编码。其经典调控机制为HK感受胞外信号并自体磷酸化、HK将磷酸基团转移给RR使之活化、活化的RR结合到靶序列从而调控相关基因的转录表达。研究发现,细菌TCSs中的HK除能激活同源RR外,还能激活非同源RR,或两个不同的HK可以激活同一RR,即形成one-to-many 或 many-to-one的交叉调控模式,这有利于菌体更快更好地适应外界环境[6]。Lm的TCSs的交叉调控模式是怎样的呢?目前关于Lm的TCSs研究主要针对单对TCS、单个HK或RR的研究,关于Lm的TCSs交叉调控方面的系统化研究则鲜有报道。研究TCSs的交叉调控网络,首先需要全面统计分析和了解目标菌株所含有的TCSs的数量、结构和功能。本研究以本实验室已完成全基因组测序的Lm食品分离株LM201[7]为研究对象,应用生物信息学方法,对LM201的TCSs进行了数量统计、结构分析和功能预测,从而为进行Lm的TCSs交叉调控网络的研究打下基础。
1 材料与方法
1.1 材料
基因组序列:Lm菌株LM201是从冷冻肉中分离得到,经本课题组测定,其血清型为4b,有中等生物被膜形成能力,小鼠LD50为6.3×106CFU。本课题组已完成LM201全基因组测序工作,其序列(AYPT00000000)[7]已提交至NCBI。L.innocuaClip11262 (NC_003212.1)、L.ivanoviisubsp.ivanoviiPAM 55(FR687253.1)、Bacillussubtilis168 (NZ_CP010052.1)和L.monocytogenesEGD-e (AL591824.1)的全基因组序列均来自GenBank (http://www.ncbi.nlm.nih.gov/genome)。
1.2 方法
1.2.1 双组分信号转导系统的预测 利用Pfam数据库(http://pfam.xfam.org/)中HKs的保守结构域HATPase_c(Pfam02518)搜寻LM201的HKs,用RRs的保守结构域 Response_reg (Pfam00072)搜寻RRs,通过NCBI的BLAST确认搜到的HKs和RRs。HATPase_c是HK的催化结构域,是ATP结合的激酶功能区,位于 HK 的 C-末端;Response_reg是RR的磷酸受体结构域,位于 RR 的 N-末端,属于REC超家族的成员。
1.2.2 结构与功能分析 功能域分析利用SMART (http://smart.embl-heidelberg.de/)来完成。蛋白质跨膜螺旋结构域用TMHMM Server v.2.0 (http://www.cbs.dtu.dk/services /TMHMM-2.0/)进行预测。利用ClustalW软件进行多重序列比对。采用BLASTP (www.ncbi.nlm.nih.gov/blast/)进行同源性比对。
2 结果与分析
2.1 LM201的TCSs统计及序列比对分析
利用Pfam数据库对LM201基因组的TCSs进行了搜寻和统计。从数量上讲, LM201有14对HK/RR和2个孤儿RR,与同种同血清型的L.monocytogenesF2365 (4b型)数量一致;比同种不同血清型的L.monocytogenesEGD-e (1/2a型)少一对双组分(lmo1061/lmo1060);与同属不同种的L.ivanovii和L.innocua的TCSs数量相近;与同目不同属的Bacillussubtilis数量则相距甚远(表1)。
通过序列比对分析发现,LM201的TCSs与同种同血清型菌株F2365的TCSs的组成和结构一致;与同种不同血清型菌株EGD-e的TCSs有一一对应关系(结果图略)。
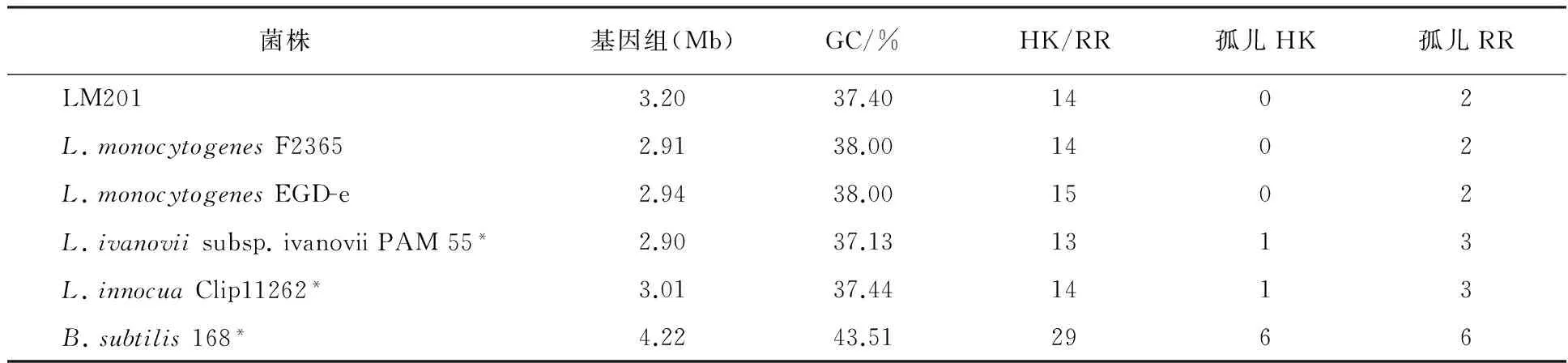
表1 LM201基因组中TCS数目的预测结果
注:*作为对照
2.2 HK的结构域组成
利用SMART对预测的14个HKs进行了结构域的分析(表2)。LM201的HKs具有11种不同组成结构,所有HK的C端均具有典型结构域HATPase_c (激酶催化结构域);N端具有不同结构域,包括HAMP、PAS、PAC和GAF等。HAMP主要存在于热休克蛋白HSP90、甲基转移酶等与ATP结合的蛋白质中,其主要功能可能是感应与细菌细胞渗透相关的信号[8];PAS (Per-Arnt-SIM)含有与血红素蛋白和光敏黄蛋白结合的固定区域,可感应氧化还原电位、光及氧等信号,参与调控细菌氧化还原反应[9];PAC 可以修饰PAS,可能有助于PAS的折叠[10];GAF存在于光合细菌及非光合细菌中,主要通过与核苷酸及其他小分子配体结合,改变配体构象而发挥催化调节活性功能[11]。
HK02 (RS05925)的N端为HPT结构域,中部有H-Kinase dim区域,C端是CheW受体结构域。HPT (Histidine phosphotransfer)只存在于真细菌中,其所含有的组氨酸残基参与磷酸基团的转移反应,介导HK和RR的信号传递[12];H-Kinase dim是含有该结构域的组氨酸激酶CheA形成二聚体的区域[13];CheW受体结构域存在于参与细菌趋化性调控的TCS中,含有该结构域的组氨酸激酶CheA可以和连接蛋白CheW偶联,CheW参与细菌的趋化性信号传导[14]。

表2 LM201的HKs的编号、基因组上的位置和结构域组成
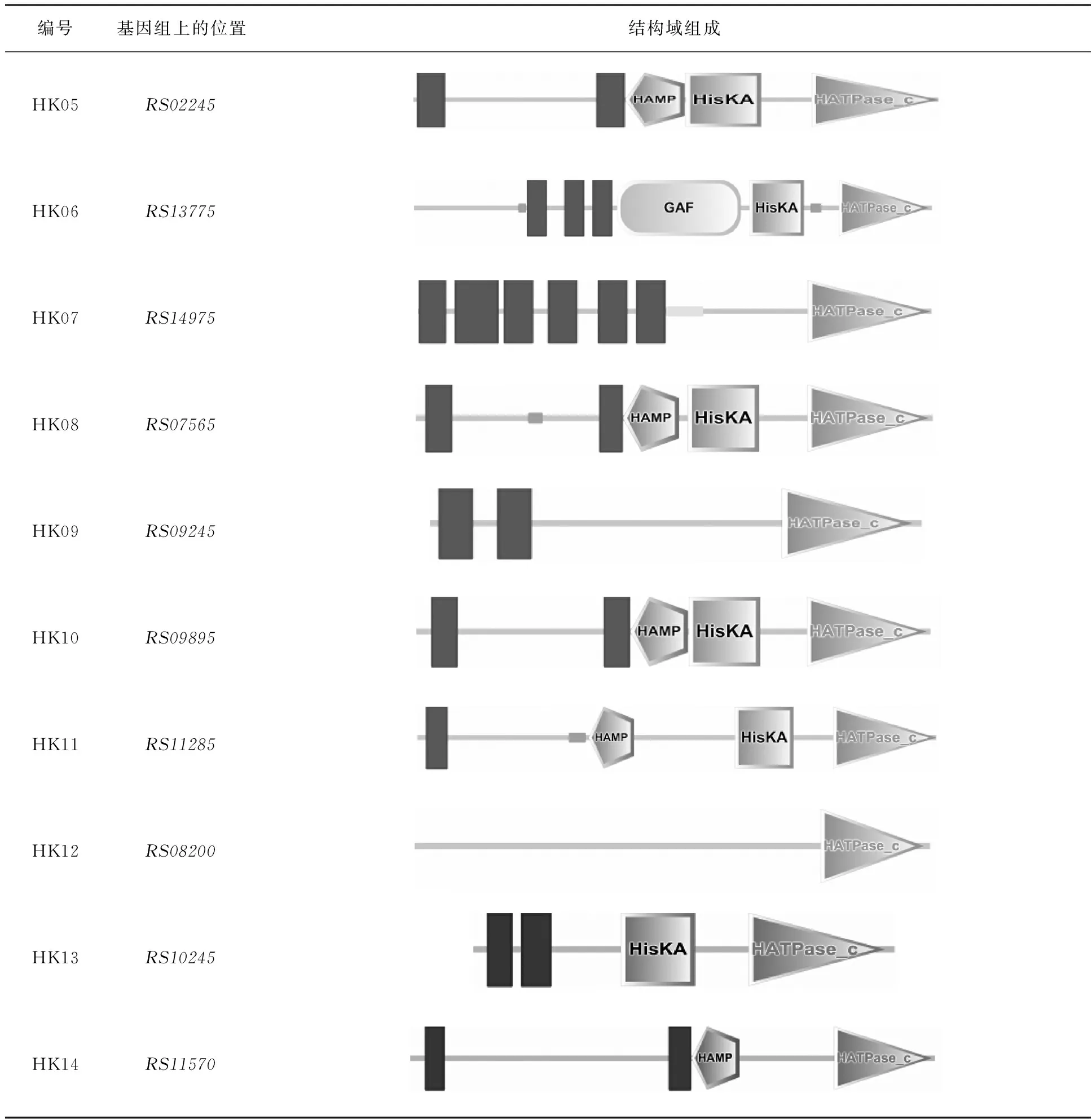
续表2
本研究通过TMHMM Server v.2.0对LM201中的HKs进行了跨膜结构分析(结果图略)。除HK11(RS11285)外,其余HKs的分析结果与表2第三栏所示的跨膜螺旋区(Transmembrane helix region,蓝色矩形所示)结构一致。TMHMM Server v.2.0分析结果显示,HK11有两个跨膜螺旋区,但用SMART预测时,只显示了一个跨膜螺旋区(表2),原因是另一个跨膜区与低复杂性结构域(Low compositional complexity,粉红色矩形所示)重叠了。
2.3 RR的结构域组成与分类
RR的结构域组成与分类利用SMART,本研究对预测的16个RRs的组成结构和亚家族的归属进行了分析(表3)。LM201的 RRs的N端都含有基本结构-磷酸受体结构域REC(RR15 (RS05910)的REC在C端),此结构域中含有保守的Asp (天冬氨酸残基)磷酸化位点,即RR的磷酸化位点;RRs的C端为输出区域,具有多样性,但多数RR的输出区域为典型结构Trans reg_C。通过分析RRs的输出区域,发现LM201的16个RRs分属于OmpR、CheV、CheY、NarL、LytTR、YesN和AmiR_NasR这7个亚家族(表3)。
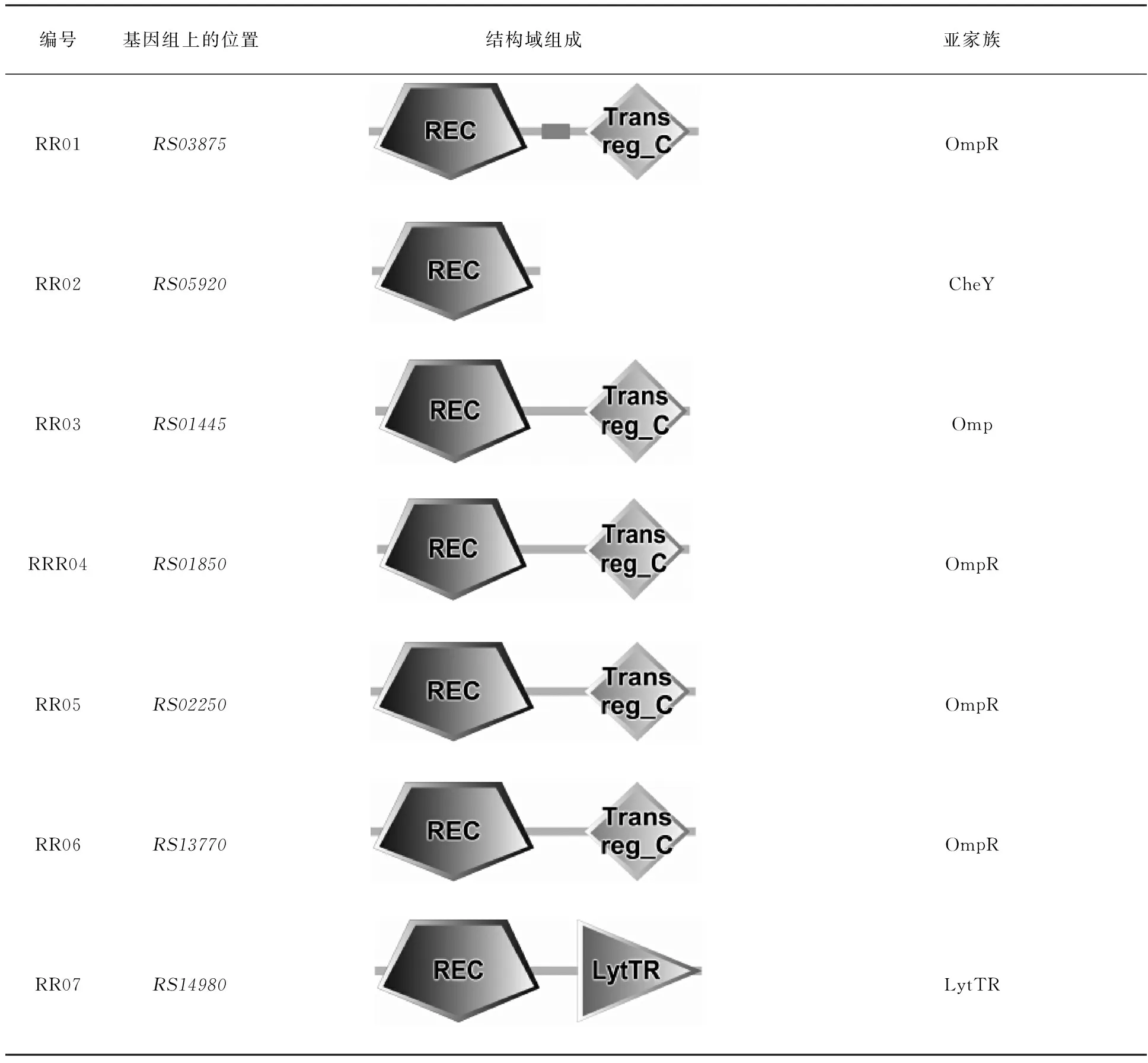
表3 LM201的RRs的编号、基因组上的位置、结构域组成和亚家族分类
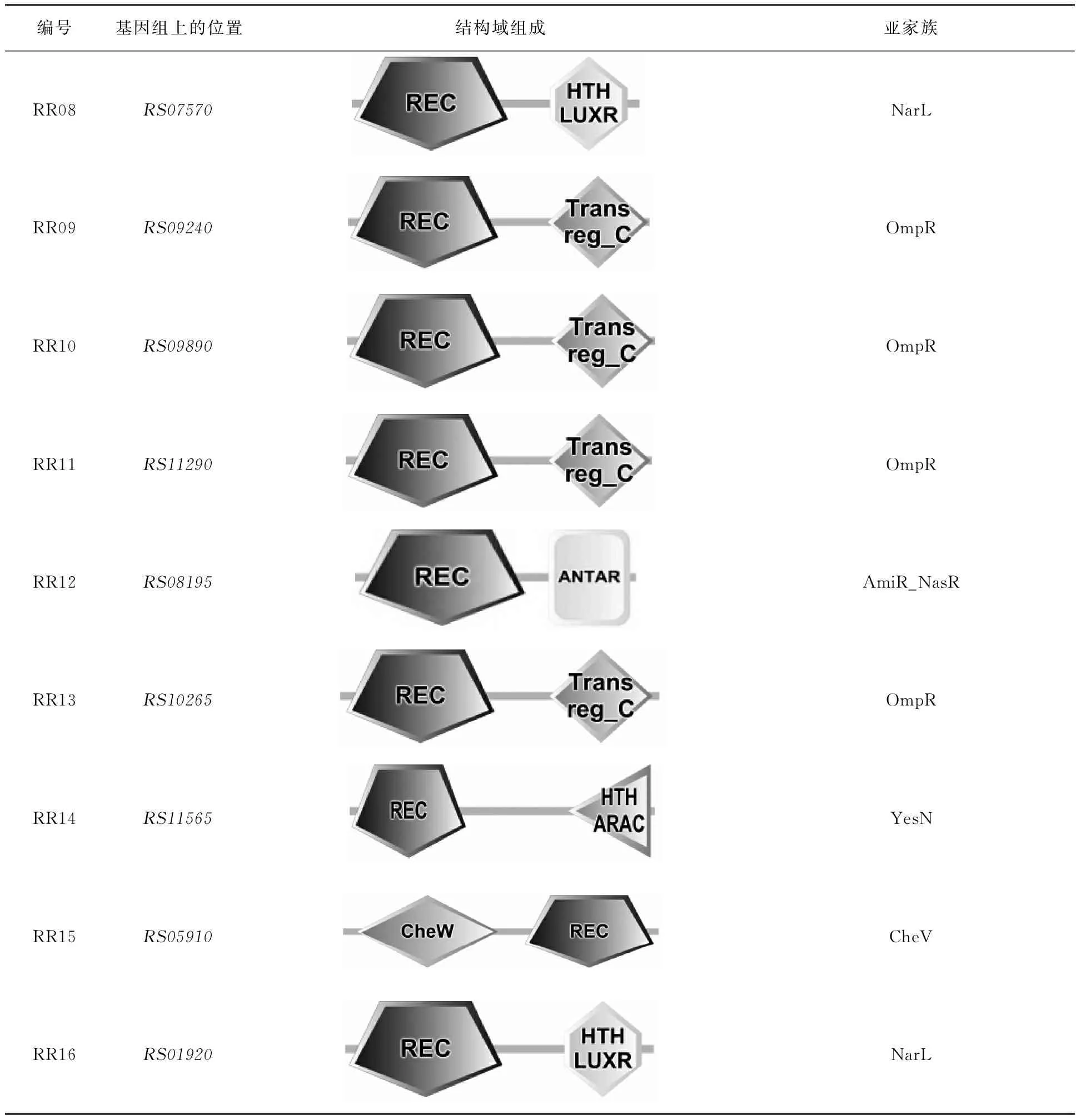
续表3
其中RR01 (RS03875)、RR03 (RS01445)、RR04 (RS01850)、RR05 (RS02250)、RR06 (RS13770)、RR09 (RS09240)、RR10 (RS09890)、RR11 (RS11290)和RR13 (RS10265)这9个RRs的输出区域为Trans reg_C,具有典型的翼状螺旋-转角-螺旋 (HTH) 结构,属OmpR亚家族,占LM201总RRs的56%。OmpR亚家族是目前已知数目最多的一类应答调节蛋白[15]。RR08 (RS07570)和RR16 (RS01920)输出区域为HTH-LuxR,属NarL亚家族,是DNA结合区域,通常发挥转录激活作用[16]。RR07 (RS14980)输出区域为LytTR,是一种新型的保守DNA结合域,属于AlgR/AgrA/LytR转录调控亚家族,此类结构域在GC含量较少的革兰阴性菌中常见,参与细胞的自溶调控[17]。RR12 (RS08195)输出区域为ANTAR,属AmiR_NasR亚家族,目前发现只在细菌中有ANTAR,其是RNA结合域,通过接受磷酸基团发挥转录抗终止作用[18]。RR14 (RS11565)输出区域为HTH-ARAC,属YesN亚家族,HTH为螺旋-转角-螺旋结构,是大多数细菌转录调控蛋白结合DNA的模体,AraC是阿拉伯糖操纵子编码的转录调节蛋白,是含有HTH结构比较多的一类转录调控蛋白,其包含的两个HTH结构形成凹槽以结合同源的DNA[19]。RR15 (RS05910)输出区域为CheW,位于N端,能与CheA发生偶联,属于CheV亚家族,主要影响趋化性调控的稳定性[20]。
由表3可知,LM201中除RR02 (RS05920,属CheY亚家族)无输出区域外,其他的RRs都有C端输出区域。C端输出区域通常与靶DNA或靶蛋白结合,发挥转录因子功能。当RR缺乏输出区域时,其通过其他方式发挥作用,例如,在大肠埃希菌中,与LM201的 RR02同类的RR蛋白CheY被磷酸化后,虽然CheY没有C端输出区域,但其直接结合到被调控蛋白—鞭毛运动调控蛋白FliM或者FliN上,使后者构象发生改变,从而改变细菌鞭毛的运动方向[21]。
2.4 功能预测
功能预测主要根据氨基酸序列一致性(identity)和蛋白质保守结构域进行预测。以上HK和RR的结构分析结果为进行TCS的功能预测打下了基础。利用BLASTP和参考相关文献对搜寻到的TCSs进行了功能预测,结果见表4。比对结果选取一致性最高且功能验证过的TCSs的功能作为参考。
LM201中有14对HK/RR和2个孤儿RR。其中,RS03880/RS03875、RS02245/RS02250和RS09895/RS09890编码的3对TCSs以及RS05910编码的孤儿RR的预测功能在Lm中未见报道,但在其他菌中有报道(表4**);RS11570/RS11565编码的TCS的功能为未知,在Lm和其他菌中均未见报道(表4***);剩余的10对TCSs和一个孤儿RR (RS01920)的预测功能在Lm中已报道(表4*)。
Lm中已报道的TCSs的功能主要涉及毒力(VirSR,DegU)、抗生素抗性(CesKR,AgrAC)、细胞壁渗透压(KdpDE,LiaSR)、氧化还原(ResDE)、趋化性(CheAY)和磷酸酶合成(PhoPR)等。
YycGF、HssSR、YclKJ和CheV的功能在Lm中未见报道,但在其他菌中已有报道,如:YycGF在枯草芽胞杆菌中调控细胞壁的合成[22];HssSR在金黄色葡萄球菌中调控胞内血红素平衡和毒力,将该双组分的RR缺失后,金黄色葡萄球菌对抗菌肽—菌丝霉素的抗性增强,而Lm的亲本野生株与其HssSR的RR缺失突变株对该抗菌肽均不敏感[23],可见,同类TCS在不同细菌中可能有不同的调控作用;YclKJ在枯草芽胞杆菌中调控厌氧生长[24];CheV在枯草芽胞杆菌中调控趋化性[25],与在Lm中已报道的CheAY位于同一个操纵子上,因此,推测CheV在Lm中也可能参与趋化性调控。YycGF、HssSR、YclKJ和CheV在Lm中的功能有待研究。
表4显示,RS11570/RS11565编码的双组分的预测功能为未知,在Lm和其他菌中均未见报道。我们发现,RS11570编码的HK在C端229~1734处存在一个YesM保守结构域,RS11565基因编码的RR含有一个YesN保守结构域,对该双组分进行一致性分析发现,其与枯草芽胞杆菌的YesM/YesN的一致性最高,分别为27%和40%,但其功能在枯草芽胞杆菌中未见报道。所以RS11570/RS11565编码的双组分在Lm中的功能有待探索。

表4 LM201的TCS一致性比较和功能预测
注:Bs:枯草芽胞杆菌(Bacillussubtilis);Lm:单增李斯特菌(Listeriamonocytogenes);Sa:金黃色葡萄球菌(Staphylococcusaureus);*:Lm中功能已被报道的双组分;**:Lm中功能未见报道的双组分,但该双组分功能在其他菌中有报道;***:功能未知的双组分
3 讨 论
细菌耐药性问题一直困扰着人们。细菌有TCSs,哺乳动物没有TCSs,故以TCSs为靶标研发新型抗菌药物是解决细菌耐药问题的有效办法之一。探索Lm的TCSs的交叉调控模式能为该应用前景提供理论依据。本研究通过生物信息学方法探明了Lm菌株LM201中TCSs的数量、结构、分类和预测功能,该结果为构建Lm的TCSs的交叉调控网络提供参考。
[1] Fuchs TM, Eisenreich W, Kern T, et al. Toward a Systemic Understanding ofListeriamonocytogenesMetabolism during Infection [J]. Frontiers in Microbiology, 2012, 3: 23.
[2] 连凯,谈卫军,赵丹,等,2002-2012年人和动物李斯特菌感染报告数据流行病学分析[J].中国人兽共患病学报,2014,10:1033-1038.
[3] Toledo-Arana A, Dussurget O, Nikitas G, et al. The Listeria transcriptional landscape from saprophytism to virulence [J]. Nature, 2009, 459(7249):950-956.
[4] Mascher T, Helmann JD, Unden G. Stimulus perception in bacterial signal-transducing histidine kinases[J]. Microbiology and Molecular Biology Reviews, 2006, 70(4):910-938.
[5] Mandin P, Fsihi H, Dussurget O, et al. VirR, a response regulator critical forListeriamonocytogenesvirulence[J]. Molecular Microbiology, 2005, 57(5):1367-1380.
[6] Agrawal R, Sahoo BK, Saini DK. Cross-talk and specificity in two-component signal transduction pathways[J]. Future Microbiology, 2016, 11:685-697.
[7] Wu Y, Zheng J, Wang Y, et al. Draft Genome Sequence ofListeriamonocytogenesLM201, Isolated from Foodstuff[J]. Genome Announcements, 2015, 3(1): e01417
[8] Williams SB, Stewart V. Functional similarities among two-component sensors and methyl-accepting chemotaxis proteins suggest a role for linker region amphipathic helices in transmembrane signal transduction[J]. Molecular Microbiology, 1999, 33(6):1093-1102.
[9] Ponting CP, Aravind L. PAS: a multifunctional domain family comes to light[J]. Current Biology, 1997, 7(11):R674-677.
[10] Zhulin IB, Taylor BL, Dixon R. PAS domain S-boxes in Archaea, Bacteria and sensors for oxygen and redox[J]. Trends in Biochemical Sciences, 1997, 22(9):331-333.
[11] Aravind L, Ponting CP. The GAF domain: an evolutionary link between diverse phototransducing proteins[J]. Trends in Biochemical Sciences, 1997, 22(12):458-459.
[12] Salvado B, Vilaprinyo E, Sorribas A, et al. A survey of HK, HPt, and RR domains and their organization in two-component systems and phosphorelay proteins of organisms with fully sequenced genomes[J]. Peer J, 2015, 3:e1183.
[13] Wang X, Vallurupalli P, Vu A, et al. The linker between the dimerization and catalytic domains of the CheA histidine kinase propagates changes in structure and dynamics that are important for enzymatic activity[J]. Biochemistry, 2014, 53(5):855-861.
[14] Gegner JA, Graham DR, Roth AF, et al. Assembly of an MCP receptor, CheW, and kinase CheA complex in the bacterial chemotaxis signal transduction pathway[J]. Cell, 1992, 70(6):975-982.
[15] Itou H, Tanaka I. The OmpR-family of proteins: insight into the tertiary structure and functions of two-component regulator proteins[J]. Journal of Biochemistry, 2001, 129(3):343-350.
[16] Fuqua WC, Winans SC, Greenberg EP. Quorum sensing in bacteria: the LuxR-LuxI family of cell density-responsive transcriptional regulators[J]. Journal of Bacteriology, 1994,176 (2):269-275.
[17] Nikolskaya AN, Galperin MY. A novel type of conserved DNA-binding domain in the transcriptional regulators of the AlgR/AgrA/LytR family [J]. Nucleic Acids Research, 2002, 30 (11):2453-2459.
[18] Morth JP, Feng V, Perry LJ, et al. The crystal and solution structure of a putative transcriptional antiterminator fromMycobacteriumtuberculosis[J]. Structure, 2004, 12(9):1595-1605.
[19] Gallegos MT, Michan C, Ramos JL. The XylS/AraC family of regulators[J]. Nucleic Acids Research, 1993, 21(4):807-810.
[20] Alexander RP, Lowenthal AC, Harshey RM, et al. CheV: CheW-like coupling proteins at the core of the chemotaxis signaling network[J]. Trends in Microbiology, 2010, 18(11):494-503.
[21] Sarkar MK, Paul K, Blair D. Chemotaxis signaling protein CheY binds to the rotor protein FliN to control the direction of flagellar rotation inEscherichiacoli[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(20):9370-9375.
[22] Howell A, Dubrac S, Andersen KK, et al. Genes controlled by the essential YycG/YycF two-component system ofBacillussubtilisrevealed through a novel hybrid regulator approach[J]. Molecular Microbiology, 2003, 49(6):1639-1655.
[23] Thomsen LE, Gottlieb CT, Gottschalk S, et al. The heme sensing response regulator HssR inStaphylococcusaureusbut not the homologous RR23 inListeriamonocytogenesmodulates susceptibility to the antimicrobial peptide plectasin[J]. BMC Microbiology, 2010, 10:307.
[24] Hartig E, Geng H, Hartmann A, et al.BacillussubtilisResD induces expression of the potential regulatory genes yclJK upon oxygen limitation[J]. Journal of Bacteriology, 2004, 186(19):6477-6484.
[25] Karatan E, Saulmon MM, Bunn MW, et al. Phosphorylation of the response regulator CheV is required for adaptation to attractants duringBacillussubtilischemotaxis[J]. The Journal of Biological Chemistry, 2001,276 (47):43618-43626.
