哮喘患者气道上皮细胞关键基因的生物信息学分析
2018-03-20周冬梅孔灵菲
周冬梅,孔灵菲
(中国医科大学附属第一医院 1. 老年呼吸感染科; 2. 呼吸内科,沈阳 110001)
支气管哮喘 (简称哮喘) 是一种以慢性气道炎症、黏液高分泌和气道高反应性为主要特征的全世界发病率最高的慢性疾病之一。气道上皮细胞是气道的第一道屏障,大量研究[1]表明,哮喘患者的气道上皮细胞存在功能障碍,其在哮喘的发生发展过程中起到重要作用。气道上皮屏障功能损伤的严重程度与支气管哮喘急性发作的严重程度相关。气道上皮细胞损伤后通过加强促炎活性,释放生长因子等参与支气管哮喘气道炎症与气道重塑[2],找出哮喘患者气道上皮与正常人的差异,有助于哮喘分型及找出新的治疗策略。本研究对正常人与重症哮喘患者气道上皮基因芯片的数据进行分析处理,旨在找出支气管哮喘气道上皮细胞在调控哮喘疾病进展中的关键基因。
1 材料与方法
1.1 基因芯片数据
基因表达数据库 (Gene Expression Omnibus,GEO) http://www.ncbi. nlm.nih.gov/geo是当今最大最全面的公共基因表达数据的储存地址,例如基因芯片和二代测序。从GEO数据库获得关于支气管哮喘患者气道上皮细胞的基因芯片数据GSE43696,实验平台为GPL6480,是美国Agilent公司的全人类基因组芯片。GSE43696包含108个样本,对其中的20例正常对照组和38例重度哮喘的数据进行分析处理,以获得突出的差异表达基因。
1.2 获取差异表达基因
GEO2R是一个交互式网络工具,允许用户进行2组或多组GEO系列样本比较以识别差异基因。调整后P值Adj.p.val<0.05,差异倍数| logFC |>0.6认为其具有表达差异。
1.3 差异表达基因的基因本体及通路富集分析
DAVID (http://david.abcc,ncifcrf.gov) 是一个基于网络的基因功能富集分析软件、整合生物学数据以及功能注释和信息链接为贴点的覆盖广泛的分析工具。使用DAVID在线分析软件对筛选出的差异表达基因进行基因本体 (gene oncology,GO) 分析和京都基因与基因组百科全书 (kyoto encyclopedia of genes and genomes,KEGG) 通路富集分析。P < 0.05为差异有统计学意义。
1.4 差异表达基因的蛋白相互作用网络图及获取关键模块
STRING数据库 (http://http://www.string-db.org/)是一个关于已知或预测蛋白质间相互作用数据组成的数据库。使用STRING对差异基因编码的蛋白进行蛋白-蛋白相互作用网络分析。并将结果上传至Cytoscape中的MOCODE插件,用以获取关键模块。
2 结果
2.1 差异表达基因分析
通过GEO2R对GSE43696中20个正常样本及38个重症哮喘样本进行差异表达基因的分析。Adj.p.val<0.05,| logFC |>0.6作为差异表达基因的标准。共获得355个差异表达基因,其中130个上调基因,225个下调基因,见表1。上调及下调差异表达基因的前50个做热图,见图1。
2.2 差异表达基因的基因本体分析
将所有差异表达基因上传至DAVID进行GO分析及KEGG分析。GO分析结果 (表2) 显示,上调的差异表达基因在生物学过程中主要涉及细胞运动,较多基因定位于膜小泡和胞外区,在分子功能方面主要涉及生长因子结合和蛋白二聚化。下调的差异表达基因在生物学过程中主要涉及离子转运和转运调节,细胞成分主要涉及突触和细胞连接,在分子功能方面主要涉及通道活性。
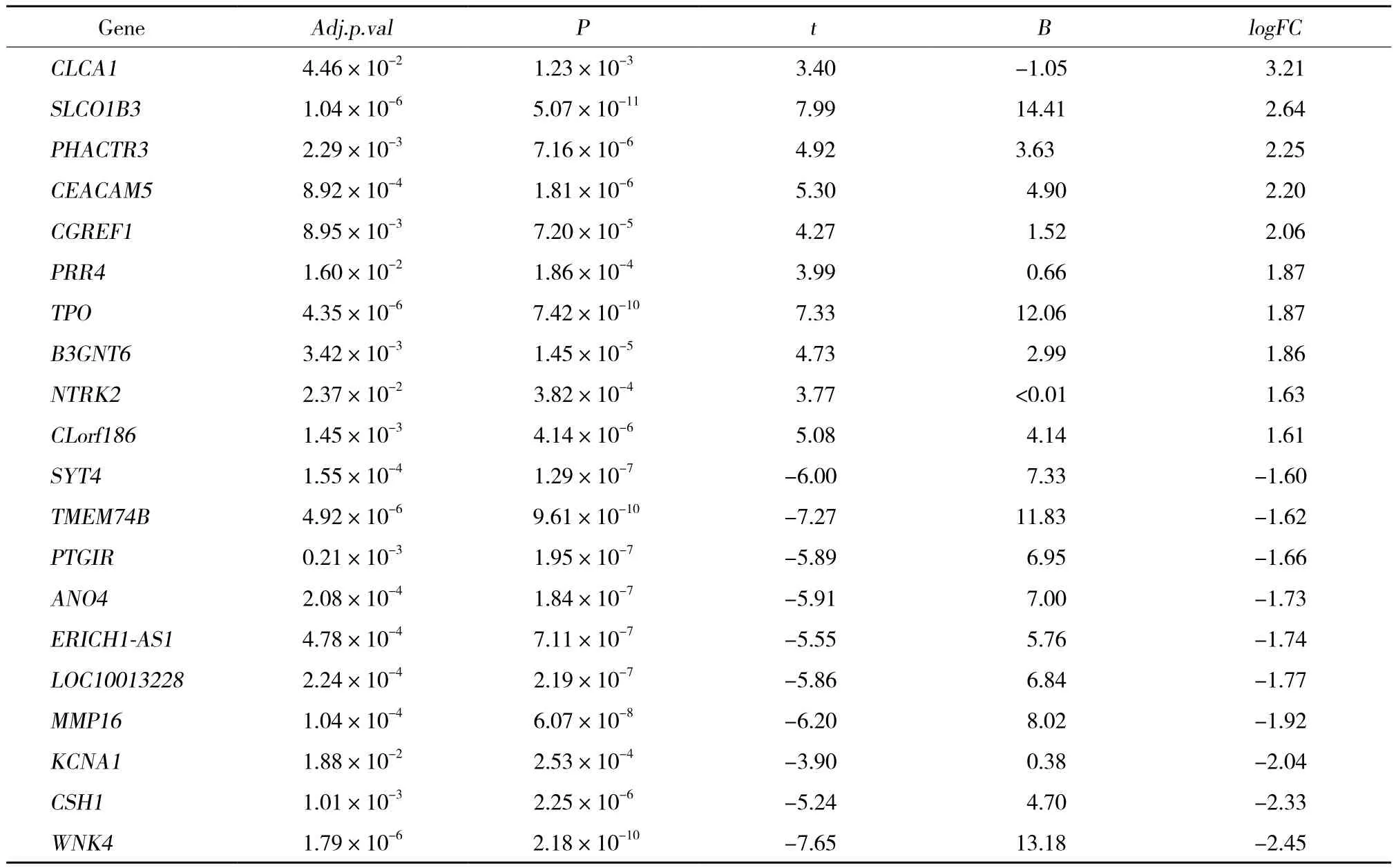
表1 前10名上调及下调差异表达基因Tab.1 Top 10 up-regulated and down-regulated DEGs

图1 前50名上调和下调差异表达基因的热图Fig.1 Heat map of top 50 up-regulated and down-regulated DEGs
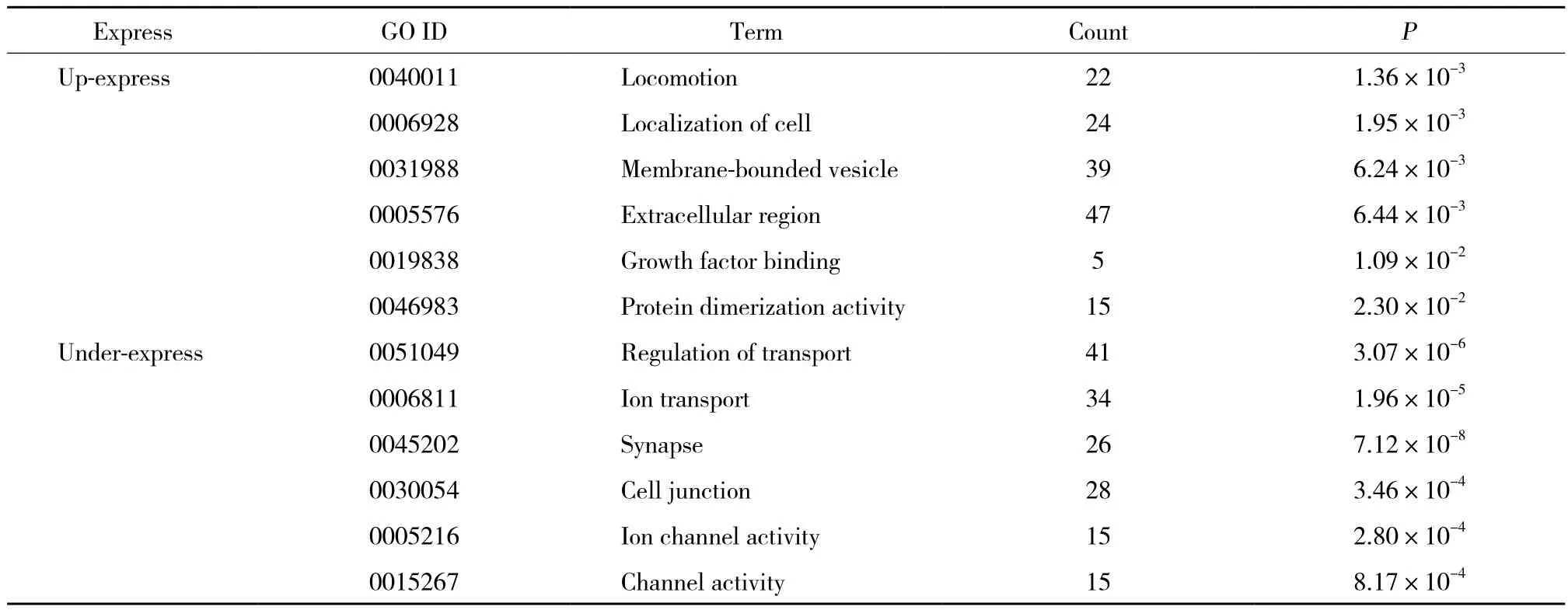
表2 差异表达基因的基因本体分析Tab.2 Gene oncology analysis of DEGs
2.3 差异表达基因的通路富集分析
KEGG分析结果显示,上调的差异表达基因主要涉及半乳糖、果糖和甘露糖代谢,而下调的差异表达基因主要涉及色氨酸代谢和神经活性受体及配体相互作用,见表3。
2.4 蛋白相互作用网络建设及模块的选择
差异表达基因的蛋白相互作用网络包含329个节点和 337个边 (图2A) 。关联值≥10作为关键基因的标准。共筛出 9个关键基因,包括脑源性神经生长因子受体 (brain derived neurotrophic factor,BDNF) ,原癌基因-受体酪氨酸激酶 (proto-oncogene receptor tyrosine kinase,KIT) ,腺苷酸环化酶4 (adenylate cyclase 4,ADCY4) ,神经介素U (neuromedin U,NMU),ERB-B2受体酪氨酸激酶2 (erb-b2 receptor tyrosine kinase 2,ERBB2) ,白细胞介素 (interleukin,IL) 6,血管内皮生长因子A (vascular endothelial growth factor A,VEGFA) ,蛋白激酶依赖性Ⅱ型调节亚单位β (protein kinase cAMP-dependent typeⅡregulatory subunit beta,PRKAR2B) ,趋化因子受体6 (C-C motif chemokine receptor 6,CCR6) 。通 过Cytoscape下 的MCODE,筛出了一个最显著的模块,包含9个节点,36个边 (图2 B) ,主要涉及环磷酸腺苷 (cyclic adenosine monophosphate,cAMP) 通路及趋化因子信号通路。

表3 差异表达基因的通路富集分析Tab.3 Pathway enrichment analysis of DEGs
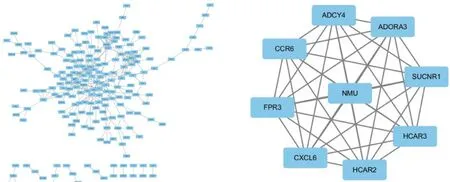
图2 差异表达基因的蛋白互作网络图及关键模块Fig.2 Significant modules in the protein-protein interaction network of DEGs in bronchial asthma
3 讨论
气道上皮细胞是气道的第一道屏障,通过纤毛摆动,释放抗微生物肽、趋化因子和细胞因子,招募并激活其他类型的免疫细胞来清除病原体。在支气管哮喘中,气道上皮细胞的屏障功能障碍,多项研究[3-5]均强调哮喘中气道上皮细胞是气道炎症的来源之一,有望成为治疗哮喘的靶细胞。近年来,基因芯片技术被广泛用于揭示疾病进展过程中的遗传改变。对哮喘气道上皮细胞基因芯片数据进行分析,有助于发掘针对气道上皮细胞治疗哮喘的靶基因。
利用GEO2R对GSE43696基因芯片数据进行分析,获得重症哮喘与正常人气道上皮细胞差异表达的355个基因,其中130个上调基因,225个下调基因。通过蛋白相互作用网络,筛选出9个关键基因,包 括BDNF、ERBB2、IL6、VEGFA、KIT、ADCY4、PRKAR2B、CCR6和NMU。IL-6是重要的Th2型炎症介质,CCR6可诱导淋巴细胞归巢,BDNF的上调可导致气道高反应,与哮喘的严重程度和Th2型气道炎症相关[6],而ERBB2是表皮生长因子受体之一,介导慢性哮喘小鼠模型气道高反应和重塑[7]。NMU可以诱导肥大细胞脱颗粒,NMU敲除的哮喘小鼠嗜酸性粒细胞浸润显著下降[8]。在儿童哮喘中,VEGFA的基因多态性与哮喘预后相关[9],而ADCYAP1R1的基因多态性与哮喘患病率相关[10]。对哮喘小鼠注射KIT基因的干扰RNA,可抑制气道炎症、IL-4和IL-5的产生[11]。PRKAR2B基因编码AMP依赖的蛋白激酶类型Ⅱ型调节亚单位β,近期研究[12]显示,树突细胞通过cAMP途径调节Th2型免疫反应,其中ADCY3增加,ADCY4降低。但尚没有ADCY4与哮喘的具体研究。
MECODE筛选的关键模块涉及的关键通路为cAMP及趋化因子信号通路。其中,SUCNR1 (succinate receptor 1) 为琥珀酸受体1。琥珀酸是三羧酸循环的中间产物,在线粒体ATP代谢过程中起至关重要的作用。SUCNR1代谢功能的障碍可导致肥胖相关糖尿病及肾功能障碍[13],但除了代谢功能外,SUCNR1还参与炎症反应的调节[14]。疾病研究中,敲除SUCNR1可以增强肥大细胞的活性,减轻关节炎,但在哮喘中其作用尚不清楚。
通过生物信息学方法,分析哮喘气道上皮细胞潜在的差异基因及信号通路,找到9个关键基因。BDNF、ERBB2、IL6、VEGFA、KIT、CCR6和NMU在支气管哮喘已多有研究,但有些基因在气道上皮细胞中的作用还有待进一步探索。趋化因子信号通路是哮喘的气道上皮细胞的重要信号通路,亦多有研究,但cAMP通路在哮喘中还有待更进一步探索。这些实验研究结果表明通过挖掘数据,并利用生物信息学分析工具可预测哮喘气道上皮功能障碍中的关键基因及通路,为哮喘的诊治提供潜在的治疗目标。
[1] XIAO C,PUDDICOMBE SM,FIELD S,et al. Defective epithelial barrier function in asthma[ J]. J Allergy Clin Immunol,2011,128( 3) :549-556. DOI:10.1016/j.jaci.2011.05.038.
[2] NAWIJN MC,HACKETT TL,POSTMA DS,et al. E-cadherin:gatekeeper of airway mucosa and allergic sensitization[ J]. Trends Immunol,2011,32( 6) :248-255. DOI:10.1016/j.it.2011.03.004.
[3] PARK JA,KIM JH,BI D,et al. Unjamming and cell shape in the asthmatic airway epithelium[ J]. Nat Mater,2015,14( 10) :1040-1048. DOI:10.1038/nmat4357.
[4] FREISHTAT RJ,WATSON AM,BENTON AS,et al. Asthmatic airway epithelium is intrinsically inflammatory and mitotically dyssynchronous[ J]. Am J Respir Cell Mol Biol,2011,44( 6) :863-869.DOI:10.1165/rcmb.2010-0029OC.
[5] GRAS D,CHANEZ P,VACHIER I,et al. Bronchial epithelium as a target for innovative treatments in asthma[ J]. Pharmacol Ther,2013,140( 3) :290-305. DOI:10.1016/j.pharmthera.2013.07.008.
[6] WATANABE T,FAJT ML,TRUDEAU JB,et al. Brain-derived neurotrophic factor expression in Asthma. Association with severity and type 2 inflammatory processes[ J]. Am J Respir Cell Mol Biol,2015,53( 6) :844-852. DOI:10.1165/rcmb.2015-0015OC.
[7] LE CRAS TD,ACCIANI TH,MUSHABEN EM,et al. Epithelial EGF receptor signaling mediates airway hyperreactivity and remodeling in a mouse model of chronic asthma[ J]. Am J Physiol Lung Cell Mol Physiol,2011,300( 3) :L414-421. DOI:10.1152/ajplung.00346.2010.
[8] MORIYAMA M,FUKUYAMA S,INOUE H,et al. The neuropeptide neuromedin U activates eosinophils and is involved in allergen-induced eosinophilia[ J]. Am J Physiol Lung Cell Mol Physiol,2006,290( 5) :L971-977. DOI:10.1152/ajplung.00345.2005.
[9] BALANTIC M,RIJAVEC M,SKERBINJEK KAVALAR M,et al.Asthma treatment outcome in children is associated with vascular endothelial growth factor A ( VEGFA) polymorphisms[ J]. Mol Diagn Ther,2012,16( 3) :173-180. DOI:10.2165/11631710-000000000-00000.
[10] CHEN W,BOUTAOUI N,BREHM JM,et al. ADCYAP1R1 and asthma in Puerto Rican children[ J]. Am J Respir Crit Care Med,2013,187( 6) :584-588. DOI:10.1164/rccm.201210-1789OC.
[11] WU W,WANG T,DONG JJ,et al. Silencing of c-kit with small interference RNA attenuates inflammation in a murine model of allergic asthma[ J]. Int J Mol Med,2012,30( 1) :63-68. DOI:10.3892/ijmm.2012.963.
[12] CHINN AM,LEE J,HERDMAN S,et al. Involvement of the cyclic amp pathway in dendritic cell regulation of Th2 immune responses[J]. FASEB J,2016,30( 969) :19.
[13] CARMONE C,ROBBEN J,DIEPEN JV,et al. The succinate receptor contributes to obesity-induced diabetes type-ii and chronic kidney disease[ J]. FASEB J,2015,29( 961) :3.
[14] MILLS E,O’NEILL LA. Succinate:a metabolic signal in inflammation[ J]. Trends Cell Biol,2014,24( 5) :313-320. DOI:10.1016/j.tcb.2013.11.008.
