五个地方绵羊种群mtDNA D-loop区系统进化及遗传多样性分析
2018-03-15梁瑞圆陈晓勇孙洪新孙少华敦伟涛沈志强刘军峰
梁瑞圆,陈晓勇,孙洪新,孙少华,敦伟涛,沈志强,刘军峰,王 珏
(1.河北省畜牧兽医研究所,河北 保定 071000;2.河北农业大学 动物科技学院,河北 保定 071000;3.山东省滨州畜牧兽医研究院,山东 滨州 256600)
我国地方绵羊种质资源非常丰富[1],其中包括以高产羔数著称的湖羊;以肉质好著称的苏尼特羊[2];以常年发情和高繁殖力著称的小尾寒羊[3]等地方绵羊品种,但由于对地方品种的特性认识和保护不够以及受到外来品种引入的影响,这些地方良种绵羊品种资源数量和种类都在不同程度上面临着严重的危机[4],如兰州大尾羊、湖羊濒临灭绝[4-5],甚至枣北大尾羊已经灭绝[6]。因此,开展现有绵羊品种的起源、遗传分类、遗传归属和品种保护,了解地方绵羊品种间和品种内的亲缘关系、起源进化和遗传多样性对于品种保护和开发利用尤为重要。小尾寒羊是我国以繁殖力著称的地方品种,小尾寒羊主要分布在山东省南部、河北黑龙港流域、河南省北部等地,主要分为2个类型:山东小尾寒羊和河北小尾寒羊,山东小尾寒羊主要特征是腿高、体格大;河北小尾寒羊主要分布在河北省黑龙港流域,四肢较山东小尾寒羊短,但尾巴较圆大,且公母羊均无角[7]。此外,湖羊、洼地绵羊和苏尼特羊均以繁殖性能好为主要特征。随着肉羊杂交利用和大量外来品种的引入,不同地方品种绵羊均存在较为广泛的血统交流,目前,上述地方品种是否存在血统交流,是否存在与外来品种血统交叉,以及遗传多样性如何等问题均有待分析。
线粒体DNA(Mitochondrial DNA,mtDNA)是动物体内唯一长期稳定存在的核外遗传物质,因其具有重组率低、替换速度快、拷贝数高、严格母系遗传、结构稳定等特点被广泛用于动物起源进化和遗传多样性等研究[8-13]。因此,本研究分析了5个地方绵羊种群mtDNA 控制区(Displacement loop,D-loop)遗传变异及序列特征、遗传多样性和种间、种内遗传距离,目的是为深入挖掘我国地方品种绵羊起源进化、种质资源和品种保护利用提供理论依据。
1 材料和方法
1.1 样品准备
每个地方绵羊种群血液样品均取自健康的不同母源家系(表1)。从NCBI网站下载3条绵羊序列作为参照序列:滩羊GenBank 登录号 KF938336.1(简称:TAN);阿勒泰GenBank 登录号 KF938320.1(简称:ALT);山东大尾寒羊GenBank 登录号 KP981380.1(简称:SDW)。
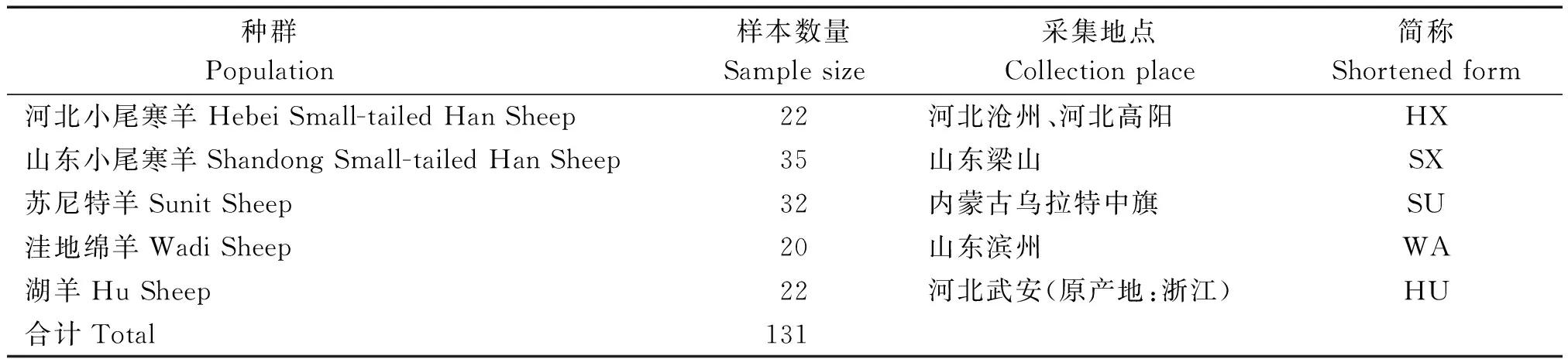
表1 样本信息Tab.1 Information of sample
1.2 DNA提取
参照康为世纪的血液基因组柱式试剂盒说明书。
1.3 PCR扩增及测序
引物信息参照文献[14]中的P18。PCR反应体系20 μL:DNA 1.6 μL,10×PCR Buffer 2 μL,dNTP(10 mmol/L)0.4 μL,TaqDNA聚合酶(5 U/μL)0.12 μL,上、下游引物(10 mmol/L)0.8 μL,双蒸水14.28 μL。PCR反应条件:95 ℃预变性5 min;95 ℃变性20 s,58 ℃退火30 s,72 ℃延伸90 s,35个循环;72 ℃延伸5 min。将扩增产物送上海生工进行双向测序。
1.4 数据分析
将测序获得的样本序列与参考序列(GenBank:AF010406)进行比对,确定样本序列的序列长度和突变位点信息。使用BioEdit 7.0和DNAStar-SeqMan软件根据测序峰图的实际情况,将测序获得的DNA序列进行比对、拼接及人工校正,利用ClustalX[15]软件进行序列比对后保存比对结果。使用DnaSP 5.10[16]软件进行分析序列间的碱基差异,获得各个样品的单倍型信息,计算单倍型多样度、核酸多样度、平均核苷酸差异数等参数,并进行中性检验。利用MEGA 6.0[17]软件确定单倍型,计算序列长度、碱基组成、突变位点数、遗传距离等参数,并且基于Kumar双参数模型进行种群内遗传距离及分子系统分析,构建邻接法(Neighbor-joining,NJ)、最小进化法(Minimum-evolution method,ME)和非加权组平均法(Unweighted pair-group method with arithmetic means,UPGMA)系统发育树,利用Bootstrap法重复1 000次。对DnaSP 5.10获得的Haplotype进行网络分析,使用Network 5.0软件构建网络关系图。
2 结果与分析
2.1 PCR扩增结果
对D-loop序列PCR扩增后电泳检测结果条带清晰可见,且条带大小1 291 bp与引物设计长度一致(图1)。
2.2 序列特征分析
5个绵羊种群131只个体mtDNA D-loop区序列长度为1 106~1 242 bp,其中长度为1 106 bp 的个体有6只,1 180 bp 的有45只,1 181 bp 的有58只,1 182 bp 的有10只,1 183 bp 的有6只,其他长度个体有6只(表2)。不同长度差异的主要原因是75 bp串联重复序列的重复次数不同,少数为插入和缺失引起的变异。
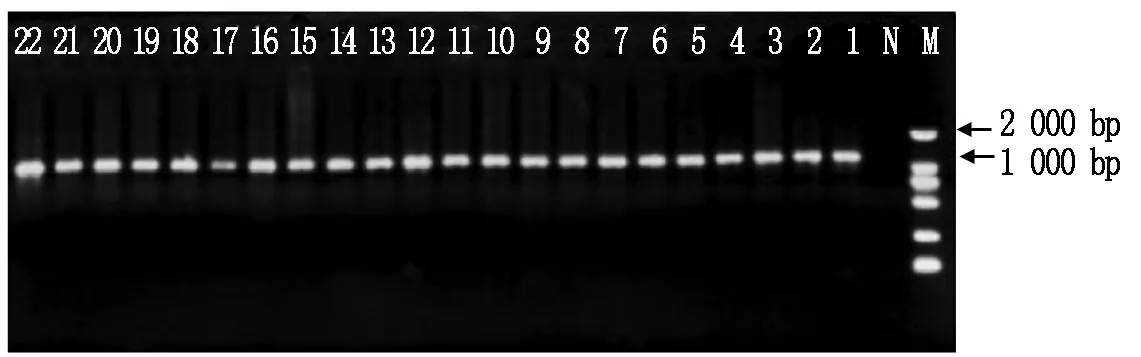
N.空白对照;M.Marker;1~22.河北小尾寒羊22个样品D-loop区扩增结果。N.Blank control;M.Marker;1-22.Amplification production for D-loop of Hebei Small-tailed Han Sheep.
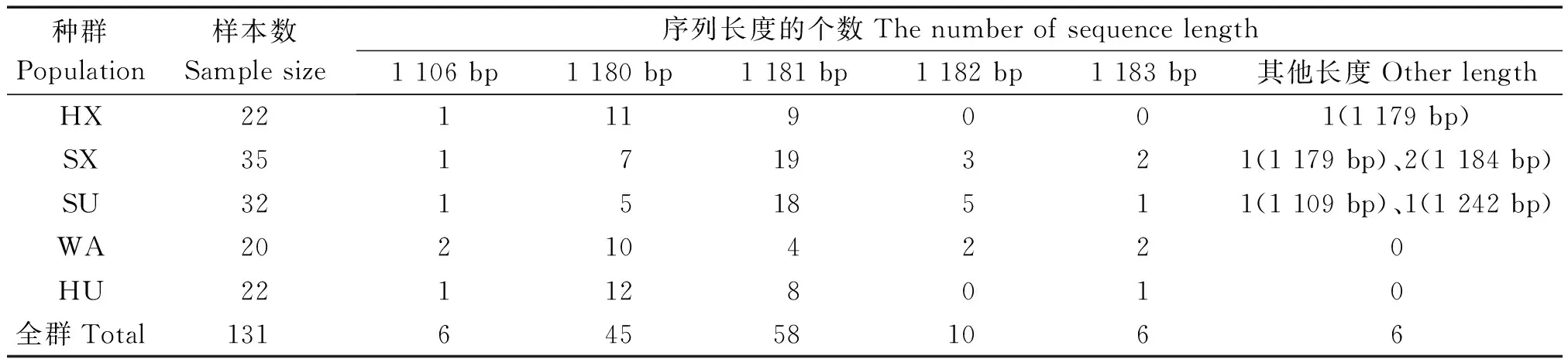
表2 五个中国地方绵羊群体D-loop区序列特征Tab.2 The features of D-loop sequence in five Chinese local sheep population 只
2.3 多态性及单倍型分析
mtDNA D-loop区序列A、T、C、G碱基的平均含量分别为33.1%,29.7%,22.9%,14.3%,其中AT含量总和高于CG含量总和(62.8%>37.2%),表现出AT偏移性。mtDNA D-loop区序列共包含135个多态变异位点,其中包括37个单一多态位点,占总数的27.4%。98个简约多态位点,占总数的72.6%。所有多态位点中除了插入缺失外,共出现了132次转换,3次颠换。在5个种群中,山东小尾寒羊多态位点最多,湖羊多态位点最少(表3)。
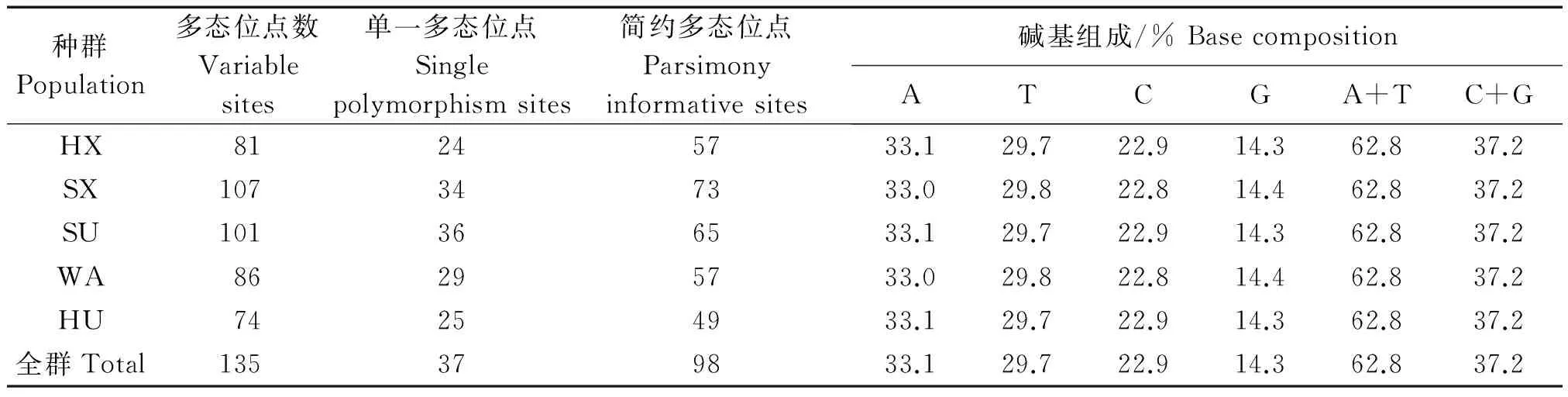
表3 五个中国地方绵羊群体D-loop 区多态性及碱基组成Tab.3 Polymorphism and base composition of D-loop in five Chinese local sheep population
5个中国地方绵羊群体D-loop区序列中共含有108种单倍型,单倍型比例为82.4%,绵羊种群单倍型多样度为0.995 0±0.002 1,其中山东小尾寒羊最高,为0.998 0,其次是河北小尾寒羊、洼地绵羊、苏尼特羊分别为0.996 0,0.995 0,0.992 0,最少的是湖羊为0.974 0;核苷酸多样度为0.017 20~0.022 22,其中河北小尾寒羊最高,为0.022 22。其次是山东小尾寒羊、湖羊、洼地绵羊分别为0.022 21,0.021 72,0.021 57,苏尼特羊最低,为0.017 20。全群平均核苷酸差异数K为20.795(表4)。所有数据均表明遗传多样性相对丰富。Tajima′s D中性检验结果(值变化-1.101 05~0.725 08)均不显著(P>0.10),说明状态平稳,符合中性突变(表4)。
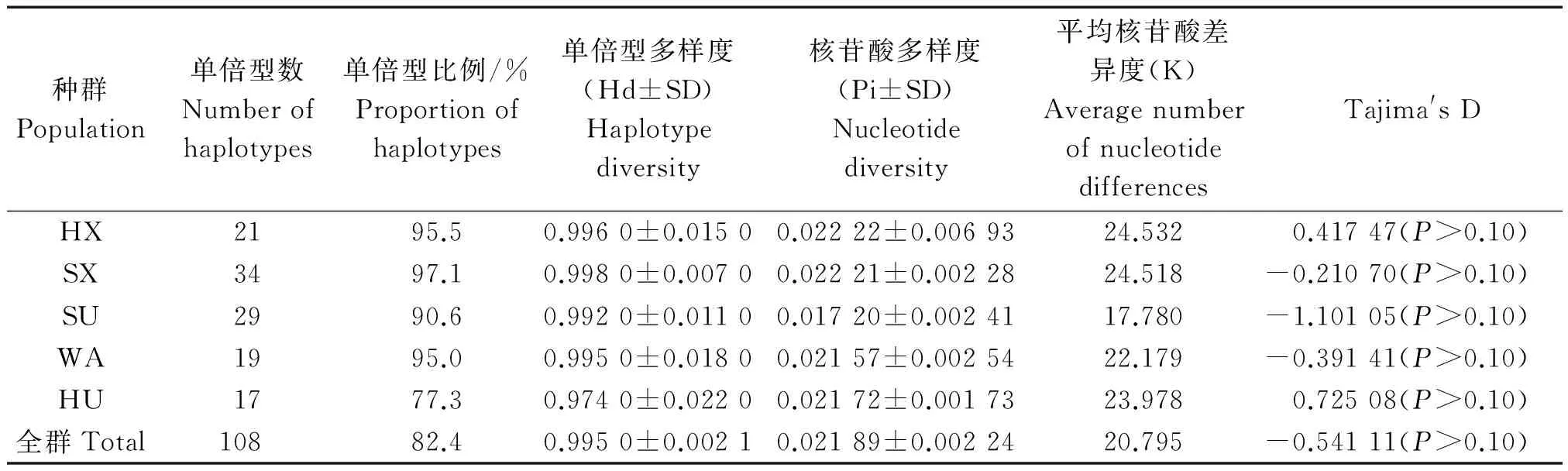
表4 五个中国地方绵羊群体D-loop 区遗传多样性参数Tab.4 Genetic diversity parameters of D-loop in five Chinese local sheep population
2.4 各种群间亲缘关系分析
利用MEGA 6.0 软件获得5个地方绵羊种群种内及种间遗传距离,种间遗传距离为0.019 5~0.027 7,山东小尾寒羊和苏尼特羊之间的遗传距离最近,为0.019 5;洼地绵羊和苏尼特羊之间的遗传距离最远,为0.027 7。种内遗传距离为0.016 5~0.021 8,苏尼特羊种内遗传距离较近,为0.016 5;河北小尾寒羊种内遗传距离较远,为0.021 8(表5)。各种群间遗传距离的系统发育分析表明,河北小尾寒羊与洼地绵羊先聚在一起,再与湖羊聚在一起,最后与苏尼特羊和山东小尾寒羊聚在一起,说明河北小尾寒羊与洼地绵羊遗传距离很近(图2)。
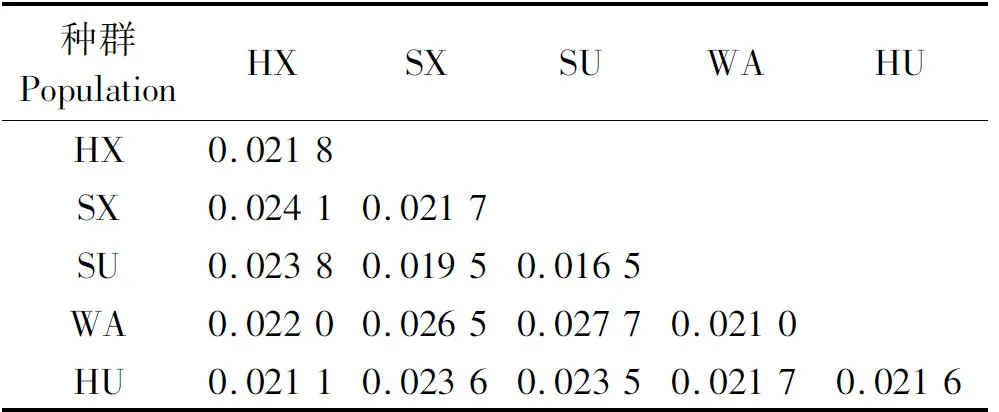
表5 五个绵羊种群间及种内D-loop区遗传距离Tab.5 Genetic distance five of D-loop varieties and intraspecific in five sheep population
注:对角线为种群内遗传距离,对角线以下为种间遗传距离。
Notes:On the diagonal was genetic distance within breeds,under the diagonal was genetic distance between breeds.

图2 基于D-loop区构建的5个绵羊群体遗传距离NJ树Fig.2 Genetic distance NJ tree of D-loop in five sheep population
2.5 系统发育分析
通过MEGA 6.0采用Kumar双参数Bootstrap重复1 000次检验,对5个绵羊群体和山东大尾寒羊(SDW)、阿勒泰羊(ALT)、滩羊(TAN)D-loop控制区构建NJ(图3)、ME树(图4)以及UPGMA聚类图(图5),在mtDNA D-loop区所有的聚类图中,均表明支系A包括70只绵羊个体,B包括48只个体,C包括13只个体,3个支系分别占53.4%,36.7%,9.9%。其中ALT和SDW均与支系C聚在一起,TAN与支系A聚在一起。除支系C外,支系A和支系B均有河北小尾寒羊分布,表明该种群与其他支系存在基因交流。
利用Network 5.0绘制D-loop区序列的单倍型网络关系图,结果显示,131只个体D-loop区序列的108种单倍型聚为3个单倍型群,与NJ树、ME树及UPGMA聚类图分析结果一致,A单倍型群包括70只个体,其中苏尼特羊26只,山东小尾寒羊23只,湖羊9只,河北小尾寒羊9只,洼地绵羊3只;B单倍型群包括48只个体,其中河北小尾寒羊13只,山东小尾寒羊8只,苏尼特羊5只,湖羊12只,洼地绵羊10只;C单倍型群包括13只个体,其中山东小尾寒羊4只,苏尼特羊1只,湖羊1只,洼地绵羊7只。其中单倍型H_8和H_23分别在A、B单倍型群的中心位置,是2个主要的共享单倍型。另外,单倍型H_14、H_27、H_30、H_34、H_43、H_45、H_49、H_51、H_85、H_89均为共享单倍型(图6)。表明河北小尾寒羊与其他上述地方绵羊种群间存在一定的基因交流。
3 讨论与结论
中国养羊历史悠久,绵羊种类繁多,广泛分布于从高海拔的青藏高原到地势较低的东部地区。根据地理分布和遗传关系,中国家养绵羊可划分为三大系谱:藏系绵羊、蒙古系绵羊、哈萨克系绵羊[1]。其中蒙古系绵羊是由中亚山脉地区的野生原羊演化而来,同羊、小尾寒羊、湖羊、滩羊等品种是其亚种。本研究所选5个地方绵羊种群均有蒙古羊血统,其中河北小尾寒羊无论从外貌特征还是生产性能方面均不同于山东小尾寒羊,而与洼地绵羊较为相似,故将其作为独立种群进行分析,从分子方面证实其起源及归属问题。
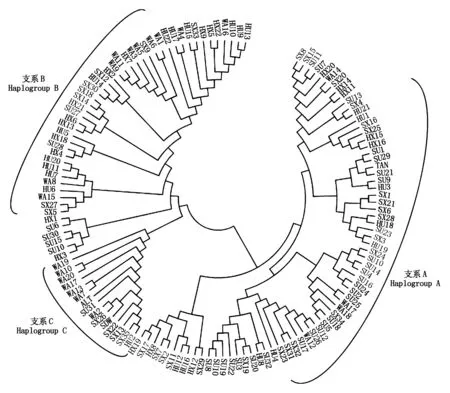
图3 五个绵羊群体mtDNA D-loop区NJ树Fig.3 NJ tree of mtDNA D-loop in five sheep population
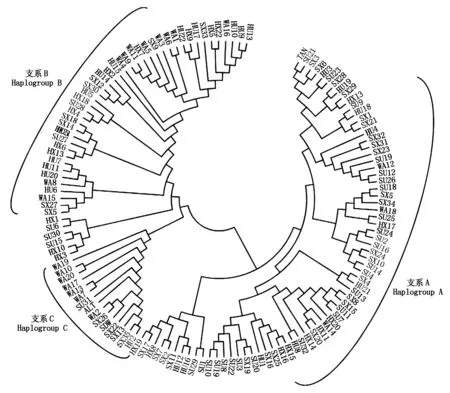
图4 五个绵羊群体mtDNA D-loop区ME树Fig.4 ME tree of mtDNA D-loop in five sheep population
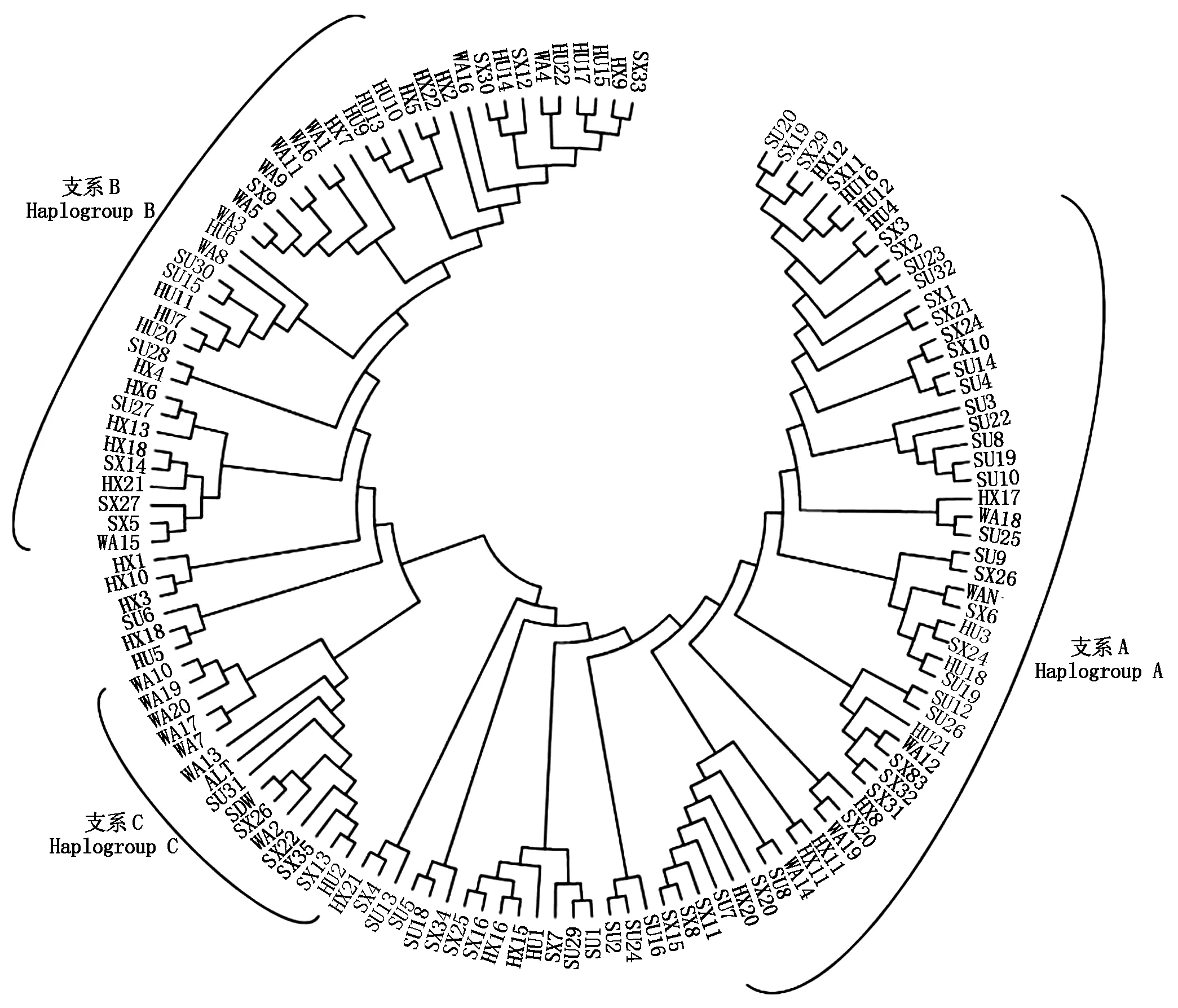
图5 五个绵羊群体mtDNA D-loop区UPGMA聚类图Fig.5 UPGMA cluster of mtDNA D-loop in five sheep population
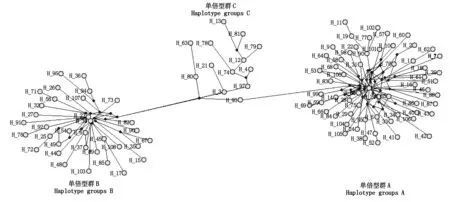
图6 五个绵羊群体mtDNA D-loop单倍型网络图Fig.6 The haplotypes network of mtDNA D-loop in five sheep population
mtDNA结构简单且稳定,严格遵守母系遗传,并在世代传递中没有重组,因此,可以用于探讨绵羊的起源进化,分析绵羊群体的遗传多样性。本研究分别对河北小尾寒羊、山东小尾寒羊、洼地绵羊、湖羊、苏尼特羊等5个种群的线粒体DNA D-loop区序列分析,序列长度在1 106~1 242 bp,长度差异的主要原因是75 bp串联重复序列的重复次数不同,少数为插入和缺失引起的变异,与Hiendleder[18]的研究一致。mtDNA D-loop区AT含量总和高于CG含量总和(62.8%>37.2%),表现出明显的AT偏移性,这与脊椎动物mtDNA D-loop 区碱基组成的特点相符合。单倍型多样度(Hd)和核苷酸多样度(Pi)是衡量一个群体mtDNA遗传分化和遗传多样性的2个重要指标,结果值越大,群体的遗传多样性越丰富,反之则越贫乏。其中单倍型多样度反映的是群体变异的程度,即群体的多样性,而核苷酸多样度是序列内核苷酸变异的程度,即个体碱基突变的多样性。本研究对5个绵羊群体131只个体的mtDNA D-loop区全序列包含135个多态位点,108种单倍型,单倍型多样性和核苷酸多样性分别为0.995 0和0.021 89,这表明遗传多样性丰富,这与韩旭等[19]的研究结果一致,其中苏尼特羊单倍型多样性和核苷酸多样性低于其他种群,说明本研究中苏尼特羊群体遗传多样性贫乏。
种间遗传距离表明不同种群之间的亲缘关系远近,种内遗传距离一定程度上反映了该种群自身的遗传多样性。通过mtDNA D-loop区遗传距离分析,结果表明,山东小尾寒羊和苏尼特羊之间的遗传距离最近,这与山东小尾寒羊的起源有关。洼地绵羊和苏尼特羊之间的遗传距离相对较远,这与娄渊根等[20]利用微卫星分析DNA遗传多样性的研究结果一致。从体型外貌来看,蒙古羊体型较大,公羊有螺旋形大角,尾巴较小,尾尖卷曲呈S形,而洼地绵羊体型适中,公母均无角,尾巴肥厚,呈方圆形,尾宽大于尾长。来自中亚的脂尾绵羊被移民中的回民带到了滨州[21],与元朝留下的蒙古羊杂交后,逐渐形成了洼地绵羊种群。因此,洼地绵羊具有蒙古羊和中亚脂尾绵羊血统。洼地绵羊与河北小尾寒羊遗传距离很近,从地理位置上来看,洼地绵羊主要产区位于山东省滨州,与河北小尾寒羊的主要产区河北省黑龙港流域相临。2种绵羊的体型外貌特征及生产性能等各方面均比较相似,可能两者因距离较近而有一定的基因交流;经历史考证,湖羊属于蒙古羊系统[22],湖羊的湖最初是胡服的胡,这可能是由“胡人、胡马”等引申而来,后来才改写成现在的湖,这也证明了湖羊来源于蒙古羊;D-loop种内遗传距离分析表明,苏尼特羊种内遗传距离较近,说明了苏尼特羊的种内遗传多样性相对贫乏。
通过构建聚类图,在mtDNA D-loop区NJ、ME树以及UPGMA图中,均表明阿勒泰羊和山东大尾寒羊均与支系C聚在一起,说明该支系与ALT和SDW有基因交流。TAN与支系A聚在一起。除支系C中不存在河北小尾寒羊个体外,其余各种群均在3个支系中有分布,表明各个种群之间有一定的基因交流,绵羊种群个体之间的碱基差异不明显,不足以完全区分开不同种群个体,这可能与历史的迁移和畜禽流动有关。
河北小尾寒羊、山东小尾寒羊、苏尼特羊、湖羊、洼地绵羊遗传多样性较为丰富,河北小尾寒羊与山东小尾寒羊存在一定遗传差异,而与洼地绵羊更为接近。
致谢:感谢中国农业科学院北京畜牧兽医研究所储明星团队提供的苏尼特羊血液样品,感谢中国科学院遗传与发育生物学研究所向海博士后在数据分析中给予的帮助。
[1] 国家畜禽遗传资源会组.羊志[M].北京:中国农业出版社,2011:62-65.
[2] 魏彩虹,杜立新.我国肉用绵羊育种现状与未来发展方向[J].中国草食动物科学,2012(S):454-457.
[3] 敦伟涛.怎样提高肉羊舍饲效益[M].北京:金盾出版社,2016:46-47.
[4] 高凤明,白乙尔图,刘 金,等.苏尼特羊及羊肉的品质与营养[J].中国畜牧兽医文摘,2014(12):43-44.
[5] 吕宝铨.抢救湖羊品种资源已刻不容缓[J].畜牧与兽医,2007,39(1):23-25.
[6] 阴卫兵.畜禽品种资源多样性的保护[J].郑州牧业工程高等专科学校学报,1999(4):283-284.
[7] 刘肇清.河北省家畜家禽品种志[M].北京:中国农业出版社,2009:20-23.
[8] Zhao E H,Yu Q,Zhang N Y,et al.Mitochondrial DNA diversity and the origin of Chinese indigenous sheep[J].Tropical Animal Health and Production,2013,45(8):1715-1722.
[9] 张 冰,廖海洪,农素群,等.广西5个猪种和国外3个猪种mtDNA D-loop序列遗传多样性及进化分析[J].中国畜牧兽医,2015,42(7):1807-1815.
[10] 郭彦斌,刘丑生,王 慧,等.利用mtDNA D-loop区研究中国10个绵羊品种的遗传多样性与起源[J].农业生物技术学报,2012,20(7):799-806.
[11] Arora R,Yadav H S,Mishra B P.Mitochondrial DNA diversity in Indian sheep[J].Livestock Science,2013,153(1/3):50-55.
[12] Othman O E,Pariset L,Balabel E A,et al.Genetic characterization of Egyptian and Italian sheep breeds using mitochondrial DNA[J].Journal of Genetic Engineering & Biotechnology,2015,81(1):79-86.
[13] 苗永旺,孙利民,王素芳,等.云南3个地方鸡群体mtDNA D-loop遗传多样性分析[J].云南农业大学学报:自然科学版,2015,30(3):395-401.
[14] 梁瑞圆,陈晓勇,孙洪新,等.河北小尾寒羊mtDNA变异及遗传多样性分析[J].畜牧与兽医,2017,49(4):12-18.
[15] Thompson J D,Gibson T J,Plewniak F,et al.The ClustalX Windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools.Nucleic Acids Res[J].Nucleic Acids Research,1997,25(24):4876-4882.
[16] Librado P,Rozas J.DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J].Bioinformatics,2009,25(11):1451-1452.
[17] Tamura K,Stecher G,Peterson D,et al.MEGA6:molecular evolutionary genetics analysis version 6.0[J].Molecular Biology and Evolution,2013,30(12):2725-2729.
[18] Hiendleder S.Molecular characterization of the sheep mitochondrial genome[J].Journal of Animal Breeding & Genetics,1996,113(1/6):293-302.
[19] 韩 旭,刘丑生,刘 刚,等.中国地方绵羊线粒体DNAD-loop区遗传多样性研究[J].中国畜牧杂志,2015,51(7):10-15.
[20] 娄渊根,刘丑生,刘建学,等.9个绵羊品种微卫星DNA遗传多样性分析[J].中国畜牧兽医,2011,38(10):106-112.
[21] 冉汝俊,李光兰,徐德范,等.洼地绵羊形成历史考证[J].中国养羊,1998(3):13-14.
[22] 山东省畜牧局编写委员会.山东省畜禽品种志[M].深圳:海天出版社,1999:51-53.
