基于植物DNA条形码技术对杏仁露中花生源性成分的鉴别研究
2018-03-13韩晴王赞章晶晶李月华杨岚周巍张岩张志胜
韩晴,王赞,章晶晶,李月华,杨岚,周巍,张岩,张志胜
(1.河北农业大学食品科技学院,河北保定 071000)(2.河北省食品检验研究院,河北省食品安全重点实验室,河北石家庄 050071)
DNA条形码类似于商品条形码,可以有效的辨别各种动物、植物[1~3]。它是现今可用于生物多样性、生态学、食品检测、生物检验检疫等方面的一种新兴技术。其在动物源性食品中的应用已相对成熟,目前常用于动物DNA条形码的基因为COI。而在植物源性食品中的应用正在迅猛发展[4~9],常用于植物DNA条形码的基因组有ITS、matK、rbcL、trnH-psbA,它们被认为是潜在的优秀的植物 DNA条形码候选序列[10~12]。DNA条形码技术虽多在动植物分类学上有所应用[13,14],但在国外该技术也成功的应用在了某些食品中植物源性成分的鉴定[15,16]。
杏仁露作为植物蛋白饮料中的一种,老少皆宜。同时也是一种深加工食品,有着复杂的加工工艺,使它失去了本身的性状,应用传统的分类学无法对其辨别。这就给了不法商家可乘之机,进行掺假造假,损害消费者的利益。找到行之有效的鉴别方法,对保护广大消费者的权益意义重大。
目前对于工艺复杂的深加工食品掺假检测常采用设计特异性引物对提取的样品总基因组 DNA进行PCR扩增的方法,但需要根据可能掺入的物种去设计不同的特异性引物,如魏晓璐等[17]对核桃露中花生和大豆源性成分进行检测,分别设计了核桃、花生、大豆的特异性引物;覃芳芳等[18]用了大豆和杏仁的内源基因设计引物对杏仁产品中杏仁成分检测。而应用植物DNA条形码技术设计的通用引物可以达到一次检测出结果的目的,更加方便快捷、检测效率更高。在以上研究基础上,本研究利用植物DNA条形码技术对杏仁露中花生源性成分进行鉴别,建立一种检测方法,同时也为相关领域检测提供一定的参考。
1 材料与方法
1.1 材料与试剂
杏仁、花生、核桃、大豆、芝麻、榛子,力兴源杏仁露、热河杏仁露、太平洋杏仁露、桦维杏仁露、露美达杏仁露、露露杏仁露、哈露杏仁露、乐野杏仁露:石家庄农贸市场、超市。
深加工食品DNA提取试剂盒:天根公司;酚:氯仿:异戊醇(25:24:1):北京酷来博科技有限公司;异丙醇(分析纯):天津市光复科技发展有限公司;无水乙醇(分析纯):天津欧博凯化工有限公司;Premix TaqTM:TaKaRa;2xSuperReal PreMix(Probe):天根公司;杏仁、花生特异性引物和探针的合成、PCR扩增所用通用引物合成、PCR产物测序:上海生工公司;GelRedTM:Biotium;50x TAE Buffe:生工公司;琼脂糖:Vivantis;100 bp、1 kb DNA Ladder:Thermo。
1.2 仪器与设备
1.3 方法
1.3.1 基因组DNA的提取
样本基因组DNA的提取方法,均采用优化后的试剂盒法进行提取:(1)取经液氮充分研磨后的样本组织约100 mg至灭菌离心管中,加入500 μL缓冲液GMO1 和 20 μL 的 Proteinase K(20 mg/μL),涡旋震荡1 min;(2)56 ℃孵育1 h,其间每隔15 min将其上下颠倒震荡一次;(3)等体积加入酚:氯仿:异戊醇(25:24:1),充分混匀,12000 r/min离心5 min;(4)将上层水相转移至新的离心管中,加入200 μL的缓冲液GMO2,充分混匀,涡旋震荡1 min,静置10 min;(5)12000 r/min离心5 min,将上清转移至新的离心管中;(6)向上清液中加入0.7倍体积的异丙醇,充分混匀,-20 ℃静置30 min,12000 r/min离心3 min,弃上清,保留沉淀;(7)加入700 μL 的70%乙醇,涡旋震荡5 s,12000 r/min离心2 min,弃上清;(8)重复(7);(9)开盖,室温放置至彻底晾干残留的乙醇;(10)加入50 μL的洗脱缓冲液TE,涡旋震荡1 min,即为提取的基因组DNA溶液。杏仁露样品的提取方法:先对杏仁露进行预处理(1)取2 mL样品于离心管中,加入等体积异丙醇,混匀后静置15 min,12000 r/min离心10 min,弃上清;(2)重复步骤(1);其余步骤同上述提取过程。
1.3.2 样本准确性的测定
为保证样本准确性,利用NanoDrop Lite测定提取的基因组DNA的A260/A280值和浓度。以提取的基因组DNA为模板,以杏仁、花生、核桃、大豆、芝麻、榛子的特异性引物和探针(参见标准 SN/T 1961.9-2013、SN/T 1961.2-2007、SN/T 1961.6-2013、SN/T 1961.19-2013、SN/T 1961.12-2013和 SN/T 1961.8-2013)进行实时荧光PCR扩增,观察增幅情况,以确定样本的准确性。采用25 μL的扩增体系中添加12.5 μL 2xSuperReal PreMix(Probe)、0.75 μL 上游引物(10 μmol/L)、0.75 μL 下游引物(10 μmol/L)、0.5 μL 探针(10 μmol/L)、2.0 μL DNA 模板(30 μg/mL~100 μg/mL)、8.5 μL ddH2O。反应程序为95 ℃预变性15 min;95 ℃变性3 s,60 ℃退火/延伸20 s采集荧光信号,共40个循环。
1.3.3 引物设计
引物 ITS2-1、ITS2-2、ITS2-3、matK-2、matK-3、rbcL-1、trnH-psbA-1、trnH-psbA-2:参考于文献[19~24],其中引物trnH-psbA-1参考于对陆生植物DNA条形码的相关研究,引物rbcL-1适用于除早熟禾、莴苣属、佩兰属、栎属、蒲黃、桦木属外物种的研究,引物ITS2-1、ITS2-2、ITS2-3、matK-2、matK-3、trnH-psbA-2分别为对芸香科植物、杜鹃属植物、蒲黃、萹蓄、茄属茄科、苦苣菜属和石榴这些物种的研究。引物序列见表 1。引物 matK-1、rbcL-2:通过 BOLD 数据库(http://www.barcodinglife.org)引物检索筛选Primer,引 物序列见表1。

表1 基因引物序列Table 1 Primer sequences of genes
1.3.4 PCR扩增
虽然现金流动性较强,但其盈利能力并不高。Jenson(1986)提出的自由现金流量假说认为,如果公司持有过多不必要的现金,管理者会不知不觉地实施现金滥用,从而盲目地进行投资项目,这在无形之间增加了企业经营期间的财务风险,也是对股东权益损害较大的一种自私自利的行为。由此可知,董事会需要抑制这种恶性投资行为来提高现金配置效率。但是当前现状是不论出于什么样的原因,很多公司都愿意超额持有现金量以满足自己的财务报表的需求。
利用设计的各引物与各样本进行 PCR扩增,50 μL 的扩增体系中添加25 μL Premix TaqTM、2 μL 上游引物(10 μmol/L)、2 μL 下游引物(10 μmol/L)、4 μL DNA 模板(30 μg/mL~100 μg/mL)、17 μL ddH2O。反应程序为95 ℃预变性5 min;然后进入循环:95 ℃变性30 s、50 ℃ (ITS2-1、ITS2-3、matK-1、matK-2、matK-3、rbcL-1)或 55 ℃ (ITS2-2、rbcL-2、trnH-psbA-1、trnH-psbA-2)退火30 s、72 ℃延伸40 s,共40个循环;最后72 ℃总延伸10 min。待反应完成后取7 μL PCR扩增产物用琼脂糖凝胶进行电泳检测。
1.3.5 PCR产物测序及比对
将扩增后的PCR产物置于-20 ℃冷冻,然后装入提前加入冰袋的保温箱中送上海生工公司进行双向测序,测序引物同于扩增引物。所得的测序结果删除两端引物序列,在NCBI上进行BLAST比对,同时结合BOLD数据库(http://www.boldsystems.org)对样品的序列进行鉴定分析。
2 结果与分析
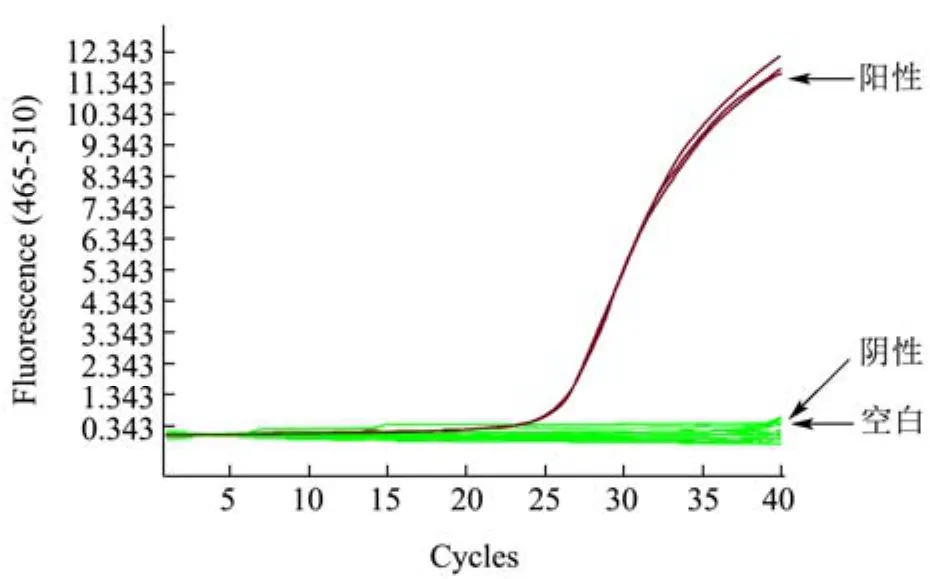
图1 实时荧光PCR检测提取的杏仁基因组DNA结果Fig.1 Almond genome DNA by real time fluorescence PCR
2.1 样本准确性的测定

图2 实时荧光PCR检测提取的花生基因组DNA结果Fig.2 Peanut genome DNA by real time fluorescence PCR
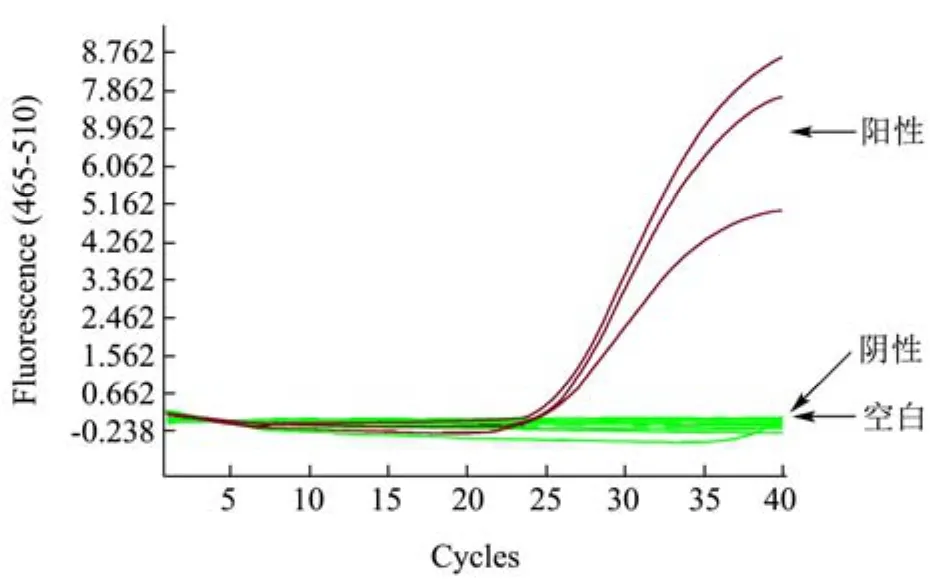
图3 实时荧光PCR检测提取的核桃基因组DNA结果Fig.3 Walnut genome DNA by real time fluorescence PCR
对提取的基因组DNA的A260/A280值和浓度进行测定,A260/A280值为 1.7~2.0,浓度控制在 30 μg/mL~100 μg/mL,有利于试验后续进行[24]。为保证样本的准确性,利用实时荧光 PCR技术对提取的杏仁、花生、核桃、大豆、芝麻和榛子基因组DNA进行鉴定,结果见图1~6所示。
根据标准当Ct值<30.0(杏仁、芝麻)、Ct值≤35.0(核桃、大豆、榛子)、Ct值≤40.0(花生),则判定为阳性,由图1和图6显示Ct值均小于30.0,阴性和空白对照均无起跳,证明本试验的样本均准确,可用于后续试验。

图4 实时荧光PCR检测提取的大豆基因组DNA结果Fig.4 Soybean genome DNA by real time fluorescence PCR

图5 实时荧光PCR检测提取的芝麻基因组DNA结果Fig.5 Sesame genome DNA by real time fluorescence PCR

图6 实时荧光PCR检测提取的榛子基因组DNA结果Fig.6 Hazelnut genome DNA by real time fluorescence PCR
2.2 PCR扩增效果
分别以文献和BLOD提供的引物与六类物种的基因组DNA进行PCR扩增,并以无菌双蒸水作为空白对照[25],扩增产物用琼脂糖凝胶电泳进行检测。试验条件经过多次优化后,结果表明引物ITS2-1、ITS2-3、matK-1、matK-2、matK-3、rbcL-1不能完全扩增出六个物种,这几对引物通用性较差,在后续实验中被排除。引物 ITS2-2、rbcL-2、trnH-psbA-1、trnH-psbA-2能全部扩增出六个物种,经过琼脂糖凝胶电泳检测后结果见图7~10。

图7 引物ITS2-2与六个样本DNA扩增的结果Fig.7 Result of primer ITS2-2 in six samples

图8 引物rbcL-2与六个样本DNA扩增的结果Fig.8 Result of primer rbcL-2 in six samples

图9 引物trnH-psbA-1与六个样本DNA扩增的结果Fig.9 Result of primer trnH-psbA-1 in six samples

图10 引物trnH-psbA-2与六个样本DNA扩增的结果Fig.10 Result of primer trnH-psbA-2 in six samples
2.3 PCR产物测序及分析
将引物ITS2-2、rbcL-2、trnH-psbA-1、trnH-psbA-2与六个样本扩增后 PCR产物送上海生工公司进行双向测序,测序结果删除两端引物序列,提交NCBI进行BLAST比对,同时使用BOLD数据库比对分析[26]。
结果表明比对结果均成功,其中引物trnH-psbA-2比对结果相似度在90%以下,其余引物比对结果相似度均在97%及以上,后续试验排除引物trnH-psbA-2;因引物rbcL-2扩增的目的片段较长,超过1000 bp,不便于测序工作进行,故后续试验中也被排除。经过以上试验过程与结果分析,引物ITS2-2、trnH-psbA-1可应用于六类物种的鉴别。
2.4 掺假模型的建立
因试验不可能完全模拟实际生产,试验所用样品量较少,若直接通过原料水平掺假必然造成很大的误差,所以本试验中采取DNA水平掺假,再经换算得到原料水平掺假。
在杏仁基因组 DNA中人为掺入花生基因组DNA,其中花生基因组DNA占总基因组DNA的比例分别为90%、80%、70%、60%、50%、40%、30%、20%和 10%[27]。
分别提取125 mg、100 mg、75 mg、50 mg 和25 mg杏仁和花生基因组DNA,提取方法参照1.3.1所述方法,不同质量的样本均按比例添加各试剂。通过对样本提取率的计算,可从DNA水平推算到原料水平。根据公式1,计算各样本的提取率T。

式中:T为提取率;mDNA为提取的样本总DNA量;m为样本质量。
对不同质量的杏仁和花生提取基因组DNA时,均做三组平行试验,提取率见表2所示。
由表 2可知在该方法下对杏仁和花生的基因组DNA 进行提取,所得杏仁提取率为 5.91×10-4、花生的提取率为8.81×10-4。
根据杏仁、花生的提取率和公式1进行DNA水平到原料水平的换算,换算公式见公式2:

式中:T杏仁为杏仁提取率;mDNA杏仁为提取的杏仁样本总DNA量;m杏仁为杏仁质量;T花生为花生提取率;mDNA花生为提取的花生样本总DNA量;m花生为花生质量。
其中杏仁的提取率为5.91×10-4、花生的提取率为8.81×10-4,将二者带入公式2得到公式3。

将DNA水平的各掺假比例换算到原料水平后的比例,见表3。
由筛选出的通用引物分别与不同水平掺假的基因组DNA模型进行PCR扩增,扩增产物经琼脂糖凝胶电泳检测,将扩增出清晰明亮条带的样品送测序公司进行测序。

表2 杏仁、花生基因组DNA提取率Table 2 Extraction rate of peanut genome DNA and almond genome DNA

1 0 7 0 3 7.5 0 9 5 5 1 2.5 0 1 0 3 6 5 0.0 0 9 2 2 6 5.0 0 1 0 2 0 6 6.6 7 1 2 5 8.1 7×1 0-2 9 5 8 0 6.6 7 1 0 0 9.5 8×1 0-2 8 9 8 7 5.0 0花生1 0 5 2 8 0.0 0 6 2 1 9 0.0 0 6 1 8 1 1.2 5 7 5 8.2 4×1 0-2 6 3 1 8 3.7 5 8.8 1×1 0-2 6 0 0 6 0.0 0 3 7 5 9 5.0 0 4 1 7 4 6.6 7 5 0 8.3 5×1 0-2 4 3 9 7 5.0 0 4 3 6 7 0.0 0 2 3 0 4 2.5 0 2 4 2 4 5.8 3 2 5 9.7 0×1 0-2 2 7 4 4 7.5 0 2 2 2 4 7.5 0

表3 DNA水平到原料水平换算和引物ITS2-2、trnH-psbA-1检测结果Table 3 Conversion of DNA level to raw material level and test results of primes ITS2-2 and trnH-psbA-1
结果见表3,由表3可知用引物ITS2-2检测掺入90%及以下花生基因组DNA(85.80%及以下花生原料)时测序结果为杏仁,虽然该引物与单一一个物种均能扩增成功,但掺假后对花生的检测灵敏度低,出现这样的结果可能是杏仁较花生对该引物竞争性更强,该引物更容易与杏仁进行扩增;
用引物trnH-psbA-1检测掺入10%及以上花生基因组 DNA (6.94%及以上花生原料)时测序结果为花生,说明此引物对花生检测的灵敏度高,即对杏仁产品中掺入花生原料时检出限为 6.94%,可用于对杏仁产品中花生源性成分的检测。为保证试验的准确性,采用引物ITS2-2、trnH-psbA-1二者组合作为通用引物使用,提高结果的可信度。
2.5 采样进行检测及分析
对采集的 11个不同批次杏仁露待测样品进行基因组DNA的提取,以真核生物18S rRNA基因对提取的杏仁露基因组DNA进行实时荧光PCR扩增,结果见图11,由图可知各样品均发生起跳,且Ct值<30.0,说明样品基因组DNA符合PCR扩增要求。
分别以引物ITS2-2、trnH-psbA-1与提取的杏仁露基因组DNA进行扩增,无菌双蒸水做空白对照[17]。PCR产物经琼脂糖凝胶电泳进行检测,结果见图12~13。
由图12~13可知11个样品与两对引物分别扩增出清晰明亮的条带,其中样品2~11与引物trnH-psbA-1扩增两个条带,其余样品均扩增出一个条带。将PCR扩增产物送上海生工公司进行双向测序并分析测序结果。测序结果见表4。
由表4可知引物ITS2-2、trnH-psbA-1与各样品扩增产物测序结果一致,其中样品2~11检测结果均为杏仁,说明这几个批次杏仁露没有掺假与产品标示相符。样品1检测结果均为花生,与产品标示不符,属于欺瞒消费者行为。
分析产生这种结果可能有以下原因:不同品牌的杏仁露加工工艺不同,对原料的破坏程度不同,使得在相同方法下提取的基因组DNA有所差异;不同批次的产品均一性不好;所测杏仁露中不含杏仁源性成分或者含量极少,添加了其他成分代替杏仁露的口感或者掺杂了除杏仁和花生外的其他物种欺瞒消费者。
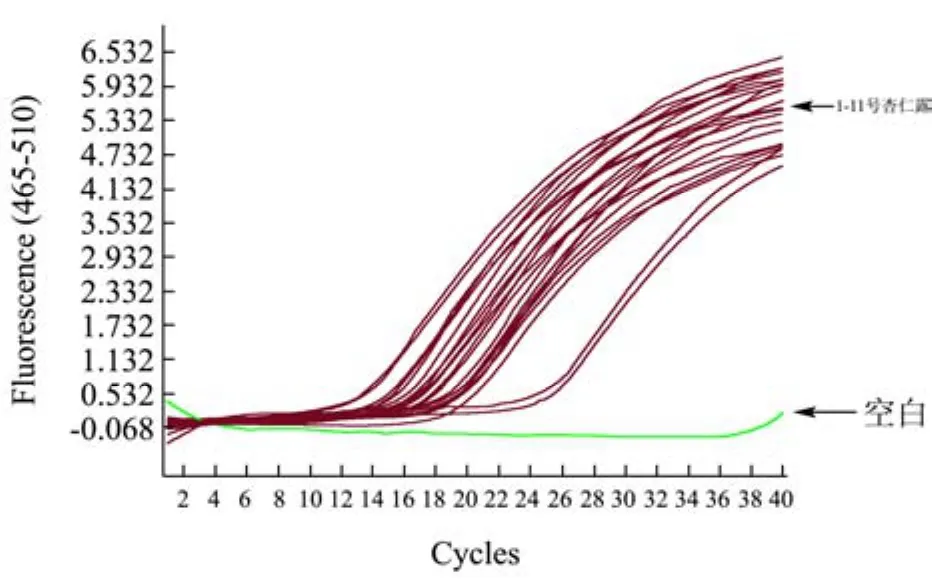
图11 实时荧光PCR检测提取的杏仁露基因组DNA结果Fig.11 Almond genome DNA by real time fluorescence PCR

图12 引物ITS2-2对不同杏仁露样品DNA扩增的结果Fig.12 PCR result of primer ITS2-2 in different almond juice

图13 引物trnH-psbA-1对不同杏仁露样品DNA扩增的结果Fig.13 PCR result of primer trnH-psbA-1 in different almond juice

表4 采样产品鉴定结果Table 4 Identification results of samples
3 结论
3.1 随着DNA条形码技术的发展,在植物界的研究范围越来越广,目前有一些应用较多的基因序列,如ITS、matK、rbcL和trnH-psbA等,但仍未达到成熟阶段。本试验针对杏仁和花生两个物种,通过参考文献和数据库下载的10对引物分别对它们进行PCR扩增,其中有两对引物ITS2-2、trnH-psbA-1对它们的扩增成功率与测序成功率较高,可作为通用引物使用。
3.2 目前对于植物蛋白饮料鉴定真伪的相关技术报道较少,该类产品因加工工艺导致难以用常规的分类学手段辨别,而利用植物DNA条形码技术应用到此类食品的检测上,可有效地辨识出所含物种。本试验模拟人为在杏仁中掺入花生,利用引物ITS2-2检测掺入90%及以下花生基因组DNA (85.80%及以下花生原料)时测序结果为杏仁;用引物trnH-psbA-1检测掺入10%及以上花生基因组 DNA(6.94%及以上花生原料)时测序结果为花生,可将二者结合应用于杏仁露产品中花生源性成分掺假的检测。
3.3 本试验虽找到两对引物,但对于某些含杏仁、花生源性成分很低的产品的检测灵敏度仍然不够,有待继续研究,找到灵敏度更高的通用引物。同时二者作为过敏原对于某些人群来说误食将有损身体健康,该研究也可为相关食品检测其中的过敏原成分提供一定的理论基础。
[1]刘宇婧,刘越,黄耀江,等.植物DNA条形码技术的发展及应用[J].植物资源与环境学报,2011,20(1):74-82,93 LIU Jing-yu, LIU Yue, HUANG Yao-jiang, et al. Progress and application of DNA barcoding technique in plants [J].Journal of Plants and Environment, 2011, 20(1): 74-82, 93
[2]LIU J, SHI L, HAN J, et al. Identification of species in the angiosperm family apiaceae using DNA barcodes [J].Molecular Ecology Resources,2014, 16(6): 1231-8
[3]Galimberti A, De Mattia F, Lose A, et al. DNA barcoding as a new tool for food traceability [J]. Food Sesearch International,2013, 50(1): 55-63
[4]吕冬梅,黄原,文慧,等.DNA条形码技术在食品鉴定中的应用[J].食品科学,2015,36(9):248-253 LV Dong-mei, HUANG Yuan, WEN Hui, et al. Application of DNA barcoding in authentication of food product [J]. Food Science, 2015, 36(9): 248-253
[5]张裕君,刘跃庭,廖芳,等.DNA条形码技术研究进展及其在植物检疫中的应用展望[J].中国植保导刊,2010,30(4):15-17 ZHANG Yu-jun, LIU Yue-ting, LIAO Fang, et al. Research progress of DNA bar code technology and prospect of its application in plant quarantine [J]. China Plant Protection,2010, 30(4): 15-17
[6]侯新东,尹帅,盛桂莲,等.基于ITS序列探讨10种荨麻科植物的系统发育关系[J].生物技术通报,2013,8:68-73 HOU Xin-dong, YIN Shuai, SHENG Gui-lian, et al. Study on phylogenetic relationship of ten urticaceae species based on nrDNA ITS sequences [J]. Biotechnology Bulletin, 2013, 8:68-73
[7]Haye P A, Segovia R D, Ventoleroa M F H, et al. Detection of mislabeled commercial fishery by products in the philippines using DNA barcodes and its implications to food traceability and safety [J]. Food Control, 2013, 33(1): 119-125
[8]LIU ZH, ZHENG X, YANG D, et al. Applying DNA barcodes for identification of plant species in the family araliaceae [J]. Gene, 2012, 499(1): 76-80
[9]裴男才,陈步峰.生物 DNA条形码:十年发展历程、研究尺度和功能[J].生物多样性,2013,21(5):616-627 PEI Nan-cai, CHEN Bu-feng. DNA barcoding of life: a classification of uses according to function and scale after ten years of development [J]. Biodiversity Science, 2013, 21(5):616-627
[10]伏建国,杨晓军,钱路,等.植物DNA条形码技术在出入境检验检疫领域的应用[J].植物检疫,2012,26(2):64-69 FU Jian-guo, YANG Xiao-jun, QIAN Lu, et al. Application of plant DNA barcoding in inspection and quarantine [J].Plant Quarantine, 2012, 26(2): 64-69
[11]王晓明,姬可平,牛宪力,等.DNA 条形码与动植物分类学的研究[J].生物信息学,2012,10(2):83-86 WANG Xiao-ming, JI Ke-ping, NIU Xian-li, et al. Study of DNA barcoding obout taxonomy of plants and animals [J].Chinese Journal of Bioinformatics, 2012, 10(2): 83-86
[12]陈亚辉,朱海军,生静雅,等.DNA条形码序列对不同品种美国山核桃的鉴定[J].江苏农业学报,2013,29(6):1445-1450 CHEN Ya-hui, ZHU Hai-jun, SHENG Jing-ya, et al.Identification of pecan varieties by DNA barcoding [J].Jiangsu Journal of Agricultural Sciences, 2013, 29(6):1445-1450
[13]李新光,王璐,赵峰,等.DNA 条形码技术在鱼肉及其制品鉴别中的应用[J].食品科学,2013,34(18):337-342 LI Xin-guang, WANG Lu, ZHAO Feng, et al. Application of DNA barcoding to identify commercial fish and fish products[J]. Food Science, 2013, 34(18): 337-342
[14]欧阳解秀,王立贤.DNA条形码技术在地方猪种质资源保护中的应用[J].农业生物技术学报,2013,21(3):348-354 OUYANG Xie-xiu, WANG Li-xian. The progress and application of DNA barcoding in genetic resources conservation for local pig [J]. Journal of Agricultural Biotechnology, 2013, 21(3): 348-354
[15]Cutarelli A, Amoroso M G, De Roma A, et al. Italian market fish species identification and commercial frauds revealing by DNA sequencing [J]. Food Control, 2014, 37: 46-50
[16]ZHU S, FU J, WANG Q, et al. Identification of Channa Species Using the Partial Cytochrome Coxidase SubunitⅠ(COⅠ) Gene as a DNA Barcoding marker [J].Biochemical Systematics and Ecology, 2013, 51: 117-122
[17]魏晓璐,黄鑫,冯悦,等.核桃乳(露)饮品中花生、大豆成分的PCR检测方法[J].食品工业科技,2014,35(13):288-291,295 WEI Xiao-lu, HUANG Xin, FENG Yue, et al. Detection of peanut and soybean in walnut milk using PCR [J]. Science and Technology of Food Industry, 2014, 35(13): 288-291,295
[18]覃芳芳,邓鸿铃,罗海英,等.杏仁产品杏仁成分的PCR检测方法研究[J].现代食品科技,2008,24(6):603-605 TAN Fang-fang, DENG Hong-ling, LUO Hai-ying, et al.Application of PCR assays in detection of amygdaline components in amygdaline food [J]. Modern Food Science and Technology, 2008, 24(6): 603-605
[19]罗焜,陈士林,陈科力,等.基于芸香科的植物通用DNA条形码研究[J].中国科学:生命科学,2010,40(4):342-358 LUO Kun, CHEN Shi-lin, CHEN Ke-li, et al. Assessment of candidate plant DNA barcodes using the Rutaceae family [J].Sci. China Life Sci., 2010, 40(4): 342-358
[20]林春,梁宗锁,韩建萍,等.基于杜娟属植物的DNA条形码序列筛选[J].世界科学技术-中医药现代化,2009,11(1): 54-57 SHI Lin-chun, LIANG Zong-suo, HAN Jian-ping, et al.Screening potentia DNA barcode regions in rhododendron [J].Modemization of Traditional Chinese Medicine and Materia Media-World Science and Technology, 2009, 11(1): 54-57
[21]张裕君,刘跃庭,廖芳,等.基于rbcL基因序列的欧洲菟丝子分子检测[J].植物保护,2009,35(4):110-113 ZHANG Yu-jun, LIU Yue-ting, LIAO Fang, et al. Molecular detection of Cuscuta Eurepean based on rbcL sequence [J].Plant Protection, 2009, 35(4): 110-113
[22]Kress W J, Erickson D L. A Two-locus Global DNA Barcode for Land Plants: the coding rbcL gene complements the non-coding trnH-psbA spacer region [J]. PLoS One, 2007,2(6): e508
[23]Fazekas A J, Burgess K S, Kesanakurti P R, et al. Multiple multilocus DNA barcodes from the plastid genome discriminate plant species equally well [J]. PLoS One, 2008,3: e2802
[24]高连明,刘杰,蔡杰,等.关于植物DNA条形码研究技术规范[J].植物分类与资源学报,2012,34(6):592-606 GAO Lian-ming, LIU Jie, CAI Jie, et al. A synopsis of technical notes on the standards for plant DNA barcoding [J].Plant Diversity and Resources, 2012, 34(6): 592-606
[25]王爽,李永波,马超峰,等.DNA条形码COI序列在常见肉类鉴别中的应用研究[J].现代食品科技,2016,32(1):188-193 WANG Shuang, LI Yong-bo, MA Chao-feng, et al. Study on selection of efficient DNA barcoding COI for identification of common meat species [J]. Modern Food Science and Technology, 2016, 32(1): 188-193
[26]侯新东,韩大永,曾莉,等.荨麻科植物DNA条形码的筛选[J].南方农业学报,2014,45(2):178-183 HOU Xin-dong, HAN Da-yong, ZENG Li, et al. Screening potential DNA barcode regions for urticaceae plants [J].Journal of Southern Agricultural, 2014, 45(2): 178-183
[27]田晨曦,周巍,王爽,等.基于DNA条形码技术常见肉类掺假鉴别技术的研究[J].现代食品科技,2016,32(8):295-301 TIAN Chen-xi, ZHOU Wei, WANG Shuang, et al.Techniques for identifying common meat adulterations based on DNA barcoding [J]. Modern Food Science and Technology, 2016, 32(8): 295-301
