神经细胞分化中microRNA-134靶基因网络功能分析*
2018-03-08苏立宁尹海峰宋小青魏会平
苏立宁,尹海峰,宋小青,魏会平
(河北北方学院基础医学院,河北 张家口 075000)
骨髓间充质干细胞(bone marrow mesenchymal stem cells,BMSCs)是一种重要的多功能干细胞,具有取材方便,分化易控制等特点。研究表明,在合适的诱导条件下BMSCs可向成骨细胞、肌细胞、软骨细胞、肝细胞、脂肪细胞、平滑肌细胞和内皮细胞等多方向分化[1-7]。研究[8-9]发现在特殊的培养条件下,BMSCs可分化为神经细胞,但有关其分子机制还有待进一步研究。
MicroRNAs,是一种短的非编码单链RNA,长约20~24个核苷酸,通过碱基互补配对原则,与mRNA结合,直接靶向切割mRNA或抑制靶基因的翻译对转录后基因表达进行调控。MicroRNAs通过与mRNA结合可调控神经系统发育和功能[10],脑特异性microRNA-134在神经突生成及突触成熟等发育阶段发挥调控作用[10-11]。研究[12]表明microRNA-134通过抑制单丝氨酸蛋白激酶1(LIM-kinase 1,LIMK1)控制神经突的生成或脊柱的生长,通过降低胚胎干细胞关键蛋白NANOG和肝受体类似物LRH1的转录后表达调节小鼠BMSCs的分化[13]。本实验推测microRNA-134在神经分化过程中发挥重要作用。
为了进一步验证microRNA-134在BMSCs向神经分化过程中的功能,本实验检测了神经分化过程中microRNA-134表达量的变化,利用生物信息学软件对其靶基因进行预测,结合所研究的目标,分析与神经分化相关的基因。
1 材料与方法
1.1 实验动物
4~6周龄大鼠3只,购自河北北方学院实验动物中心。
1.2 主要试剂
DMEM购自美国Gibco公司,碱性成纤维细胞生长因子(basic fibroblast growth factor,bFGF)和表皮生长因子(epidermal growth factor,EGF)购自上海PrimeGene Bio-Tech公司,RNA提取试剂盒和兔抗神经元特异性烯醇化酶(NSE)引物购自日本TaKaRa公司,兔抗巢蛋白(nestin)和NSE购自北京博奥森生物技术有限公司。
1.3 BMSCs的分离与培养
颈椎脱臼法处死大鼠,75%的酒精浸泡10 min消毒,无菌条件下取出股骨,置无菌PBS液中清洗2次,露出骨髓腔,用5 ml的无菌注射器吸取含有10%胎牛血清的DMEM培养液冲洗骨髓腔,收集洗液,置无菌培养瓶中培养,72 h后换液去除未贴壁的细胞,每隔3 d换液1次,待细胞密度达80%左右时按1∶2进行传代,直至第4代。流式细胞仪检测BMSCs的表面标志物。
1.4 BMSCs诱导分化为神经细胞
取第4代的细胞分为两组,对照组不加任何因子,诱导组加入含20 ng/ml EGF和20 ng/ml bFGF的诱导液。对照组每隔3 d换液1次,诱导组每隔2 d半量换液。
1.5 MicroRNA-134表达量分析
收集对照组细胞和诱导组细胞(诱导1、2、3、4 d)。利用RNA提取试剂盒分别提取两组总RNA。采用microRNA-134逆转录引物,逆转录为cDNA,采用实时荧光定量聚合酶链反应(quantitative real-time polymerase chain reaction,qRT-PCR)检测microRNA-134在两组不同时间点的表达量变化,反应体系为95℃ 5 min,94℃ 30 s 40个循环,60℃30 s,72℃ 30 s。成熟 microRNA-134的正向扩增引物为5'-CGTGTGACTGGTTGACC-3',反向引物5'-GAGCAGGCTGGAGAA-3'。内参β-actin 的正向引物为5'-CCCATCTATGAGGGTTACGC-3',反向引物为5'-TTTAATGTCACGCACGATTTC-3'。采用Ct值比较法[14],比较两组的表达差异。
1.6 MicroRNA-134靶基因预测及GO功能富集
利用数据库miRWalk[15]对microRNA-134靶基因进行预测。预测得到的靶基因上传至DAVID数据库进行Gene Ontology(GO)功能富集分析,筛选与神经分化相关的基因,应用Fisher’s exact test方法选取P<0.05注释基因条目。
1.7 神经分化相关基因蛋白互作网络构建及分析
将选取的神经分化相关基因输入到STRING 10[16]数据库中,选取交互作用评分<0.7(高等可信度)的互作关系构建神经分化相关基因的蛋白互作网 络(protein- protein interaction network,PPI)。 网络分析采用生物图表可视化工具Cytoscape3.2.1软件[17]进行,且利用“Network Analyzer”工具对各节点的“度”进行计算。“度”代表了与目标蛋白相互作用的蛋白的数量。网络中,节点越大,“度”越大。利用 Cytoscape3.2.1软件中的“ClusterONE”工具对蛋白质相互作用网络进行功能模块分析,筛选标准为minimum size=6,minimum density=0.05,P<0.05。
1.8 统计学方法
采用SPSS17.0软件进行数据分析,计量资料以均数±标准差(±s)表示,各指标间比较采用重复测量设计的方差分析,P<0.05为差异有统计学意义。
2 结果
2.1 BMSCs的鉴定结果
大鼠骨髓中获取的间充质干细胞置于含10%胎牛血清的DMEM培养基中,培养48 h后半量换液,待细胞密度达80%~90%传代,3代细胞生长较稳定,密度较均匀,形态以长梭形为主,符合BMSCs的细胞形态特征。待细胞传至4代,流式细胞仪检测细胞的表面标志物,检测结果显示,标志物CD29和CD90表达阳性,CD34和CD45表达弱阳性,符合骨髓间充质干细胞的特点(见图1)。

图1 BMSLs的鉴定结果
2.2 BMSCs向神经细胞的诱导分化结果
向传至第4代的BMSCs中加入含20 ng/ml EGF和20 ng/ml bFGF的诱导液,诱导2~3 d后,出现少量的神经样细胞;继续诱导4 d,大部分已分化为神经样细胞(见图2A)。免疫组织化学法检测分化后神经细胞标志物nestin和NSE的表达量,结果为阳性(见图2B)。收集对照组诱导1~4 d的细胞,利用qRTPCR进行NSE基因表达量的检测,结果显示诱导4 d后,NSE基因的表达量达到高峰。与对照组相比,诱导组1~4 d后表达量分别升高1.08、1.25、2.01和2.94倍(见图2C),其中,诱导组第3和4天表达量与对照组比较,差异有统计学意义(P<0.05)。
2.3 BMSCs诱导分化后microRNA-134表达量的变化
诱导组与对照组细胞培养1、2、3及4 d的microRNA-134表达量比较,采用重复测量设计的方差分析,结果:①不同时间点间的microRNA-134表达量有差别(F=69.711,P=0.003),②诱导组与对照组细胞的microRNA-134表达量有差别(F=18.096,P=0.021),诱导组与对照组相比microRNA-134表达量较高,可诱导BMSCs细胞向神经细胞分化,③不同诱导时间和诱导方法间存在交互作用,诱导组与对照组的不同诱导时间有差别(F=5.390,P=0.014)。见表1。
2.4 MicroRNA-134靶基因网络相互作用分析
为了进一步研究microRNA-134在神经分化中的作用,利用在线数据库miRWalk预测microRNA-134靶基因,得到1 220个靶基因。将靶基因输入到DAVID软件6.7中进行GO(P<0.05)功能富集分析,选取与神经分化相关的GO功能组:GO 0030182和GO 0048667(见表2)。
2.5 GO 0030182和GO 0048667 PPI网络分析
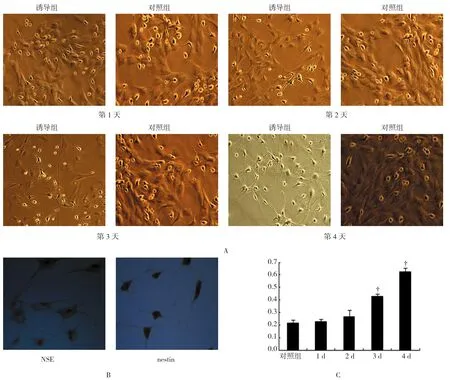
图2 BMSCs分化为神经细胞的鉴定结果
表1 microRNA-134表达量的变化 (±s)
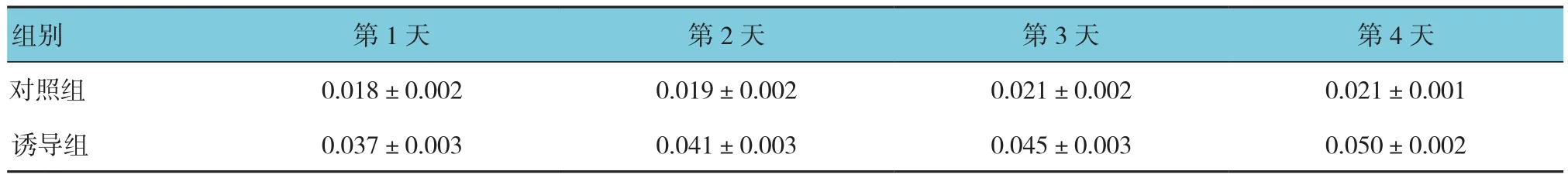
表1 microRNA-134表达量的变化 (±s)
组别 第1天 第2天 第3天 第4天对照组 0.018±0.002 0.019±0.002 0.021±0.002 0.021±0.001诱导组 0.037±0.003 0.041±0.003 0.045±0.003 0.050±0.002

表2 与神经分化相关的microRNA-134靶基因
为了预测与神经元分化相关的靶基因(GO 0030182和GO 0048667)及与其他基因之间的蛋白质相互作用,利用STRING10数据库构建PPI网络,然后利用生物图表可视化工具Cytoscape3.2.1软件进行网络分析,且利用“Network Analyzer”工具对各节点的“度”进行计算(见图3)。构建结果显示,21个靶基因蛋白均与STRING数据库中蛋白质匹配,这21个基因为半胱氨酸天冬氨酸蛋白酶3(Caspase-3,CASP3)、靶向整合素β1(Integrinβ1,ITGB1)、血小板激活因子乙酰水解酶bata1(plateletactivating factor acetylhydrolase,PAFAH1B1)、蛋白激酶C(Protein kinase C alpha,PRKCA)、酪氨酸蛋白激酶A4(tyrosine protein kinase A4,EPHA4)、肝配蛋白A2(Ephrin-A2,EFNA2)、NK6 同源框蛋白 1(NKX6-1)、神经细胞黏着分子(neural cell adhesion molecule,NRCAM)、突触融合蛋白结合蛋白1基因(synaptic fusion protein binding protein 1 gene,STXBP1)、钾离子通道相关作用蛋白2(Kv channel-interacting protein 2,KCNIP2)、蛋白磷酸酶ABI2、腺嘌呤核苷A2a受体(adenine nucleoside both A2a receptors,ADORA2A)、接触蛋白2(contactin-2,CNTN2)、锌指转录因子DST、叉头状转录因子 FOXA1(forkhead box A1)、SEPT2(septin-2)、微管相关蛋白 Doublecortin(DCX)、ETS变异基因1(ETS gene variant 1,ETV1)、腱蛋白样蛋白1(cordin-like 1,CHRDL1)、生长休止蛋白7(growth arrest-specific,GAS7)和蛋白酪氨酸磷酸酶受体M(protein tyrosine phosphatase receptor type M,PTPRM)。利用Cytoscape3.2.1软件中的“ClusterONE”工具进行了模块分析,结果12个基因被聚集在模块中(见图4)。

图3 MicroRNA-134靶基因PPI网络分析


图4 MicroRNA-134靶基因相互作用网络的11个模块 黄色节点代表靶基因,红色节点代表与靶基因蛋白相互作用的蛋白。节点越大,“度”越大
3 讨论
MicroRNAs通过作用于靶基因调控细胞分化等各种生物学过程。
MicroRNA-134具有脑特异性,在神经中枢中高表达。而microRNA-134在BMSCs诱导分化为神经细胞过程中的作用机制还有待研究。
许多研究已证明EGF和bFGF联合作用可以诱导BMSCs向神经细胞分化[18],因此本实验选择的诱导剂为EGF和bFGF。利用qRT-PCR检测microRNA-134在各实验组的表达量变化,诱导组随诱导时间的递增表达量升高。因此初步假设microRNA-134在促进BMSCs分化为神经细胞过程中发挥作用。以往的研究证明[19],microRNA-134通过降低转录因子蛋白FOXM1的表达量调控人类多能干细胞分化发挥作用。神经系统中,LIMK1通过抑制microRNA-134的表达量调节神经突的生成、脊柱的生长及树突棘的大小[12,20]。另外还有研究发现沉默信息调节因子2相关酶1通过转录抑制因子复合体作用于microRNA-134调控神经突触的可塑性[21]。本实验初步验证了在神经分化过程中microRNA-134表达量升高,与前人的结论一致。MicroRNA-134以哪些基因为靶基因调控神经分化,在本文中进行了理论的预测。
本文以miRWalk和DAVID数据库为依据,以GO功能为基础筛选了与神经分化相关的microRNA-134靶基因(GO 0030182和GO 0048667),并构建了PPI网络图,通过ClusterONE功能模块分析发现CASP3,ITGB1,PAFAH1B1,PRKCA,EFNA2,NKX6-1,NRCAM,STXBP1,KCNIP2,ABI2,CNTN2,FOXA1等12个基因聚集在11个模块中发挥作用,说明这12个基因作用在神经分化中比较重要。NKX6-1,是一种同源框转录因子,通过调控3个神经元轴突导向分子的表达量控制神经元轴突的生长[22],且通过与PR结构域蛋白12的交叉抑制作用促进中间神经元的生长[23]。FOXA1,作为一种转录因子,可调控中脑多巴胺能神经元的分化[24],CNTN2在早期脑神经节和脊髓运动神经元中不表达,而在成熟神经元中表达[25]。
综上所述,本文初步验证了microRNA-134在促进BMSCs向神经细胞分化的过程中发挥重要作用,应用生物信息学的方法分析了在神经分化中发挥作用的microRNA-134的靶基因,并筛选了与神经分化相关的12个关键蛋白,但这些蛋白的作用还需实验的进一步验证。
[1]ZHENG G,LIU Y,JING Q,et al.Differentiation of human umbilical cord-derived mesenchymal stem cells into hepatocytes in vitro[J].Biomed Mater Eng,2015,25(Suppl): 145-157.
[2]JIQING C,YAQIN L,YINGYIN L,et al.BMP4 inhibits myogenic differentiation of bone marrow-derived mesenchymal stromal cells in mdx mice[J].Cytotherapy,2015,17(9): 1213-1219.
[3]HU N,FENG C,JIANG Y,et al.Regulative effect of mir-205 on osteogenic differentiation of bone mesenchymal stem cells(BMSCs): possible role of SATB2/Runx2 and ERK/MAPK pathway[J].Int J Mol Sci,2015,16(5): 10491-10506.
[4]GHALI O,BROUX O,FALGAYRAC G,et al.Dexamethasone in osteogenic medium strongly induces adipocyte differentiation of mouse bone marrow stromal cells and increases osteoblast differentiation[J].BMC Cell Biol,2015,16(9): 1-15.
[5]HIROTA N,MCCUAIG S,O’SULLIVAN M J,et al.Serotonin augments smooth muscle differentiation of bone marrow stromal cells[J].Stem Cell Res,2014,12(3): 599-609.
[6]OUYANG J F,LOU J,YAN C,et al.In-vitro promoted differentiation of mesenchymal stem cells towards hepatocytes induced by salidroside[J].J Pharm Pharmacol,2010,62(4): 530-538.
[7]CHOI Y A,LIM J,KIM K M,et al.Secretome analysis of human BMSCs and identi fi cation of SMOC1 as an important ECM protein in osteoblast differentiation[J].J Proteome Res,2010,9(6): 2946-2956.
[8]CAPKIN M,CAKMAK S,KURT F O,et al.Random/aligned electrospun PCL/PCL-collagen nanofibrous membranes:comparison of neural differentiation of rat AdMSCs and BMSCs[J].Biomed Mater,2012,7(4): 045013.
[9]JIANG X,XU R,GUO Z,et al.Neurobiochemistry and neuroelectrophysiology of neuron-like cells differentiated from neural BMSCs-D-NSCs[J].Conf Proc IEEE Eng Med Biol Soc,2005,5: 5215-5218.
[10]MAES O C,CHERTKOW H M,WANG E,et al.MicroRNA:implications for alzheimer disease and other human CNS disorders[J].Curr Genomics,2009,10(3): 154-168.
[11]SUN E,SHI Y.MicroRNAs: Small molecules with big roles in neurodevelopment and diseases[J].Exp Neurol,2007,268: 46-53.
[12]COOLEN M,BALLY-CUIF L.MicroRNAs in brain development and physiology[J].Curr Opin Neurobiol,2009,19(5): 461-470.
[13]TAY Y M,TAM W L,ANG Y S,et al.MicroRNA-134 modulates the differentiation of mouse embryonic stem cells,where it causes post-transcriptional attenuation of Nanog and LRH1[J].Stem Cells,2008,26(1): 17-29.
[14]QU K,WANG Z,LIN X L,et al.MicroRNAs: Key regulators of endothelial progenitor cell functions[J].Clin Chim Acta,2015,448: 65-73.
[15]DWEEP H,STICHT C,PANDEY P,et al.MiRWalk-database:prediction of possible miRNA binding sites by “walking” the genes of three genomes[J].J Biomed Inform,2011,44(5): 839-847.
[16]VON MERING C,HUYNEN M,JAEGGI D,et al.STRING: a database of predicted functional associations between proteins[J].Nucleic Acids Res,2003,31(1): 258-261.
[17]SHANNON P,MARKIEL A,OZIER O.Cytoscape: a software environment for integrated models of biomolecular interaction networks[J].Genome Res,2003,13(11): 2498-2504.
[18]HUAT T J,KHAN A A,ABDULLAH J M,et al.MicroRNA expression pro fi le of neural progenitor-Like cells derived from rat bone marrow mesenchymal stem cells under the in fl uence of IGF-1,bFGF and EGF[J].Int J Mol Sci,2015,16(5): 9693-9718.
[19]CHEN Y,MENG L,YU Q,et al.The miR-134 attenuates the expression of transcription factor FOXM1 during pluripotent NT2/D1 embryonal carcinoma cell differentiation[J].Exp Cell Res,2015,330(2): 442-450.
[20]SCHRATT G M,TUEBING F,NIGH E A,et al.A brain-speci fi c microRNA regulates dendritic spine development[J].Nature,2006,439(7074): 283-289.
[21]GAO J,WANG W Y,MAO Y W,et al.A novel pathway regulates memory and plasticity via SIRT1 and miR-134[J].Nature,2010,466(7310): 1105-1109.
[22]PRAKASH N,PUELLES E,FREUDE K,et al.Nkx6-1 controls the identity and fate of red nucleus and oculomotor neurons in the mouse midbrain[J].Development,2009,136(15): 2545-2555.
[23]THELIE A,DESIDERIO S,HANOTEL J,et al.Prdm12 speci fi es V1 interneurons through cross-repressive interactions with Dbx1 and Nkx6 genes in Xenopus[J].Development,2015,142(19):3416-3428.
[24]METZAKOPIAN E,BOUHALI K,ALVAREZ-SAAVEDRA M,et al.Genome-wide characterisation of foxa1 binding sites reveals several mechanisms for regulating neuronal differentiation in midbrain dopamine cells[J].Development,2015,142(7): 1315-1324.
[25]RATIE L,WARE M,JAGLINE H,et al.Dynamic expression of notch-dependent neurogenic markers in the chick embryonic nervous system[J].Front Neuroanat,2014,8: 158.
