基于干旱胁迫下杉木转录组序列的EST-SSR分子标记开发1)
2018-03-07吴夏雷董黎孙宇涵胡瑞阳郑会全胡德活李云
吴夏雷 董黎 孙宇涵 胡瑞阳 郑会全 胡德活 李云
(北京林业大学,北京,100083) (中国林业科学研究院华北林业实验中心) (广东省林业科学研究院) (北京林业大学)
杉木(Cuiminghamialanceolata(Lamb.) HooK)是我国南方特有的多年生针叶用材树种,距今已有3 000多年的栽培历史,主要分布于我国秦岭地区、长江流域以南以及台湾山区,南北约跨10个纬度,东西约15个经度,海拔最高可达1 800 m以上[1]。杉木具有生长快、材性优良的特点,是我国重要的商品材,用途广泛[2]。近年来对杉木的研究多集中在杂交育种、良种繁育、材性提高等方面,而杉木的分子遗传学研究基础比较薄弱,早期开发出来的分子标记,因其标记缺乏共显性、多态性不高、操作复杂等原因,很大程度上限制了杉木分子育种的发展,气候环境的变化急需集合速生、抗逆等特性的新种质。
简单重复序列(SSR),又称微卫星,是重复序列为2~6 bp的单元,它们均匀分布于基因组中,是重复序列中主要组成部分之一[3-5]。简单重复序列具有高度的保守性和专一性,可以特异地定位于染色体的某一位置,扩增所得产物可以在高分辨率的8%非变性聚丙烯酰胺凝胶上电泳加以分离,可以检测出不同个体在每个SSR位点上遗传结构的差异[6-7]。
SSR标记主要包括基于基因组序列开发的SSR标记(Genomic-SSR)和基于表达序列开发的SSR标记(EST-SSR)。据研究表明,相对于目前使用的大多数分子标记[8-11],SSR标记具有多态性高、重复性好、共显性遗传等优点,被广泛应用于DNA指纹图谱的构建、遗传多样性分析、分子标记辅助育种等方面[12],但从基因组开发SSR标记,存在步骤繁杂、成本高、效率低等缺点。表达序列标记(EST)来源于基因的转录区,与功能基因紧密连锁,能够直接反映基因的表达信息,同时也避免了构建基因组DNA文库等繁杂的步骤[13-14]。目前杉木已经开发得到的标记主要针对遗传图谱、核心种质资源构建、材性等经济性状,缺少与抗逆相关的标记。本试验利用高通量测序技术获得的干旱胁迫下杉木的转录组数据,结合毛细管电泳检测技术,开发得到一组多态性较好的杉木EST-SSR标记,为今后杉木的遗传多样性分析、逆境胁迫响应等研究奠定了基础。
1 材料与方法
杉木材料来自中国广东的杉木良种基因型“GZ7”首先经组织培养扩繁得到组培苗,并种植于营养钵(营养钵底部直径13 cm、高17 cm、上口直径18 cm),种植所用营养土由V(泥炭土)∶V(沙)∶V(蛭石)∶V(珍珠岩)=3∶1∶1∶1的均匀混合而成。试验以基因型“GZ7”15~20 cm高的1年生杉木组培苗为干旱胁迫处理材料。
SSR扩增所用材料取自2014年在中国广东省龙山国家森林农场收集的300份杉木核心种质中大于0.02遗传距离的24份种质,取当年生杉木嫩叶,液氮速冻后保存于冰箱中-80 ℃备用。初筛引物所用的8份杉木种质随机取自上述24份种质。
1.1 干旱胁迫处理
试验所需的25株组培苗培养于北京林业大学人工气候室,培养条件为70%相对湿度,25 ℃ 16 h光照与20 ℃ 8 h黑暗交替,光照强度为200~250 μmol·m-2·s-1。将25株的组培苗平均分为5组,包括正常浇水的5株苗作为对照,3个不同程度的干旱胁迫处理组(轻度、中度、重度),和一个复水处理组。从对照及各个处理中随机挑选3株组培苗,取顶端约5 cm长带针叶茎段进行液氮速冻之后放到冰箱-80 ℃保存用于总RNA提取。
1.2 转录组测序
将对照及各处理的3株组培苗茎段在液氮中研磨成粉末,等量混合后提取总RNA。依据EASYspin植物RNA提取试剂盒使用说明抽提总RNA,总RNA质量经电泳及RNA完整度检验合格后进行转录组测序。
杉木转录组数据以二代高通量测序技术,在Solexa mRNA-Seq平台上对试验材料提取的总RNA进行测序[15]。使用CLC Genomics Workbench软件(version:6.0.4)[16-18]对预处理后的杉木转录组数据进行从头测序拼接,序列的质量控制参数为:字长=45,最小重叠长度≥300,得到初始非重复序列。再应用CAP3 EST拼接软件对初始非重复序列进行第二次拼接,得到一个最终非重复序列集合。
1.3 EST-SSR挖掘和引物设计
用MISA软件对得到的非重复序列进行EST-SSR分析,查找标准:二、三、四、五、六核苷酸的重复数分别大于6、5、5、5、5次;EST-SSR位点两侧序列长度≥50 bp。
随机挑选120个EST-SSR,用Primer Premier 5软件设计引物,并用Oligo 7进行验证。引物设计时设置的主要参数为:GC含量40%~70%,退火温度55~63 ℃,引物长15~30 bp,预期扩增产物长度200~400 bp,无二级结构和二聚体。
EST-SSR引物于2015年11月由北京睿博兴科生物技术有限公司合成,并利用合适的荧光进行修饰得到可用于毛细管电泳检测的荧光引物,引物用LX加序号命名,如LX-1。挑选遗传距离较远的8份杉木DNA样品通过PCR扩增和8%非变性聚丙烯酰胺凝胶电泳对120对引物进行初步筛选。
1.4 PCR扩增及产物检测
用大于0.02遗传距离的24份杉木DNA材料进行PCR扩增,扩增混合物体系为20 μL,其中含有10 μL 2×2xTSINGKE®Master Mix(蓝色)(北京擎科生物科技有限公司),7 μL无RNase水,1 μL正向引物(含有荧光标记FAM和HEX)、1 μL反向引物和1 μL基因组DNA(30 ng/L)。PCR的程序如下:94 ℃预变性5 min,随后进行35个循环,每个循环包括94 ℃变性30 s,55~63 ℃退火30 s(其中55~63 ℃是选择其中最佳的退火温度),72 ℃下延伸30 s。最后,在72 ℃下延伸10 min,4 ℃保温。所得扩增产物交由北京擎科生物科技有限公司进行毛细管电泳检测。
1.5 数据统计与分析
利用GeneMarker V2.2.0软件分析毛细管电泳结果,得到基因型数据(如果只扩增出1条带,则按纯合基因型处理);利用Convert v1.31软件转换数据格式,用于后续分析;利用POPGEN v1.32软件计算等位基因数;有效等位基因数;观测杂合度、期望杂合度和多态性信息指数。
2 结果与分析
2.1 干旱胁迫下杉木转录组序列拼接
对杉木转录组序列拼接结果进行统计(表1),共得到75 357个片段重叠群,总长度65.29 Mb,平均长度867 bp,总序列的GC含量为39.4%。核苷酸长度在200~400 bp的片段重叠群数量最多,有24 948个,占总片段重叠群数的33.11%,其次是核苷酸长度在400~600 bp的片段重叠群数量,有19 633个,占总片段重叠群数的26.05%。拼接序列中最大拼接长度为26 392 bp,N75长度为552 bp,N50长度为1 289 bp,N25长度为2 430 bp。
2.2 干旱胁迫下杉木转录组中EST-SSR位点的分布特点
在转录组共75 357条非重复序列中有7 326个潜在EST-SSR位点,分布频率(与总非重复序列数量之比)为9.72%,其中单核苷酸型位点有4 943个。在总计2 383个复合型和二、三、四、五、六核苷酸重复类型的EST-SSR中,复合型有384个,二、三、四、五、六核苷酸重复类型分别有925、982、67、11、14个,平均长度分别为15.70、16.96、22.85、25.45、31.71、60.37 bp。杉木转录组序列中平均47.66 kb就能发现1个EST-SSR位点,在2 383个EST-SSR位点中共包含525种重复单元,复合型、二、三、四、五、六核苷酸重复类型EST-SSR分别有384、11、61、44、11、14种,其中复合型、五、六核苷酸重复类型EST-SSR每种重复单元的数量只有1个(表2)。出现频率最高的重复单元类型是AT/TA(453次),占总EST-SSR的19.01%,其次是AG/TC(149次),占总EST-SSR的6.25%,同时,在二核苷酸重复类型中还发现了少量的CG重复(2次)。
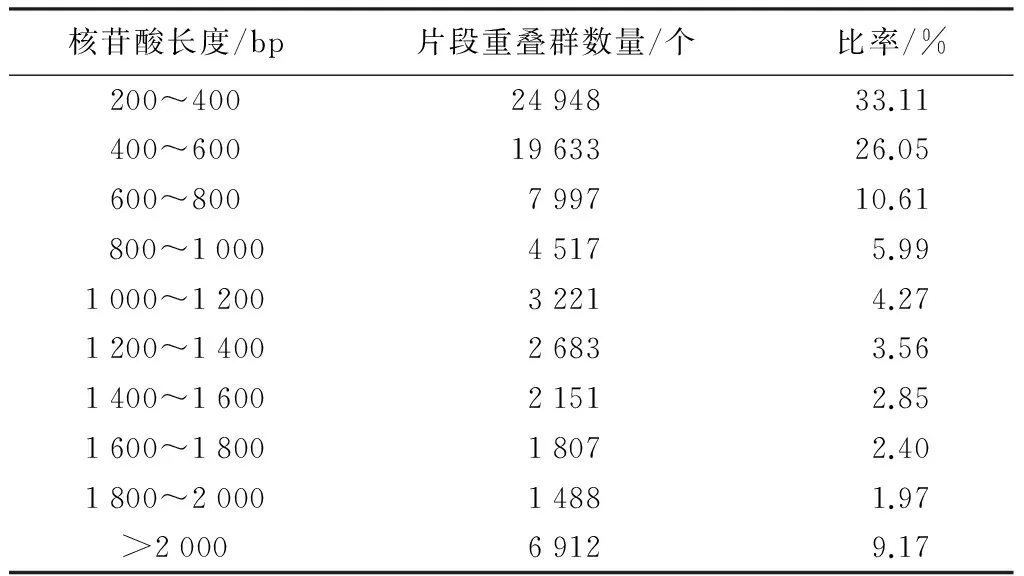
表1 杉木转录组CLC拼接结果
干旱胁迫下杉木转录组中主要的EST-SSR重复类型中,三核苷酸和二核苷酸重复类型数量最多,分别占总EST-SSR数的41.21%和38.82%;复合型EST-SSR也较多,占总EST-SSR数的16.11%;四、五、六核苷酸重复类型EST-SSR数量很少,总计占3.86%。
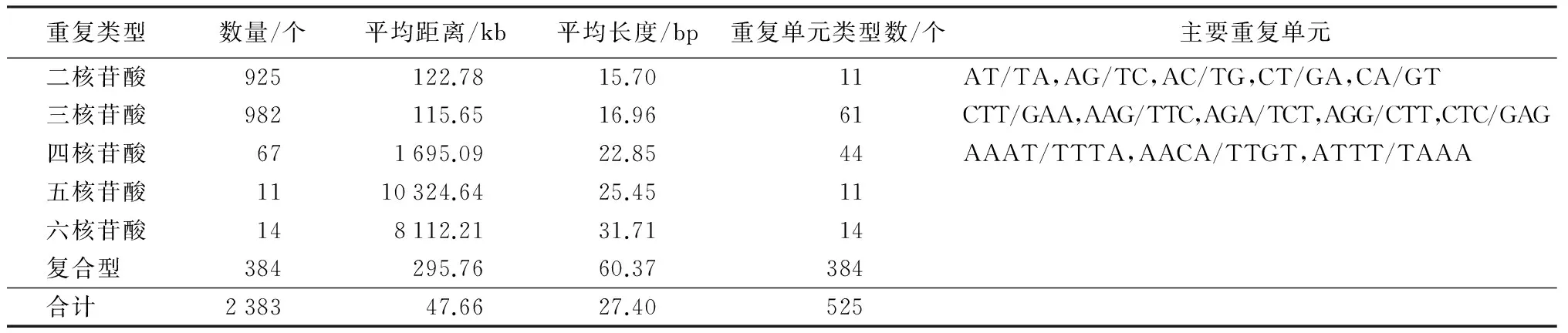
表2 杉木录组中EST-SSR重复单元的分布特征
2.3 干旱胁迫下杉木转录组EST-SSR标记的多态性分析
以8份杉木DNA样品为模板进行PCR扩增,通过8%非变性聚丙烯酰胺凝胶电泳对120对引物初步筛选,部分电泳结果如图1所示,挑选电泳条带好的引物,初选出24对引物用24个杉木DNA样品进行毛细管电泳检测,结果发现在24对引物中有23对引物检测出特异性峰(以引物LX-18为例(图2)),只有1对引物没有检测出特异性峰。

M.DL100 DNA Marker;编号1~8的为引物LX-18,编号9~16的为引物LX-19,编号17~24的为引物LX-20;编号25~32的为引物LX-21。

图2 引物LX-18多态性峰
在24份杉木样品中共检测到94个等位基因,等位基因变异数范围为2~9个,平均每个位点有4.087 0个,其中,引物LX-21、LX-77、LX-84、LX-87最少;LX-18、LX-47最多,有6对引物的等位基因数介于5~9个,9对引物的等位基因数为3个及以下;有效等位基因数范围为1.086 8(LX-77)~6.914 3(LX-47)个,平均有效等位基因数2.089 0个;观测杂合度范围为0.250 0(LX-66)~1.000 0(LX-84),平均0.648 7;期望杂合度范围为0.082 4(LX-79)~0.896 1(LX-47),平均0.429 0;多态性信息指数范围是0.173 2(LX-77)~2.035 3(LX-47),平均0.809 0,说明所开发的EST-SSR具有很高的多态性(表3)。

表3 23对EST-SSR引物在24份杉木样品中的多态性结果
3 结论与讨论
在干旱胁迫下杉木的转录组中,EST-SSR分布的频率较高,为9.72%,高于火炬松(Pinustaeda)的1.2%,海岸松(Pinuspinaster)的2.1%,日本落叶松(Larixkaempferi)的3.85%[19-20],其平均分布距离为47.66 kb。在EST-SSR重复类型中,复合型和二、三、四、五、六核苷酸重复类型都有出现,其中三核苷酸重复类型EST-SSR最多,有982个,占总EST-SSR的41.21%,很多谷类作物如小麦、水稻的EST-SSR研究中也是这种情况[21-22]。在二核苷酸重复单元中AT/TA(占总EST-SSR的19.01%)占主导地位,同时也发现了2次CG重复,表现出了明显的偏倚性。值得注意的是,在干旱胁迫下杉木转录组中出现的复合型EST-SSR较多,有384个,占EST-SSR总数的16.11%,且复合模式多为三核苷酸与三核苷酸复合或三核苷酸与六核苷酸复合,这可能与生物的信使RNA分子上3个碱基决定1个氨基酸的模式有关,EST序列位于基因的编码区,与功能基因紧密连锁,且EST-SSR在生物适应性进化中起着重要的调控作用[23-25],植物对逆境的应答反应或许很大程度上受EST-SSR变异的调控。本试验中,丰富的EST-SSR标记说明杉木可能以这些标记所对应的基因的变异来适应干旱胁迫。
毛细管电泳技术可以一次获得多个位点的多态性信息,具有效率高、成本低、产率高等优点[26],可以利用该技术提高转录组中大量的多态性位点的选择效率。本试验中,通过8%非变性聚丙烯酰胺凝胶电泳初选出的24对引物,用24个杉木DNA样品进行毛细管电泳检测后发现有23对引物能够稳定扩增出有多态性的目的片段,只有1对引物的扩增产物无多态性,这可能是该引物附近的序列复杂性比较低,也有可能是引物所在序列属于多基因家族所致。在23对具有多态性的引物中,引物LX-18、LX-47的等位基因变异数(Na)最多(9个),且这两个引物的重复类型都为三核苷酸重复,验证了低级重复单元SSR的多态性普遍比高级重复单元高的推断[27],因此在设计SSR引物时尽量多选择含低级重复单元的序列。
在杉木的遗传改良过程中,利用EST-SSR对种子园内花粉种类、花粉量的大小及其分布的进行监测,从而有效地提高种子园的经营管理水平。EST-SSR分子标记也具有高可靠性的品种间鉴别能力,通过构建主栽品种的EST-SSR分子标记数据库和设置合理的品种鉴别相似性阈值,能够有效的排除品种内变异对分类鉴定结果的干扰,更加客观和准确的对品种进行分类和鉴别。在林木育种中,EST-SSR标记的应用可以起到加快育种进程、提高选择效率的作用。试验得到的一组杉木EST-SSR分子标记为研究杉木的遗传多样性、种群结构、DNA指纹数据库构建和遗传信息的保存提供了基础,对杉木遗传结构的演化方式及方向、遗传改良策略的研究也具有重要意义。
[1] 中国科学院中国植物志编辑委员会.中国植物志:裸子植物门:7卷[M].北京:科学出版社,1978.
[2] 施季森.林木生物技术育种未来10年若干科学问题展望[J].南京林业大学学报(自然科学版),2012,36(5):1-13.
[3] 王娟娟,赵明,韩雨威,等.微卫星DNA标记开发技术进展及其在经济植物研究中的应用[J].生命科学研究,2016,20(3):260-266.
[4] 余其,王振波,沈真辉,等.分子标记技术及其在大麦传育种中的应用[J].大麦与谷类科学,2016,34(2):1-5.
[5] NOOMOHAMMDI Z, SHEIDAIi M, GHASEMZADEH-BARAKI S, et al. Genetic diversity analysis in Opal cotton hybrids based on SSR, ISSR, and RAPD markers[J]. Genetics & Molecular Research,2013,12(1):256-69.
[6] 梅利那,范付华,崔博文,等.基于马尾松转录组的SSR分子标记开发及种质鉴定[J].农业生物技术学报,2017,25(6):991-1002.
[7] ZHOU Q, LUO D, MA L, et al. Development and cross-species transferability of EST-SSR markers in Siberian wildrye (ElymussibiricusL.) using Illumina sequencing[J]. Scientific Reports,2016,6:20549.
[8] 程晓凤,黄福江,刘明典,等.454测序技术开发微卫星标记的研究进展[J].生物技术通报,2011(8):82-90.
[9] 邓紫宇,项东云,熊涛,等.引种粗皮桉种源遗传多样性的RAPD与SSR分析[J].西北林学院学报,2017,32(1):131-136.
[10] 李明,黄木生,苏烁烁,等.福建杉木王种质资源的遗传多样性[J].森林与环境学报,2016,36(3):312-318.
[11] 郝博搏,邹帆,胡尚力,等.基于ISSR标记的杉木种源遗传多样性分析[J].广西林业科学,2014,91(1):17-23.
[12] 罗冉,吴委林,张旸,等.SSR分子标记在作物遗传育种中的应用[J].基因组学与应用生物学,2010,29(1):137-143.
[13] SAKAIi C, IWANO S, SHIMIZU M, et al. Analysis of gene expression for microminipig liver transcriptomes using parallel long-read technology and short-read sequencing[J]. Biopharmaceutics & Drug Disposition,2016,37(4):220.
[14] 徐阳,陈金慧,李亚,等.杉木EST-SSR与基因组SSR引物开发[J].南京林业大学学报(自然科学版),2014,38(1):9-14.
[15] HU R, WU B, ZHENG H, et al. Global reprogramming of transcription in chinese fir (Cunninghamia lanceolata) during progressive drought stress and after rewatering[J]. International Journal of Molecular Sciences,2015,16(7):15194-15219.
[16] SU C, CHAO Y T, CHANG Y C A, et al. De novo assembly of expressed transcripts and global analysis of the phalaenopsis aphrodite transcriptome[J]. Plant & Cell Physiology,2011,52(9):1501-14.
[17] GARG R, PATEL R K, TYAGI A K, et al. De novo assembly of chickpea transcriptome using short reads for gene discovery and marker Identification[J]. DNA Research,2011,18(1):53-63.
[18] RAMANATHAN B, JINDAL H M, CHENG F L, et al. Next generation sequencing reveals the antibiotic resistant variants in the genome of Pseudomonas aeruginosa[J]. Plos One,2017,12(8):232-241.
[19] 黄海燕,杜红岩,乌云塔娜,等.基于杜仲转录组序列的SSR分子标记的开发[J].林业科学,2013,49(5):176-181.
[20] 杨秀艳,孙晓梅,张守攻,等.日本落叶松EST-SSR标记开发及二代优树遗传多样性分析[J].林业科学,2011,47(11):52-58.
[21] 潘海涛,汪俊君,王盈盈,等.小麦EST-SSR标记的开发和遗传作图[J].中国农业科学,2010,43(3):452-461.
[22] BORA A, CHOUDHURY P R, PANDE V, et al. Assessment of genetic purity in rice (OryzasativaL.) hybrids using microsatellite markers[J]. Biotech,2016,6(1):1-7.
[23] 徐照龙,易金鑫,余桂红,等.藜科6种耐盐植物遗传多样性的EST-SSR分析[J].植物遗传资源学报,2011,12(1):113-120.
[24] XU Y, ZHENG R H, WANG Z J, et al. Identification and characterization of genic microsatellites inCunninghamialanceolata(Lamb.) Hook (Taxodiaceae)[J]. Archives of Biological Sciences,2016,68(2):417-425.
[25] WRIGHTt E P, HUPPERT J L, WALLER Z A. Identification of multiple genomic DNA sequences which form i-motif structures at neutral pH[J]. Nucleic Acids Research,2017,45(6):4673-4680.
[26] YANG Q, DU Z, YANG S, et al. Size-selective separation and overall-amplification of cell-free fetal DNA fragments using PCR-based enrichment[J]. Scientific Reports,2017,7(8):591-602.
[27] ZESHAN A, AFZALl M, ALGHAMDI S S, et al. Evaluation of genetic diversity amongthe pakistani wheat (TriticumaestivumL.) lines through random molecular markers[J]. Braz Arch Biol Technol,2016,59(4):863-874.
