葡萄miR159家族生物信息学分析及靶基因预测分析
2018-03-02葛金涛王丽丽赵统利刘兴满
葛金涛,王丽丽,赵统利,刘兴满
微小 RNA(MicroRNAs)是一类非编码小分子RNA,无论在植物还是动物的生长发育过程中都发挥着重要的调控作用[1]。miR159是已经被证实的8个高度保守的miRNA之一,其在GA信号途径中起到重要的调控作用[2]。
植物MiR159家族对应的靶基因主要为MYB-like和MYB。研究表明在拟南芥(Arabidopsis thaliana)中,已经通过实验验证有8个MYB基因受到miR159的调控,其中有7个属于GAMYB家族[3]。另外,在拟南芥中,miR159通过间接调控靶基因MYB33、MYB65以及 MYB101来控制拟南芥的根长[4];在拟南芥中过表达 miR159a,其靶基因 MYB33和MYB65的表达量均呈下降趋势,说明两者均受到miR159a的调控;在水稻(Oryza sativa)中共有3个GAMYB-like的同源基因,靶基因预测结果表明,miR159与这3个基因均存在相互作用的位点;另外,在水稻中miR159的表达量与GAMYB-like1和GAMYB的表达量呈负相关[5]。在谷物绒毡层细胞中有较多的受GA调控的GAMYB蛋白,两者相互作用共同参与调节细胞程序性死亡,在植物花药及种子的发育中有重要的调控作用[6]。miR159的表达并非贯穿于水稻的整个生长发育过程中,在水稻种子阶段未检测到其表达情况。在GA信号转导过程中,GA调控LFY基因的表达是通过作用结合在LFY基因启动子上的GAMYB作用元件实现的。另外研究表明,在短日照下施用GA可激活LFY基因的启动子活性,从而促进植物开花[7]。大岩桐 miR159与SsGAMYB的相互作用影响了大岩桐的开花时间[8]。因此表明,miR159与其靶基因GAMYB转录因子之间的互作可能在植物种子萌发、成花诱导等过程中有重要的作用。
在金鱼草(Antirrhinum majus L.)等植物的研究中发现,在植物花发育过程中,GA的信号转导是其中重要的一个环节,而GAMYB是GA信号转导中关键作用因子,miR159通过介导其表达量间接发挥调控作用[9]。葡萄(Vitis vinifera)是具有较高经济价值的果树之一,但目前有关葡萄miR159和GAMYB的相关研究尚未见详细报道。随着生物信息学与分子生物学的快速发展,对植物全基因组数据中非编码基因及其靶基因的挖掘、检测、定位等已越来越成为研究的热点。本研究利用生物信息学分析方法初步分析了葡萄miR159家族的系统发生规律、结构预测、靶基因预测等,进而推测其功能与葡萄生长发育过程之间的关系,以期从非编码基因角度为今后葡萄的遗传改良育种提供新的理论依据。
1 材料和方法
1.1 序列获得
在数据库miRbase 21.0中下载葡萄、水稻、大豆(Glycine max)、碧桃(Prunus persica)、二穗短柄草(Brachypodium distachyon)、番茄(Solanum lycopersicum)的miR159家族成员的成熟序列和前体序列。
1.2 vvi-miR159家族的基因定位
利用NCBI数据库BLAST在线工具,在葡萄基因组数据库中对其miR159前体序列进行相似序列搜索(BLAST Assembled RefSeq Genomes)。根据最终的序列匹配结果,对葡萄miR159家族成员进行基因组定位。
1.3 vvi-miR159家族进化分析及碱基保守性分析
利用ClustalW2在线工具对相关miR159基因家族前体序列进行比对。在WebLogo在线平台输入葡萄miR159家族的成熟序列,分析其碱基保守性,并绘制序列Logo图。利用MEGA 6.0多序列比对结果,采用邻近法(Neighbor-joining,NJ)构建相关植物物种miR159家族的系统发育进化树,重复值设置为1000。
1.4 vvi-miR159家族前体二级结构预测分析
在RNAfold WebServer平台上预测葡萄miR159家族成员的前体二级茎环结构,采用默认参数值。
1.5 vvi-miR159家族的靶基因预测
通过psRNATarget平台对靶基因进行预测。提交葡萄miR159家族的成熟序列,选择psRNATarget软件中提供的葡萄JGI数据库作为目标基因搜索库,采用默认参数,预测其目标基因;另外在GenBank中对目标基因功能进行注释,初步确定其功能。
2 结果与分析
2.1 葡萄miR159家族的基因定位
由表1可知,葡萄miR159基因主要分布于葡萄基因组的两条染色体(Chr)上;其中 vvi-miR159a和vvi-miR159b均分布在 Chr15 上,而 vvi-miR159c分布于Chr17上。
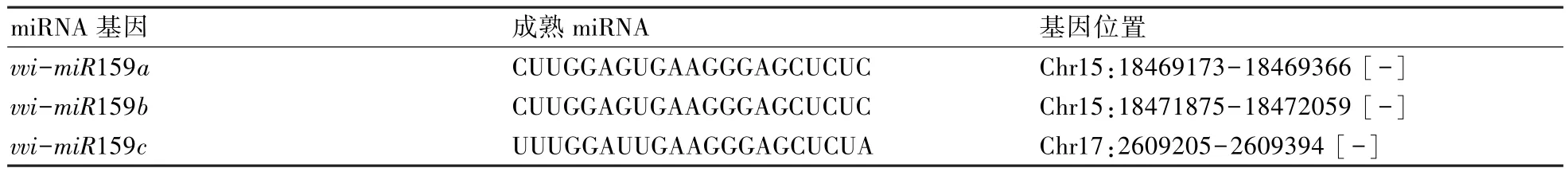
表1 葡萄vvi-miR159基因家族的成熟序列及其基因定位
2.2 进化树构建结果
建立基于水稻、葡萄等物种miR159家族的系统发育树。结果(图1)显示:水稻osa-miR159和玉米zma-miR159家族成员分别聚类到一起;葡萄 vvimiR159家族3个成员(已标注)中vvi-miR159a和vvi-miR159b聚类在一起,与大豆 gma-miR159家族成员的遗传关系较近;而vvi-miR159c分布于另一个进化分支上,与碧桃ppe-miR159聚类在一起,与葡萄家族的另外两个成员间遗传距离相对较远。结合vvimiR159基因定位结果,发现位于同一条染色体(Chr15)上的 vvi-miR159a 和 vvi-miR159b聚类在一个分支,而位于染色体(Chr17)上的 vvi-miR159c则聚类在较远的分支,两者结果一致,符合植物基本进化规律,同时也说明染色体间的miR159基因复制应该早于同一条染色体上的基因复制。
2.3 二级结构预测结果
二级结构预测结果(图2)表明,葡萄vvi-miR159家族成员的前体序列都可以形成稳定的茎环结构,且其3个成员的成熟序列都产生在其对应前体序列的5′端臂上。
2.4 成熟序列碱基的保守性分析
对植物miR159的91个家族成员(图3A)以及葡萄的3个家族成员(图3B)的成熟序列进行碱基保守性分析。结果表明,植物miR159家族的标准序列为 5′-UUUGGAUUGAAGGGAGCUCUA-3′,且在 2~20位碱基处保守性整体较高。葡萄vvi-miR159家族成员成熟序列的碱基种类基本上与标准序列一致(图3B),其中vvi-miR159c的碱基种类与标准序列完全一致,vvi-miR159a和 vvi-miR159b成熟序列的碱基种类与标准序列基本一致,仅在第1、7以及21位碱基与标准序列存在差异(U转为C;U转为G;A转为C)。说明葡萄miR159家族成员成熟序列的碱基保守性较高。

图1 基于前体序列构建的系统进化树

图2 二级结构预测结果
2.5 靶基因预测结果
经在线平台预测,在葡萄 JGI基因库中,vvimiR159家族成员都预测到有目标基因存在,对应的目标基因有2个,分别为GSVIVT01012447001和GSVIVT01037362001(表2)。 在NCBI中通过 BLAST进行功能预测,结果表明GSVIVT01012447001为GAMYB转录因子家族成员,而GSVIVT01037362001的功能目前尚未得到确认。据此可初步推断,葡萄miR159与其靶基因之间的互作在植物花期调控、根发育等过程中发挥重要调控作用。
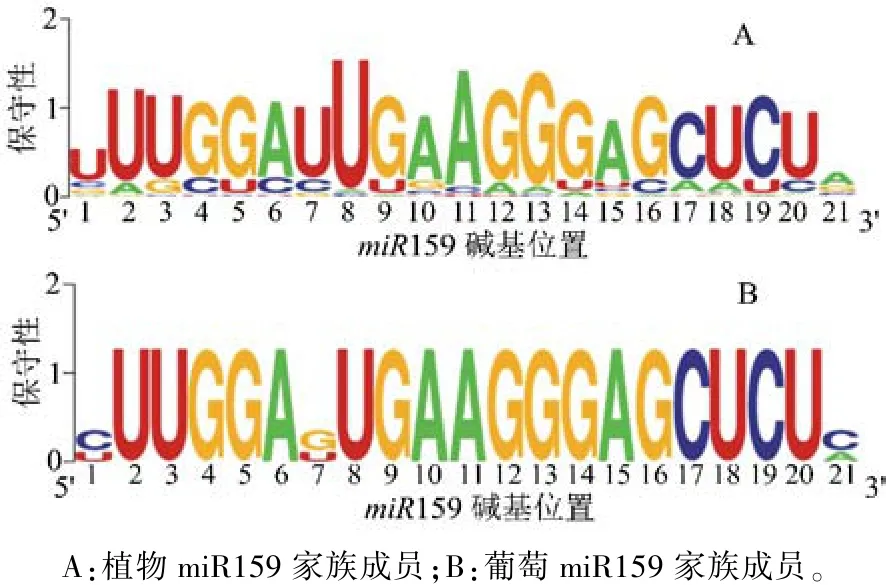
图3 植物miR159家族成员及葡萄miR159家族成员成熟序列的碱基保守性分析结果
3 结论与讨论
自从Llave等[10]2002年首次在拟南芥中克隆到miRNA后,越来越多的研究发现,miRNA不仅在植物应对逆境胁迫反应过程中有重要的调控作用,而且还参与调控植物的生长发育过程,包括植物叶片形态建成、花期[11]以及花器官[12]的变化等。 目前,主要有三类miRNA在植物开花过程中发挥主要作用,分别为miR172、miR159和 miR156。 这3种 miRNA是植物在成花期重要的调控因子,其中miR159主要通过GA信号途径介导相关开花基因的表达[13]。
本文通过对葡萄vvi-miR159家族成员的前体序列进行系统发育进化分析,结果发现与基因定位分析结果一致;另外对葡萄vvi-miR159的前体序列进行二级结构预测,对其成熟序列的碱基保守性以及目标基因进行分析预测,结果显示,葡萄miR159家族成员的前体序列都可形成稳定的二级结构,且其对应成熟序列的碱基基本上与miR159家族的标准序列保持一致,即植物miR159的成熟序列在不同物种中差异很小,这与李晓燕等[14]在大岩桐中的研究结果相同。靶基因预测结果表明葡萄miR159家族的主要目标基因为GSVIVT01012447001,经功能预测分析,发现其为GAMYB转录因子家族成员,这与在大麦(Hordeum vulgare)、水稻[5]、拟南芥[15]、马铃 薯 ( Solanum tuberosum)[16]、草莓(Fragaria ananassa Duch.)[17]以及落叶松属[18]植物中的研究结果相一致。

表2 葡萄miR159家族成员与其对应的靶基因
但近期有实验表明,在短日照条件下,拟南芥miR159ab双突变体中,MYB33和MYB65两个基因的表达量都上调,开花时间出现了延迟现象,而在myb33-myb65突变体植株中,花期相对于野生型植株无变化;外施GA后,myb33-myb65功能缺失突变体表型也没有发生变化,开花时间提前20 d左右,但miR159ab双突变植株花期没有变化,说明在拟南芥中MYB33和MYB65并不是控制开花途径的关键基因。水稻gamyb突变植株在正常培养条件下,抽穗期和花期都未发生变化,据此表明GAMYB在植物花期调控中的作用较为复杂,仍有待于探索[19]。
另外,miRNA及其靶基因之间的调控是一个极为复杂的网络;另外,最新研究表明,circular RNA(环形RNA)中富含miRNA的结合位点,可在细胞中结合相应的miRNA,从而影响miRNA靶基因的表达水平[20]。本研究中发现葡萄 vvi-miR159的主要靶基因是GAMYB家族,但是葡萄vvi-miR159c对应的靶基因功能尚未确定,因此葡萄miR159c是否还参与对其他类型的靶基因的调控,是否还参与葡萄其他生长发育的调控过程还有待进一步确认。此外,有相关研究[21]表明,植物miR159成熟序列5′端的第10和第11位碱基之间存在对目标基因的剪切作用位点,但在葡萄中miR159的作用位点是否有时空与物种特异性,这有待深入探索分析。本文首次对葡萄vvimiR159家族进行了初步的生物信息学分析,并对其靶基因进行了初步在线预测。今后仍有必要进一步采用转基因技术对葡萄vvi-miR159及其靶基因的功能与互作关系进行更加深入的探讨,从而从非编码基因角度为今后葡萄新品种改良与选育提供新的参考。
[1] Guo C,Xu Y,Shi M,et al.Repression of miR156 by miR159 regulates the timing of the juvenile-to-adult transition in Arabidopsis[J].Plant Cell, 2017, 29(6): 1293-1304.
[2] Allen R S, Li J,Stahle M J, et al.Genetic analysis reveals functional redundancy and the major target genes of the Arabidopsis miR159 family [J].Proc Natl Acad Sci U S A, 2007(104): 16371-16376.
[3] Allen R S,Li J,Alonso-Peral M M,et al.MicroR159 regulation of most conserved targets in Arabidopsis has negligible phenotypic effects [J].Silence, 2010(1): 18.
[4] Xue T,Liu Z,Dai X,et al.Primary root growth in Arabidopsis thaliana is inhibited by the miR159 mediated repression of MYB33, MYB65 and MYB101 [J].Plant Sci, 2017, 262:182-189.
[5] Tsuji H, Aya K,Ueguchi-Tanaka M, et al.GAMYB controls different setsof genes and isdifferentially regulated by microRNA in aleurone cells and anthers[J].Plant J,2006,47(3):427-444.
[6] Aya K,Ueguchi-Tanaka M,Kondo M, et al.Gibberellin modulates anther development in rice via the transcriptional regulation of GAMYB [ J].Plant Cell, 2009, 21:1453-1472.
[7]Achard P,Herr A,Baulcombe D C,et al.Modulation of floral development by a gibberellin-regulated microRNA [J].Development, 2004, 131: 3357-3365.
[8] Li X,Bian H,Song D,et al.Flowering time control in ornamental gloxinia (Sinningia speciosa) by manipulation of miR159 expression [J].Annals of Botany,2013,111: 791-799.
[9] Millar A A, Gubler F.The GAMYB like genes MYB33 and MYB65,are microRNA regulated genes that redundantly facilitate anther development [J].Plant Cell,2005,17: 705-721.
[10]Llave C,Xie Z,Kasschau K D,et al.Cleavage of scarecrow-like mRNA targets directed by a class of Arabidopsis miRNA[J].Science, 2002, 297(5589): 2053-2056.
[11] Vaucheret H,Vazquez F,Crete P,et al.The action of ARGONAUTE1 in the miRNA pathway and its regulation by the miRNA pathway are crucial for plant development[J].Genes Dev, 2004, 18: 1187-1197.
[12] Kidner C A, Martienssen R A.The role of ARGONAUTE1(AGO1) in meristem formation and identity [J].Dev Biol,2005, 280: 504-517.
[13] Li Y,Alonso-Peral M, Wong G, et al.Ubiquitous miR159 repression of MYB33/65 in Arabidopsis rosettes is robust and is not perturbed by a wide range of stresses [J].BMC Plant Biol, 2016, 16(1): 179.
[14]李晓燕.MiR159和miR172表达对大岩桐花发育的调控作用及其机理研究[D].杭州:浙江大学,2012.
[15]Palatnik J F, Wollmann H, Schommer C, et al.Sequence and expression differences underlie functional specializaton of Arabidopsis microRNAs miR159 and miR319 [J].Dev Cell,2007, 13: 115-125.
[16] Yang J,Zhang N,Mi X,et al.Identification of miR159s and their target genes and expression analysis under drought stress in potato [J].Comput Biol Chem,2014, 53: 204-213.
[17]Csukasi F, Donaire L, Casanal A, et al.Two strawberry miR159 family members display developmental-special expression patternsin the fruit receptacle and cooperatively regulate Fa-GAMYB [J].New Phytol, 2012, 195: 47-57.
[18] Li W F,Zhang SG,Han SY,et al.Regulation of LaMYB33 by miR159 during maintenance in Larix kaempferi (Lamb.)[J].Carr Plant Cell Tiss Organ Cult, 2013, 113: 131-136.[19] Sun X, Zhang Y, Zhu X, et al.Advances in identification and validation of plant microRNAs and their target genes[J].Physiologia Plantarum,2014,152:203-218.
[20] Chu Q, Zhang X, Zhu X, et al.PlantcircBase: a database for plant circular RNAs [J].Mol Plant,2017, 10(8):1126-1128.
[21] Wang C, Han J, Korir N K,et al.Characterization of target mRNAs for grapevine microRNAs with an integrated strategy of modified RLM-RACE,newly developed PPM-RACE and qPCRs[J].Journal Plant Physiology, 170: 943-957.
