Taq酶体外合成DNA时模板错位机制的探讨
2018-03-01李慧慧
李慧慧,王 涛
(1.中国科学院 合肥物质科学研究院 强磁场科学中心,安徽 合肥 230031;2.中国科学技术大学 生命科学学院,安徽 合肥 230026)
利用Taq DNA聚合酶体外合成DNA过程中,当反应体系中缺少与模板链互补配对的dNTP底物时,产物合成并不会在底物缺失位点处终止,聚合反应继续进行.为研究此复制缺陷现象,设计一系列模板用于DNA体外酶促合成.除了已知的碱基错配机制,笔者发现存在另一种“模板错位”机制,即模板中与底物非Watson-Crick互补配对的碱基位点首先进行收缩滑动,形成模板bulge结构后再继续进行酶促合成反应.这项研究有助于提高DNA样品合成保真度以及继续深入探索体外DNA合成的详细机制.
Taq DNA聚合酶;复制缺陷;模板错位;模板bulge结构;互补dNTP底物缺失
Taq DNA聚合酶(以下简称Taq酶)作为分子克隆以及DNA体外合成的重要工具酶,具有非常广泛的应用[1].实验室常用的DNA聚合酶有Taq酶、pfu酶、Klenow Fragment等,其中Taq酶是所发现的耐热DNA聚合酶中活性最高的一种,为DNA快速高效扩增奠定了坚实的基础[2].但Taq酶没有3′-5′外切酶活性,因而在合成中对错误引入的核苷酸没有校正功能[3-4],使得其合成产物保真度相对较低,并具有在目的产物序列的3′-端额外再加上一个核苷酸尾巴的特性,尤其是腺苷酸的加尾现象更常见[5-6].对于Taq酶的研究,一方面致力于对相关催化结构域进行改造而提高其保真性[7-10];另一方面,利用Taq酶保真性不高的特点发展易错PCR技术,即通过改变PCR条件来提高扩增产物中错误碱基引入的频率,从而扩增基因文库[11-13].提高基因扩增中碱基错配频率的主要方法有以下几种:1)使用4种非最优浓度配比的dNTP底物池;2)添加Mn2+;3)提高Mg2+浓度等.其中基因扩增中的错误率与各dNTP底物偏离最优浓度配比的程度密切相关,因此人为控制4种dNTP底物的组分使其偏离最优浓度配比是诱导发生碱基错配的重要手段之一[11].通常在易错PCR体系中,共同存在着4种碱基dNTP底物,但当某特定碱基dNTP底物出现缺失,即各底物浓度配比达到极端偏离时,DNA酶促反应还能否继续进行以及相关复制机制均值得进一步探究.
为研究这种极端条件下的DNA合成反应,笔者选用了体外DNA酶促合成反应体系作为研究对象.该体系只包括PCR过程中的一个循环,与PCR相比更加简明,也能避免多轮循环引起的错误累积而使复杂程度扩大,便于简化实验结果的分析,更适合研究DNA合成反应中的复制缺陷现象和机制.同时该研究也将有助于帮助改进酶促合成方法,提高样品合成质量[14-15].在该研究中选择合成体系相对简明的TBA-11(d(GGTTGGTGTGG))作为目的产物.TBA-11序列在经典的抗凝血酶适配体TBA(thrombin binding aptamer)完整序列基础上截短了4个碱基,可形成稳定的G-三链体结构[16].TBA-11序列只含有G和T 2种碱基,对于该序列的完整复制合成反应来说,dGTP和dTTP底物都是必不可少的.然而发现当向合成体系中只添加单一底物dTTP时,合成反应并未因另一底物dGTP缺失而中断.在缺失互补底物的相应复制位点处,产物链还可以继续延伸1~2个碱基.基于这一现象,推测可能存在3种合成机制:1)非模板依赖的加尾机制(如图1A);2)碱基错配机制(如图1B);3)模板错位机制(如图1C),即模板中与底物未互补配对的碱基位点首先进行收缩滑动,形成模板bulge结构后合成反应再继续进行.为探索具体是采用了哪种机制,笔者设计了一系列非TBA-11序列的模板进行DNA合成反应.首先设计平末端模板加入相对应的底物验证非模板依赖的加尾机制;其次在模板中设置2对已错配的碱基加入底物进行合成反应验证碱基错配机制(如图1B);最后通过含有无碱基但仍保留糖磷骨架的相应模板进行合成反应验证模板错位机制.该研究是针对Taq酶体外合成中出现的模板错位机制进行探讨,将为后续探索详细的合成机制打下基础,并有望将该方法应用于DNA样品合成,有助于规避DNA体外合成错误,提高样品的合成效率、纯度以及产量.
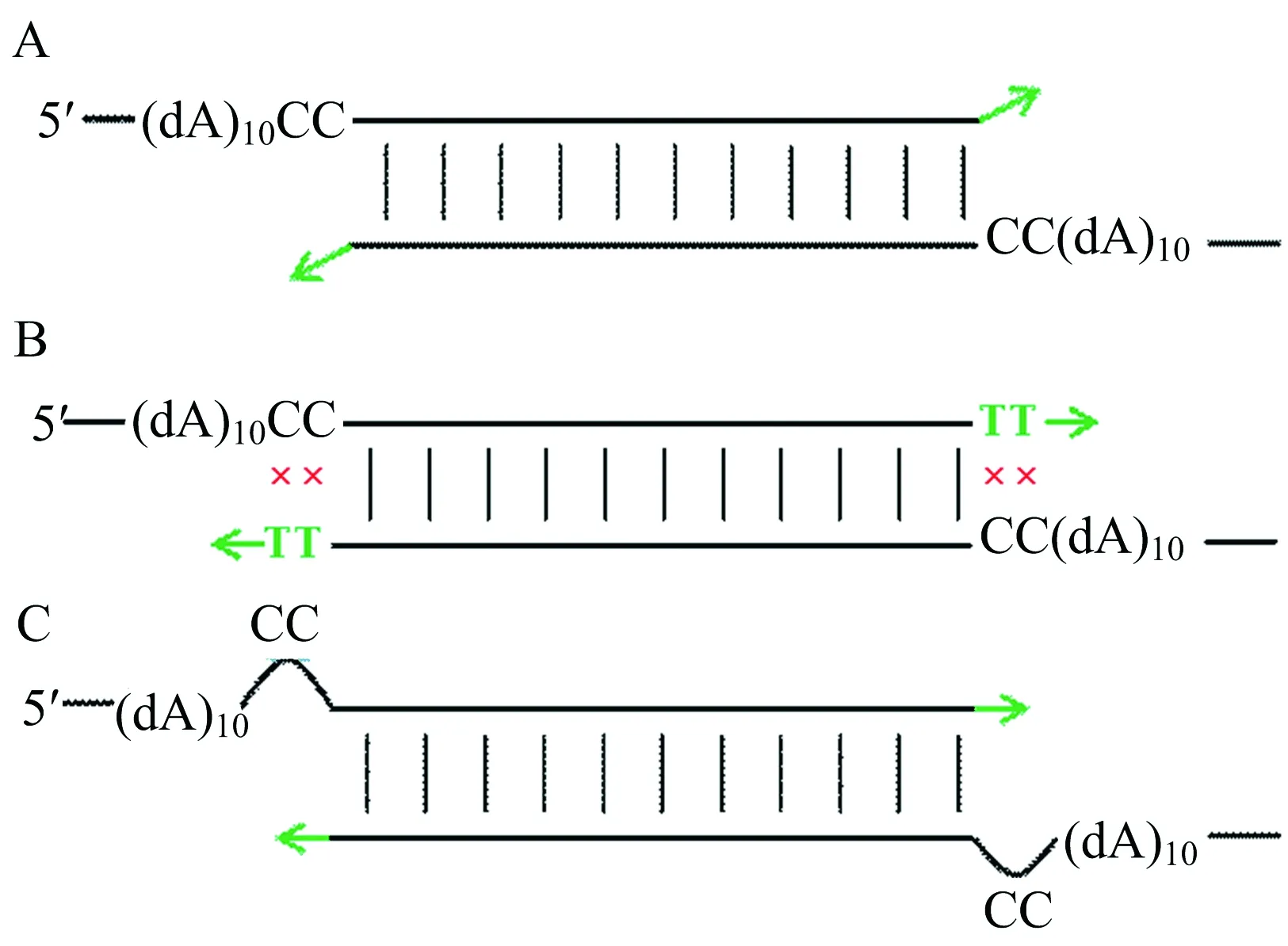
A:非模板依赖加尾机制;B:碱基先错配再延续合成反应;C:模板中非互补匹配碱基先形成bulge再发生合成反应(黑色代表模板链;绿色代表产物;红色表示此处碱基错配;蓝色表示未配对碱基).图1 互补底物缺失仍延续酶促聚合反应时所涉及的复制机制猜测
1 材料与方法
1.1 材 料
用于该实验的Taq DNA聚合酶购自生工生物工程(上海)股份有限公司.
1.2 方 法
1.2.1 引物设计
表1为实验过程中设计的全部DNA合成模板序列及电泳中用到的Marker,表中所列全部序列均由生工生物工程(上海)股份有限公司化学合成.

表1 模板及Marker序列
注:下划线表示该段序列可以形成回文结构;F:full-length;FR:fragment; mAB:该序列第11位为无碱基的糖磷骨架;dAB:该序列第11位和第12位为无碱基的糖磷骨架;/idSP/:脱碱基修饰位点.
1.2.2 DNA合成反应
20 μL的DNA合成体系为:10×聚合酶缓冲液((500 mmol·L-1KCl, 100 mmol·L-1Tris-HCl, pH 9.0, 1% Triton X-100)0.22 μm膜过滤,4 ℃保存)2 μL,10 mmol·L-1MgCl21 μL,Taq酶1 U,4种dNTP底物母液浓度为1 mmol·L-1,dNTP的比例参照目的产物序列的碱基组成,反应体积用ddH2O补至20 μL.DNA合成反应温度和时间:95 ℃,5 min热激处理后72 ℃反应1 h或6 h.反应产物进行20%变性PAGE电泳检测,300 V恒压2 h左右(跑胶时间具体参照反应条带的大小);Marker均采用由化学固相合成制备的DNA寡核酸链,序列为酶促反应中所用模板以及酶促反应不同阶段的产物.
2 结 果
2.1 DNA酶促反应过程中出现复制缺陷现象
为研究dNTP浓度配比极端变化条件下Taq酶催化DNA合成的反应过程,首先使用体外合成TBA-11的反应体系.实验涉及引物-模板序列的构建(24nt-S除外)采用单独一条寡核苷酸链,通过引物部分中回文序列自我配对形成二聚体,构成引物-模板复合物,如图1A所示.这样设计的引物-模板复合物,避免了酶促反应体系中含有任何多余引物或模板而干扰实验结果.
以TBA-11的模板序列进行DNA酶促合成反应,复制产物序列第一位正常情况下应为G;而当dTTP为唯一底物时,如图2A所示,虽然在该复制位点底物dTTP与模板序列(C)并不互补匹配,但PAGE电泳实验结果显示酶促反应并未在此复制位点立即终止,引物在原长度上最长仍可延伸2个碱基,即有2个dTTP加至引物3′末端(图2A:lane 1,2).然而酶促聚合反应并未能连续进行到底,没有得到全长完整的复制产物,这可能是因为TBA-11产物序列多处复制位点为碱基G.为避免该因素对于实验结果的干扰,设计模板C2A10:前2个与底物dTTP不能互补配对的碱基C位点紧邻引物3′-端下游,从第3位起后续为10个能正常互补碱基配对的T位点序列.反应结果如图2B:1 h内会发生合成反应,反应产物条带有多条(图2B:lane 3),6 h所得产物在1 h产物的基础上继续延伸,最长条带接近全长(图2B:lane 4).从可能涉及的机制上考虑(如图1所示),连续的非模板加尾,碱基的错配,或者是将模板中与底物未配对的碱基位点首先进行收缩滑动,形成模板bulge结构后再继续进行合成反应,都可能产生上述观察到的实验现象.那么究竟采用了哪种机制呢?
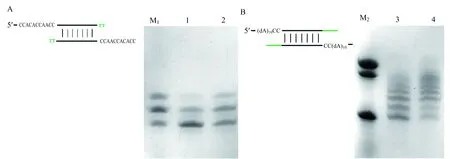
A:TBA-11体系中只加入底物dTTP 的反应;B:C2A10体系中只加入底物dTTP 的反应(黑色代表模板链;绿色代表产物).M1:TBA-11-T + TBA-11-T+1 + TBA-11-T+2;1:TBA-11体系中只加dTTP底物反应1 h;2:TBA-11体系中只加dTTP底物反应6 h; M2:C2A10 + C2A10-FG + C2A10-F;3:C2A10体系中只加dTTP底物反应1 h;4:C2A10体系中只加dTTP底物反应6 h(M1和M2为每个反应体系的Marker,为上述所列序列的混合物,具体序列见表1).图2 DNA酶促反应中的复制缺陷现象
2.2 验证排除连续性非模板依赖加尾机制
为进一步验证是否是由于连续非模板依赖性加尾造成的产物链延伸,设计一系列模板(24nt-S,24nt-P,24nt-P+T),序列见表1.运用与TBA-11合成体系相同的酶促反应条件,DNA合成结果如图3所示.
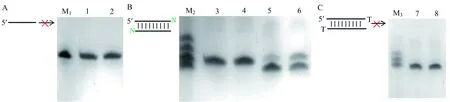
A:随机单链DNA模板24nt-S的合成反应;B:平末端模板24nt-P的合成反应;C:已加尾模板24nt-P+T的合成反应(黑色代表模板链;绿色代表产物;红色代表未发生反应). M1:24nt-S;1:24nt-S体系中只加dTTP底物反应1 h;2:24nt-S体系中只加dTTP底物反应6 h; M2:24nt-P + 24nt-P+1+ 24nt-P+2+ 24nt-P+3;3:24nt-P体系中只加dATP底物反应1 h;4:24nt-P体系中只加dATP底物反应6 h;5:24nt-P体系中只加dTTP底物反应1 h;6:24nt-P体系中只加dTTP底物反应6 h;M3:24nt-P+T + 24nt-P+2+ 24nt-P+3;7:24nt-P+T体系中只加dTTP底物反应1 h;8:24nt-P+T体系中只加dTTP底物反应6 h;M:Marker.图3 排除非模板加尾的验证实验
对于一条随机单链(24nt-S,该序列不能自我配对)来说,任何合成反应都没有发生(图3A:lane 1,2),而平末端序列(24nt-P,该序列为完整的回文结构,可以全部自身配对形成平端互补双链)会加上1个碱基T或A的尾巴(图3B:lane3-6),但是碱基T的加尾速率(图3B:lane 5,6)比碱基A慢(图3B:lane 3,4).对于已经加上一个碱基T的模板(24nt-P+T)来说,不能再进一步继续加尾(图3C:lane 7,8).这就排除了上述模板C2A10发生的DNA合成反应由连续非模板依赖性加尾促成的可能.
2.3 验证排除碱基错配机制
如果模板C2A10反应的进行不是由连续非模板依赖性加尾引起,那么是否是通过模板中C碱基与底物T发生错配之后再进行产物合成的呢?为验证猜想,设计如下模板进行实验:当引物的3′端直接与模板形成两对C-T碱基错配(模板CTA10),模拟错配模板下的合成情况.实验结果如图4所示.
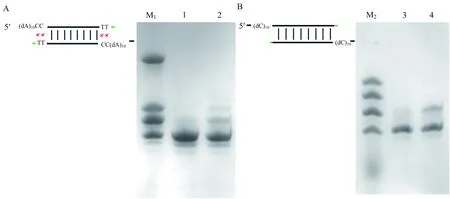
A:模板中有2对C-T碱基错配(CTA10)的合成;B:模板C10的合成(黑色代表模板链;绿色代表产物;红色表示此位点处碱基错配). M1:CTA10+ CTA10+1+ CTA10+2+ CTA10-F;1:CTA10体系中只加dTTP底物反应1 h;2:CTA10体系中只加dTTP底物反应6 h;M2:C10+ C10+1+ C10+2+ C10+4;3:C10体系中只加dTTP底物反应1 h;4:C10体系中只加dTTP底物反应6 h;M:Marker.图4 排除错配机制的验证实验
如图4A所示:1 h内不发生DNA合成反应(图4A:lane 1),6 h只有部分模板发生加上1~2个碱基的合成反应(图4A:lane 2),这与以C2A10为模板的反应结果差异很大(图2B:lane 3,4).而当模板中合成起始位点处是10个连续碱基C位点时(模板C10),加入dTTP后进行合成反应,1 h内无法进行DNA合成(图4B:lane 3),6 h内只有部分模板加上1个碱基(图4B:lane 4),说明通过C-T碱基错配的酶促合成效率非常低.以上实验结果排除了C2A10模板反应产物是通过碱基错配生成的可能.
2.4 脱碱基模板合成验证存在模板错位机制
用脱碱基序列mAB-A10和dAB-A10作为模板进行合成反应(mAB-A10代表序列中有1个位点为无碱基的糖磷骨架;dAB-A10代表序列中有连续2个位点为无碱基的糖磷骨架).以模板mAB-A10和dAB-A10进行DNA合成反应,结果如图5所示.
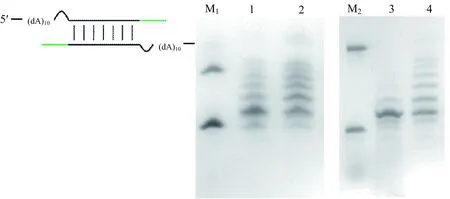
黑色代表模板链;绿色代表产物.M1:mAB-A10+ A10-F1;1:mAB-A10体系中只加dTTP底物反应1 h;2:mAB-A10体系中只加dTTP底物反应6 h;M2:dAB-A10+ A10-F2;3:dAB-A10体系中只加dTTP底物反应1 h;4:dAB-A10体系中只加dTTP底物反应6 h;M:Marker.图5 脱碱基模板的DNA合成实验
图5显示,1 h内都发生了合成反应(lane 1,3),6 h合成反应继续延伸(lane 2,4).但是dAB-A10合成效率相比于mAB-A10略有降低.该模板的设计直接规避了碱基错配的可能性,实验结果进一步证明了bulge结构的存在.
2.5 不同bulge长度影响合成进行
设计不同bulge长度的模板进行合成反应,bulge处均为C碱基,长度分别为2个碱基(模板C2A10)、5个碱基(模板C5A10)以及10个碱基(模板C10A10).底物只有dTTP时,DNA合成反应结果如图6所示.
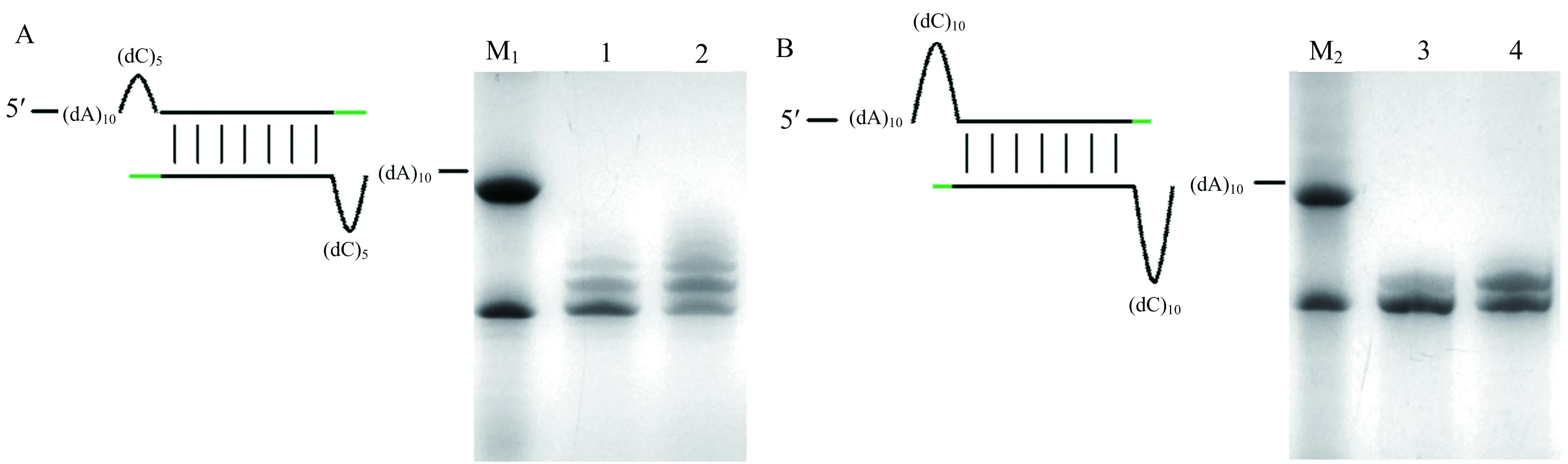
A:bulge处为5个C碱基(模板C5A10);B:bulge处为10个C碱基(模板C10A10) (黑色代表模板链;绿色代表产物;蓝色表示未配对碱基).M1:C5A10+ C5A10-FG;1:C5A10体系中只加dTTP底物反应1 h;2:C5A10体系中只加dTTP底物反应6 h;M2:C10A10+ C10A10-FG;3:C5A10体系中只加dTTP底物反应1 h;4:C5A10体系中只加dTTP底物反应6 h;M:Marker.图6 不同bulge长度模板的DNA合成实验
图6显示,当bulge越长,所能合成的产物最大长度越短,C5A10模板只能出现加入2个碱基的产物条带(图6A:lane 1, 2),而C10A10模板的产物只有部分可以加上1个碱基(图6B:lane 3, 4),合成反应效率与模板C2A10(图2B:lane3, 4)相比大幅度降低.如果模板合成产物只是单纯经过C-T碱基错配机制或连续性非模板依赖加尾机制,那么在同样的反应时间内,C10A10模板应该也可以像C5A10模板那样,生成加上2个错配碱基的产物条带,但是并没有观察到这样的实验现象.这就从侧面支持了模板错位机制,同时也表明形成bulge结构的大小是有限制的.
3 讨 论
笔者通过设计一系列不同模板,研究了互补dNTP底物缺失极端条件下Taq酶催化体外合成DNA的反应过程,提出模板错位的机制.通过该机制,合理解释了观察到的复制缺陷现象.首先观测到在非TBA-11序列的模板中,当dNTP底物中不含有与复制模板链互补配对的碱基时,反应并不会在相应的非互补匹配位点终止,产物链仍能继续延伸,但所得产物表现出模板复制时碱基缺失现象.而在紧邻引物链3′-端的复制位点处,当采用含有无碱基但仍保留糖磷骨架的相应模板进行合成反应时,发现合成反应仍能进行,说明bulge结构的存在.另外,还发现bulge长度可能受到Taq酶结合底物的活性位点处空间大小限制,bulge太长则与Taq酶活性位点的空间大小不匹配从而并不容许该结构的形成[17-20].但是在脱碱基序列作为模板的DNA合成实验中,反应效率较参照组合成反应体系降低,这可能是由脱碱基位点结构的不稳定性造成的.此外,由于模板错位机制中bulge结构的存在,使得Taq酶在DNA链上的延续滑动将要受到bulge阻碍,所以其酶促DNA合成反应的效率相应也较正常复制时降低.在该研究基础上笔者后续准备进一步对模板错位机制开展研究:一方面,对于bulge结构分别位于引物链或模板链的位置、bulge的长度和序列等因素对于反应的影响进行更深入的研究;另一方面,比较具有不同保真度的DNA聚合酶对于底物缺失该极端实验条件下的合成情况,明确pfu DNA聚合酶等实验室常用的高保真酶是否也存在笔者提出的底物缺失极端条件时的模板错位机制.该研究有助于探索体内DNA复制修复等相关的DNA生物合成过程.此外对DNA酶促合成反应机制的研究也有助于规避DNA体外合成错误的发生,提高体外聚合酶催化DNA合成的反应效率、合成样品的纯度以及产量等.
4 结束语
以Taq酶为研究材料,发现了DNA合成存在区别于碱基错配的模板错位机制,即模板中与底物非Watson-Crick互补配对的碱基位点首先进行收缩滑动,形成模板bulge结构后再继续进行合成反应.
[1] GARG P, BURGERS P M J. DNA polymerases that propagate the eukaryotic DNA replication fork[J]. Crit Rev Biochem Mol Biol, 2005, 40 (2): 115-128.
[2] TINDALL K R, KUNKEL T A. Fidelity of DNA synthesis by the Thermus aquaticus DNA polymerase[J]. Biochemistry, 1988, 27 (16): 6008-6013.
[3] MCCULLOCH S D, KUNKEL T A. The fidelity of DNA synthesis by eukaryotic replicative and translesion synthesis polymerases[J]. Cell Res, 2008, 18 (1): 148-161.
[4] ZHAO G, GUAN Y. Polymerization behavior of Klenow fragment and Taq DNA polymerase in short primer extension reactions[J]. Acta Biochim Biophys Sin (Shanghai), 2010, 42 (10): 722-728.
[5] CULJKOVIC B, SAVIC D, STOJKOVIC O, et al. Poly(A) tailing of ancient DNA: a method for reproducible microsatellite genotyping[J]. Anal Biochem, 2003, 318 (1): 124-131.
[6] OHTSUBO Y, NAGATA Y, TSUDA M. Efficient N-tailing of blunt DNA ends by Moloney murine leukemia virus reverse transcriptase[J]. Sci Rep, 2017, 7: 41769.
[7] YAMAGAMI T, ISHINO S, KAWARABAYASI Y, et al. Mutant Taq DNA polymerases with improved elongation ability as a useful reagent for genetic engineering[J]. Front Microbiol, 2014, 5: 461.
[8] AREZI B, MCKINNEY N, HANSEN C, et al. Compartmentalized self-replication under fast PCR cycling conditions yields Taq DNA polymerase mutants with increased DNA-binding affinity and blood resistance[J]. Frontiers in Microbiology, 2014, 5: 408.
[9] STRERATH M, GLOECKNER C, SCHNUR A, et al. Directed DNA polymerase evolution: effects of mutations in motif C on the mismatch-extension selectivity of Thermus aquaticus DNA polymerase[J]. Chembiochem, 2007, 8 (4): 395-401.
[10] ASCHENBRENNER J, MARX A. Direct and site-specific quantification of RNA 2′-O-methylation by PCR with an engineered DNA polymerase[J]. Nucleic Acids Res, 2016, 44 (8): 3495-3502.
[11] 高义平, 赵和, 吕孟雨, 等. 易错PCR研究进展及应用[J]. 核农学报, 2013, 27 (5): 607-612.
[12] CLARK J M. Novel non-templated nucleotide addition reactions catalyzed by procaryotic and eucaryotic DNA polymerases[J]. Nucleic Acids Res, 1988, 16 (20): 9677-9686.
[13] KULP C. Reviews of science for science librarians: PCR: the discovery that powered the genomic revolution in modern science[J]. Science & Technology Libraries, 2014, 33 (2): 124-142.
[14] ELIAS A A, CISNEROS G A. Computational study of putative residues involved in DNA synthesis fidelity checking in Thermus aquaticus DNA polymerase I[J]. Adv Protein Chem Struct Biol, 2014, 96: 39-75.
[15] NELISSEN F H, GIRARD F C, TESSARI M, et al. Preparation of selective and segmentally labeled single-stranded DNA for NMR by self-primed PCR and asymmetrical endonuclease double digestion[J]. Nucleic Acids Res, 2009, 37 (17): e114.
[16] LIMONGELLI V, DETITO S, CEROFOLINI L, et al. The G-triplex DNA[J]. Angew Chem Int Ed Engl, 2013, 52 (8): 2269-2273.
[17] LIU B Y, XUE Q Z, TANG Y, et al. Mechanisms of mutagenesis: DNA replication in the presence of DNA damage[J]. Mutation Research/Reviews in Mutation Research, 2016, 768: 53-67.
[18] LOEB L A, MONNAT R J JR. DNA polymerases and human disease[J]. Nat Rev Genet, 2008, 9 (8): 594-604.
[19] CHEN J M, FEREC C, COOPER D N. Complex multiple-nucleotide substitution mutations causing human inherited disease reveal novel insights into the action of translesion synthesis DNA polymerases[J]. Human Mutation, 2015, 36 (11): 1034-1038.
[20] KIM Y, EOM S H, WANG J, et al. Crystal structure of Thermus aquaticus DNA polymerase[J]. Nature, 1995, 376 (6541): 612-616.
