陕西关中地区羊乳中细菌多样性的分析
2018-02-28高佳媛邵玉宇雷飞艳王毕妮张富新
高佳媛,邵玉宇,雷飞艳,王毕妮,张富新
(陕西师范大学 食品工程与营养科学学院,陕西 西安,710119)
羊乳营养价值较高,富含蛋白质、脂肪、乳糖、氨基酸、矿物质以及多种维生素,有“白色血液”之称,是人类主要的乳源之一[1]。由于羊乳的成分丰富,为不同的细菌提供了优良的生存环境,是细菌生长繁殖的天然培养基。羊乳中的细菌种类和数量对乳品的营养品质和保质期有着重要影响,完整的解析羊乳中细菌微生物的多样性,对有效利用原料羊乳中有益菌、防止有害菌滋生及提高羊乳安全和质量具有重要意义。
自然界大多数微生物处于活的非可培养状态,且环境中的微生物群落信息比较丰富和复杂,采用常规的分离、纯化、培养及鉴定方法不易获取样品中完整的微生物群落结构信息[2-3]。随着分子生物学方法的发展,焦磷酸测序、AB SOLiD sequencer、单分子测序技术和Illumina Solexa高通量测序平台[4]等新的检测技术受到广大学者的关注,这些方法测序通量大、速度快、精度高、成本较低,不需要对微生物进行纯培养,可直接从样品中提取DNA,进行特异性扩增后测序,为更好的挖掘环境中微生物新资源提供了很好的平台,成为环境样品中菌种鉴定、研究微生物群落组成结构的重要手段[5-7]。
目前,对于食品中细菌多样性的研究主要集中在发酵乳和泡菜,对于原料乳的细菌多样性研究也以牛乳为主,对羊乳中细菌群落组成研究较少。本研究以陕西关中地区的羊乳为原料,提取羊乳中微生物宏基因组DNA,对16S rRNA基因V3-V4区进行扩增、高通量测序,研究羊乳中细菌的组成及丰度,解析羊乳中细菌微生物的多样性,对羊乳中有害微生物的防控具有指导价值,同时为进一步开发和利用羊乳中微生物资源提供研究基础。
1 材料与方法
1.1 羊乳
样品采自陕西省杨凌、宝鸡、渭南3个地区的乳样。人工挤乳后将样品快速置于无菌管中,编号后立即将样品置于冰盒中运回实验室于-40 ℃冰箱中保存。
每个地区分别取两个牧区的混合乳样进行多样性分析。杨凌西农羊场、宝鸡地区和渭南地区各采取2个乳样,编号分别为Y1、Y2、B1、B2、W1和W2。
1.2 实验试剂
粪便DNA基因组提取试剂盒,QIAGEN公司;DNA Marke、PCR扩增试剂盒,天根生化科技(北京)有限公司;PCR引物,上海生工生物工程公司。
1.3 仪器设备
L5S L6/L6S紫外可见分光光度计,上海仪电分析仪器有限公司;DYY-12电泳仪,北京六一仪器厂;BIO-BEST200E凝胶成像仪,美国西蒙;ARITIK型PCR扩增仪,美国热电公司;LDZX-30KBS手轮式自动不锈钢立式压力蒸汽灭菌器,上海申安医疗器械厂。
1.4 实验方法
1.4.1 羊乳中总DNA提取及纯度测定
1.4.2 16S rRNA基因的V3-V4区特异性扩增
本试验以样品总DNA稀释液(l00 ng/uL)作为PCR扩增反应的模板进行目的片段的扩增,以341F:5’-CCTAYGGGRBGCASCAG-3’和907R:5’-GGACTACNNGGGTATCTAAT-3’为引物,对细菌16S rRNA基因的V3-V4区进行扩增。扩增体系(25 μL)为:10×PCR buffer (Mg2+) 2.5 μL,dNTP mix (10 mmol/L) 2.0 μL,引物(10 pmol/μL)各1 μL,Tap DNA polymerase (5 U/μL) 0.3 μL,模板DNA (100 ng/μL) 1μL,用ddH2O补充至25 μL。PCR反应条件为:94 ℃预变性5 min后,进行30次循环(94 ℃变性1 min,58 ℃退火1 min,72 ℃延伸2 min),最后72 ℃末端延伸10 min,4 ℃保温。扩增完毕后,用1.5%浓度的琼脂糖凝胶进行电泳检测,30 min后在凝胶成像仪紫外下查看结果。
1.4.3 PCR产物的混样和纯化及测序
将扩增产物等量混合后检测扩增产物,成功后回收扩增产物。使用TruSeq®DNA PCR-Free Sample Preparation Kit建库试剂盒进行文库构建,构建好的文库经过Qubit和Q-PCR定量,文库合格后,用HiSeq2500 PE250执行测序。
1.4.4 高序列质量控制
参照Qiime(V1.7.0,http://qiime.org/scripts/split_libraries_fastq.html)[10]的Tags质量控制流程来处理原始数据(Raw Tags)。截去Barcode和引物序列后,使用FLASH(V1.2.7, http://ccb.jhu.edu/software/FLASH/)[11]对每个样品的reads进行拼接、质控[12],得到有效数据(Clean Tags)、去除嵌合体序列[13-14]后得到高质量序列(Effective Tags)进行多样性分析。
1.4.5 序列生物信息学分析
用Uparse软件(Uparse v7.0.1001,http://drive5.com/uparse/)[15]对所有乳样的全部有效Tags进行聚类,按照序列一致性为97%将序列聚类成为操作分类单元(OTUs,Operational Taxonomic Units),同时选取OTUs代表性序列并进行物种注释,通过使用Mothur方法和SILVA(http://www.arb-silva.de/)[16]的SSUrRNA数据库用来进行物种注释(阈值设定为0.8~1),从而得到分类学信息并在各个水平进行分类。Chao1,Simpson,Shannon,ACE指数用Qiime软件来计算。
2 结果与分析
2.1 样品高通量测序结果
将提取好的羊乳总DNA的16S rRNA基因进行V3-V4区扩增,合格后进行高通量测序。通过 Illumina HiSeq高通量测序,原始数据质控、去除嵌合体处理后,本研究共从6个测序样品中获得了225 424条高质量的16 SrRNA基因序列(Effective Tags),将所有样品的Effective Tags进行聚类,以97%的一致性将序列聚类成OUT,然后对OTUs的代表序列进行物种注释,平均每个样品聚成673个OTUs。各乳样测序信息见表1。由表1可知,本测序结果Q20都大于98%,表示有98%以上的碱基测序的正确率均达到了99%,表明测序结果准确可靠,可进行下一步分析。
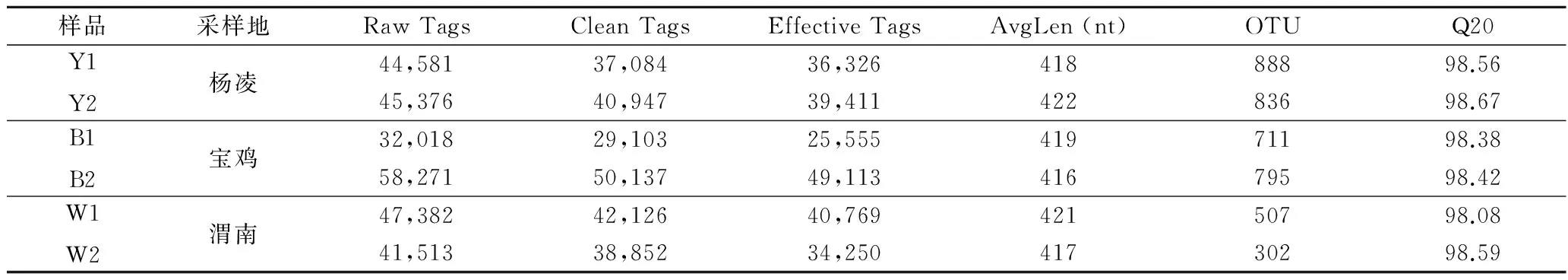
表1 各样品的测序标签
注:Y、B、W分别代表陕西杨凌市西农羊场、宝鸡市千阳县、渭南市富平县3个地区的乳样,下同。
2.2 羊乳中细菌群落构成
2.2.1 特定物种分类树
通过对所有乳样进行物种分类,筛选特别关注的物种(相对丰度排名前10的属)进行物种分类树统计[17],结果见图1。从左至右的分类水平分别为界、门、纲、目、科、属、种;不同灰度的圆圈表示不同的乳样,对应左侧图例;圆圈中扇形的大小代表在该分类水平上细菌的相对丰度大小;细菌名称下方的两个数字均表示相对丰度百分率,前者表示该分类占该样品中所有分类物种的百分率,后者则表示该分类占该样品所选取的分类物种中的百分率。从图1可以看出,样品中在门水平上丰度大于1%的物种依次为Proteobacteria、Firmicutes和Bacteroidetes;在纲水平上丰度较大的物种有Gammaproteobacteria、Betaproteobacteria和Bacilli等;在目分类水平上含量较多的细菌目有Pseudomonadales、Enterobacteriales和Burkholderiales等;含量超过1%的细菌科有Enterobacteriaceae、Moraxellaceae、Pseudomonadaceae和Alcaligenaceae等;在属水平上丰度较大的物种依次为Enterobacter、Acinetobacter、Pseudomonas、Staphylococcus、Massilia和Stenotrophomonas等。
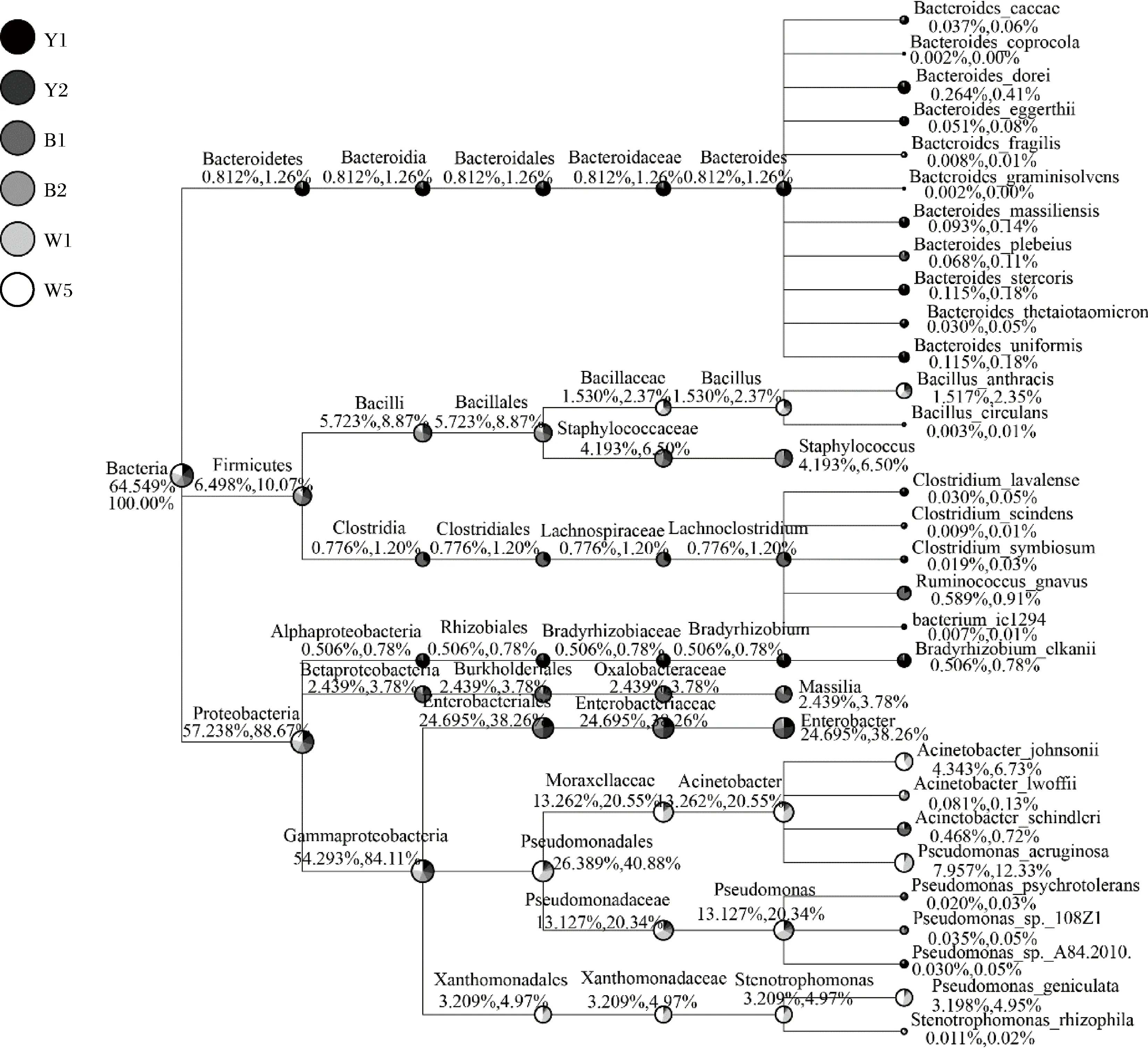
图1 各乳样中特定物种分类树Fig.1 Classification tree of specific species in samples
2.2.2 物种相对丰度展示
选取每个乳样或分组在属水平上最大丰度排名前10的物种,生成物种相对丰度柱形累加图,以便直观查看各样品在属水平上,相对丰度较高的物种及其比例。物种相对丰度柱形图如图2和图3。
从图2和图3中可以看出,6个样品在属水平上物种丰度大小依次均为Enterobacter、Acinetobacter、Pseudomonas、Staphylococcu、Stenotrophomonas和Massilia等,其中杨凌地区Enterobacter最高,占总菌属37.4%,在Y1和Y2两个牧场中分别占32.9%和41.9%;在宝鸡两个牧场分别占33.1%和34.5%;Enterobacter在渭南地区的乳样所占比例最小,仅占2.87%。渭南地区的优势细菌属为Acinetobacter,占总细菌数的33.9%,W1和W2两个牧场各占28.9%和38.8%。
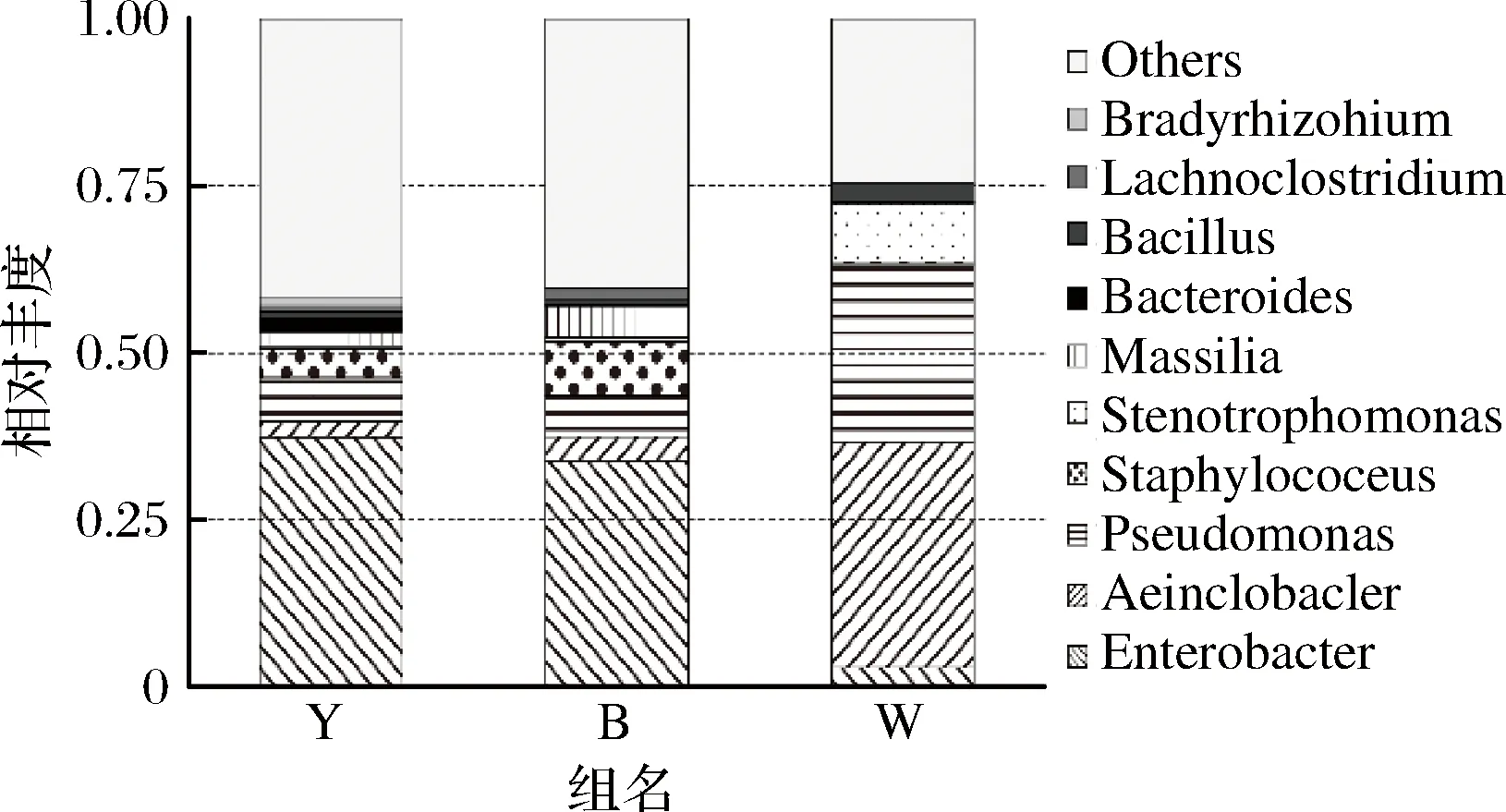
图2 各分组在属水平上的物种相对丰度柱形图Fig.2 The relative abundance of species at the genus level between groups
图3 各样品在属水平上的物种相对丰度柱形图Fig.3 The relative abundance of species at the genus level between samples
2.2.3 羊乳中细菌群落门水平分布
本研究中,在所有羊乳样品中共有27种细菌门被鉴定出,含量超过0.1%的细菌门有Proteobacteria、Firmicutes、Bacteroidetes、Actinobacteria、Thaumarchaeota和Cyanobacteria,各个样品中的细菌群落门分布见表2。
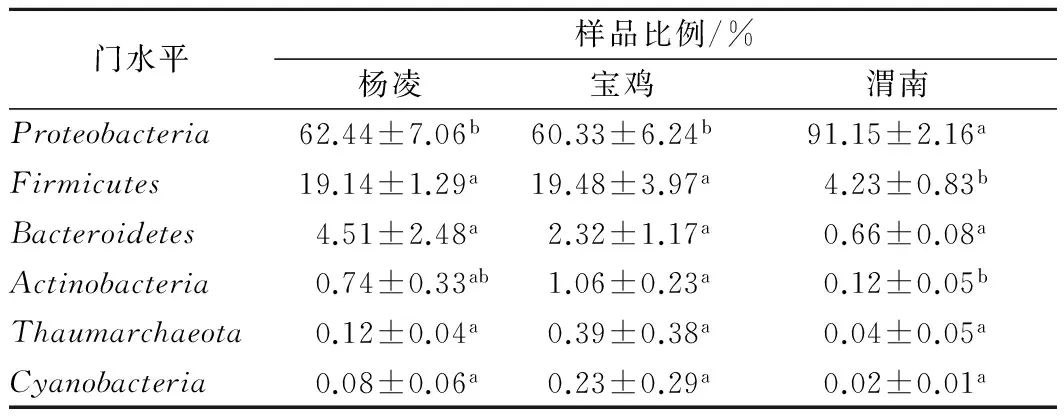
表2 羊乳中细菌群落在门水平分类
由表2可知,3个地区羊乳中的细菌门组成存在一定的差异。Proteobacteria为陕西关中地区羊乳中的优势菌群门,其中渭南地区所占比例最高,为91.15%,在杨凌和宝鸡地区分别占62.44%和60.33%,且渭南地区的Proteobacteria含量和其他两个地区具有显著性差异。Firmicutes在杨凌和宝鸡地区所占比例较高,分别为19.14%和19.48%,在杨凌地区仅占4.23%,且杨凌、宝鸡地区和渭南地区的Firmicutes含量具有显著性差异。Bacteroidetes在杨凌、宝鸡和渭南地区所占比例分别为4.51%、2.32%和0.66%,各地区间无显著性差异。Actinobacteria、Thaumarchaeota和Cyanobacteria在3个地区的细菌门中所占比例较低,且杨凌和宝鸡地区这3种细菌门的含量均高于渭南地区。
2.2.4 羊乳中细菌群落属水平分布
在所有羊乳中共鉴定出了354种细菌属,含量超过0.5%的细菌属有Enterobacte、Acinetobacter、Pseudomonas、Staphylococcus、Stenotrophomonas、Massilia、Bacillus、Streptococcus、Bacteroides、Delftia、Lachnoclostridium、E.Shigella、Janthinobacterium、Lactococcus、Sphingomonas和Bradyrhizobium。排名前5的细菌属在3个地区的相对含量分布见表3。

表3 羊乳中细菌群落在属水平分类
由表3可以看出,杨凌两个羊场主要以Enterobacter、Pseudomonas和Acinetobacter等细菌种属为主,宝鸡两个羊场主要以Enterobacter、Staphylococcus和Pseudomonas等细菌种属为主,渭南两个羊场主要以Acinetobacter、Pseudomonas和Stenotrophomonas等细菌种属为主。Enterobacter是关中地区羊乳中的优势细菌属,在总细菌属中含量最多,3个地区所占比例分别为37.41%、33.82%和2.87%。Acinetobacter在渭南地区所占比例较高,为33.85%,在杨凌和宝鸡地区所占比例较小,分别为2.31%和3.63%。Pseudomonas在渭南地区的相对含量高于杨凌和宝鸡地区,分别为6.54%、6.26%和26.59%,杨凌和渭南地区、宝鸡和渭南地区的Pseudomonas含量具有显著性差异,杨凌和宝鸡地区的Pseudomonas含量无显著性差异。Staphylococcus在杨凌、宝鸡和渭南地区所占比例分别为4.27%、7.94%和0.37%,各地区间无显著性差异。Stenotrophomonas在渭南地区含量较高,占8.5%,在杨凌和宝鸡地区仅占0.51%和0.63%。
2.2.5 羊乳中细菌群落种水平分布
在陕西3个地区所有羊乳中共鉴定出了细菌种有826种,含量超过0.5%的细菌种有P.aeruginosa、A.johnsonii、P.geniculata、B.anthracis、D.tsuruhatensis、E.coli、J.lividum、L.lactis、R.gnavus、S.thermophilus和B.elkanii,3个地区排名前5的细菌种相对含量分布见表4。

表4 羊乳中细菌群落在种水平分类
由表4可以看出,菌种P.aeruginosa在3个地区中含量最高,为陕西关中地区的优势菌株。P.aeruginosa在杨凌和宝鸡地区所占比例较小,分别占1.17%和0.81%,在渭南地区的乳样中所占比例达21.8%,显著高于其他两个地区。菌种A.johnsonii在渭南地区所占比例也较高,而在杨凌和宝鸡地区仅占0.45%和1.26%。渭南地区的P.geniculata、B.anthracis和D.tsuruhatensis含量均显著高于杨凌和宝鸡地区。
2.3 不同地区羊乳样品细菌群体间的多样性分析
2.3.1 不同地区羊乳中细菌的多样性指数
一般来说,序列相似性在97%以上的聚类成为一个OTU的序列,可断定可能是源自于同一个种的序列。本实验对不同乳样在97%相似性下的多样性进行分析,用Chao,Shannon, Simpson,Observed species和ACE这5个多样性指数,综合体现羊乳中细菌群落的丰富度和均匀度,结果见表5。
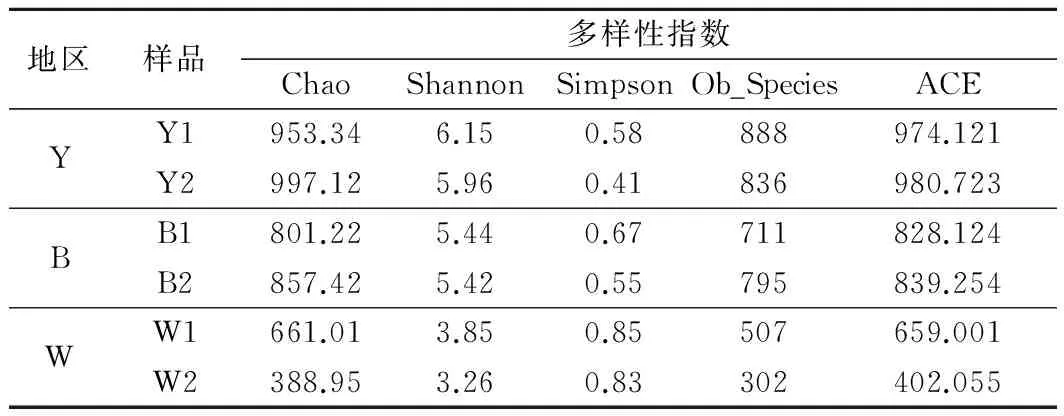
表5 不同地区羊乳样品的多样性指数
群落丰富度由Chao或ACE指数决定,值越高,表明越丰富越高;群落多样性则由Shannon和Simpson值决定;Shannon值越高、Simpson值越低,则表明群落多样性越高,Observed species表示实际测到的OTU数量。由表5可以看出,不同地区羊乳样品中细菌的多样性各有差异。杨凌和宝鸡地区羊乳中细菌Chao指数、ACE指数和Shannon指数值明显高于渭南地区,Simpson值低于渭南地区,表明杨凌和宝鸡这两个地区羊乳中细菌群落丰富度和多样性较高,渭南地区羊乳中细菌群落丰富度和多样性较低。杨凌和宝鸡地区羊乳的OTUs数也高于渭南地区,说明杨凌和宝鸡地区羊乳样品的细菌种类较为丰富。
2.3.2 UPGMA聚类树
为了研究陕西3个地区不同乳样间细菌群落结构的相似性,使用UPGMA(Unweighted pair-group method with arithmetic mean)聚类分析方法对乳样进行了聚类分析,将聚类结果与各乳样在门水平上的物种丰度进行整合从而构建出样品的Weighted Unifrac聚类树[18-19],结果见图4。由图4可知,所有样品共聚成两大类和三小类。其中第一小类包括Y1和B2,第二小类包括Y2和B1,第三小类包括W1和W2。总体来说,不同地区之间的羊乳菌群有一定的差异性,聚类之间的不同可能与不同的采样地点有关;但是,样品之间微生物群落结构差距不大,有一定的相似性,这可能是由于采样地区都位于陕西省关中地带,乳山羊的品种、饮食情况、所处环境气候等因素都比较相似,导致其乳样的菌群结构比较接近。
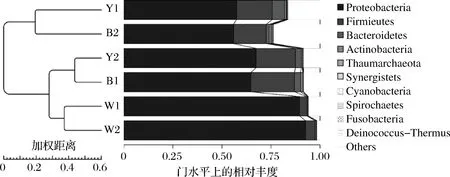
图4 基于Weighted Unifrac距离的UPGMA聚类树Fig.4 UPGMA clustering tree based on Weighted Unifrac distance
3 讨论
高通量测序技术具有高灵敏度、低运行成本、测序深度高等突出优势,目前已经广泛于应用食品工业,主要针对其中的微生物多样性进行分析,使人们对食品中微生物群落结构有更多的了解。目前,国内外对羊乳细菌多样性研究比较少,本研究通过高通量测序技术对陕西关中3个地区(渭南富平、宝鸡千阳、杨凌西农羊场)羊乳的菌群结构和微生物多样性进行研究,在所有羊乳样品中共鉴定出了13种细菌门,含量超过0.5%的细菌门分别为Proteobacteria(71.31%)、Firmicutes(14.28%)、Bacteroidetes(2.50%)和Actinobacteria(0.65%),其中Proteobacteria是优势菌门,这4种菌门的含量占到总数的88.73%。在属的水平上,在所有羊乳样品中含量均超过0.5%分别为Enterobacter、Acinetobacter、Pseudomonas和Staphylococcus等。TORMO[20]等对法国朗格多克-鲁西永比利牛斯的羊乳中细菌多样性进行分析,发现优势菌属有Staphylococcus、Arthrobacter和Serratia,与本试验结果共有的优势菌属是Staphylococcus。总体而言,羊乳中有相似的细菌菌群,但是由于地区不同,其饮食,海拔,温度,湿度等有很大的差异,导致不同地区之间羊乳中微生物群落结构有差异。
通过高通量测序对其他原料乳中微生物多样性的研究也有报道。韩瑞芳[21]对山东和宁夏地区牛乳中细菌多样性进行分析,结果发现山东地区乳样中优势种属细菌菌群是Acinetobactersp和Klebsiellasp,宁夏地区在乳样中21株优势种属细菌菌群是L.lactissubsp.lactis和Acinetobactersp。张敏[22]等对新疆地区不同原料乳中微生物多样性分析,发现牛乳中优势菌属是Pseudomonas、Enterobacter和Lactococcus,驼乳优势菌属是E.Shigella、Enterobacter和Lactococcus,马乳优势菌属是Leuconostoc、Acinetobacter和Lactococcus。COLLADO[23]等通过实时定量PCR对健康人乳中细菌多样性进行分析,结果发现优势菌群是Staphylococcus、Streptococcus、Bifidobacterium和Lactobacillus。目前,研究牛乳中微生物多样性已经非常普遍。通过高通量测序,我们了解到牛乳中优势菌群主要是Acinetobactersp、Klebsiella、Enterobacte和Lactococcus等,和羊乳中微生物结构具有一定的差异性,可能是由于羊乳中酪蛋白含量、脂肪球直径、乳糖比例和各种氨基酸含量都不同于牛乳[24],乳成分的差异可能导致其中微生物组成的差异。
此外,在对羊乳中的细菌进行分析时,除了P.aeruginos和A.johnsonii等含量较多的菌种外,我们发现还存在一定量的L.reuteri、W.cibaria、P.aeruginosa菌种及细菌属Staphylococcus、Alcaligenes、Psychrobacte等。KRASSE[25]等通过对患有牙龈炎的患者服用L.reuteri,发现L.reuteri可以有效治疗牙龈出血和牙龈炎症。熊涛[26]等发现L.reuteriNCU801发酵液对E.coliCMCC444有明显的抑制作用。姚沛琳[27]等通过对引起龋齿的一种主要病原菌S.mutansATCC 25175的研究,发现了W.cibaria对龋齿具有一定的预防作用。这些细菌的益生性能的研究为以后羊乳中益生菌的提取和开发利用提供一定的科学依据。同时,Staphylococcus经常被检测出来与食品中毒有关[28],主要是由于食用了含有S.aureus的食品,而对于原料乳则是常见的感染来源[29],S.aureus污染会导致乳产品质量下降,造成巨大经济损失[30]。羊乳中存在着一些在贮藏期间会产生耐热脂肪酶和耐热蛋白酶的嗜冷菌,如细菌属Alcaligenes[31]、Psychrobacter[32],细菌种P.psychrotoleran[33]、P.celer[34]等,这些细菌在巴氏杀菌处理后仍然会有残留,在贮藏期间产生的耐热脂肪酶能分解脂肪产生游离的脂肪酸,耐热蛋白酶能分解蛋白导致酪蛋白胶束不稳定,使乳发生凝固现象,且产生的氨基酸会使产品发生褐变,导致乳及乳制品的风味劣化[35]。研究这些有害菌,对于防止羊乳中有害菌滋生,提高羊乳安全和质量具有重要意义。
4 结论
以陕西地区3个牧场的羊乳样品为研究对象,利用高通量测序技术研究羊乳中细菌多样性和群落的组成结构。对不同地区羊乳中细菌群落构成分析显示,所有羊乳样品中细菌门有27种,以变形菌门(Proteobacteria)和厚壁菌门(Firmicutes)为主,分别占71.31%和14.28%;细菌属有354种,以肠杆菌属(Enterobacter)、不动杆菌属(Acinetobacter)和假单胞菌属(Pseudomonas)为主,分别占24.69%、13.26%和13.13%;细菌种有826种,优势细菌种有铜绿假单胞菌(P.aeruginos)、约氏不动杆菌(A.johnsonii)和弯曲假单胞菌(P.geniculata),分别占7.96%、4.34%和3.20%。
[1] 李贺, 马莺. 羊乳营养及其功能性特性[J]. 中国乳品工业, 2017, 45(1): 29-33.
[2] GIRAFFA G, NEVIANI E. DNA-based, culture-independent strategies for evaluating microbial communities in food-associated ecosystems[J]. International Journal of Food Microbiology, 2001, 67(1): 19-34.
[3] 吴咚咚. 基于活的但非可培养细菌降解毒死蜱及其酶降解效果[D]. 金华: 浙江师范大学, 2015.
[4] 秦楠, 栗东芳, 杨瑞馥. 高通量测序技术及其在微生物学研究中的应用[J]. 微生物学报, 2011, 51(4): 445-457.
[5] CAPORASO J G, LAUBER C L, WALTERS W A, et al. Colloquium paper: global patterns of 16S rRNA diversity at a depth of millions of sequences per sample[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(Suppl 1): 4 516.
[6] YOUSSEF N, SHEIK C S, KRUMHOLZ L R, et al. Comparison of species richness estimates obtained using nearly complete fragments and simulated pyrosequencing-generated fragments in 16S rRNA gene-based environmental surveys[J]. Applied & Environmental Microbiology, 2009, 75(16): 5 227-5 236.
[7] HESS M, SCZYRBA A, EGAN R, et al. Metagenomic discovery of biomass-degrading genes and genomes from cow rumen[J]. Science, 2011, 331(6 016): 463-467.
[8] CABRERA-RUBIO R, MIRA-PASCUAL L, MIRA A, et al. Impact of mode of delivery on the milk microbiota composition of healthy women[J]. Journal of Developmental Origins of Health & Disease, 2016, 7(1): 54-60.
[10] CAPORASO J G, KUCZYNSKI J, STOMBAUGH J. QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7(5): 335-336.
[12] BOKULICH N A, SUBRAMANIAN S, FAITH J J, et al. Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing[J]. Nature Methods, 2013, 10(1): 57-59.
[13] EDGAR R C, HAAS B J, Clemente J C, et al. Uchime improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011, 27(16): 2 194-2 200.
[14] HAAS B J, GEVERS D, EARL A M, et al. Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons[J]. Genome Research, 2011, 21(3): 494-504.
[15] EDGAR R C. Uparse: highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998.
[16] WANG Q, GARRITY G M, TIEDJE J M, et al. Naive bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied & Environmental Microbiology, 2007, 73(16): 5 261-5 267.
[17] JR D S, HUGENHOLTZ P, KELLER K, et al. Nast: a multiple sequence alignment server for comparative analysis of 16S rRNA genes[J]. Nucleic Acids Research, 2006, 34(Web Server issue): 394-399.
[18] LOZUPONE C, LLADSER M E, DAN K, et al. Unifrac: an effective distance metric for microbial community comparison[J]. Isme Journal, 2011, 5(2): 169-172.
[19] LOZUPONE C A, HAMADY M, KELLEY S T, et al. Quantitative and qualitative β diversity measures lead to different insights into factors that structure microbial communities[J]. Applied & Environmental Microbiology, 2007, 73(5): 1 576-1 585.
[20] TORMO H, DELACROIX A, LOPEZ C, et al. Farm management practices and diversity of the dominant bacterial species in raw goat′s milk[J]. International Journal of Dairy Science, 2007, 6(1): 29-43.
[21] 韩瑞芳. 利用分子学方法研究生鲜乳中细菌的多样性[D]. 济南: 齐鲁工业大学, 2015.
[22] 张敏, 张艳, 黄丽丽,等. 基于16S rDNA高通量测序方法比较新疆西北部地区乳品中微生物的多样性[J]. 食品科学, 2017,38(20):27-33.
[23] COLLADO M C, DELGADO S, MALDONADO A, et al. Assessment of the bacterial diversity of breast milk of healthy women by quantitative real-time PCR[J]. Letters in Applied Microbiology, 2009, 48(5): 523-528.
[24] 刘蒙佳, 周强. 羊乳与牛乳理化特性比较[J]. 饮料工业, 2010, 13(4): 11-13.
[25] KRASSE P, CARLSSON B, DAHL C, et al. Decreased gum bleeding and reduced gingivitis by the probioticLactobacillusreuteri[J]. Swedish Dental Journal, 2005, 30(30): 55-60.
[26] 熊涛, 邓耀军, 廖良坤, 等. 罗伊氏乳杆菌NCU801的鉴定及抑菌性能研究[J]. 食品与发酵工业, 2015, 41(2): 24-29.
[27] 姚沛琳. 乳酸菌抑制变异链球菌生物膜形成的研究[D]. 无锡: 江南大学, 2015.
[28] HENNEKINNE J A, BRUN V, BUYSER M L D, et al. Innovative application of mass spectrometry for the characterization of staphylococcal enterotoxins involved in food poisoning outbreaks[J]. Appl Environ Microbiol, 2009, 75(3): 882-884.
[29] PELES F, WAGNER M, VARGA L, et al. Characterization ofStaphylococcusaureusstrains isolated from bovine milk in Hungary[J]. International Journal of Food Microbiology, 2007, 118(2): 186-193.
[30] SPANU V, SCARANO C, VIRDIS S, et al. Population structure ofStaphylococcusaureusisolated from bulk tank goat's milk[J]. Foodborne Pathogens & Disease, 2013, 10(4): 310-315.
[31] 王娇. 原料乳中嗜冷菌的变化和控制研究[D]. 哈尔滨: 黑龙江东方学院, 2014.
[32] 王伟军, 李延华. 乳源嗜冷菌产胞外耐热酶检测方法的对比分析[J]. 食品研究与开发, 2015, (15): 87-91.
[32] 彭炜. 绿脓杆菌蛋白酶IV的cDNA克隆及表达[D]. 沈阳: 沈阳药科大学, 2008.
[34] 卜宪娜, 孙谧, 郝建华,等. 嗜冷杆菌EastSeaG5-1415脂肪酶基因的克隆和序列测定[J]. 高技术通讯, 2005, 15(2): 94-99.
[35] ERCOLINI D, RUSSO F, FERROCINO I, et al. Molecular identification of mesophilic and psychrotrophic bacteria from raw cow's milk[J]. Food Microbiology, 2009, 26(2): 228-231.
