一种快速鉴定猪肺炎支原体免疫显性蛋白抗原的ELISA方法的建立
2018-02-28周瑶琴游凤钟杰王豪举丁红雷
周瑶琴,游凤,钟杰,王豪举,丁红雷
西南大学 兽医传染病学实验室,重庆 400715
猪支原体肺炎 (Mycoplasmal pneumonia of swine,MPS) 是由猪肺炎支原体 (Mycoplasma hyopneumoniae,Mhp) 引起的一种慢性、接触性呼吸道传染病,广泛分布于世界各地[1]。我国每年由此病造成的直接经济损失超过 100亿元[2]。现阶段我国大部分猪场使用MPS灭活疫苗,还有部分猪场使用弱毒疫苗控制MPS。这些疫苗能够在一定程度减轻临床症状和肺部病理损伤[3-4],提高日增重[4],但其免疫保护效果和人们的期望仍有差距[5-6]。基因工程疫苗是未来 MPS疫苗的一个发展方向。基因工程疫苗保护性抗原的筛选通常采用体外表达纯化病原蛋白后再免疫实验动物或宿主,测定实验动物或宿主的免疫学指标和评价蛋白抗原对实验动物或宿主的免疫保护来实现。这种方法工作量大,花费不菲,且对抗原的选择盲目性很大,抗原筛选的成功率不高[7]。
若能在病原菌基因组编码的所有蛋白信息基础上,高通量大规模鉴定疫苗蛋白抗原,则能够降低因只关注某几个潜在抗原蛋白而造成抗原筛选的遗漏和偏差,增加基因工程疫苗研发成功的概率和提高疫苗免疫保护效果。建立快速鉴定Mhp免疫显性抗原的方法在高通量大规模鉴定MPS疫苗候选蛋白抗原工作中十分必要,这在很大程度上能够减少抗原筛选的工作量,加快工作进度。
Mhp366是一种能够在Mhp自然感染条件下,刺激宿主产生强烈体液免疫的蛋白[8]。本研究以Mhp366蛋白为例,建立了一种基于间接酶联免疫吸附试验 (Indirect enzyme-linked immunosorbent assay,Indirect ELISA) 原理的快速鉴定Mhp血清体液免疫显性蛋白抗原的方法,并用 Mhp156和 Mhp364蛋白验证了该方法的有效性[9]。该方法的建立能够在基因组水平高通量筛选 Mhp血清体液免疫显性蛋白抗原,并为Mhp初乳体液免疫和粘膜免疫显性蛋白抗原鉴定方法的建立奠定基础。
1 材料与方法
1.1 菌株和质粒
大肠杆菌 BL21(DE3)、大肠杆菌 XL-1 Blue感受态和pGEX-6P-1载体由本实验室保存。
mhp366、mhp364和mhp156基因由生工生物工程 (上海) 股份有限公司合成。在合成时将mhp366中第 835–837、967–969、1224–1226 和1312–1314 位,mhp364中第 676–678、979–981、1456–1458 位,mhp156中第 133–135、169–171、193–195、235–237、256–258 位 TGA 密码子修改为TGG。合成的基因连接pGEX-6P-1载体,构建pGEX-6P-1-mhp366、pGEX-6P-1-mhp364、pGEX-6P-1-mhp156重组质粒,转化入大肠杆菌DH5α。
1.2 血清
Mhp阳性猪场血清采集自重庆市和四川省两个猪场具有咳嗽、气喘等典型猪支原体肺炎临床症状的猪,共35份。这两个猪场的猪群在寒冷季节和气候骤变时表现出明显的 MPS临床症状;Mhp阴性血清采集自贵州省某猪场,共30份。该猪场具有良好的管理模式,猪群没有MPS临床症状,在过去两年的血清学监测中,Mhp抗体均为阴性。同时采集对应的猪的支气管肺泡灌洗液。
1.3 主要试剂和菌株
核酸marker购自天根生化科技 (北京) 有限公司。质粒小量提取试剂盒购自 Omega公司。BamHⅠ、XhoⅠ、SalⅠ、蛋白 marker购自宝生物工程 (大连) 有限公司。谷胱甘肽琼脂糖珠购自GE Healthcare公司。谷胱甘肽包被板、HRP标记兔抗猪IgG、胎牛血清 (FBS) 购自Thermo Scientific公司。猪肺炎支原体抗体ELISA检测试剂盒购自美国IDEXX公司。ELISA底物液A、B和终止液购自武汉科前生物股份有限公司。鼠抗GST-Tag单抗购自Bioworld公司。HRP标记羊抗鼠IgG购自Proteintech公司。PVDF膜购自Roche Diagnostics公司。eECL购自康为世纪生物科技有限公司。PreScission Protease由第三军医大学微生物与生化药学实验室惠赠。
1.4 重组蛋白的原核表达与纯化
提取大肠杆菌DH5α中的pGEX-6P-1-mhp366、pGEX-6P-1-mhp364和pGEX-6P-1-mhp156质粒,pGEX-6P-1-mhp366和 pGEX-6P-1-mhp364经BamHⅠ、XhoⅠ双酶切鉴定,pGEX-6P-1-mhp156经BamHⅠ、SalⅠ双酶切鉴定。鉴定正确后,pGEX-6P-1-mhp366、pGEX-6P-1-mhp364转化大肠杆菌BL21(DE3) 感受态细胞,pGEX-6P-1-mhp156转化大肠杆菌XL-1 Blue感受态细胞,筛选阳性重组子,分别再次经BamHⅠ+XhoⅠ、BamHⅠ+XhoⅠ以及BamHⅠ+SalⅠ双酶切鉴定。鉴定正确的重组菌分别命名为GST-Mhp366重组菌、GST-Mhp364重组菌和GST-Mhp156重组菌。
GST-Mhp366重组菌、GST-Mhp364重组菌和GST-Mhp156重组菌分别接种于含100 μg/mL氨苄西林的LB液体培养基,37 ℃摇床培养至OD600为0.7–0.9时,加入IPTG使终浓度为1 mmol/L,GST-Mhp366和GST-Mhp364 30 ℃诱导表达目的蛋白4 h,GST-Mhp156 16 ℃诱导表达目的蛋白20 h。收集菌液,4 ℃、5 000 r/min离心15 min。弃上清后加入细菌裂解液 (PBS中加入1% Triton X-100、1 mmol/L苯甲基磺酰氟、75 IU/mL抑肽酶、20 μmol/L亮肽素、1.6 μmol/L抑肽素,pH 7.2),超声 (220 V、110 W) 破碎细菌30次 (工作 3 s,间歇 4 s)。4 ℃、13 000 r/min 离心 15 min,取上清,与谷胱甘肽琼脂糖珠室温旋转 (25 r/min)孵育1 h,经PBST洗涤2次和PBS洗涤1次后离心。沉淀经SDS-PAGE检测蛋白表达情况。
与谷胱甘肽琼脂糖珠孵育结合的蛋白加入适量PBS和PreScission Protease,室温酶切5 h,4 ℃、10 000 r/min离心1 min,取上清;在沉淀中再次加入适量 PBS,振荡后再次离心取上清,如此重复2次。将上清液和剩余的沉淀经SDS-PAGE检测蛋白纯化情况。
pGEX-6P-1载体转化大肠杆菌BL21(DE3)感受态细胞,提取质粒鉴定,该基因工程菌命名为GST工程菌,作为阴性对照。其诱导表达温度和IPTG浓度同GST-Mhp366重组菌。
1.5 重组蛋白的鉴定
将纯化的 GST-Mhp366、GST-Mhp364、GST-Mhp156蛋白进行 Western blotting鉴定。SDS-PAGE后转PVDF膜,5%脱脂奶粉的TBST封闭 2 h,TBST洗涤 3次,每次 5 min;鼠抗GST-Tag单抗 (1∶8 000稀释) 室温孵育 1 h,TBST洗涤3次,每次5 min;HRP标记羊抗鼠IgG(1∶20 000稀释) 室温孵育1 h,TBST洗涤3次,每次5 min,eECL显色。
1.6 Mhp阳性血清和阴性血清的准备
Mhp阳性猪场血清和阴性猪场血清经Mhp抗体检测试剂盒检测,支气管肺泡灌洗液经套式PCR检测[10]。在Mhp阳性猪场中随机选取17份Mhp抗体阳性和核酸阳性猪血清,在Mhp阴性猪场中随机选取13份Mhp抗体阴性和核酸阴性猪血清。
GST工程菌经IPTG诱导表达后制备细菌裂解液,按GST细菌裂解液∶血清=1∶50的比例加入Mhp阳性血清/阴性血清,4 ℃过夜旋转 (25 r/min)孵育,对血清进行预吸附。
1.7 最佳抗原包被浓度的选择
GST-Mhp366重组菌经诱导表达后离心,加入细菌裂解液超声破碎,25 mL菌液对应1 mL细菌裂解液,4 ℃、13 000 r/min离心15 min去除细胞碎片。细菌裂解液按原液、1∶5、1∶10比例稀释后,谷胱甘肽包被板每孔加入200 μL,4 ℃包被过夜,PBST洗涤5次,每次 3 min。加入 200 μL含0.25%脱脂奶粉和10% FBS的PBS作为封闭液室温封闭1 h,PBST洗涤5次,每次3 min。将猪血清用封闭液按1∶500比例稀释后,每孔加入100 μL,室温孵育2 h,PBST洗涤5次,每次3 min。HRP标记兔抗猪IgG用封闭液1∶40 000比例稀释后,每孔加入100 μL,37 ℃孵育1 h,PBST洗涤5 次,每次3 min。每孔加入底物液A和底物液B各50 μL,混匀,室温避光显色10 min后加入50 μL终止液,10 min内酶标仪测定OD630值。
GST工程菌诱导表达后按GST-Mhp366重组菌条件进行ELISA试验,测定相应孔的OD630值。
1.8 最佳封闭液的确定
按已确定的GST-Mhp366重组菌和GST工程菌的最佳细菌裂解液浓度加入谷胱甘肽包被板,分别以PBS+10% FBS、PBS+2.5%脱脂奶粉、PBS+10%FBS+2.5%脱脂奶粉封闭,每孔加入200 μL,室温封闭1 h,进行ELISA试验,测量相应孔的OD630值。
1.9 最佳血清稀释度和最佳酶标二抗工作浓度的确定
按已确定的GST-Mhp366重组菌和GST工程菌的最佳细菌裂解液浓度加入谷胱甘肽包被板,使用最佳封闭液进行封闭。血清按 1∶100、1∶200、1∶500、1∶1000稀释,HRP标记兔抗猪IgG酶标二抗按照1∶20 000、1∶40 000、1∶80 000稀释。测量相应孔的OD630值。
1.10 间接ELISA反应条件的判定标准
根据不同的反应条件进行间接 ELISA。GST-Mhp366细菌裂解液和GST细菌裂解液分别与13份 Mhp阴性血清反应,计算 GST-Mhp366细菌裂解液和GST细菌裂解液与每份阴性血清反应的OD630值之差,该差值的平均值为x,标准差为s,将+2s所得的值定为用Mhp366蛋白判定阴阳血清的界限。
GST-Mhp366细菌裂解液和GST细菌裂解液分别与 17份 Mhp阳性血清反应。同理,计算GST-Mhp366细菌裂解液和GST细菌裂解液与每份阳性血清反应的OD630值之差,该差值命名为A。若A≥+2s,则判定为Mhp366抗体阳性。选择A值≥+2s对应的血清数量最多的反应条件,确定为 Mhp血清体液免疫显性蛋白抗原ELISA的最佳反应条件。
1.11 间接ELISA方法的验证
GST-Mhp364重组菌和 GST-Mhp156重组菌经诱导表达后离心收集菌体,加入细菌裂解液超声破碎,25 mL菌液对应1 mL细菌裂解液,离心去除细胞碎片。用GST-Mhp366蛋白建立的间接ELISA反应条件[9]检测 Mhp364抗体阳性血清数量和Mhp156抗体阳性血清数量。若某蛋白抗体阳性血清数量超过Mhp阳性血清数量的一半,则将该蛋白判定为血清体液免疫显性蛋白抗原。
2 结果与分析
2.1 GST-Mhp366、GST-Mhp364、GST-Mhp156和GST蛋白的原核表达、纯化与鉴定
提取 GST-Mhp366、GST-Mhp364和 GSTMhp156重组菌中的重组质粒,分别经双酶切后,得到4 984 bp的载体片段和3个载体中的1 668 bp、1 599 bp、1 062 bp 的mhp366、mhp364、mhp156基因片段 (图1)。所有片段和预期条带大小相符,说明 pGEX-6P-1-mhp366、pGEX-6P-1-mhp364、pGEX-6P-1-mhp156重组质粒构建正确,并在大肠杆菌BL21(DE3)/XL-1 Blue中成功转化。GST重组菌中也能提取出大小约5 000 bp的质粒,说明pGEX-6P-1质粒转化大肠杆菌BL21(DE3)成功。
GST-Mhp366、GST-Mhp364、GST-Mhp156重组菌经1 mmol/L IPTG诱导后超声破菌,以13 000 r/min离心取上清检测蛋白表达情况并进行后续试验。由于包涵体蛋白12 000 ×g离心后沉淀[11],因此,3个重组蛋白均能够以可溶形式表达。
经谷胱甘肽琼脂糖珠吸附的GST-Mhp366破菌上清SDS-PAGE后可见在90 kDa和26 kDa处各有一条明显的条带 (图2,第1泳道)。90 kDa处为与预期蛋白大小相同的GST-Mhp366融合表达蛋白,26 kDa处为 GST蛋白。融合蛋白经PreScission Protease酶切后,得到约 70 kDa的Mhp366 (图 2,第2、3、4、5泳道),比预期蛋白大小 (64 kDa) 偏大。经酶切之后的谷胱甘肽琼脂糖珠-融合蛋白复合物中仍有未被酶切的融合蛋白、被酶切的Mhp366蛋白、GST蛋白和46 kDa的PreScission Protease (图2,第6泳道)。
经吸附的GST-Mhp364破菌上清电泳后可见与预期蛋白大小相同的87 kDa GST-Mhp364融合蛋白和26 kDa GST蛋白 (图2,第7泳道),经PreScission Protease酶切后,得到61 kDa与预期蛋白大小相同的Mhp364 (图2,第8、9、10、11泳道)。经酶切之后的谷胱甘肽琼脂糖珠-融合蛋白复合物中仍有未被酶切的融合蛋白、被酶切的Mhp364蛋白、GST蛋白和46 kDa的PreScission Protease (图 2,第 12泳道)。
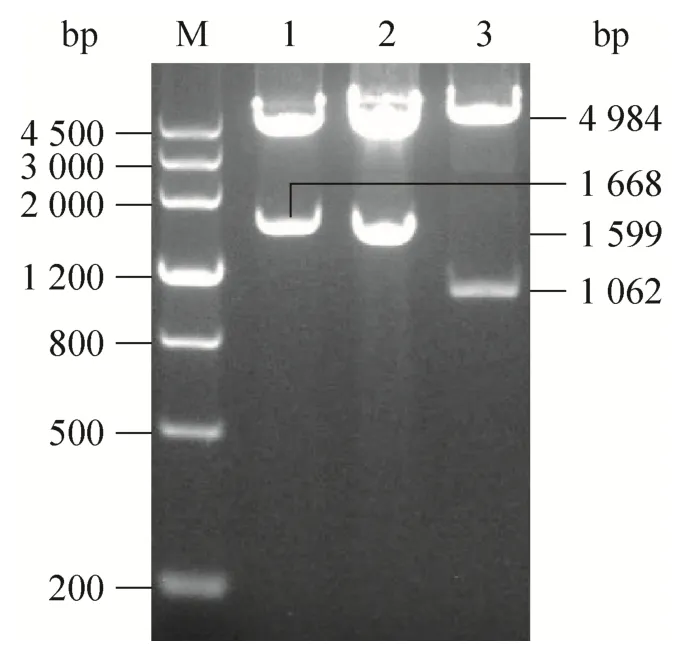
图 1 pGEX-6P-1-mhp366、pGEX-6P-1-mhp364、pGEX-6P-1-mhp156重组质粒的双酶切鉴定Fig. 1 Identification of recombinant plasmids pGEX-6P-1-mhp366, pGEX-6P-1-mhp364 and pGEX-6P-1-mhp156 by double enzyme digestion. M: DNA marker Ⅲ; Lane 1:pGEX-6P-1-mhp366 was digested with BamHⅠ and XhoⅠ. Lane 2: pGEX-6P-1-mhp364 was digested with BamHⅠ and XhoⅠ. Lane 3: pGEX-6P-1-mhp156 was digested with BamHⅠ and SalⅠ.

图2 GST-Mhp366、GST-Mhp364、GST-Mhp156重组蛋白表达纯化及蛋白酶切分析的电泳图Fig. 2 Recombinant proteins GST-Mhp366, GST-Mhp364 and GST-Mhp156 were purified by Glutathione SepharoseTM 4B and digested by PreScission Protease. M: protein molecular weight marker; 1: GST-Mhp366 fusion protein; 2: the Mhp366 supernatant was harvested after the cleavage; 3–5: the Mhp366 beads were sequentially washed three times with PBS; 6: the remaining Mhp366 bead sample after digestion and washing; 7: GST-Mhp364 fusion protein; 8: the Mhp364 supernatant was harvested after the cleavage; 9–11: the Mhp364 beads were sequentially washed three times with PBS; 12: the remaining Mhp364 bead sample after digestion and washing; 13: GST-Mhp156 fusion protein; 14: the Mhp156 supernatant was harvested after the cleavage; 15–17: the Mhp156 beads were sequentially washed three times with PBS; 18: the remaining Mhp156 bead sample after digestion and washing.
经吸附的GST-Mhp156破菌上清电泳后可见与预期蛋白大小相同的62 kDa GST-Mhp156融合蛋白和26 kDa GST蛋白 (图2,第13泳道),经PreScission Protease酶切后,得到 36 kDa的Mhp156 (图 2,第 14、15、16、17 泳道),与预期蛋白大小相同。该蛋白极易降解。经酶切之后的谷胱甘肽琼脂糖珠-融合蛋白复合物中仍有未被酶切的融合蛋白、被酶切的Mhp156蛋白、GST蛋白和46 kDa的PreScission Protease (图2,第18泳道)。
图2泳道1–18中的杂带为非特异结合在谷胱甘肽琼脂糖珠的杂蛋白。
用抗GST标签单抗进行Western blotting,结果表明GST-Mhp366、GST-Mhp364、GST-Mhp156融合蛋白大小分别为90、87、62 kDa (图3),均与预期大小相符,进一步表明这3个重组蛋白均成功表达。
2.2 间接ELISA反应条件的确定
2.2.1 最佳抗原包被浓度的选择
GST-Mhp366和GST细菌裂解液原液按1∶5、1∶10比例稀释后进行抗原包被,原液和按1∶5稀释时 A≥+2s对应的 Mhp366抗体阳性血清数最多 (图4)。考虑到在今后Mhp血清免疫显性蛋白抗原的筛选试验中,部分重组蛋白抗原的表达量可能低于GST-Mhp366重组菌中重组蛋白的表达量,因此将细菌裂解液原液作为最佳抗原包被浓度。
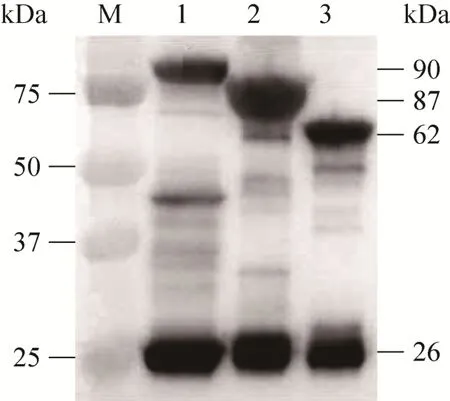
图3 GST-Mhp366、GST-Mhp364、GST-Mhp156重组蛋白进行Western blotting分析Fig. 3 Western blotting analysis of recombinant proteins GST-Mhp366, GST-Mhp364 and GST-Mhp156. M:precision Plus Protein™ Dual Color Standards; 1: GSTMhp366 fusion protein; 2: GST-Mhp364 fusion protein;3: GST-Mhp156 fusion protein.
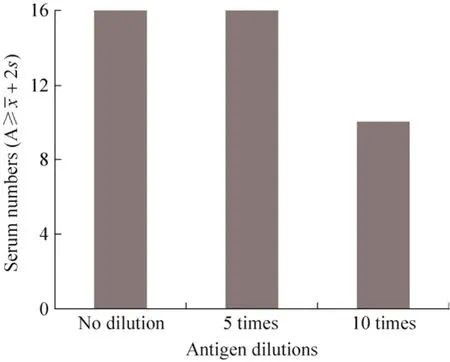
图4 抗原最佳包被浓度的选择Fig. 4 Optimization for the concentration of coating antigen with indirect ELISA.
2.2.2 最佳封闭液的确定
以最佳抗原包被浓度包被酶标板,选用PBS+10% FBS、PBS+2.5%脱脂奶粉、PBS+10%FBS+2.5%脱脂奶粉3种封闭液比较封闭效果,结果显示PBS+10% FBS+2.5%脱脂奶粉作为封闭液进行ELISA,A值≥+2s对应的Mhp366抗体阳性血清数最多 (图5)。因此选择PBS+10% FBS+2.5%脱脂奶粉作为封闭液。
2.2.3 最佳血清稀释度和最佳酶标二抗工作浓度的确定
血清1∶500稀释,酶标二抗1∶40 000、1∶80 000稀释时均有16个Mhp366抗体阳性血清,因此选择血清1∶500稀释为最佳血清稀释度;考虑到酶标二抗稀释度越高试验误差越大,因此将酶标二抗按照1∶40 000稀释作为最佳工作浓度 (图6)。
2.2.4 间接ELISA方法的验证
采用本试验建立的方法,GST-Mhp364和GSTMhp156重组菌裂解液分别与17份Mhp抗体阳性血清和13份抗体阴性血清反应后,Mhp364抗体阳性血清有9份,Mhp156抗体阳性血清有12份,二者的抗体阳性血清数量均超过所用Mhp抗体阳性血清数量的一半。因此,Mhp364和Mhp156蛋白均为血清体液免疫显性蛋白抗原,这与Simionatto等报道的结果一致 (Mhp364和Mhp156在7 448株中编号分别为MHP0353和MHP0223)[9]。
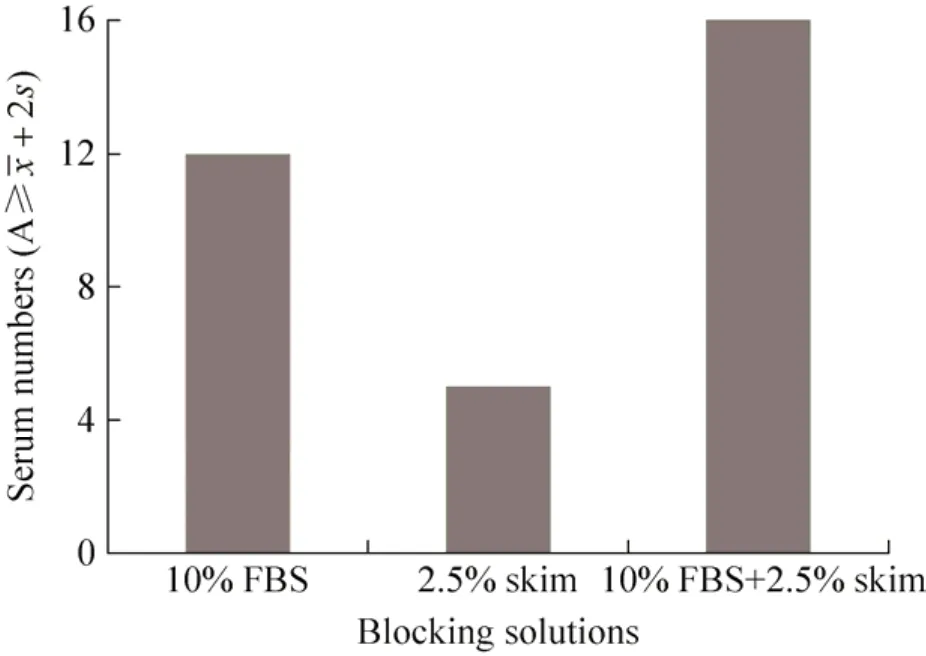
图5 最佳封闭液的确定Fig. 5 Optimization of blocking buffer.
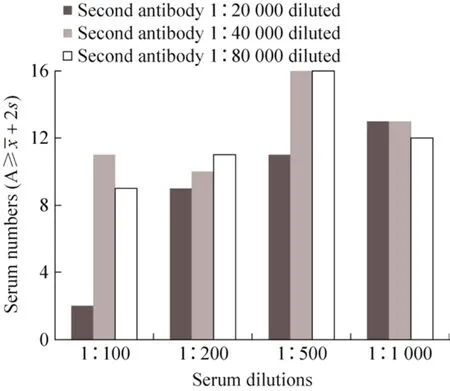
图6 最佳血清稀释度和最佳酶标二抗工作浓度的确定Fig. 6 Determination of optimal dilution of serum and HRP-conjugated IgG.
3 讨论
本研究中的 Mhp366蛋白在自然感染菌株中能够刺激机体产生强烈的体液免疫反应;体外无细胞培养时,表达量很低,几乎不能刺激机体产生体液免疫反应,也几乎不与Mhp高免血清发生反应[8]。因此,本研究以 Mhp366蛋白为基础,建立筛选猪肺炎支原体血清体液免疫显性蛋白抗原方法。
在真核生物和大部分原核生物中色氨酸由TGG编码,在支原体中色氨酸由TGA编码[12]。TGA在真核生物和大部分原核生物中编码终止密码子。在mhp366、mhp364、mhp156中,分别有4个、3个、5个编码色氨酸的TGA密码子。因此,若在大肠杆菌中表达 Mhp366、Mhp364、Mhp156蛋白,需要将TGA突变为TGG。考虑到PCR扩增mhp366、mhp364和mhp156后再将TGA密码子突变为TGG,工作量很大,因此,本研究直接合成了3个基因,并将TGA密码子直接修改为TGG。
本试验采用 pGEX-6P-1载体连接mhp366、mhp364、mhp156基因分别构建重组质粒。因为pGEX-6P-1载体含有GST基因,重组菌表达的GST蛋白具有极强的亲水性,和 Mhp366、Mhp364、Mhp156分别形成融合蛋白后能最大限度地保证融合蛋白以具有天然构象的可溶形式表达[12]。另外GST蛋白能和商品化谷胱甘肽包被板上的谷胱甘肽形成特异性硫键共价结合,间接地包被了目的蛋白,实现了目的蛋白的快速包被。不同重组菌目的蛋白的表达量存在差别,为了能够饱和包被板上的谷胱甘肽[13],采用重组菌裂解液原液来包被谷胱甘肽板。
我们采用重组菌裂解液包被ELISA的结果减去GST工程菌裂解液包被ELISA的结果,减少重组菌中由于非特异结合形成的背景值。另外在ELISA之前,我们还用GST工程菌裂解液吸附了血清中可能存在的GST抗体和一些其他病原感染猪形成的抗体,特别是环境中大量存在的大肠杆菌蛋白抗体,最大限度降低了血清的背景值,其结果优于不使用GST工程菌裂解液吸附。
传统的蛋白抗原筛选通常是通过纯化蛋白后免疫实验动物评价抗原的免疫应答和对实验动物的免疫保护作用[14]。许多Mhp外膜蛋白也作为疫苗候选抗原用于免疫小鼠,并对相应的免疫学指标进行了检测[15-17]。但抗原对小鼠的免疫应答不能代表抗原对猪的免疫应答,更重要的是这种免疫应答不能代表对猪的免疫保护。反向疫苗学技术为疫苗抗原的筛选提供了全新的思路,可以基于微生物学基因组编码的所有蛋白,通过与感染病原的本动物血清反应,筛选具有高免疫反应性的普遍存在于病原的保守蛋白抗原[9,18],但这些方法比较费时费力。已有实验室采用反向疫苗学方法,利用谷胱甘肽包被板与携带GST标签的沙眼衣原体重组蛋白结合,筛选免疫显性蛋白抗原,建立了沙眼衣原体免疫显性抗原组[19-21],该实验室还利用此方法筛选了肺炎衣原体[22]和鼠衣原体[23-24]免疫显性蛋白抗原。那么,这种方法能否用于Mhp免疫显性蛋白抗原的检测?本试验在可溶表达GST-Mhp366重组蛋白基础上,将重组菌细胞裂解液与谷胱甘肽包被板结合,直接与本动物血清反应,发现Mhp366蛋白几乎能与所有Mhp阳性血清反应 (16/17),证明了Meens等的结果[8],并以此建立了一种快速鉴定Mhp血清体液免疫显性蛋白抗原的方法。
Mhp364和Mhp156是两个能够在体外和体内表达、高度保守[25]且在自然感染条件下刺激猪产生体液免疫反应的Mhp蛋白[9]。本研究进一步用Mhp364和Mhp156验证了所建立筛选方法的有效性,这为Mhp基因工程疫苗蛋白抗原的筛选提供了有效的研究方法。今后将对Mhp蛋白进行大量表达,并用此方法大规模筛选Mhp血清体液免疫显性蛋白抗原。
[1]Maes D, Segales J, Meyns T, et al. Control ofMycoplasma hyopneumoniaeinfections in pigs. Vet Microbiol, 2008, 126(4): 297–309.
[2]Chen C, Hua LZ, Gan Y, et al. Research progress of MPS eradication programme. China Animal Health,2014, 16(11): 16–19 (in Chinese).陈财, 华利忠, 甘源, 等. 猪支原体肺炎净化研究进展. 中国动物保健, 2014, 16(11): 16–19.
[3]Sibila M, Bernal R, Torrents D, et al. Effect of sow vaccination againstMycoplasma hyopneumoniaeon sow and piglet colonization and seroconversion, and pig lung lesions at slaughter. Vet Microbiol, 2008,127(1/2): 165–170.
[4]Meyns T, Dewulf J, de Kruif A, et al. Comparison of transmission ofMycoplasma hyopneumoniaein vaccinated and non-vaccinated populations. Vaccine,2006, 24(49/50): 7081–7086.
[5]Villarreal I, Meyns T, Dewulf J, et al. The effect of vaccination on the transmission ofMycoplasma hyopneumoniaein pigs under field conditions. Vet J,2011, 188(1): 48–52.
[6]Villarreal I, Maes D, Vranckx K, et al. Effect of vaccination of pigs against experimental infection with high and low virulenceMycoplasma hyopneumoniaestrains. Vaccine, 2011, 29(9): 1731–1735.
[7]Singh K, Mehta S. The clinical development process for a novel preventive vaccine: an overview. J Postgrad Med, 2016, 62(1): 4–11.
[8]Meens J, Bolotin V, Frank R, et al. Characterization of a highly immunogenicMycoplasma hyopneumoniaelipoprotein Mhp366 identified by peptide-spot array.Vet Microbiol, 2010, 142(3/4): 293–302.
[9]Simionatto S, Marchioro SB, Galli V, et al.Immunological characterization ofMycoplasma hyopneumoniaerecombinant proteins. Comp Immunol Microbiol Infect Dis, 2012, 35(2): 209–216.
[10]Lu XM, Feng ZX, Liu MJ, et al. Establishment of a nested PCR assay for detection ofMycoplasmahyopneumoniae. Jiangsu J Agric Sci, 2010, 26(1):91–95 (in Chinese).逯晓敏, 冯志新, 刘茂军, 等. 猪肺炎支原体套式PCR检测方法的建立及应用. 江苏农业学报, 2010,26(1): 91–95.
[11]Ding HL, Pan J, Zhang WJ, et al.Helicobacter pyloricagT protein: expression, purification and immunogenicity. J Southwest Univ: Nat Sci Ed,2010, 32(4): 46–50 (in Chinese).丁红雷, 潘竞, 张卫军, 等. 幽门螺杆菌cagT蛋白的表达、纯化及免疫原性研究. 西南大学学报: 自然科学版, 2010, 32(4): 46–50.
[12]Yamao F, Muto A, Kawauchi Y, et al. UGA is read as tryptophan inMycoplasma capricolum. Proc Natl Acad Sci USA, 1985, 82(8): 2306–2309.
[13]Sharma J, Zhong Y, Dong F, et al. Profiling of human antibody responses toChlamydia trachomatisurogenital tract infection using microplates arrayed with 156 chlamydial fusion proteins. Infect Immun,2006, 74(3): 1490–1499.
[14]Sharon J, Rynkiewicz MJ, Lu Z, et al. Discovery of protective B-cell epitopes for development of antimicrobial vaccines and antibody therapeutics.Immunology, 2014, 142(1): 1–23.
[15]Conceição FR, Moreira AN, Dellagostin OA. A recombinant chimera composed of R1 repeat region ofMycoplasma hyopneumoniaeP97 adhesin withEscherichia coliheat-labile enterotoxin B subunit elicits immune response in mice. Vaccine, 2006,24(29/30): 5734–5743.
[16]Chen YL, Wang SN, Yang WJ, et al. Expression and immunogenicity ofMycoplasma hyopneumoniaeheat shock protein antigen P42 by DNA vaccination.Infect Immun, 2003, 71(3): 1155–1160.
[17]Galli V, Simionatto S, Marchioroa SB, et al.Immunisation of mice withMycoplasma hyopneumoniaeantigens P37, P42, P46 and P95 delivered as recombinant subunit or DNA vaccines.Vaccine, 2012, 31(1): 135–140.
[18]Marchioro SB, Simionatto S, Galli V, et al. Production and characterization of recombinant transmembrane proteins fromMycoplasma hyopneumoniae. Vet Microbiol, 2012, 155(1): 44–52.
[19]Li Z, Chen C, Chen D, et al. Characterization of fifty putative inclusion membrane proteins encoded in theChlamydia trachomatisgenome. Infect Immun, 2008,76(6): 2746–2757.
[20]Wang J, Zhang Y, Lu C, et al. A genome-wide profiling of the humoral immune response toChlamydia trachomatisinfection reveals vaccine candidate antigens expressed in humans. J Immunol,2010, 185(3):1670–1680.
[21]Wang J, Chen L, Chen F, et al. A chlamydial type III-secreted effector protein (Tarp) is predominantly recognized by antibodies from humans infected withChlamydia trachomatisand induces protective immunity against upper genital tract pathologies in mice. Vaccine, 2009, 27(22): 2967–2980.
[22]Luo J, Liu G, Zhong Y, et al. Characterization of hypothetical proteins Cpn0146, 0147, 0284 & 0285 that are predicted to be in theChlamydia pneumoniaeinclusion membrane. BMC Microbiol, 2007, 7: 38.
[23]Zeng H, Gong S, Hou S, et al. Identification of antigen-specific antibody responses associated with upper genital tract pathology in mice infected withChlamydia muridarum. Infect Immun, 2012, 80(3):1098–1106.
[24]Zeng H, Hou S, Gong S, et al. Mapping immunodominant antigens and H-2-linked antibody responses in mice urogenitally infected withChlamydia muridarum. Microbes Infect, 2012,14(7-8): 659–665.
[25]Pendarvis K, Padula MP, Tacchi JL, et al.Proteogenomic mapping ofMycoplasma hyopneumoniaevirulent strain 232. BMC Genomics, 2014, 15: 576.
