鲟鱼肠道微生物多样性的研究
2018-02-13李小义张效平王艳艳
李小义, 张效平, 赵 凤, 孔 杰, 赵 飞, 周 洲, 王艳艳
(贵州省农业科学院水产研究所,贵州贵阳 550025)
肠道微生物是人体及动物肠道的正常组成部分,对宿主的营养和健康有重要影响。研究表明,肠道微生物可参与宿主的营养物质消化吸收、能量代谢、过敏、自身免疫疾病等多方面调节过程[1-3],对宿主的年龄亦有指示作用[4]。近年来,肠道微生物与水产动物的联系受到了越来越多的关注。Kan等将金鱼暴露在0~100 μg/L的五氯苯酚中,对金鱼肠道微生物变化进行了分析,结果表明,拟杆菌属丰度与五氯苯酚浓度呈正相关,拟杆菌门、拟杆菌属及厚壁菌门/拟杆菌门之比对金鱼在五氯苯酚环境中的体质量和肝质量均有重要影响[5]。Rolig等对斑马鱼肠神经系统进行缺失突变处理,结果发现肠道发炎个体中促炎性细菌菌群较多,而在同样做缺失突变的肠道正常个体中具有抗炎作用的细菌菌群较多,研究表明肠道微生物对斑马鱼肠道健康具有重要作用[6]。此外,动物体肠道始终处于动态变化,摄食方式和肠道微生物种类、数量及占例均有影响,动物体内肠道菌群结构合理有助于加快宿主的生长和改善肉质[7]。唐杨等在凡纳滨对虾饲料中添加蜡样芽孢杆菌,试验组凡纳滨对虾生长速度比空白组平均快15.2%,且试验组中菌群丰度变化明显,相对不添加蜡样芽孢杆菌的对照组,试验组中变形菌门丰度有所下降,而拟杆菌门丰度有所增加[8]。
鲟鱼为高蛋白、多脂肪性鱼类,是世界上优良的淡水鱼品种,也是现存于世界上最珍奇古老的冷水性生物鱼群之一,有“活化石”之称,其肉厚骨软,同时鲟鱼卵可以加工成鲟鱼子酱,具有“黑色黄金”之称。鱼体含有人体必需的多种氨基酸,具有很高的经济价值和药用价值。本研究对10月龄鲟鱼苗肠道微生物多样性进行了初步分析,获得对鲟鱼健康有利的微生物菌群信息,对益生菌在鲟鱼类饲料添加剂中的应用及减少抗生素使用具有重要意义。
1 材料与方法
1.1 材料
2016年5月,从贵州省水产研究所惠水试验基地采集10月龄鲟鱼苗数条、养殖水体3 L(标记为S)及饲料20 g(标记为SL)。迅速带回实验室,不投饵暂养3 d。
1.2 试剂与仪器
DNA Marker、细菌DNA提取纯化等试剂,购自上海生工生物工程股份有限;Gel-Doc2000凝胶成像分析仪,购自 BIO-RAD公司;电泳仪,购自北京市六一仪器厂。
1.3 鲟鱼肠道采集
待鲟鱼肠粪便排空后,在超净台中对鲟鱼进行解剖取样。用75%乙醇对鲟鱼体表消毒后用解剖剪沿肛门朝前对鲟鱼进行解剖。清理出肠道,75%乙醇擦拭消化道外壁,无菌PBS缓冲液冲洗,5 mL灭菌离心管分别收集前、中、后肠并作标记,Q4、Q5为前肠样品,Z4、Z5为中肠样品,H4、H5为后肠样品。
1.4 肠道微生物DNA提取
对解剖取得的鲟鱼肠道样品,使用天根试剂盒提取DNA,具体方法见试剂盒说明书。用1.5%琼脂糖凝胶对提取获得的DNA样品质量进行检测,并将合格样品干冰保存寄送北京诺禾致源生物信息科技有限公司进行测序分析。
1.5 16S rDNA文库构建及多样性分析
利用16S V4区引物(515F:5′-GTGCCAGCMGCCGCGG TAA-3′和806R:5′-GGACTACHVGGGTWTCTAAT-3′)对肠道微生物DNA进行扩增。采用Illumina Hiseq平台对扩增获得的测序结果进行分析。截去Barcode和引物序列后使用FLASH对每个样品的reads进行拼接,后去除嵌合体序列,得到最终有效数据。利用Uparse软件(Uparse v7.0.1001)对所有样品全部有效数据进行聚类,默认以97%的一致性将序列聚类成为OTU。对OTUs代表序列进行物种注释,用Mothur方法与SILVA的SSUrRNA数据库进行物种注释分析(设定阈值为0.8~1.0),获得分类学信息并分别在各分类水平:kingdom(界)、phylum(门)、class(纲)、order(目)、family(科)、genus(属)、species(种)统计各样本的群落组成。使用PyNAST软件(Version 1.2)与GreenGene数据库中的“Core Set”数据信息进行快速多序列比对,得到所有OTUs代表序列的系统关系。最后以样品数据量最少的为标准对各样品数据进行均一化处理。使用Qiime软件(Version 1.7.0)计算Observed-species、Shannon指数、Goods-coverage、Unifrac距离及构建UPGMA样品聚类树,使用R软件(Version 2.15.3)绘制稀释曲线,Rank abundance曲线,物种累积曲线,进行Alpha多样性指数组间差异分析并绘制PCA、PCoA、NMDS图。
2 结果
2.1 细菌16S rRNA基因测序结果分析
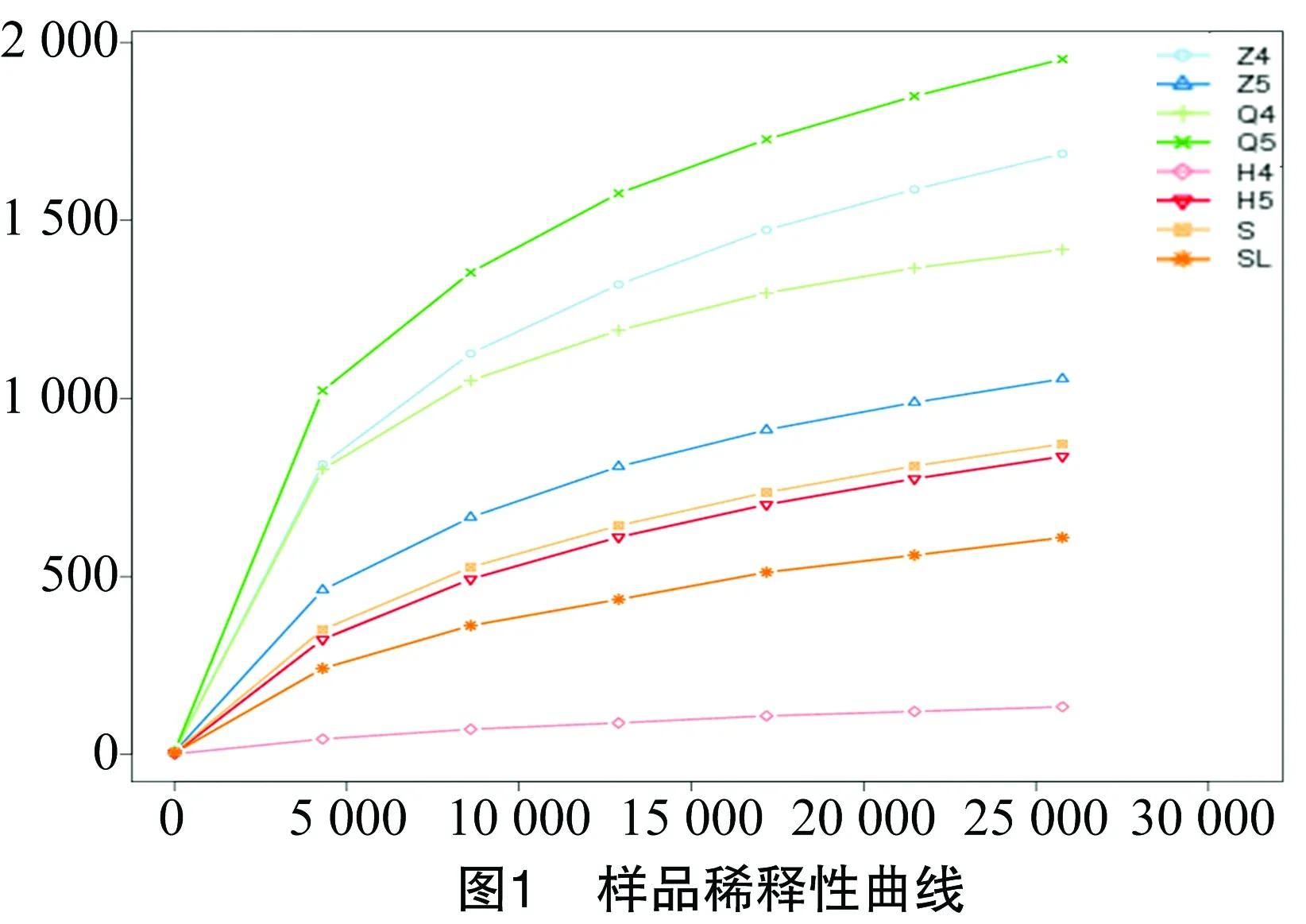
以序列相似性97%一致性(identity)的原则将序列聚类成为1个OTU(operational taxonomic unit)。鲟鱼肠道微生物样品共获得8 239个OUT,其中Q4、Q5样品分别含有 1 432、1 771个OUTs,Z4、Z5样品分别含有1 702、1 064个OUTs,H4、H5样品分别包含165、847个OUTs(表1)。从养殖水体S样品和饲料SL样品中分别获得861、602个OUTs。覆盖率(good’s coverage)是测序获得的序列占整个基因组的比例,用以评估抽样完整性。本研究中所有样品的覆盖率为97.7%~99.7%,表明本次检测结果具有较好的覆盖性。通过单样本的多样性分析(Alpha多样性)可以反映样品内的微生物群落的丰富度和多样性,Shannon是用来估算样品中微生物多样性指数之一,其值越大表明群落多样性越高,本研究中鲟鱼肠道前肠样品Shannon指数最高,表明鲟鱼前肠细菌多样性丰富,后肠样品Shannon指数最低,为0.365。结果表明,养殖水体中细菌多样性较饲料高,而鲟鱼肠道细菌微生物多样性表现为:前肠>中肠>后肠(表1)。稀释曲线是从样品中随机抽取一定测序量的数据,统计它们所代表物种数目(即OTUs数目),以抽取的测序数据量与对应的物种数来构建曲线。稀释曲线可直接反映测序数据量的合理性,并间接反映样品中物种的丰富程度,本研究各样品稀释曲线较为平缓(图1),说明测序数据量逐渐趋于合理。

表1 基于16S rRNA基因序列的细菌多样性指数
2.2 细菌多样性分析
根据物种注释结果在门水平对物种相对丰度进行统计分析,由图2可知,其中相对丰度排前10的物种包括变形菌门(Proteobacteria)、蓝细菌门(Cyanobacteria)、梭杆菌门(Fusobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、绿弯菌门(Chloroflexi)、酸杆菌门(Acidobacteria)、芽单胞菌门(Gemmatimonadetes)及硝化螺旋菌门(Nitrospirae)。前肠样品组G6中变形菌门相对丰度最高,为 75.0%,其次为拟杆菌门(12.9%)、厚壁菌门(5.2%)、放线菌门(2.3%)、酸杆菌门(1.3%)。中肠样品组G7中亦包含较多的变形菌门(57.4%),其次为拟杆菌门(18.6%)、厚壁菌门(10.2%)、放线菌门(4.3%)、酸杆菌门(3.5%)。后肠样品组G8中,除了变形菌门(66.0%)丰度较高以外,梭杆菌门(30.7%)明显高于前肠组(0.71%)和中肠组(0.77%)。水体样品G4(S)中,丰度较高的物种包括变形菌门(50.8%)、放线菌门(26.2%)、梭杆菌门(11.7%)、拟杆菌门(7.7%)、厚壁菌门(1.8%)。饲料样品G5(SL)中丰度较高的物种包括蓝细菌门(59.5%)、变形菌门(29.8%)、厚壁菌门(8.1%)、拟杆菌门(1.1%)。前中后肠、水样及饲料样品中变形菌门均为主要优势物种。前中肠细菌在门水平物种类型及丰度差异不大,后肠细菌和前中肠内细菌差异明显。
在菌属水平上对每个样品的菌群结构及分布进行统计分析,由图3可知,5个样品组丰度前10的菌属包括:假单胞菌属(Pseudomonas)、气单胞菌属(Aeromonas)、不动杆菌属(Acinetobacter)、黄杆菌属(Flavobacterium)、unidentified Marinilabiaceae、Sedimenticola、地杆菌属(Geobacter)、Polynucleobacter、unidentified Chloroplast及unidentified Mitochondria。前肠样品组G6中,假单胞菌属丰度最高,为23.18%,其次为气单胞菌属(7.23%)、不动杆菌属(3.73%)、黄杆菌属(2.47%)、unidentified Marinilabiaceae(2.43%)、Sedimenticola(2.20%)。中肠样品组G7中,丰度排名前6的菌属分别为unidentified Marinilabiaceae(4.22%)、Sedimenticola(3.13%)、地杆菌属(2.60%)、假单胞菌属(2.37%)、不动杆菌属(1.74%)、气单胞菌属(1.40%)。后肠样品组G8中,主要包括气单胞菌属(0.52%)、假单胞菌属(0.47%)、不动杆菌属(0.13%)、Sedimenticola(0.07%)、unidentified Marinilabiaceae(0.06%)、unidentified Chloroplast(0.03%)。养殖水体样品组G4中,Polynucleobacter丰度最高为4.06%,其次为黄杆菌属(1.27%)、unidentified Chloroplast(0.70%)、假单胞菌属(0.51%)、unidentified Marinilabiaceae(0.43%)、气单胞菌属(0.29%)。饲料样品组G5中,unidentified Chloroplast丰度最高(59.50%),其次为unidentified Mitochondria(21.33%)、假单胞菌属(1.35%)、气单胞菌属(0.77%)、unidentified Marinilabiaceae(0.27%)、不动杆菌属(0.21%)。不同来源样品组中细菌丰度各不相同,后肠样品组G8细菌多样性与其他组相差较大,不含unidentified Mitochondria、Polynucleobacter及Geobacter等3个属的细菌。


2.3 核心微生物菌落分析
绘制成韦恩图对不同样品(组)之间共有、特有的OUT进行分析,由图4可知,其中前肠2个克隆共有OUTs 980个,前肠2个克隆样本及养殖水体和饲料样品共有OUTs 307个,优势菌属包括Marinifilum(2.61%)、假单胞菌属(2.28%)、黄杆菌属(1.63%)。中肠2个克隆样本共有OUTs 852个,中肠2个克隆样本及养殖水体和饲料样品共有OUTs315个,主要包括Marinifilum(2.54%)、假单胞菌属(2.22%)、黄杆菌属(1.59%)、芽孢杆菌属(1.27%)、unidentified_Marinilabiaceae(1.27%)及Bacteroides(1.27%)等菌属。后肠2个克隆样本共有OUTs 79个,后肠样品及养殖水体和饲料共有OUTs 26个,在门水平上主要为变形菌门(73.08%)和厚壁菌门(15.38%)。

3 讨论
近年来,研究者对人体[9-10]、斑马鱼[11]、虹鳟鱼[12]、果蝇[13]及蝴蝶[14]等脊椎和非脊椎动物肠道微生物多样性均进行了研究。Ni等对24条草鱼肠道微生物多样性进行了研究,共发现1 228种细菌(116种古细菌和1 112种细菌),其中大部分属于厚壁菌门、变形杆菌门及梭杆菌门,此外笔者还发现,以黑麦草为饲料喂养的草鱼肠道微生物在草鱼肠道碳代谢、氨基酸代谢及脂肪酸代谢过程中作用显著[15]。Tzeng等研究发现,变形杆菌门是日本沼虾肠道微生物丰度最高的细菌门类,其次为厚壁菌门和放线菌门[16]。李建柱等研究发现鱼菜共生模式下的草鱼、鲫、鲢及鳙4种鲤科鱼类肠道微生物优势菌均为鲸杆菌属、梭状芽孢杆菌属、拟杆菌属及芽孢杆菌属,表明鱼类肠道微生物组成不完全由食性决定[17]。兰阿峰等研究发现大鲵肠道微生物以变形菌门为主,其次还包含梭菌门、芽孢杆菌门及衣原体门[18]。
目前,对鲟鱼肠道微生物的研究较少。Ghorbani等对闪光鲟胃肠道可培养细菌进行了分离鉴定, 其中分离获得的细菌以肠杆菌科中的气单胞菌、假单胞菌及沙门氏菌为主[19]。Zahra等人从西伯利亚鲟肠道中分离纯化获得129株单菌,并利用5种致病菌对这129株单菌的益生效果进行了研究,其中乳酸球菌属(7株)、芽孢杆菌属(2株)及柠檬酸杆菌属(1株)对致病菌的抵抗作用效果明显[20]。
采用高通量测序分析方法,本研究对鲟鱼肠道微生物多样性进行了分析。门水平分析,前中肠样品组主要包括变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)及放线菌门(Actinobacteria),该结果与王纯等研究的草鱼肠道微生物组成相似[21]。后肠样品组中,主要以变形菌门(Proteobacteria)和梭杆菌门(Fusobacteria)为主。有研究表明,宿主健康状况及发育阶段的不同均会对肠道微生物组成产生影响[22-23]。本研究中,前中后肠微生物差异原因可能与食物在前中后肠中处于不同状态有关,具体原因有待进一步研究。
