基于mtDNA控制区的波纹唇鱼的4个不同地理群体的遗传多样性
2018-01-11李雨欣王秀英张国庆
李雨欣,王秀英,张国庆
(1.海南大学 海洋学院,海南 海口 570228; 2.海南省水产品质量安全检测中心,海南 海口 570126)
基于mtDNA控制区的波纹唇鱼的4个不同地理群体的遗传多样性
李雨欣1,王秀英2,张国庆1
(1.海南大学 海洋学院,海南 海口 570228; 2.海南省水产品质量安全检测中心,海南 海口 570126)
以海南陵水、西沙、南沙以及马来西亚4个地理区域的101尾波纹唇鱼作为研究对象,利用mtDNA控制区序列对波纹唇鱼进行了遗传多样性分析,并通过扩增、克隆与序列测定技术,获得了mtDNA控制区905 bp的序列,其多态性遗传参数的统计显示,在101尾个体中存在110个变异位点和71个单倍型;总群体的单倍型多样性(Hd)为0. 981,核苷酸的多样性指数(Pi)为0.005 79,表明其遗传多样性处于较低水平.Tajima’s D中性检测均为负值,Fst值属于较低水平,仅为0.031 95,分子方差分析(AMOVA)和Kimuar 2参数模型的分析表明,这4个不同地理群体的波纹唇鱼是由一个小而高效的种群迅速发展而来的,且其遗传多样性和遗传分化水平较低.
波纹唇鱼; mtDNA控制区; 遗传多样性;遗传分化
波纹唇鱼(Cheilinusundulatus)俗称苏眉鱼,属鲈形目(Pereifomes),隆头鱼科(Labridae),唇鱼属(Cheilinus)[1],主要分布于我国东海南部及南海海域;在世界范围主要分布于太平洋西部热带海域、印度洋西北部、大堡礁及红海[2].波纹唇鱼的幼鱼常生活在珊瑚礁礁盘的内侧浅水区,其成鱼则多生活于珊瑚礁礁盘的外侧深水水域[3].波纹唇鱼的肉质富含蛋白质、不饱和脂肪酸和多种微量元素,其肉味鲜美,因而导致了人们对其的过度捕捞[4].近年来,国际动物保护组织已将波纹唇鱼列入濒危物种的名录中(IUCN,CITES等)[5].因此,开展波纹唇鱼种质资源的调查可为该物种的保护提供有价值的参考资料.
线粒体DNA(mtDNA)可作为鱼类遗传分化的一种标记,它是研究因地理隔离而产生遗传差异的主要标识之一[6].底栖鱼类的活动范围有限,其生活的水系亦存在差异,因而造成其群体间存在明显的地理隔离及分化,究其原因是因为mtDNA单倍型发生变异;因此,探究鱼类不同地理群体的mtDNA,这是探索鱼类群体遗传多样性和获知其相关遗传生物学信息的有效途径.传统的鱼类种群识别是基于形态、细胞、同工酶等标记,但重要遗传变异的分子机制是传统手段无法揭示的,而大量存在于mtDNA控制区的分子标记却可作为区分种群的有力标识.mtDNA的存活时间较长,而且遗传自母亲,因此以其来确认家庭关系十分理想[7],且以它作为一种分子遗传标记,其检测十分简便[8].通过mtDNA控制区部分序列和微卫星的分析,Beheregaray揭示了在来自于海洋和江河口的银汉鱼群中所存在的明显的遗传分化[9];Grunwald等亦分析了短鼻鲟群体mtDNA控制区的部分序列,揭示了该群体的地理遗传差异[10].因此,通过分析mtDNA控制区的全部或部分序列,可获得种群间的亲缘关系、起源、分化以及遗传距离等重要的遗传分化信息.
为了获得群体分化与遗传结构的综合评价,本研究通过分子标记的手段,对南海4个不同地理区域的波纹唇鱼的mtDNA控制区进行了评估(陵水、西沙、南沙以及马来西亚),旨在分析该区域波纹唇鱼的序列变异和序列多态性,探讨该物种种质资源的分布状况,以便为该物种的保护和遗传育种奠定理论基础.
1 材料与方法
1.1样品采集如图1所示,实验样品来自南海海域的4个野生群体,海南陵水(LC)有31尾,西沙(XC)有14尾,南沙(NC)有36尾,马来西亚(MC)有20尾,共计101尾波纹唇鱼.实验是通过采集鱼体的尾鳍组织来提取DNA的.
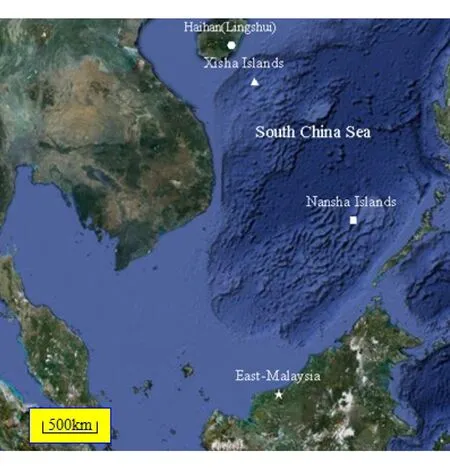
图1 波纹唇鱼群体采集分布注:陵水(●), 西沙 (▲), 南沙(■), 马来西亚 (★)
1.2DNA的提取、扩增、克隆及测序分别取0.1 g尾鳍组织来提取基因组DNA,提取采用的是“酚-氯仿”法,并在50 μL的TE中溶解,于-20 ℃保存(备用).通过PCR扩增可获取mtDNA控制区域,PCR 反应总体积均为25 μL,其中包括2.5 μL 10×PCR Buffer,2 μL Mg2+(25 mmol/L),2 μL dNTP(2 mmol/L),正反向引物各1 μL(10 μmol/L,见表1,由上海捷瑞生物工程有限公司合成),0.3 μL Taq DNA polymerase (5 U/μL),14.2 μL ddH2O,模板2 μL.PCR 反应程序为:94 ℃预变性5 min;热循环94 ℃、30 s,54 ℃、30 s,72 ℃、1.5 min;共30 cycle,最后72 ℃延伸10 min.
扩增所得的产物一部分送上海华大基因公司进行测序鉴定,另一部分则与载体(pMDTM18-T Vector)连接,并导入感受态细胞(DH5ɑ).接着,挑取目的菌落并将其转入含有特定抗生素(氨苄)的液体培养基中进行扩增,然后测序和鉴定.
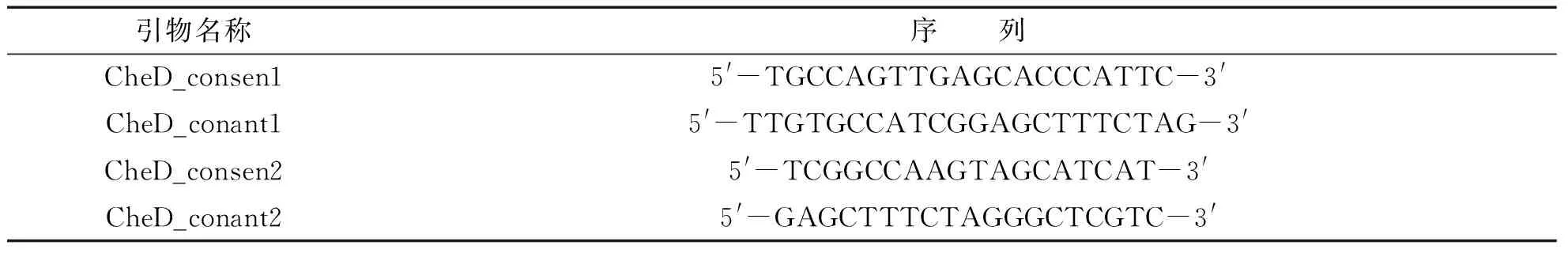
表1 引物序列
1.3数据处理将两种测序结果与波纹唇鱼mtDNA序列(accession number: GU296101)进行比对(blast),以确认mtDNA控制区域,然后运用软件(MEGA5.0)来构建所得序列的NJ系统树,各分支的置信度由1 000次Bootstrap法来检验[11],并计算群体内/间的平均遗传距离;遗传多样性参数,如核苷酸多样性Pi以及单倍型多样性Hd等则是通过运用Dnasp 5.0软件[12]来计算得出;遗传分化参数,如4个群体的单倍型数目、类型及分布、碱基含量等则是由Areliquin 3.11软件[13]计算得出,并经AMOVA分析和Tajima’s D值中性检验.
2 结 果
2.1序列多态性对4个群体(共101尾波纹唇鱼个体)的mtDNA控制区域进行扩增和测序后,获得了905 bp的序列,揭示出110个突变位点(见表2).在这些多态性位点中的碱基被取代最多,仅少部分为缺失和插入.在LC,MC,XC和NC群体中分别存在63,35,14和53个突变位点,总群体中4个碱基的平均含量分别为24.42%(T), 28.47%(C), 27.88%(A)和19.23%(G)(见表3).

表2 波纹唇鱼群体mtDNA控制区的遗传多样性参数
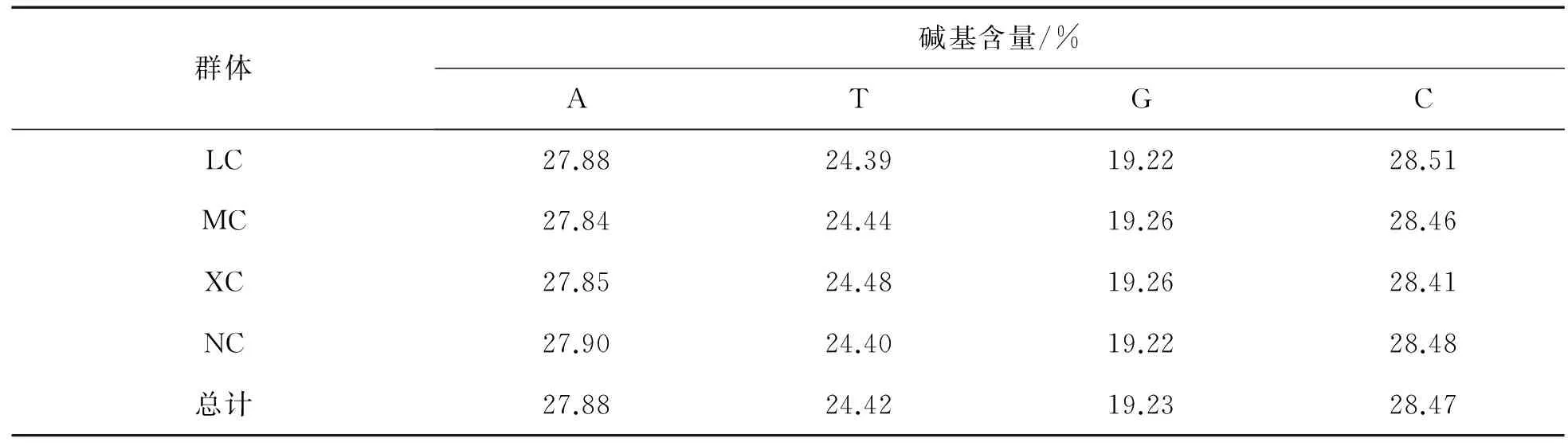
表3 波纹唇鱼群体mtDNA控制区的碱基含量
2.2单倍型和遗传群体分析基于碱基突变,101条序列被划分为71个单倍型,4个群体的单倍型数量分别是29,17,9,29(见表4),每一个群体都拥有其特异的单倍型,NJ进化树显示(见图2),这4个群体的71个单倍型分成了2个较为明显的分支,基于上述单倍型之间的分析,结果表明:群体的遗传差异并不显著.由于许多分支的BP(bootstrap proportion)值小于50%,所以该进化树中几乎没有相似的单倍型.该种群的总核苷酸多样性值较低,仅为0.005 79,但其单倍体多样性却很高,达到了0.981,且所有种群的Tajima’s D值均为负值(XC,P> 0.05;P< 0.05).基于Kimuar 2参数模型,因地理差异而导致的2个种群间的遗传距离从0.005 02到0.006 70不等,种群内部的遗传距离分布在0.005 25~0.006 98的范围(见表5).总体上,LC,MC,XC以及NC种群内部的平均遗传距离分别为0.006 13,0.005 46,0.006 70和0.005 02,然而,一些种群内部的遗传距离甚至大于种间的遗传距离,LC与MC、LC与XC、LC与NC的遗传距离分别为0.005 87,0.006 98和0.005 55;MC与XC和MC与NC的遗传距离分别为0.006 58和0.005 25,XC与NC的遗传距离为0.006 57.
2.3群体结构Fst分析结果显示,HC与XC之间、HC与NC之间的Fst值均大于0.15,但所有种群间的差异并不显著 (P> 0.05).当运用AMOVA方法对波纹唇鱼群体的方差分量进行解析时,结果显示,种间发生的分子变异比例达到了96.81%,种群内的变异仅为3.19%的(见表6).
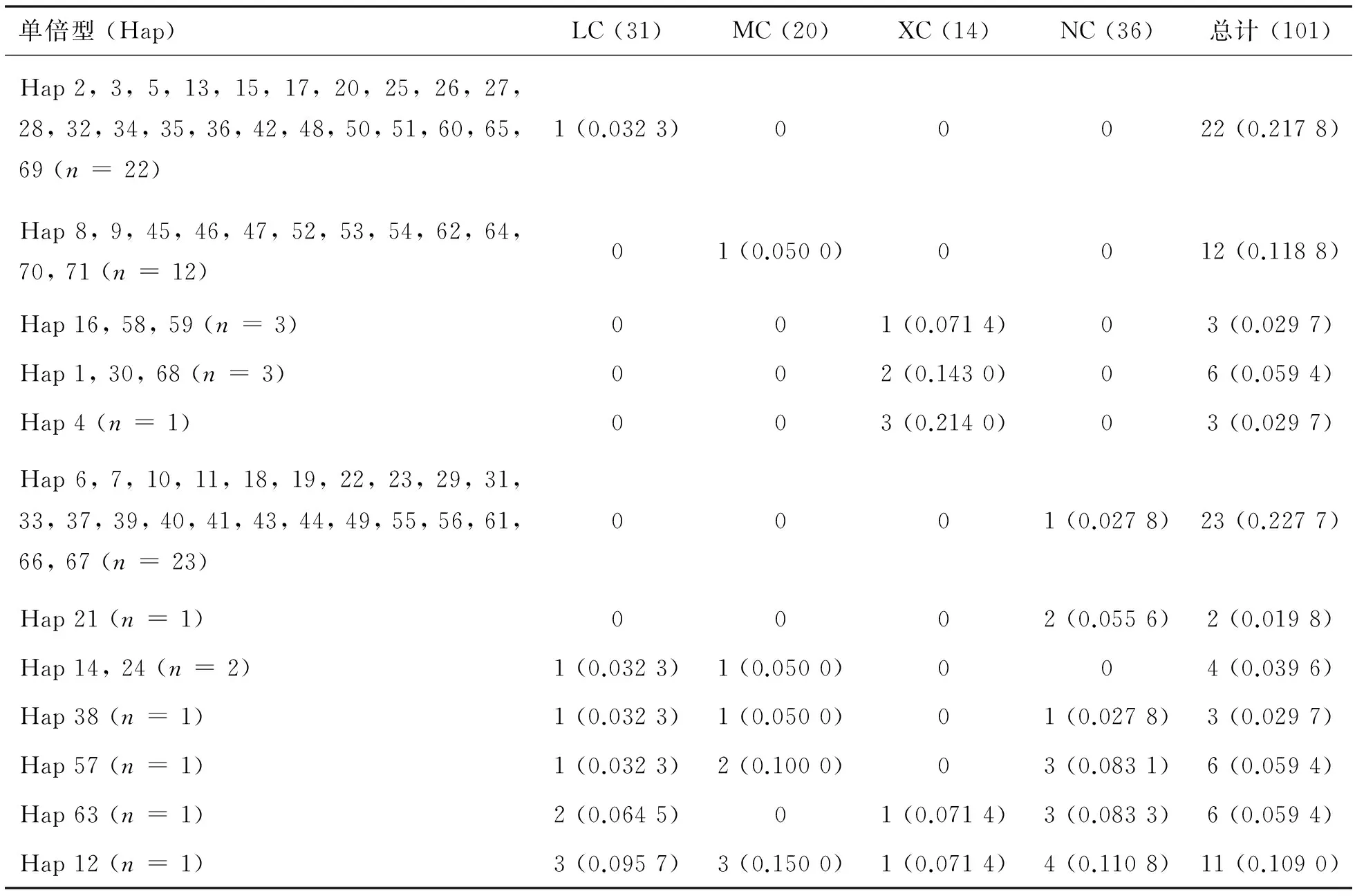
表4 波纹唇鱼4个群体的单倍型

表5 群体间/内遗传距离及分化
“*”为群体内的Kimura 2-parameter 遗传距离.

表6 波纹唇鱼四个群体mtDNA控制区的AMOVA分析
3 讨 论
波纹唇鱼mtDNA控制区的单倍型多样性较高(0.981),但核苷酸多样性属于一个中等偏低的水平[14](0.005 79),此结果与对短鼻鲟鱼mtDNA控制区序列的单倍型和核苷酸多样性的分析很相似[10].不同的是,波纹唇鱼的单倍体和核苷酸多样性远远高于大马哈鱼的(Hd:0.63,Pi:0.003 7)单倍体和核苷酸多样性[15],这或许是序列长度的差异所造成.波纹唇鱼的mtDNA控制区(905 bp)与大马哈鱼[15]的mtDNA控制区(500 bp)和短鼻鲟[16]的mtDNA控制区(440 bp)相比较,其可能能揭示出更多的变异位点;然而,与短鼻鲟鱼和大马哈鱼的大种群相比,波纹唇鱼是一种濒危物种,其种群数量有限,这可能会限制单倍型多样性和核苷酸多样性的评估分析.
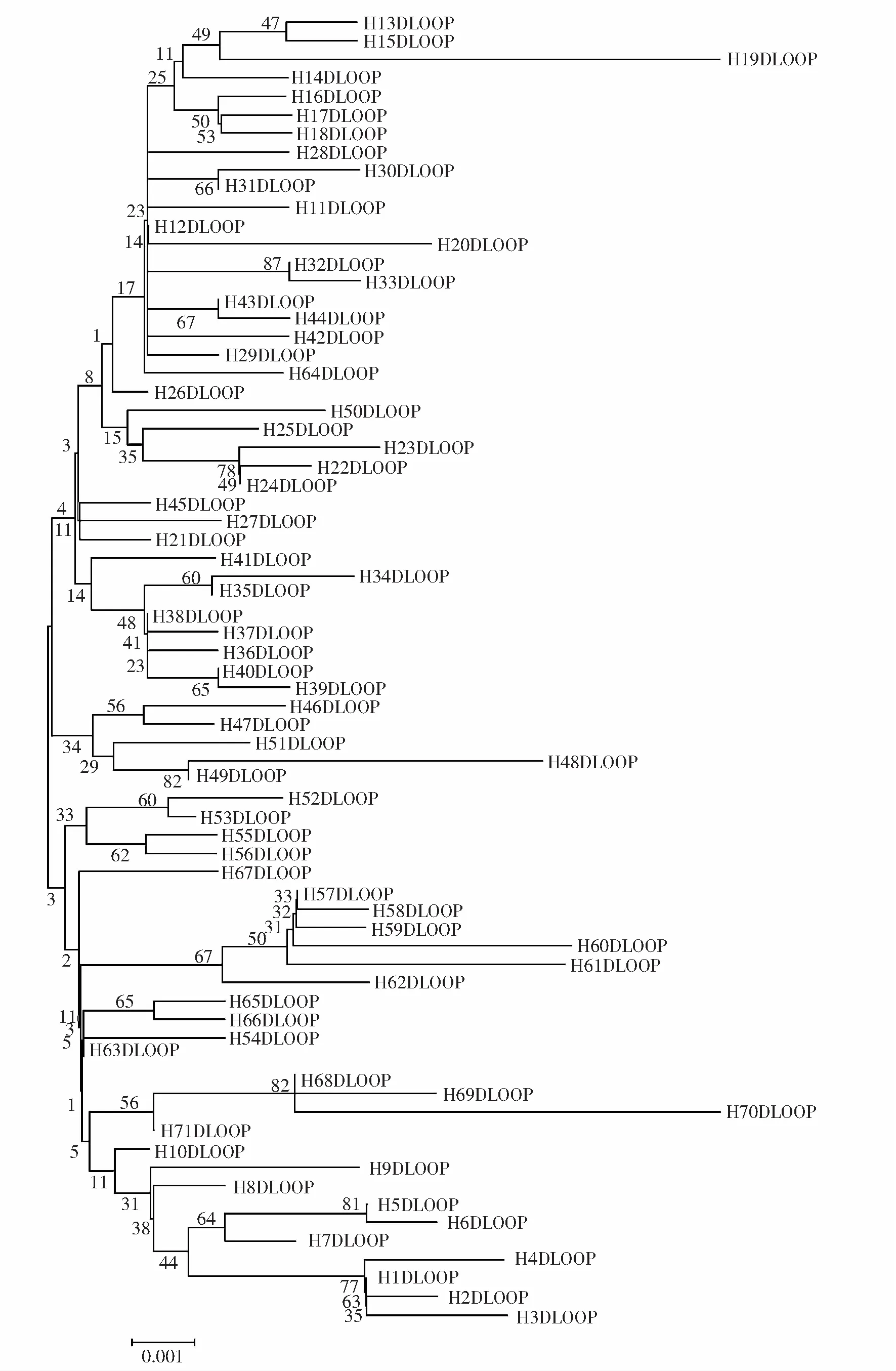
图2 波纹唇鱼mtDNA控制区单倍型NJ进化树
波纹唇鱼4个群体的Fst值水平较低(0.031 95),这表明其群体间的遗传分化较低[17],此外,总群体中的Tajima's D值均为负值,这可能是种群扩增现象的有力依据,同时,它在某种程度上解释了高单倍型但低核苷酸多样性的群体分化现象.结果表明,海南陵水、西沙、南沙以及马来西亚的波纹唇鱼群体是由一个“精”而高效的群体迅速发展而来,因此,它无法在仓促时间内积淀较为重要的核苷酸变异[19];所以其种群和个体间的遗传距离几乎处于同一水平,前者比后者稍低一些.AMOVA的分析结果表明,该物种4个群体的遗传分化较低,但基因交换率却较高.
基于波纹唇鱼mtDNA控制区的分析结果表明,这4个不同地理区域的波纹唇鱼的群体遗传多样性和遗传分化程度较低,这或许是因为群体有限而导致近交频率升高的缘故,进而导致了遗传多样性降低.波纹唇鱼分布的地域较窄,且还伴随着捕捞量剧增,这使得基因之间的交换频率降低,从而造成遗传多样性降低.所以,加强针对该物种的研究和保护是非常有必要的,可见,我们的数据和统计分析对这一物种的人工繁殖和保护是有用的.
[1] Colin P L. Aggregation and spawning of the humphead wrasseCheilinusundulatus(Pisces: Labridae): general aspects of spawning behaviour[J]. Journal of Fish Biology,2010,76(4): 987-1007.
[2] 沈世杰. 台湾鱼类志[M].台北:台湾大学动物学系,1993:143-144.
[3] 佘长宁. 波纹唇鱼神经肽Y基因的克隆及原核表达[D]. 海口:海南大学,2014.
[4] Sadovy Y, Kulbicki M, Labrosse P, et al.,Thehumphead wrasse,Cheilinusundulatus:synopsis of a threatened and poorly known giant coral reef fish [J]. Reviews in Fish Biology and Ficheries, 2003,13:327-364.
[5] 陈猛猛, 骆剑, 陈国华,等. 波纹唇鱼(Cheilinusundulatus)的胚胎发育及初孵仔鱼的形态观察[J]. 渔业科学进展, 2015, 36(5):38-44.
[6] 陈四海,区又君,李加儿.鱼类线粒体DNA及其研究进展[J].生物技术通报, 2011(3):13-20.
[7] 尹绍武, 黄海, 雷从改,等. DNA标记技术在鱼类遗传育种中的应用[J]. 水产科学, 2005, 24(11):42-45.
[8] 孙嘉仪, 杨超, 胡晓雯,等. 一种基于多重PCR的人类线粒体基因组高通量测序方法[J]. 中国科学:生命科学, 2017,47(4):396-402.
[9] Beheregaray L B, Sunnucks P. Fine-scale genetic structure, estuarine colonization and incipient speciation in the marine silverside fishOdontesthesargentinensis.[J]. Molecular Ecology, 2001, 10(12):2849-2866.
[10] Grunwald C, Stabile J, Waldman J R, et al. Population genetics of shortnose sturgeonAcipenserbrevirostrumbased on mitochondrial DNA control region sequences[J]. Molecular Ecology, 2002, 11(10):1885-1898.
[11] Tamura K, Peterson D, Peterson N, et al. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28: 2731-2739.
[12] Librado P, Rozas J. Dnasp v5: a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25:1451-1452.
[13] Excoffier L, Laval G, Schneider S.Arlequin Ver.3.0: an integrated software package for population genetics data analysis [J]. Evolutionary Bioinformatics, 2005, 1:47-50.
[14] Lan H, Shi L M. The origin and genetic differentiation of native breeds of pigs in south west China: An approach from mitochondrial DNA polymorphism [J]. Biochemical Genetics,1993, 31: 51-60.
[15] Sato S, Kojima H, Ando J, et al. Genetic population structure of chum salmon in the Pacific Rim inferred from mitochondrial DNA sequence variation[J]. Environmental Biology of Fishes,2004, 69:37-50.
[16] 刘红艳, 余来宁, 张繁荣. 鱼类线粒体DNA控制区的分子结构及应用进展[J]. 水生态学杂志, 2008, 28(2):4-8.
[17] Ahmed M N, Yu Q, Chanfi M I, et al. Genetic diversity and population differentiation of small giant clamTridacnamaximain Comoros islands assessed by microsatellite markers[J]. Springerplus, 2016, 5(1):2017.
[18] Han Z Q, Li Y Z, Chen G B. Population genetic structure of coral reef speciesPlectorhinchusflavomaculatusin South China Sea [J]. African Journal of Biotechnology, 2008, 7(11):1774-1781.
[19] Lapinski A G,Pavlenko M V, Solovenchuk L L, et al. Some limitations in the use of the mitochondrial DNA cytb, gene as a molecular marker for phylogenetic and population—genetic studies by the example of theApodemus, genus[J]. Russian Journal of Genetics Applied Research, 2016, 6(1):84-90.
GeneticDiversityandDivergenceofCheilinusundulatesfromFourDifferentGeographicPopulationsBasedonmtDNAControlRegion
Li Yuxin1, Wang Xiuying2, Zhang Guoqing1
(1. The Ocean College, Hainan University, Haikou 570228, China;2. Testing Center for Aquatic Product Quality and Safety in Hainan Province, Haikou 570126, China)
In the report, based on the analysis of mtDNA control region, the genetic diversity and divergence of 101Cheilinusundulatesfrom Lingshui, Malaysia, Xisha, and Nansha were analyzed. The 905 bp fragment of the mtDNA control region was obtained by amplification and cloning. There were 110 variation sites, 71 haplotypes in 101 individuals, the total population haplotype diversity (Hd) was 0.981, and the nucleotide diversity index (Pi) was 0.005 79, which indicated that the genetic diversity was low. The data of Tajimas’ D test was negative, and Fst was only 0.031 95. The results of AMOVA and Kimuar 2 parameter model analysis suggested that the different geographical populations ofC.undulatusare developed from a small and efficient population, and the genetic diversity and divergence of four geographical populations ofC.undulatuswere low.
Cheilinusundulatus; mtDNA control region; genetic diversity; genetic divergence
2017-09-25
国家自然科学基金(40966003);海南省自然科学基金(809009)
李雨欣(1991-),女,海南海口人,海南大学海洋学院2013级硕士研究生,E-mail: wenxinfish@163.com
张国庆(1986-),男,黑龙江安达人,博士,工程师,E-mail: zhangguoqing27@163.com
1004-1729(2017)04-0359-07
S917.4
ADOl10.15886/j.cnki.hdxbzkb.2017.0055
