受环境因素影响的DNA甲基化在孤独谱系障碍病因学中的研究进展
2018-01-05赵袁志周东升曹晓君胡珍玉
赵袁志,黄 凌,周东升,曹晓君,胡珍玉
(1宁波大学医学院,浙江 宁波 315211;2宁波市康宁医院,浙江 宁波 315201)
受环境因素影响的DNA甲基化在孤独谱系障碍病因学中的研究进展
赵袁志1,黄 凌1,周东升2,曹晓君1,胡珍玉2
(1宁波大学医学院,浙江 宁波 315211;2宁波市康宁医院,浙江 宁波 315201)
0 前言
孤独谱系障碍(autism spectrum disorder,ASD)是以社会沟通、交往和兴趣行为等表现偏离正常为共同特点的一组神经发育性障碍的统称[1].近十几年年来国际和国内流行病学的相关数据报告均表明,全世界各个国家ASD患病率都在明显升高,已引起相关机构和组织的强烈重视[2].如:美国孤独症及发展障碍监控网(astism and developmental disabilitties monitering,ADDM)2014年统计的数据表明,2010年,美国11个州每1000名8岁儿童中有5.7~21.9名患病,平均有 14.7 名 ASD 患儿.[3].2011 年,俞蓉蓉等[4]通过对已报道的中国10个省市ASD儿童患病率进行统计和分析,认为132 788例0~14岁儿童ASD的总患病率为每1000名孩子中有2.55名患病.
整个20世纪ASD都被认为是一种极其罕见的疾病,起初人们认为父母及家人错误的养育方式才是造成儿童ASD的主要原因.后来有人首次提出遗传和生物因素可能是导致ASD的部分原因,但由于与错误教养方式无关,当时被人们所排斥[5].20世纪60~70年代,ASD被考虑为儿童精神病或者儿童精神分裂症,这种考虑代表人们对ASD的病因学认识取得了巨大的进步.直到20世纪80年代,ASD的生物学机制才开始被人们接受,并且认为这是一种儿童的发育障碍.孤独症先后被DSM-3(the diagnostic and statistic manual of mental disorders-Ⅲ)和 ICD-10(the international classification of diseases-10)引用,定义为广泛性发育障碍 (pervasive developmental disorders,PDD)的一个亚型.在DSM下一个版本DSM-Ⅳ中,PDD被概念化为3个核心症状:社会互动障碍、定性沟通障碍和限制的兴趣爱好与重复的行为模式.其包括五个具体的亚型:孤独症(autism)、不典型孤独症(atypical autism)、阿斯伯格综合症(aspergevr's)、雷特综合症(Rett's)、童年瓦解性障碍(又称衰退性精神障碍,disintegration disorder)[6].在 2013 年 6 月发布的最新版本的DSM-5中这些子组被合并成一个统一的谱系,即孤独谱系障碍.DSM-5与 DSM-4相比,保留DSM-4中的多项内容,排除Rett综合症、童年瓦解性精神障碍.但是,DSM-5在类别、名称、障碍程度、诊断领域、社会性与感知觉内容、诊断年龄等方面有所变动[7].
ASD有很强的遗传基础,如关于双胞胎的一个前瞻性研究报道称,如果一个孩子有一个同胞患有ASD,那么其后代患病的可能性为18.7%,如果这个孩子有超过一个以上的同胞患有ASD,则可能性会达到 32.2%[8-9].ASD 遗传方面的原因很复杂,并且潜在的遗传生物学机制不明,仅用遗传学来解释ASD的病因会产生很多疑问,例如ASD相关的遗传变异也常出现在非ASD诊断的人群中[10].一个大型双队列研究证实子宫内环境因素所致ASD的风险率为30%~80%,明显高于遗传的 14%~67%[11].大量的遗传研究[12]发现,遗传因素目前只能解释大约1/3的ASD患者的病因.这些研究均表明,使用遗传因素解释ASD的病因略显证据不足,ASD产生的机制可能涉及基因和环境危险因素之间复杂的相互作用[13].近年来的主要观点是ASD的产生是遗传、表观遗传和环境多方面共同作用的结果.
1 孤独谱系障碍疾病遗传学病因
基因和基因组变异通常有不同的分类:按照病因是否明确分为“典型”变异和“特发性”变异;根据最小等位基因频率(minor allele frequency,MAF)分为常见变异、罕见变异和低频变异[14];按照机制主要分为单核苷酸多态性(single nucleotide polymorphism,SNP)和 DNA拷贝数变异(copy number variation,CNV)等.
“特发性”是用来描述不明原因引起的ASD的一个概念.这些原因包括单基因、多基因和环境等因素,许多涉及癫痫和智能低下的基因被用来解释ASD的病因.“典型”ASD通常由基因变异所引起,如结节性硬化症、脆性X综合征或其它已知综合症[15].常见变异模型被广泛应用于ASD的研究,为许多候选基因和全基因组关联研究提供了依据.通过对常见变异模型的研究找出了许多ASD易感基因,近来对于ASD罕见基因的研究也成为热门.大量研究[16-17]发现,很多常见疾病如ASD和精神分裂症的主要原因是高度渗透的罕见基因变异,而不是常见基因变异.常见基因突变来源于祖先,这些突变的基因产生的表型会减弱ASD的生育能力,且很多时候其遗传到下一代并未表现出来,这些证据表明常见基因突变并不能完全解释一些遗传问题.而罕见变异由于产生配子时出现了新发突变等因素,导致各种功能障碍和生育能力下降,如只能遗传几代,负面的影响使得这种突变永远不会成为“常见”变异.下面主要从SNP和CNV角度重点综述遗传和ASD之间的关系.
1.1 SNP SNP是指由单个核苷酸变异引起的DNA序列多态性,包括碱基的转换、颠换、插入或缺失.已知SNP增加特发性ASD的遗传风险已经通过候选基因测序的方法证实.
例如SHANK3是ASD的SNP关联性分析中研究最多的基因之一[18].通过结合孤独症测序联盟(autism sequencing consortium,ASC)和孤独症基因组计划(autism genome project,AGP)中已发表的 ASD相关的数据进行统计分析,确认了65个ASD风险基因[19].由于ASD是多基因遗传病,涉及许多 SNPs,而每一个SNP对于ASD产生的影响较小.尽管随着测序技术的高速进步,全外显子组测序(whole exome sequencing,WES)和全基因组测序(whole genome sequencing,WGS)的应用均提高了ASD的SNP检测能力,然而这些方法只能确定有限的位点,并且检测之后的结果难以复制.以上结论均表明仅用常见的SNP对ASD进行预测诊断需要更多样本的证实[18].已知ASD是一种神经发育性疾病,而从神经系统发育相关的基因角度来看ASD的发病基因,一般可大致分为两类,神经发育相关基因(如FMR1、神经元表面蛋白家族基因、MeCP2、神经配体蛋白家族基因等)以及神经变性相关基因(ApoE、Reelin和APP 等)[20].
1.2 DNA拷贝数变异 CNV指的是至少1 kb的基因组复制或删除.CNVs与其他机制相比,是一种比较温和的致病机制.新发或罕见的CNV作为ASD的遗传变异已经被确定[21].Sanders 等[22]发现显著增加的碱基对重复与ASD有关.虽然CNV是多态性的,且经常在人类中遗传,但是在ASD患者中发现的新发或罕见CNV频率更高.有一个ASD家系数量为近200个的研究表明,单一家系(家系中只有一个ASD患者)比多态性家系(家系中有2个或2个以上ASD 患者)有更多的罕见或新发 CNV 比率[23-25].另外,虽然7q31涉及CADPS2和其他7个基因的新发删除,但连锁研究发现7q31中存在几倍的复制[26].而7q31的潜在作用在CADPS2基因敲出的老鼠和在患有ASD的人体研究中都得到了证实[27].Roberts等[28]使用微阵列分析了65名ASD患者的样本,发现参与ASD最常见的染色体是15号染色体,而其上的15q、22q、16p和7q中出现相当高频率的CNVs,如15q11-13 的复制和 16p11.2 的删除和复制[29].
1.3 ASD相关的遗传病因组成 目前经过各方面统合的研究只识别出了部分遗传原因[30],已知的遗传病因在ASD总病因的比例分别为:细胞遗传学变异(<5%),拷贝数变异(即亚微观的删除和重复)(10%~20%)和与ASD相关的单基因变异 (<5%).
2 孤独谱系障碍与表观遗传相关研究
随着遗传学研究的深入,有些疾病的病因并不能很好的用遗传学异常学说解释.例如,虽然同卵双胞胎基因型相同,但他们的表型及疾病易感性大不相同[31].随着 Wu 等[32]在 1942 年对“表观”这个概念的首次提出,人们对表观遗传在解释各种疾病的发病机制的作用形成了初步的认识.表观遗传是指在环境和遗传相互作用后,不发生DNA序列变化的可遗传的基因功能改变.表观遗传作用机制主要涉及DNA共价修饰、蛋白质共价修饰、染色质重塑和非编码RNA的调控等.近些年来,随着人们对表观遗传学各个方面研究的了解加深,逐渐认识到ASD的产生与表观遗传学机制有着密切关系.Rangasamy等[33]发现神经结构及其功能的表观遗传学机制是神经系统发育的关键环节.Nguyen等[34]通过对ASD的同卵双胞胎的淋巴细胞系细胞进行的全基因组的表观遗传分析,证明ASD患者存在广泛的表观遗传失调现象.Schanen等[35]发现许多与ASD相关联的染色体位点可以受到基因组印记影响,证明表观遗传能够增加ASD发病的风险.脆性X综合征和Rett综合症是ASD的亚型,也被发现这些患者存在表观遗传失调的现象[36-37].DNA 甲基化是表观遗传修饰的最重要的方式之一,有证据证明ASD患者存在全基因组甲基化调节异常[38-39].下面主要从 ASD相关 DNA甲基化的各个角度进行分析和介绍.
2.1 DNA甲基化修饰相关背景知识简述 DNA甲基化主要是指将S-腺苷甲硫氨酸(S-adenosyl methionine,SAM)上的甲基(-CH3)转移至DNA的双核苷酸序列(CpG)的特定碱基胞嘧啶的第5位碳原子上,产生5-甲基胞嘧啶(5mC),这个过程依靠 DNMT1、DNMT3a和DNMT3b这3种主要的DNA甲基转移酶作用才能完成[40].普遍认为DNA上高甲基化可以抑制基因的转录,最终使基因沉默(即基因不能表达)[41],低甲基化则使基因高表达.然而也有研究得出相反的结论,即DNA高甲基化也可产生基因的高表达,这可能是因为高甲基化抑制了反义RNA或微小RNA的转录,而它们的功能是下调宿主基因的表达[42].基因甲基化水平和模式随着细胞类型的不同和发育阶段的不同而不同,DNA甲基化在胚胎期、儿童期和成人期均可以发生明显的变化[43],在胚胎期变化最明显.早期胚胎阶段,DNA甲基化用来消除全能性;在细胞分化期间,特定的DNA甲基化发生重调;在后期的发育中,DNA甲基化遗传模式受环境影响发生去甲基化和从头甲基化.组织类型方面,与其他组织相比,人体胚胎干细胞和脑组织有着更高程度的甲基化(>75%)[44].虽然大脑组织是研究精神疾病的最佳组织,近来的全基因组研究却证明一些大脑的表观遗传是可以在外周组织(如血液或者唾液细胞)表现的[45].
2.2 与ASD相关的DNA甲基化 2010年后,ASD相关的全基因组和特定基因的DNA甲基化实验研究总结如表1.
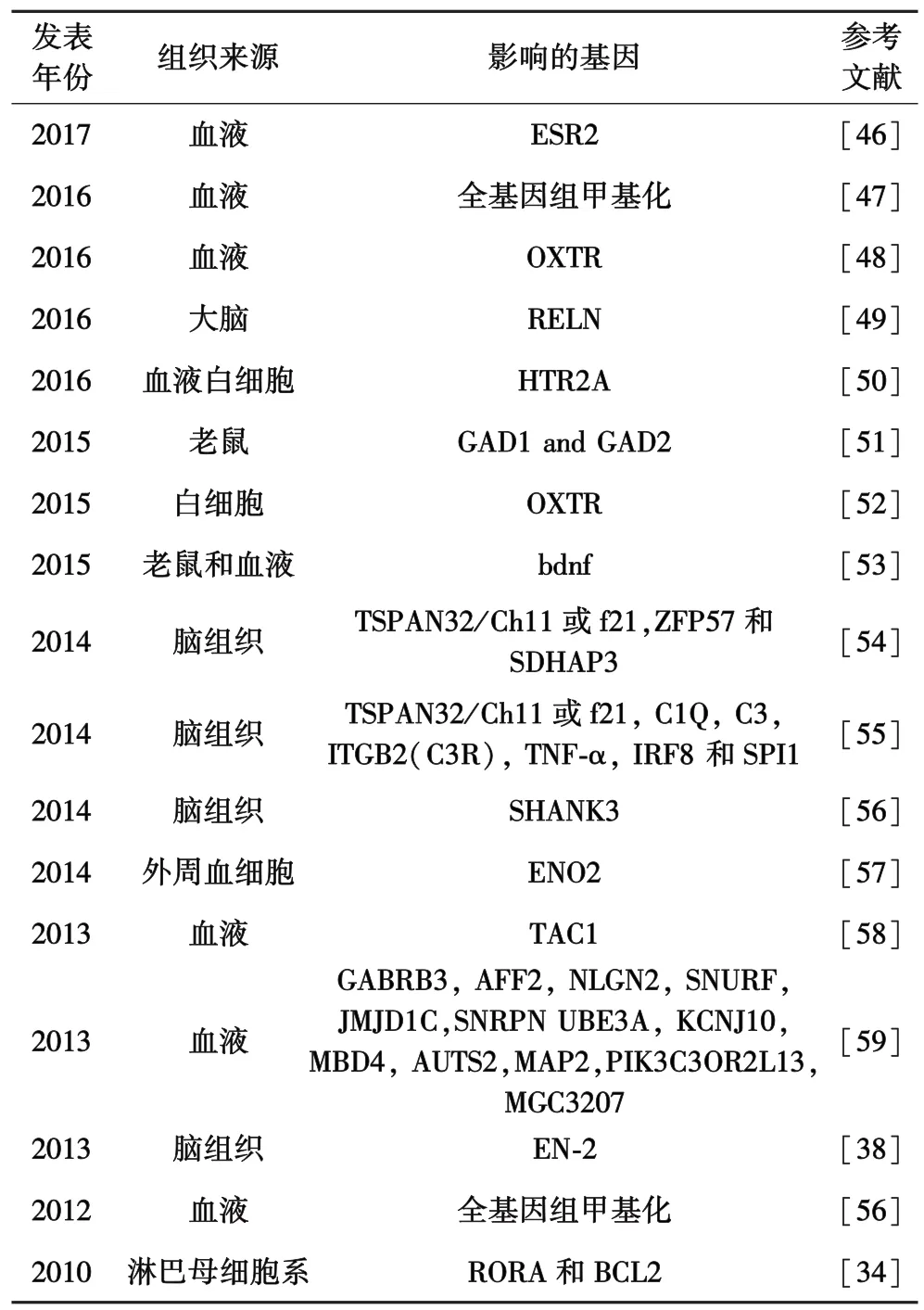
表1 与ASD相关的DNA甲基化研究
2.3 与其他表观遗传学机制的关系 DNA甲基化的过程与另外两个表观遗传机制(基因组印记和X染色体失活)相互作用.例如,启动子区域CpG岛形成的5mC在基因组印记中起重要作用[60].美国的一项研究表明,女性X染色体上有一个微小的环,环内的基因灭活,会导致基因的表达异常,从而引起其后代的发育异常,如Turner综合症、严重的精神发育迟滞等[61].
3 环境因素对ASD的DNA甲基化的影响
尽管环境因素对ASD表观遗传的影响机制并不完全清楚,但环境因素与DNA甲基化的修饰密切相关.如多个人类研究已经确定怀孕期间环境的影响和后代甲基化模式存在关联[10].与ASD相关的很多环境因素也可能导致人体内的表观遗传学变化,使基因功能发生紊乱,从而增加儿童患ASD的易感性.目前可以将对ASD的DNA甲基化产生影响的环境因素分为以下大类:感染和免疫、饮食和营养、环境毒性物质的接触、父母生理状态、生活方式和压力或氧化应激等.
3.1 感染和免疫 Nardone等[10]通过综述大量的文献得出结论:免疫激活(如母孕期的病毒感染)和孕产妇自身免疫性疾病与ASD有关.Poly I:C(聚肌胞苷酸)是一种双链模拟RNA分子,可以通过激活Toll样受体表达interferon-1触发免疫反应,可以向怀孕的啮齿动物注入Poly I:C模拟母孕期的免疫激活反应[62].Basil等[63]用这个模型模拟免疫反应,检测新生老鼠的丘脑下部组织MECP2和LINE-1基因的DNA甲基化,发现这2个基因CpGs的DNA甲基化降低.Labouesse 等[51]的实验也发现,用 Poly I:C 治疗母老鼠,其后代的额叶皮质GAD1和GAD2基因DNA甲基化增高.这些实验表明,免疫激活可以引发后代大脑中DNA甲基化模式的长期变化.
3.2 饮食和营养 在饮食补充方面,最著名的是对1944~1945年丹麦的冬天饥荒的研究,Heijmans等[64]通过检测曾经历过饥荒母亲的后代的甲基化分析发现,IGF2基因的DNA甲基化发生了失调.Tobi等[65]继续展开研究,发现在饥荒过了60多年后,之前的表观改变仍然存在,这就证明了孕产期环境因素(包括饮食的摄入)对表观遗传的影响长期持续存在.而针对ASD的DNA甲基化相关的饮食摄入的研究,迄今为止研究最多的营养物质是叶酸.DNA甲基化取决于饮食甲基供体(如叶酸、胆碱和蛋氨酸等)的可用性,而这些供体又通过一碳单位代谢相互关联.叶酸或甲基供体可以通过影响DNA甲基化能力继而间接影响神经的发育.现有的人类和动物一碳单位代谢的研究表明,母体叶酸状态通过影响DNA甲基化而影响 ASD 患病的风险因素[66].Hendren 等[67]用甲基维生素B12治疗ASD的儿童,发现临床改善与等离体蛋氨酸的增加、S-腺苷-L-高半胱氨酸(SAH)的减少和S-腺苷甲硫胺酸 (SAM)/(SAH)的增加成正相关,表明甲基维生素B12可以改善ASD患儿的细胞甲基化能力.当然除了甲基化供体物质还有其他物质的摄入也能影响甲基化的变化,如高脂肪、低碳水化合物和生酮饮食[68].
3.3 环境毒性物质 环境毒性物质会增加ASD患病风险,Deth[69]发现,环境化学物质可以改变发育的大脑的DNA甲基化模式,从而增加ASD的患病风险.这些化学物质具有代表性的是多氯联苯(PCB)、多环芳烃(PAH)、铅、双酚 A(BPA),汞和杀虫剂.研究[70-71]表明妊娠期接触 PAH,会导致后代 Bdnf IV启动子甲基化的增加和PPARγ甲基化的降低.与生殖毒性有关的BPA能够影响生长和免疫调节,在围产期饮食接触过BPA的妇女,近来发现其后代的骨髓源性肥大细胞中全基因组甲基化降低[72].空气污染作为重要的环境毒性物质之一,早在2006年就被报道与罹患ASD风险之间存在关联[73].美国的科学家发现在高浓度的超细环境颗粒环境下生活的小鼠模型的大脑出现了神经化学变化、神经胶质细胞激活和脑室体积扩大的现象[74].而中国科学家发现细颗粒物(PM2.5)可以通过影响DNA甲基化而对神经发育产生毒性作用[75].这些研究均为空气污染如何增加ASD患病风险提供了可能的发生机制.
3.4 父母的生理状态 母亲的妊娠期糖尿病(gestational diabetes)是一种常见的妊娠并发症,Wang等[76]在2017年发表了一篇meta分析,指出妊娠期糖尿病是ASD的患病风险因素之一.Barua等[77]也指出妊娠期糖尿病可以诱发几个基因的甲基化模式发生潜在的变化,这些受影响的基因参与代谢疾病的形成.多个研究发现母亲被诊断为妊娠期糖尿病,通过对其后代的脐带血进行甲基化检测,发现DNA甲基化调节异常[78-79].DNA 甲基化的干扰可以起源于精子时期,与父亲的年龄有关,这种机制通过表观缺陷而表达出来[80].
3.5 生活方式 父母最常见的不良生活方式包括吸烟和饮酒.Breton等采用“Illumina Human Methylation 27芯片”技术对产前烟雾暴露史的儿童进行甲基化分析,发现19个CpG位点的甲基化与产前吸烟显著相关,表明产前吸烟与表观遗传改变密切相关,而这种改变能够持续在整个儿童时期[81].Ladd-Acosta等[82]搜集了产前母体吸烟的572个婴儿脐带血样本,对与之相关的26个CpG位点的甲基化进行分析,这些DNA甲基化变化似乎有烟草特异性,且母亲产前接触吸烟的后代在学龄前儿童期和出生时的DNA甲基化情况基本一致,从而说明了血液DNA甲基化可能作为预测产前是否接触过吸烟的生物标志物.对老鼠胚胎研究发现,饮酒可以通过DNA甲基化改变而影响早期胚神经的形成和几个基因(参与细胞周期、生长、凋亡和印记等功能)的表达,从而最终调节早期胎儿的发育[83].尽管妊娠期吸烟饮酒也会导致甲基化,但Fujiwara等[84]通过综述大量文献得出结论,传统的危险因素如吸烟/烟草和酒精不太可能与ASD有关,因此产前的吸烟和饮酒通过调节DNA甲基化影响ASD的患病风险仍需进一步探索.
3.6 压力和氧化应激 Frye等[85]指出可以通过铁和铜代谢异常等多种氧化应激机制增加ASD的患病风险,而氧化应激总体上的的机制是内源/外源活性氧的生成增强和对活性氧的抗氧化的防御的失衡.氧化应激可以影响DNA甲基化,双酚A(bisphenol A,BPA)可以通过高水平的循环葡萄糖诱导ASD患儿的氧化应激反应[86],同时双酚 A可以抑制甲基化[72],由此我们可以推断氧化应激可能抑制ASD的甲基化水平.日本科学家指出,妊娠前3个月,产前压力或应激会通过影响表观修饰(如DNA甲基化)来增加患ASD的易感性[87].
3.7 环境因素之间的关系 与ASD相关的DNA甲基化很难只受到单一环境因素影响,实际上环境因素之间也能够相互作用,因而环境因素对ASD的DNA甲基化的影响机制相当复杂.例如上文提到的荷兰饥荒研究中,至少有两个以上的风险因素可能会相互影响后代的神经发育,如营养不良和由于饥荒、战争和许多其他压力事件(如悲惨的生活状况)带来的母亲的压力[88].与ASD有关的妊娠期糖尿病不仅可以影响DNA甲基化失调,而且还可以通过影响表观遗传来调节免疫系统的激活[10].多种环境因素的相互作用对ASD的DNA甲基化的产生影响机制仍需进一步的研究.
4 DNA甲基化对ASD的应用前景
致病性甲基化变化对外环境、内环境改变较敏感,也能成为药物作用的新靶点.目前用于表观遗传的药物分为两类:核苷类似物和非核苷类似物.①核苷类似物整合于DNA,并通过构成共价键封锁DNA甲基转移酶,现阶段正试验用于恶性血液病治疗.②非核苷类似物通过结合催化亚基封锁DNA甲基转移酶并限制其表达[89].使用Rett综合症的小鼠大脑皮层神经元和Rett综合症患者的成纤维细胞作为实验对象的研究,表明MeCp2删除可以导致BDNF的减少,异常细胞迁移和分化,而这些异常都可以被组蛋白去乙酰化酶6抑制剂(tubastatin A)干预所恢复,以上研究说明表观遗传治疗可能用于Rett综合症的治疗[90-91].同样用 DNA 甲基化抑制剂 5-氮杂胞苷(5-azacytidine)治疗相关疾病的研究也得到了类似的效果[92].
5 总结和展望
本文重点综述了与ASD相关的遗传和表观遗传方面的研究进展,尤其是环境因素对ASD相关的DNA甲基化的影响.目前国内对ASD的表观遗传的研究较少,作为ASD病因学研究的又一切入口,DNA甲基化是联系个体与环境的一个纽带.人类在母体内所处的早期环境以及后期的生存环境均可影响基因组DNA的甲基化修饰,进而调控相关基因的表达,最终影响疾病的发生和发展[93].本文的作者均是宁波市康宁医院儿童精神学组的成员,而我们此次ASD甲基化课题的目的就是想筛选出更多与ASD发病相关的DNA甲基化位点,且进一步证实其差异化的甲基化影响基因的功能和表达.未来的ASD甲基化研究重点可能集中于研究DNA甲基化对基因表达的影响机制、其他表观遗传修饰(如组蛋白修饰、非编码RNA调控)与DNA甲基化相互作用机理以及环境因素对DNA甲基化的作用机制.建立起与ASD相关的DNA甲基化的功能数据网络,以便对提高ASD疾病风险的预测能力(如预后判断、诊断分型、疗效评估、疾病发展监控),并对其进行更完善的系统评估.
[1]邹小兵,邓红珠.美国精神疾病诊断分类手册第5版“孤独症谱系障碍诊断标准”解读[J].中国实用儿科杂志,2013,28(8):561-563.
[2]李洪华,杜 琳,单 玲,等.孤独症谱系障碍流行病学研究现状[J].中华临床医师杂志:电子版,2014,8(24):4471-4474.
[3]武丽杰.我国孤独症谱系障碍流行病学现状及趋势[J].中国实用儿科杂志,2013,28(8):571-576.
[4]俞蓉蓉,林良华,许 丹,等.我国儿童孤独症患病情况分析[J].中国妇幼保健,2011,26(29):4563-4565.
[5]Cantwell DP,Baker L,Rutter M.Families of autistic and dysphasic children.I.Family life and interaction patterns[J].Arch Gen Psychiatry,1979,36(6):682-687.
[6]Gmitrowicz A, Kucharska A.[Developmental disorders in the fourth edition of the American classification:diagnostic and statistical man-ual of mental disorders (DSM IV--optional book)][J].Psychiatr Pol, 1994,28(5):509-521.
[7]李改智,江文庆,杜亚松,等.以DSM系统和ABC量表角度来看孤独症症状变化[J].中国儿童保健杂志,2015,23(3):257-260.
[8]Ronald A, Hoekstra RA.Autism spectrum disorders and autistic traits: a decade of new twin studies[J].Am J Med Genet B Neuropsychiatr Genet,2011,156B(3):255-274.
[9]Ozonoff S, Young GS, Carter A, et al.Recurrence risk for autism spectrum disorders: a baby siblings research consortium study[J].Pediatrics,2011,128(3):e488-e495.
[10]Nardone S, Elliott E.The Interaction between the immune system and epigenetics in the etiology of autism spectrum disorders[J].Front Neurosci, 2016,10:329.
[11]Hallmayer J, Cleveland S, Torres A, et al.Genetic heritability and shared environmental factors among twin pairs with autism[J].Arch Gen Psychiatry,2011,68(11):1095-1102.
[12]Yu L, Wu Y, Wu BL.Genetic architecture, epigenetic influence and environment exposure in the pathogenesis of Autism[J].Sci China Life Sci,2015,58(10):958-967.
[13]Anderson GM.Twin studies in autism: what might they say about genetic and environmental influences[J].J Autism Dev Disord,2012,42(7):1526-1527.
[14]Abrahams BS, Geschwind DH.Advances in autism genetics: on the threshold of a new neurobiology[J].Nat Rev Genet,2008,9(5):341-355.
[15]Lintas C,Persico AM.Autistic phenotypes and genetic testing:stateof-the-art for the clinical geneticist[J].J Med Genet,2009,46(1):1-8.
[16]McClellan J, King MC.Genetic heterogeneity in human disease[J].Cell,2010,141(2):210-217.
[17]McClellan J, King MC.Genomic analysis of mental illness: a changing landscape[J].JAMA,2010,303(24):2523-2524.
[18]Huguet G, Ey E, Bourgeron T.The genetic landscapes of autism spectrum disorders[J].Annu Rev Genomics Hum Genet,2013,14:191-213.
[19]Sanders SJ, He X, Willsey AJ, et al.Insights into autism spectrum disorder genomic architecture and biology from 71 risk loci[J].Neuron,2015,87(6):1215-1233.
[20]Hua R,Wei M,Zhang C.The complex genetics in autism spectrum disorders[J].Sci China Life Sci,2015,58(10):933-945.
[21]Girirajan S, Johnson RL, Tassone F, et al.Global increases in both common and rare copy number load associated with autism[J].Hum Mol Genet,2013,22(14):2870-2880.
[22]Sanders SJ, Ercan-Sencicek AG, Hus V, et al.Multiple recurrent de novo CNVs, including duplications of the 7q11.23 Williams syndrome region, are strongly associated with autism[J].Neuron,2011,70(5):863-885.
[23]Sebat J, Lakshmi B, Malhotra D, et al.Strong association of de novo copy number mutations with autism[J].Science,2007,316(5823):445-449.
[24]Stefansson H, Rujescu D, Cichon S, et al.Large recurrent microdeletions associated with schizophrenia[J].Nature,2008,455(7210):232-236.
[25]Pinto D, Pagnamenta AT, Klei L, et al.Functional impact of global rare copy number variation in autism spectrum disorders[J].Nature,2010,466(7304):368-372.
[26]Szatmari P, Paterson AD, Zwaigenbaum L, et al.Mapping autism risk loci using genetic linkage and chromosomal rearrangements[J].Nat Genet,2007,39(3):319-328.
[27]Sadakata T,Washida M,Iwayama Y,et al.Autistic-like phenotypes in Cadps2-knockout mice and aberrant CADPS2 splicing in autistic patients[J].J Clin Invest,2007,117(4):931-943.
[28]Roberts JL, Hovanes K, Dasouki M, et al.Chromosomal microarray analysis of consecutive individuals with autism spectrum disorders or learning disability presenting for genetic services[J].Gene,2014,535(1):70-78.
[29]Levy D, Ronemus M,Yamrom B, et al.Rare de novo and transmitted copy-number variation in autistic spectrum disorders[J].Neuron,2011,70(5):886-897.
[30]Ivanov HY, Stoyanova VK, Popov NT, et al.Autism spectrum disorder-a complex genetic disorder[J].Folia Med (Plovdiv),2015,57(1):19-28.
[31]Kato T, Iwamoto K, Kakiuchi C, et al.Genetic or epigenetic difference causing discordance between monozygotic twins as a clue to molecular basis of mental disorders[J].Mol Psychiatry,2005,10(7):622-630.
[32]Wu Ct, Morris JR.Genes, genetics, and epigenetics: a correspondence[J].Science,2001,293(5532):1103-1105.
[33]Rangasamy S, D'Mello SR, Narayanan V.Epigenetics, autism spectrum, and neurodevelopmental disorders[J].Neurotherapeutics,2013,10(4):742-756.
[34]Nguyen A, Rauch TA, Pfeifer GP, et al.Global methylation profiling of lymphoblastoid cell lines reveals epigenetic contributions to autism spectrum disorders and a novel autism candidate gene,RORA, whose protein product is reduced in autistic brain[J].FASEB J,2010,24(8):3036-3051.
[35]Schanen NC.Epigenetics of autism spectrum disorders[J].Hum Mol Genet,2006,15 Spec No 2:R138-R150.
[36]Penagarikano O, Mulle JG, Warren ST.The pathophysiology of fragile x syndrome[J].Annu Rev Genomics Hum Genet,2007,8:109-129.
[37]Shahbazian MD, Zoghbi HY.Rett syndrome and MeCP2: linking epigenetics and neuronal function[J].Am J Hum Genet,2002,71(6):1259-1272.
[38]James SJ, Shpyleva S, Melnyk S, et al.Complex epigenetic regulation of engrailed-2 (EN-2) homeobox gene in the autism cerebellum[J].Transl Psychiatry,2013,3:e232.
[39]Hu VW.From genes to environment: using integrative genomics to build a “systems-level” understanding of autism spectrum disorders[J].Child Dev,2013,84(1):89-103.
[40]Kar S,Deb M, Sengupta D,et al.An insight into the various regulatory mechanisms modulating human DNA methyltransferase 1 stability and function[J].Epigenetics,2012,7(9):994-1007.
[41]Shen L, Kondo Y, Guo Y, et al.Genome-wide profiling of DNA methylation reveals a class of normally methylated CpG island promoters[J].PLoS Genet,2007,3(10):2023-2036.
[42]Wang LQ, Liang R, Chim CS.Methylation of tumor suppressor microRNAs: lessons from lymphoid malignancies[J].Expert Rev Mol Diagn,2012,12(7):755-765.
[43]Numata S, Ye T, Hyde TM, et al.DNA methylation signatures in development and aging of the human prefrontal cortex[J].Am J Hum Genet,2012,90(2):260-272.
[44]Irizarry RA, Ladd-Acosta C, Wen B, et al.The human colon cancer methylome shows similar hypo-and hypermethylation at conserved tissue-specific CpG island shores[J].Nat Genet,2009,41(2):178-186.
[45]Abdolmaleky HM,Nohesara S, Ghadirivasfi M,et al.DNA hypermethylation of serotonin transporter gene promoter in drug naïve patients with schizophrenia[J].Schizophr Res,2014,152(2- 3):373-380.
[46]Wang X, Liang S, Sun Y, et al.Analysis of estrogen receptor β gene methylation in autistic males in a Chinese Han population[J].Metab Brain Dis,2017.
[47]Homs A, Codina-Solà M, Rodríguez-Santiago B, et al.Genetic and epigenetic methylation defects and implication of the ERMN gene in autism spectrum disorders[J].Transl Psychiatry,2016,6(7):e855.
[48]Elagoz Yuksel M, Yuceturk B, Karatas OF, et al.The altered promoter methylation of oxytocin receptor gene in autism[J].J Neurogenet,2016,30(3-4):280-284.
[49]Lintas C, Sacco R, Persico AM.Differential methylation at the RELN gene promoter in temporal cortex from autistic and typically developing post-puberal subjects[J].J Neurodev Disord,2016,8:18.
[50]Hranilovic D, Blazevic S, Stefulj J, et al.DNA methylation analysis of HTR2A regulatory region in leukocytes of autistic subjects[J].Autism Res,2016,9(2):204-209.
[51]Labouesse MA, Dong E, Grayson DR, et al.Maternal immune activation induces GAD1 and GAD2 promoter remodeling in the offspring prefrontal cortex[J].Epigenetics,2015,10(12):1143-1155.
[52]Behnia F, Parets SE, Kechichian T, et al.Fetal DNA methylation of autism spectrum disorders candidate genes:association with spontaneous preterm birth[J].Am J Obstet Gynecol,2015,212(4):533.e1-9.
[53]Kundakovic M, Gudsnuk K,Herbstman JB, et al.DNA methylation of BDNF as a biomarker of early-life adversity[J].Proc Natl Acad Sci U S A,2015,112(22):6807-6813.
[54]Ladd-Acosta C, Hansen KD, Briem E, et al.Common DNA methylation alterations in multiple brain regions in autism[J].Mol Psychiatry,2014,19(8):862-871.
[55]Nardone S, Sams DS,Reuveni E,et al.DNA methylation analysis of the autistic brain reveals multiple dysregulated biological pathways[J].Transl Psychiatry,2014,4:e433.
[56]Zhu L, Wang X, Li XL, et al.Epigenetic dysregulation of SHANK3 in brain tissues from individuals with autism spectrum disorders[J].Hum Mol Genet,2014,23(6):1563-1578.
[57]Wang Y, Fang Y, Zhang F, et al.Hypermethylation of the enolase gene (ENO2) in autism[J].Eur J Pediatr, 2014,173(9):1233-1244.
[58]Aldinger KA, Plummer JT,Levitt P.Comparative DNA methylation among females with neurodevelopmental disorders and seizures identifies TAC1 as a MeCP2 target gene[J].J Neurodev Disord, 2013,5(1):15.
[59]Melnyk S, Fuchs GJ, Schulz E, et al.Metabolic imbalance associated with methylation dysregulation and oxidative damage in children with autism[J].J Autism Dev Disord, 2012,42(3):367-377.
[60]James SJ, Shpyleva S, Melnyk S, et al.Elevated 5-hydroxymethylcytosine in the Engrailed-2 (EN-2) promoter is associated with increased gene expression and decreased MeCP2 binding in autism cerebellum[J].Transl Psychiatry,2014,4:e460.
[61]Migeon BR.Why females are mosaics, X-chromosome inactivation,and sex differences in disease[J].Gend Med, 2007,4(2):97-105.
[62]Alexopoulou L, Holt AC, Medzhitov R, et al.Recognition of doublestranded RNA and activation of NF-kappaB by Toll-like receptor 3[J].Nature,2001,413(6857):732-738.
[63]Basil P, Li Q, Dempster EL, et al.Prenatal maternal immune activation causes epigenetic differences in adolescent mouse brain[J].Transl Psychiatry,2014,4:e434.
[64]Heijmans BT, Tobi EW, Stein AD, et al.Persistent epigenetic differences associated with prenatal exposure to famine in humans[J].Proc Natl Acad Sci U S A,2008,105(44):17046-17049.
[65]Tobi EW, Lumey LH, Talens RP, et al.DNA methylation differences after exposure to prenatal famine are common and timing-and sex-specific[J].Hum Mol Genet,2009,18(21):4046-4053.
[66]Warzak DA, Johnson SA, Ellersieck MR, et al.Effects of post-weaning diet on metabolic parameters and DNA methylation status of the cryptic promoter in the A(vy) allele of viable yellow mice[J].J Nutr Biochem,2015,26(6):667-674.
[67]Hendren RL, James SJ, Widjaja F, et al.Randomized, placebocontrolled trial of methyl B12 for children with autism[J].J Child adolesc psychopharmacol,2016,26(9):774-783.
[68]Boison D.New insights into the mechanisms of the ketogenic diet[J].Curr Opin Neurol,2017,30(2):187-192.
[69]Deth RC.Autism: a redox/methylation disorder[J].Glob Adv Health Med,2013,2(6):68-73.
[70]Miller RL, Yan Z, Maher C, et al.Impact of prenatal polycyclic aromatic hydrocarbon exposure on behavior,cortical gene expression and DNA methylation of the Bdnf gene[J].Neuroepigenetics,2016,5:11-18.
[71]Yan Z, Zhang H, Maher C, et al.Prenatal polycyclic aromatic hydrocarbon, adiposity, peroxisome proliferator-activated receptor(PPAR) γ methylation in offspring, grand-offspring mice[J].PLoS One,2014,9(10):e110706.
[72]O'Brien E, Dolinoy DC, Mancuso P.Perinatal bisphenol A exposures increase production of pro-inflammatory mediators in bone marrow-derived mast cells of adult mice[J].J Immunotoxicol,2014,11(3):205-212.
[73]Windham GC, Zhang L, Gunier R, et al.Autism spectrum disorders in relation to distribution of hazardous air pollutants in the san francisco bay area[J].Environ Health Perspect,2006,114(9):1438-1444.
[74]Allen JL, Liu X, Pelkowski S, et al.Early postnatal exposure to ultrafine particulate matter air pollution: persistent ventriculomegaly,neurochemical disruption,and glial activation preferentially in male mice[J].Environ Health Perspect,2014,122(9):939-945.
[75]Wei H, Liang F, Meng G, et al.Redox/methylation mediated abnormal DNA methylation as regulators of ambient fine particulate matterinduced neurodevelopment related impairment in human neuronal cells[J].Sci Rep,2016,6:33402.
[76]Wang C, Geng H, Liu W, et al.Prenatal, perinatal, and postnatal factors associated with autism: a meta-analysis[J].Medicine (Baltimore),2017,96(18):e6696.
[77]Barua S, Junaid MA.Lifestyle, pregnancy and epigenetic effects[J].Epigenomics,2015,7(1):85-102.
[78]Finer S, Mathews C, Lowe R, et al.Maternal gestational diabetes is associated with genome-wide DNA methylation variation in placenta and cord blood of exposed offspring[J].Hum Mol Genet,2015,24(11):3021-3029.
[79]Quilter CR,Cooper WN,Cliffe KM,et al.Impact on offspring methylation patterns ofmaternal gestational diabetes mellitus and intrauterine growth restraint suggest common genes and pathways linked to subsequent type 2 diabetes risk[J].FASEB J,2014,28(11):4868-4879.
[80]Feinberg JI, Bakulski KM, Jaffe AE, et al.Paternal sperm DNA methylation associated with early signs of autism risk in an autismenriched cohort[J].Int J Epidemiol,2015,44(4):1199-1210.
[81]Breton CV, Siegmund KD, Joubert BR, et al.Prenatal tobacco smoke exposure is associated with childhood DNA CpG methylation[J].PLoS One,2014,9(6):e99716.
[82]Ladd-Acosta C, Shu C, Lee BK, et al.Presence of an epigenetic signature of prenatal cigarette smoke exposure in childhood[J].Environ Res,2016,144(Pt A):139-148.
[83]Liu Y, Balaraman Y, Wang G, et al.Alcohol exposure alters DNA methylation profiles in mouse embryos at early neurulation[J].Epigenetics,2009,4(7):500-511.
[84]Fujiwara T, Morisaki N, Honda Y, et al.Chemicals, nutrition, and autism spectrum disorder: a mini-review[J].Front Neurosci,2016,10:174.
[85]Frye RE, Melnyk S, Fuchs G, et al.Effectiveness of methylcobalamin and folinic Acid treatment on adaptive behavior in children with autistic disorder is related to glutathione redox status[J].Autism Res Treat,2013,2013:609705.
[86]Manikkam M,Tracey R,Guerrero-Bosagna C,et al.Plastics derived endocrine disruptors(BPA, DEHP and DBP) induce epigenetic transgenerational inheritance of obesity,reproductive disease and sperm epimutations[J].PLoS One,2013,8(1):e55387.
[87]Udagawa J, Hino K.Impact of maternal stress in pregnancy on brain function of the offspring[J].Nihon Eiseigaku Zasshi,2016,71(3):188-194.
[88]Wright EC,Johnson SA,Hao R, et al.Exposure to extrinsic stressors, social defeat or bisphenol a, eliminates sex differences in DNA methyltransferase expression in the amygdala[J].J Neuroendocrinol,2017.
[89]周 萌,唐倩倩,刘 婷,等.基于DNA甲基化的宫颈癌基因治疗进展[J].转化医学电子杂志,2017,4(5):70-73.
[90]Gold WA, Lacina TA, Cantrill LC, et al.MeCP2 deficiency is associated with reduced levels of tubulin acetylation and can be restored using HDAC6 inhibitors[J].J Mol Med (Berl),2015,93(1):63-72.
[91]Xu X, Kozikowski AP, Pozzo-Miller L.A selective histone deacetylase-6 inhibitor improves BDNF trafficking in hippocampal neurons from Mecp2 knockout mice: implications for Rett syndrome[J].Front Cell Neurosci,2014,8:68.
[92]Shvachko LP.DNA hypomethylation as Achilles'heel of tumorigenesis: a working hypothesis[J].Cell Biol Int,2009,33(8):904-910.
[93]Connor CM, Akbarian S.DNA methylation changes in schizophrenia and bipolar disorder[J].Epigenetics,2008,3(2):55-58.
Research progress on the etiology of DNA methylation affected by the environmental factors in autism spectrum disorder
ZHAO Yuan-Zhi1, HUANG Ling1, ZHOU Dong-Sheng2, CAO Xiao-Jun1,HU Zhen-Yu21Medical School of Ningbo University, Ningbo 315211, China;2Ningbo Kangning Hospital, Ningbo 315201, China
Autism spectrumdisorder(ASD) is a general term for a group of neurodevelopmental disorders characterized by deviation from normal in social communication,interaction and interest behavior and so on.At present, the treatment and effect for ASD on children is limitted and the prognosis is poor,those children who suffered from ASD fail to get effective intervention and treatment,not only will their physical and mental health be seriously affected,but also huge burden will be imposed on their family as well as society.A rapid progress on epigenetics has been made in recent years,especially the deepening research on DNA methylation,which has identified that DNA methylation is an important reason for ASD production.It will provide help for the study of etiology of ASD and provide valuable information for the prevention and treatment of ASD if the DNA methylation genes and loci of ASD abnormalities are found and the molecular biological mechanism of DNA methylation of ASD and its influencing factors(especially environmental factors) are identified.
autism spectrum disorder; genetics; epigenetics;DNA methylation;environmental factors
孤独谱系障碍(ASD)是以社会沟通、交往和兴趣行为等表现偏离正常为共同特点的一组神经发育性障碍的统称.目前对儿童ASD的治疗方法和效果有限,且预后较差.ASD患儿如果没有得到有效的干预和治疗,不仅会严重影响患儿的身心健康,也会对其家庭和社会造成巨大的负担.近些年表观遗传学迅速进展,尤其是对DNA甲基化领域研究的逐步深入,已经确定DNA甲基化是ASD产生的重要原因之一.找到ASD异常的DNA甲基化基因和位点,明确ASD的DNA甲基化的分子生物学机制及其影响因素(尤其是环境因素)可以为ASD的病因学研究提供帮助,并对ASD的预防和治疗提供宝贵的资料.
孤独谱系障碍;遗传;表观遗传;DNA甲基化;环境因素
R96
A
2095-6894(2017)12-64-04
2017-06-07;接受日期:2017-06-20
浙江省医药卫生科技项目(2017207569)
赵袁志.硕士生.研究方向:儿童精神科.E-mail:1225316340@ qq.com
胡珍玉.教授,硕士生导师.研究方向:儿童精神科.E-mail:zhaoyz2017@ 163.com
