库尔勒香梨PsLOX基因克隆及生物信息学分析
2017-12-22刘旭新徐立生吐尔逊娜依
张 军 刘旭新 徐立生 吐尔逊娜依 李 疆
(1.新疆农业职业技术学院园林科技学院,昌吉 831100; 2.新疆喀什质量技术监督局,喀什 844000; 3.新疆农业大学林学与园艺学院,乌鲁木齐 830052)
库尔勒香梨PsLOX基因克隆及生物信息学分析
张 军1刘旭新1徐立生2吐尔逊娜依1李 疆3*
(1.新疆农业职业技术学院园林科技学院,昌吉 831100;2.新疆喀什质量技术监督局,喀什 844000;3.新疆农业大学林学与园艺学院,乌鲁木齐 830052)
克隆了库尔勒香梨(PyrussinkiangensisYü)脂氧合酶(lipoxygenase,LOX)基因PsLOX,了解其在香梨果实不同发育时期的表达差异,为香梨果实香气代谢机理研究提供理论依据。以库尔勒香梨嫩叶及不同时期果实表皮为试材,利用两种不同的方法提取总RNA,通过RT-PCR技术得到目的基因PsLOX的cDNA序列,以生物信息学方法对其进行分析和功能预测。运用半定量RT-PCR(SqRT-PCR)技术,分析PsLOX基因在香梨嫩叶及果实生长发育及货架期的表达特性和差异。结果:试剂盒提取总RNA质量较高,PsLOX基因CDS序列为912 bp,编码303个氨基酸,属于脂氧合酶家族基因,与其他植物LOX基因编码的氨基酸序列有较高的同源性,与南果梨相似性最高,达到99%;PsLOX基因在香梨果实中发育过程中表达差异明显,即生长发育前期表达量很低,成熟至完熟时期表达量最高,然后开始减少。推测克隆获得PsLOX基因在香梨果实香气代谢过程中起到重要作用。
库尔勒香梨;PsLOX基因;半定量RT-PCR;香气
果实香气是一类由大量挥发性芳香物质组成的复杂混合物,能够反映出果实的风味特点,是评价果实品质的重要指标[1~2]。库尔勒香梨(PyrussinkiangensisYü. cv.‘Kuerle xiangli’)属于新疆特有梨种,果实以独特而浓郁的芳香气味而著称[3]。栽培区域仅限新疆巴州和阿克苏地区,正常年份总产量约7.0×105吨[4],其规模效益和特色优势效益日益显著。随着产业的发展,香梨品质提升日益凸显。果实香气是构成和影响果实品质以及经济价值的重要因素之一[5]。怡人的果实香气可以吸引消费者并增强果品的市场竞争力。目前,香梨由于栽培管理不当、采摘期过早等原因致使其浓郁的香气在贮藏、运输及销售过程中丧失程度明显,这一问题降低了其商品价值和市场竞争力。研究香梨果实发育成熟过程中香气动态变化和代谢机理,对确定库尔勒香梨商品果的适贮采收时间和品质评价标准,厘清果实香气变化规律与调控的途径,研究果实香气调控应用技术等具有重要的理论和实践意义。
在苹果、桃等果实中直链芳香物质的生物合成物质己经被证实主要源于以亚油酸(linoleic acid,LA)和亚麻酸(linolenic acid,LeA)等为前体的脂氧合酶(lipoxygenase,LOX)途径[6]。目前,普遍认为脂氧合酶LOXs作为LOX途径的初始关键酶,通过断裂多不饱和脂肪酸(PUFAs)来形成C6醛类物质,然后通过醇脱氢酶将醛类物质转化成醇,继而通过醇酰基转移酶生成脂类物质[7]。研究表明,在成熟的苹果、草莓、甜瓜、番茄等果实香气中超过80%以上的成分都是脂类物质[8~9]。目前已在桃[10~12]、梨[13~15]中克隆得到了LOX基因并且进行了序列分析和表达特性的研究,尽管已有学者开始关注香梨的香气,但主要集中在果实香气成分等次生代谢产物分析方面[16~18],在调控香梨果实香气的分子机制,尤其是主效基因方面却从未涉及。本研究以库尔勒香梨为试材,通过RNA提取、RT-PCR等,获得一个完整的LOX基因cDNA的CDS序列,通过生物信息学技术对该基因CDS进行了序列和结构分析。并利用半定量PCR方法分析香梨在不同时期特异LOX基因的表达情况,以期为香梨LOX基因鉴定、调控方式、功能分析等提供基础信息,并为揭示库尔勒香梨香气代谢分子机制的提供参考。
1 材料与方法
1.1 材料
供试材料为库尔勒市沙依东园艺场梨园健康植株(树龄15年左右),采集嫩叶、果实等组织后立即进行液氮处理,运至实验室放置-80℃冰箱超低温保存备用。引物委托生工生物工程(上海)股份有限公司合成。
DEPC、Pfu DNA聚合酶、dNTPs、ddH2O、10×PCR buffer(含Mg2+),日本Takara生物公司;RNAprep Pure植物总RNA提取试剂盒(DP441 TIANGEN),感受态大肠杆菌DH5α菌株,零背景PLB-Vector载体等,北京天根生物科技公司;DNA Marker DL5000、Gelview、6×Loading Buffer,北京百泰克生物技术有限公司;AP-MN-P-250质粒小量DNA提取试剂盒,北京科恩堡生物科技有限公司;K1622反转录试剂盒,赛默飞世尔科技(Thermo)公司;MEGA-bead®DNA凝胶回收试剂盒,美国Invitrogen公司;TRIzol试剂,美国Life Technologies公司;琼脂糖,Biowest Agarose西班牙公司;其余试剂为国产分析纯或化学纯。
1.2 仪器与设备
5417R台式高速冷冻离心机,BioSpectrometer微量紫外可见光度计等德国Eppendorf有限公司;BioDoc-It凝胶成像系统,美国UVP公司;ABI2720型PCR扩增仪,美国应用生物系统公司;Cubis®MSA225P-100-DI天平德国赛多利斯公司等。
1.3 方法
1.3.1 库尔勒香梨总RNA提取及cDNA的合成
试验按照TRIzol Reagent(Ambion)提供方法提取库尔勒香梨嫩叶、嫩枝等总RNA,选择植物总RNA提取试剂盒(DP441)提供的方法对比,用紫外分光光度计检测总RNA浓度,电泳检测总RNA的完整性。提取较好的总RNA用K1622反转录试剂盒指导手册进行RT-PCR,反转录得到的cDNA用于下列试验,其余放置-25℃冰箱保存备用。
1.3.2库尔勒香梨PsLOX基因引物设计和PCR扩增
根据NCBI(www.ncbi.nlm.nih.gov)的GenBank核酸库根据已公布的砂梨LOX基因片段(Ge Bank Accession No.EF215448.1),经Blast比对后在脂氧合酶基因(LOX)mRNA序列同源性较高的区域,采用Primer Premier 6.0软件设计引物,引物序列为F:5′ACGATGAATGCAAACGCAT3′,R:5′TTAGATGTTGATACTGGTGGGAACT3′。该引物由生工生物工程(上海)股份有限公司合成。
PCR总反应体系为20 μL,其中DNA模板2 μL、引物2 μL、Pfu DNA聚合酶0.3 μL、dNTPs 2 μL、10×PCR buffer(含Mg2+)4 μL ddH2O 17.7 μL混匀后于96℃的条件下预变性5 min,96℃的条件下变性30 s,56℃条件下复性30 s,72℃的条件下延伸1 min,PCR反应共设置30个循环,最后在72℃的条件下延伸7 min,反应终止于14℃。
1.3.3 PCR产物回收、克隆及测序
PCR产物经过1.0%的琼脂糖电泳之后,检测到一条在1 kB大小的条带,用凝胶回收试剂盒纯化回收,克隆至无背景PLB-Vector载体,42℃条件下90 s热击转化感受态大肠杆菌DH5α后,在羧苄青霉素抗性筛选条件下,挑取单菌落经PCR鉴定后于LB培养基中摇菌12 h培养,用试剂盒提取质粒,送生工生物工程(上海)股份有限公司进行测序。
1.3.4 目的片段的序列比对、分析
测序结果使用Mega 6.0进行拼接对照,通过GENEDOC软件对部分已经公布的LOX开放阅读框进行比较,与植物LOX基因序列进行Blast比对,检测所克隆的片断是否为LOX基因序列,确认测序结果的正确性。
1.3.5 库尔勒香梨psLOX基因生物信息学分析
对扩增得到的基因序列使用Lasergene 7.1的EditSeq软件将PsLOX基因翻译成氨基酸序列,并与NCBI的Nr数据库进行BLASTp分析,获得相似度最高的序列,预测编码蛋白质;使用GENEDOC软件对预测的氨基酸序列与相似度最高的5条序列进行保守序列分析;使用MEGA 6.0软件将预测的氨基酸序列与GenBank上的其他序列进行系统进化分析,构建系统进化树;ExPASy在线服务器的ProtParam(http://web.expasy.org/protparam/)预测蛋白质的基本理化性质;PSORT服务器http://wolfpsort.org/)预测蛋白质的亚细胞定位;SOPMA软件http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page]=/NPSA/npsa_sopma.html)进行蛋白质二级结构分析;使用SWISS-MODEL(http://swissmodel.expasy.org/)进行三维结构预测。
1.3.6半定量RT-PCR检测库尔勒香梨psLOX基因表达
对香梨嫩叶(4月中旬)、不同时期果实的果皮(5月15日、6月16日、7月18日、8月20日,9月16日、10月17日、11月21日)进行RNA提取以及cDNA第一链反转录得到的cDNA用内标基因Actin-7进行扩增验证,内标引物序列为Actin-7F:5′TGGTGTCATGGTTGGTATGG3′,Actin-7R:5′CAGGAGCAACACGAAGTTCA3′。扩增表达引物分别为F:5′CTATCTCGGACAGCGTGACA3′,R:5′TACTGGTGGGAACTCCCTTG3′。
半定量RT-PCR之前,使用紫外检测仪检测RNA浓度,将其调成一致。总反应体系为20 μL,其中cDNA模板2 μL、引物2 μL、DNA聚合酶0.3 μL、dNTPs 2 μL、10×PCR buffer(含Mg2+)4 μL ddH2O 17.7 μL混匀后于96℃的条件下预变性5 min,96℃的条件下变性30 s,56℃条件下复性30 s,72℃的条件下延伸0.5 min,PCR反应共设置30个循环,最后在72℃的条件下延伸7 min,反应终止于14℃。产物取5 μL与缓冲液经1%琼脂糖凝胶电泳,Gelview染色,紫外观察照相。
2 结果与分析
2.1 试剂盒法提取库尔勒香梨总RNA质量较好
传统TRIzol法获得香梨组织总RNA溶液呈棕褐色并有粘稠状不溶物存在,试剂盒法获得的RNA溶液呈现乳白色。用两种方法提取香梨组织的总RNA,其BioSpectrometer紫外检测浓度和纯度结果差异较大:用传统TRIzol法提取香梨幼嫩组织的总RNA的到浓度185.1 μg·mL-1,纯度A260/A280为1.04,用RNAprep Pure试剂盒提取得在总RNA浓度为1 245 μg·mL-1,纯度A260/A280为1.96,A260/A230值为2.213,说明RNA纯度较高,质量较好,可满足后续工作的需要(图1)。
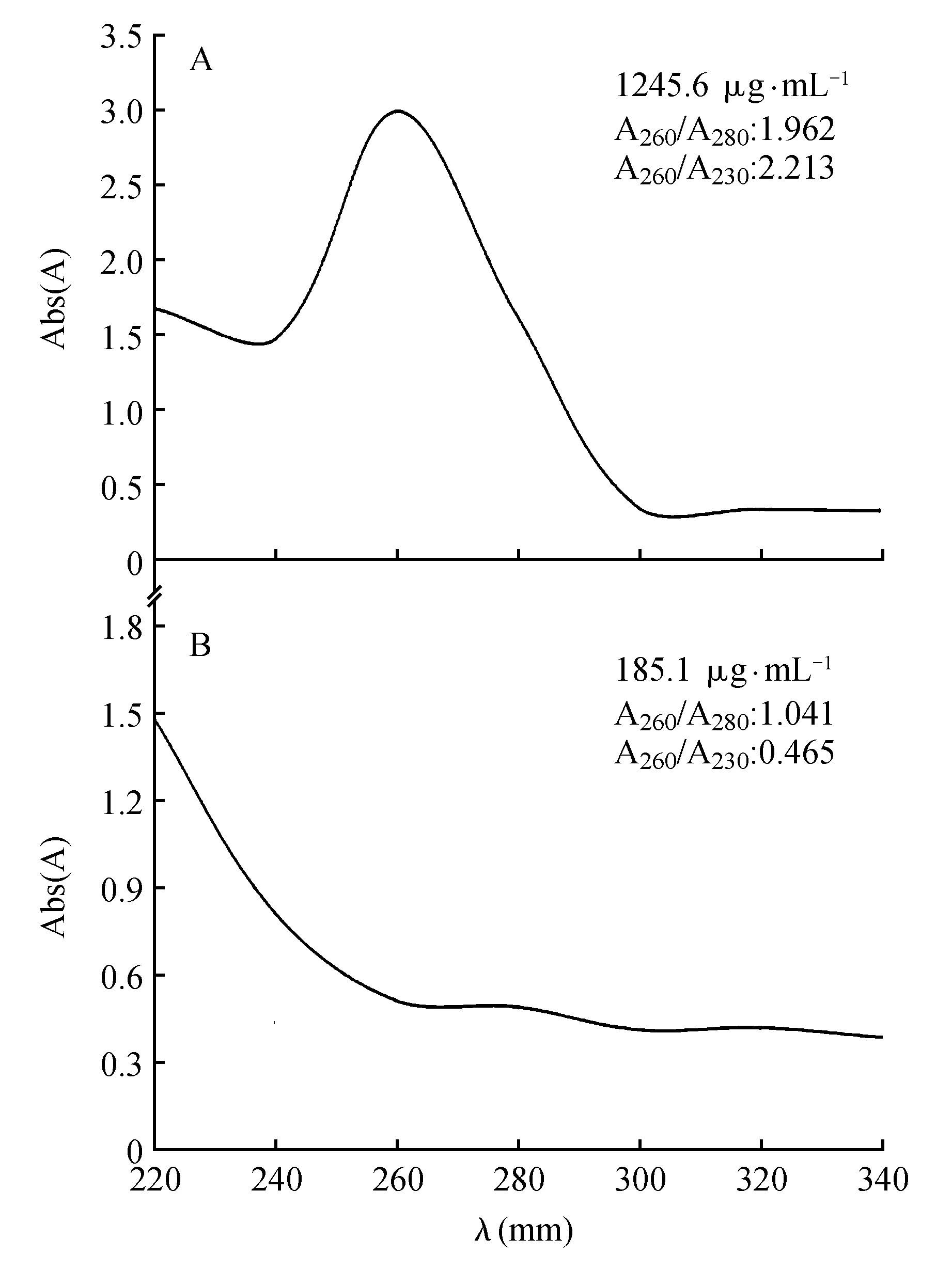
图1 紫外检测总RNA质量A.试剂盒法;B.传统TRIzol法Fig.1 Total RNA quality by UV detectionA. Kit method; B. Traditional TRIzol method
由于果树幼嫩组织中富含多糖、多酚[19],由图1可见,传统TRIzol法不适合该种类型。造成RNA提取质量较差的原因是果树幼嫩组织中的多糖理化性质与RNA相似,用乙醇或异丙醇沉淀RNA时,往往产生多糖的凝胶状沉淀,这种沉淀难溶于水[20],造成包裹部分RNA使其很难分离,多酚化合物与RNA形成不溶性复合物,造成提取总RNA的丢失[21]。A260/A280是核酸纯度的指示值,一般RNA样品的A260/A280的比值应为1.8~2.0。当A260/A280小于1.8时,表明有蛋白质、酚类等污染;当A260/A230值小于2.0时表明样品被糖类、盐类或者有机溶剂污染,RNA质量较差[22]。传统TRIzol法获得香梨组织总RNA溶液A260/A230值为0.465,小于2.0说明RNA被污染。
琼脂糖凝胶电泳检验总RNA完整性图谱由图2显示,试剂盒法提取的总RNA质量较好,呈现典型的28S、18S、5S三种带型,其条带清晰明亮、稳定,并且没有降解,基本可以满足实验的要求。而传统TRIzol法提取的总RNA没有任何条带显示,只是在5S条带下有模糊的核苷二聚体。
2.2 库尔勒香梨PsLOX基因的获得与序列分析
采用特异引物PCR扩增获得了与预期大小相符的特异条带,约为1 000 bp(图3A)。将PCR产物过大孔琼脂糖电泳,然后切胶用试剂盒处理得到浓度相对较高的目的基因(图3B)。将目的基因连接导入PLB-Vector载体,将转化的DH5α大肠杆菌平皿培养后,选择平板培养基上的24个单克隆菌落,利用特异引物对菌落进行PCR扩增。
结果表明,菌落单克隆2和8中的目标片段已经插入(图3C),将2和8单克隆菌落用液态LB培养过夜,提取重组质粒PCR鉴定电泳结果(图3D)。
质粒提取的结果符合预期,测序结果显示PsLOX基因区域全长1 256 bp,测序如图4。
测序结果使用primer premier 6.0进行拼接对照,结果显示该基因有912 bp的开放阅读框ORF(CDS),编码303个氨基酸,该基因开放阅读框序列如下图5所示。DNAman9.0预测结果为,分子重量34.446 8 kDa,等电点PI为4.83。
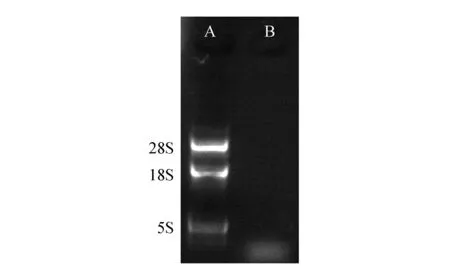
图2 电泳检测总RNA质量A.试剂盒法;B.传统TRIzol法Fig.2 Gel electrophoresis of Total RNAA. Kit method; B. Traditional TRIzol method
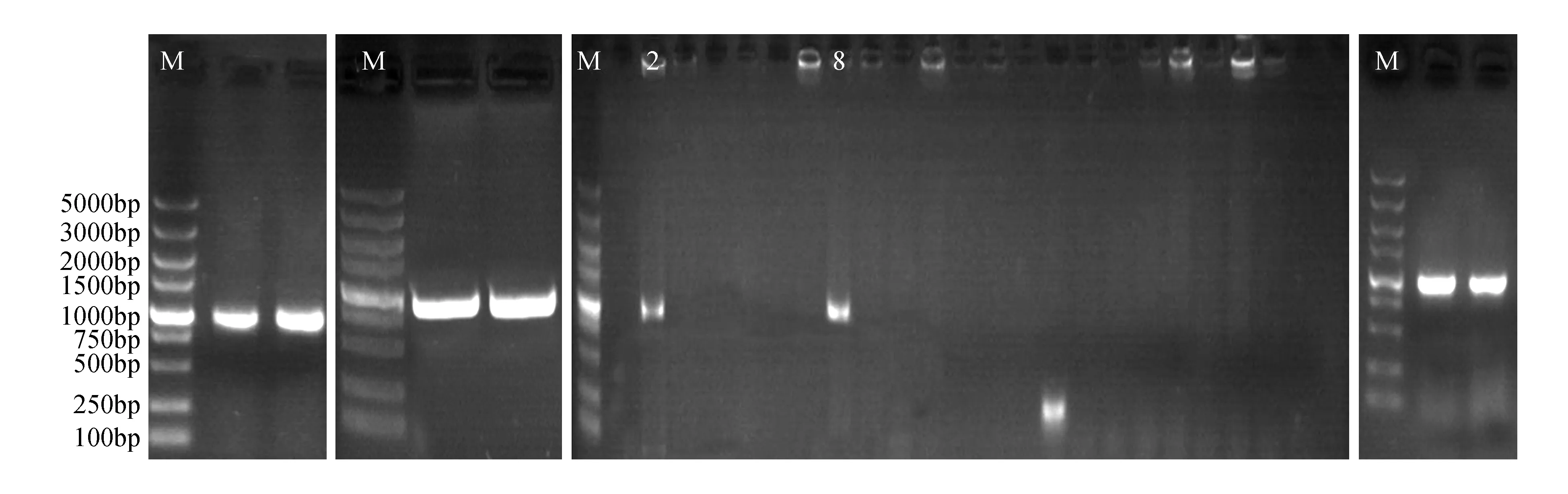
图3 库尔勒香梨PsLOX基因电泳 M. DNA Marker DL5000;2,8. 目标片段已经插入的单克隆菌落Fig.3 Electrophoresis to PsLOX gene of Korla pear M. DNA Marker DL5000;2, 8. Target fragments inserted into the monoclonal colonies
2.3 库尔勒香梨PsLOX基因氨基酸序列相似性与同源性分析
通过NCBI数据库BlasntX比对,该基因核苷酸与砂梨LOX基因的相似性为99%,且在LOX基因的CDS区段,有5个单核苷酸多态性(Single Nucleotide Polymorphisms SNP)位点,这为以后利用分子手段鉴定库尔勒香梨的提供重要依据。与南果梨、苹果、桃等LOX基因有较高的一致性,同源性80%~98%,因此认定该基因为库尔勒香梨特有LOX基因,命名为PsLOX基因。PsLOX基因核苷酸CDS序列对应氨基酸与其他植物LOX基因对应的氨基酸序列同源比对(图6)。
图4 库尔勒香梨PsLOX基因测序Fig.4 Sequencing to Korla pear PsLOX gene

图5 库尔勒香梨PsLOX基因CDS序列Fig.5 CDS sequence of Korla pear PsLOX gene

图6 库尔勒香梨PyLOX氨基酸序列与其它植物LOX氨基酸序列同源比对 库尔勒香梨PyLOX;梨XP009376684,XP018507432.1,XP009376681.1[Pyrus×bretschneideri];南果梨AJD18612.1[Pyrus ussuriensis];苹果AGI16416.1,GI16415.1,AGI16414.1,AGI16413.1[Malus domestica];杏ABZ05753.1[Prunus armeniaca];桃EMJ09301.1,ABV32552.1,ACH91370.1,[Prunus persica];扁桃CAB94852.1[Prunus dulcis];草莓CAE17327.1[Fragaria×ananassa];酸梅XP008246451.1,XP008245950.1[Prunus mume];猕猴桃ABF60001.1[Actinidia deliciosa];葡萄AGU28274.1[Vitis vinifera]Fig.6 homologous sequences comparison of Korla pear PsLOX amino acid and other plant’s Korla pear PyLOX ;pear XP009376684,XP018507432.1,XP009376681.1[Pyrus×bretschneideri];Nanguo Pear AJD18612.1[Pyrus ussuriensis];Apple AGI16416.1,GI16415.1,AGI16414.1,AGI16413.1[Malus domestica];Apricot ABZ05753.1[Prunus armeniaca];Peach EMJ09301.1,ABV32552.1,ACH91370.1,[Prunus persica];Almond CAB94852.1[Prunus dulcis];Strawberry CAE17327.1[Fragaria×ananassa];Dark plum XP008246451.1,XP008245950.1[Prunus mume];Kiwifruit ABF60001.1[Actinidia deliciosa];Grape AGU28274.1[Vitis vinifera]
利用MEGA6.0软件的最大似然法,将PyLOX基因氨基酸序列与GenBank上登录的不同物种的LOX酶氨基酸序列进行分子进化树分析,结果(图7)表明,库尔勒香梨PyLOX基因氨基酸序列与沙梨、南果梨亲缘关系相对最近,聚为一类,与苹果亲缘关系bootstrap值能到达100,单独聚为一簇。由此推测,PyLOX在表达、功能等方面与砂梨、南国梨等LOX家族成员类似。
2.4 库尔勒香梨PsLOX蛋白质结构与功能预测
通过ExPASy平台http://web.expasy.org/protparam/对PsLOX基因编码蛋白质进行理化性质分析,蛋白质分子式C1545H2366N406O469S10,氨基酸构成及其含量见下表1。
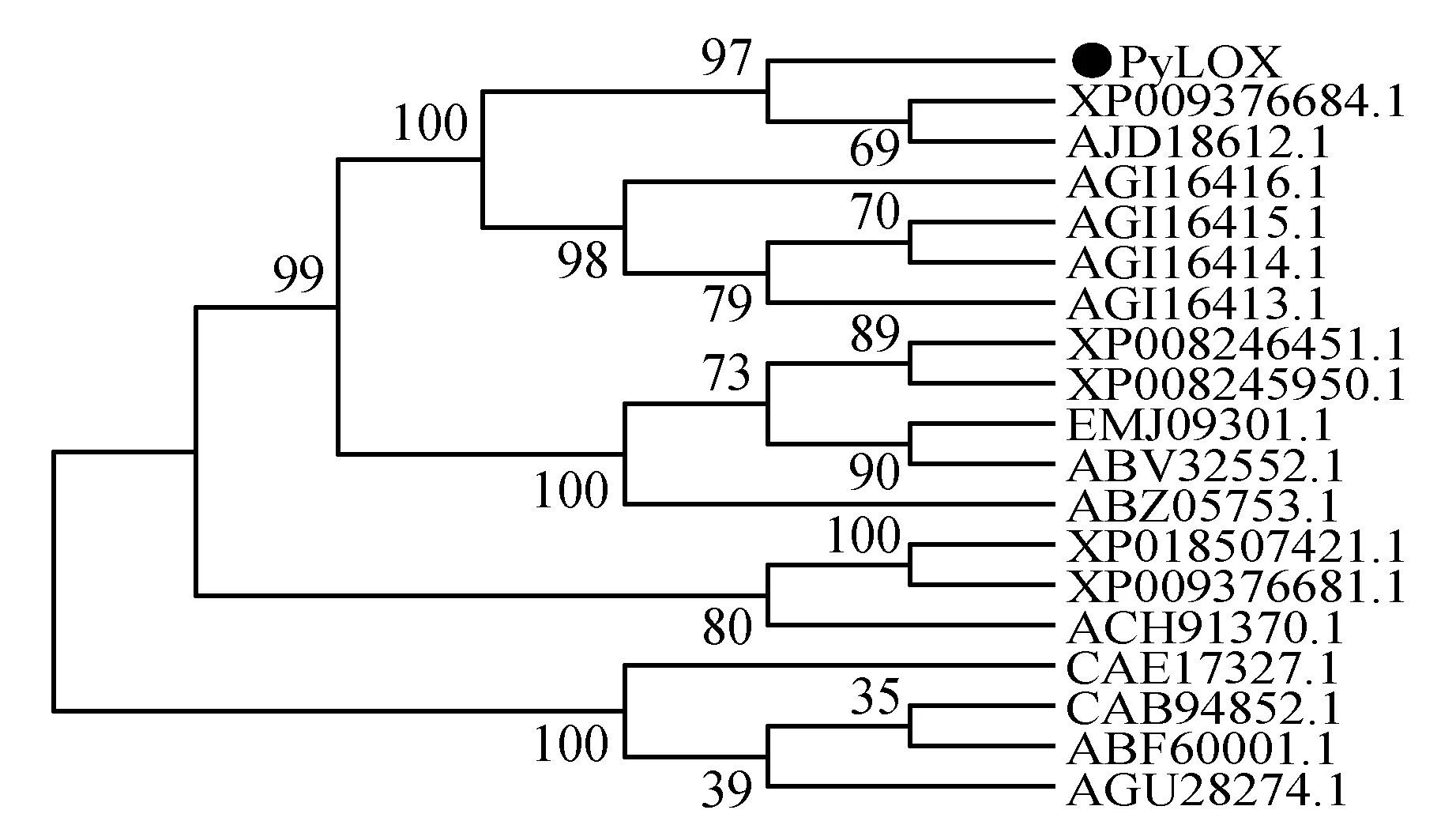
图7 库尔勒香梨PyLOX氨基酸序列与其它植物LOX氨基酸序列系统进化树Fig.7 Phylogenetic trees on LOX amino acids of Korla pear PsLOX and other plant’s

表1 PsLOX蛋白质氨基酸构成
该蛋白质总负电荷残基数(D+E)为45,总正电荷残基数(R+K)为33,PI为4.83,因此,为PsLOX基因编码蛋白质酸性蛋白质。估计半衰期在体外哺乳动物网织红细胞为30 h,在酵母体内大于20 h,在大肠杆菌体内大于10 h。不稳定系数为52.14,为不稳定蛋白。
氨基酸序列决定了蛋白质的立体结构,即肽链的折叠方式,因此研究肽链的构象有利于探索蛋白质的空间结构和功能。利用C.Geourjon,G.Deléage已经发表的蛋白质二级结构预测方法SOPMA[23],在线(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_sopm.pl)对PsLOX编码的蛋白质二级结构进行分析,结果如下图,该二级结构主要由4种形式组成,其中α-螺旋(Alpha-helix)占38.61%,无规则卷曲(Random coil)占37.62%,延伸链(Extended strand)占11.88%,β-转角(β-turn)占11.88%,α-螺旋和延伸链分别分布于蛋白的N端和C端各一个,N末端以α-螺旋的形式存在,C-末端以延伸链形式存在(图8)。
疏水/亲水平衡特征是维持蛋白质立体结构的重要因素,影响着蛋白质的诸多功能。因此,参照Kyte J.,Doolittle R.F.方法[24],利用ProtScale在线http://web.expasy.org/cgi-bin/protscale/protscale.pl对PsLOX进行了亲水性/疏水性分析,如图3所示,横坐标表示氨基酸残基的序号,纵坐标表示残基的疏水、亲水特性,正直为疏水,负值为亲水。发现PsLOX编码的氨基酸序列中,具有多个明显的亲水区域,其中269位亲水值为最大,达到3,而214,215位疏水性达到最大,均为1.711。大部分属于亲水性氨基酸,占整个比例的71.28%。因此,PsLOX基因翻译产物属于亲水性蛋白。

图8 库尔勒香梨PsLOX编码蛋白质二级结构预测 MNA等是氨基酸缩写;h.螺旋;e.延伸链;t.转角;c.卷曲Fig.8 The predicated secondary structure of Korla pear PyLOX encoding protein MNA etc. is the abbreviation for amino acids; hetc. Helix; e. Extended strand; t. Turn; c. Coil
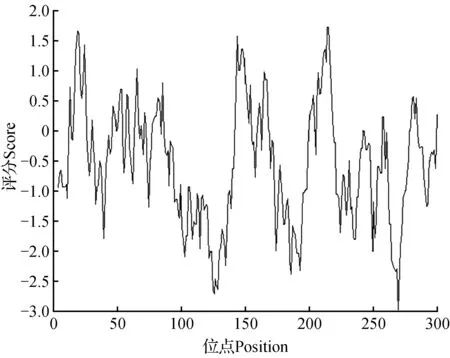
图9 PsLOX氨基酸亲水性/疏水性分析Fig.9 Distributing in hydropathy of the PsLOX amino acid
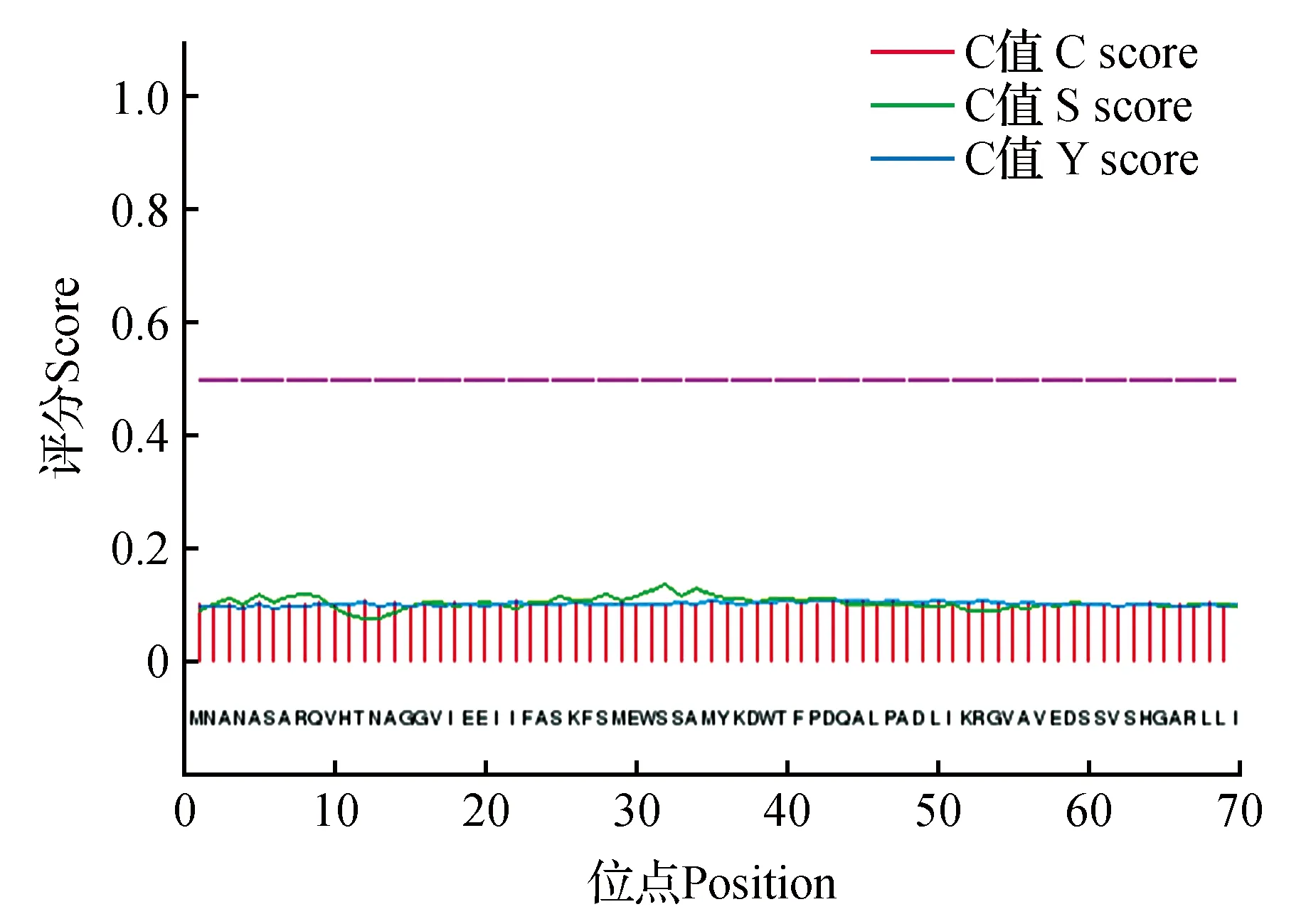
图10 PsLOX蛋白信号肽预测图Fig.10 Prediction to signal peptide of PsLOX protein
利用SignalP 4.1 Server软件,Petersen TN.等的方法[25]在线对PsLOX的蛋白质信号肽进行预测(http://www.cbs.dtu.dk/services/SignalP),选择神经网络法NN和隐马可夫模型HMM两种方式联合,可预测是否含有信号肽以及信号肽位点。信号肽辨别率D-score是S-mean和Y-max的平均值,是区分是否含有信号肽的重要指标。由图所示,D-score为0.109,小于临界值0.45,所以没有信号肽存在(图10)。
通过TMHMM Server2.0在线软件http://www.cbs.dtu.dk/services/TMHMM分析(图11),曲线的纵坐标是概率,横坐标是序列,一共203个氨基酸,红色表示跨膜区,几乎都在5%概率下,蓝色在膜内部,概率极低,相反紫色细线表示在膜外的概率,几乎100%。在线给出结论是:1到203氨基酸全部位于膜外,该蛋白质无跨膜区域,说明它可能位于胞质或核(胞浆)中。又通过http://www.csbio.sjtu.edu.cn/cgi-bin/PlantPLoc.cgi在线软件亚细胞定位[26],结果显示该蛋白在细胞质中(Cytoplasm)。
利用https://swissmodel.expasy.org/interactive/z6yrUY/models/同源建模得出PsLOX基因编码蛋白质的三维结构见图12。
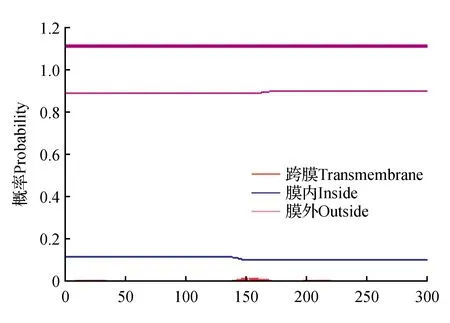
图11 PyLOX蛋白的跨膜区分析Fig.11 Transmembrane analysis of PyLOX protein

图12 PyLOX蛋白的三级结构Fig.12 The model of tertiary structure by PyLOX protein
2.5 库尔勒香梨PsLOX基因表达
以库尔勒香梨嫩叶、花后30天的果实8个时期的cDNA为模板,利用Actin-7基因为的引物进行半定量RT-PCR检测,琼脂糖电泳均能检测到符合预期大小的Actin-7基因片段,亮度基本一致,条带清晰,无非特异性扩增带出现(图13)。说明RT-PCR得到的模板cDNA浓度基本一致。从图13中可以看出,香梨PsLOX基因在库尔勒香梨成熟期(条带5~7)表现为的表达量大于成长期(条带2~4),而当香梨摘后常温贮藏至第二个月(条带8),psLOX表达明显减弱,条带变得模糊,由于PsLOX基因是脂类等次生代谢物的一个主要调控基因,故可初步得出:香梨PsLOX基因可能在成熟期时香梨香气合成代谢起到非常重要的作用。
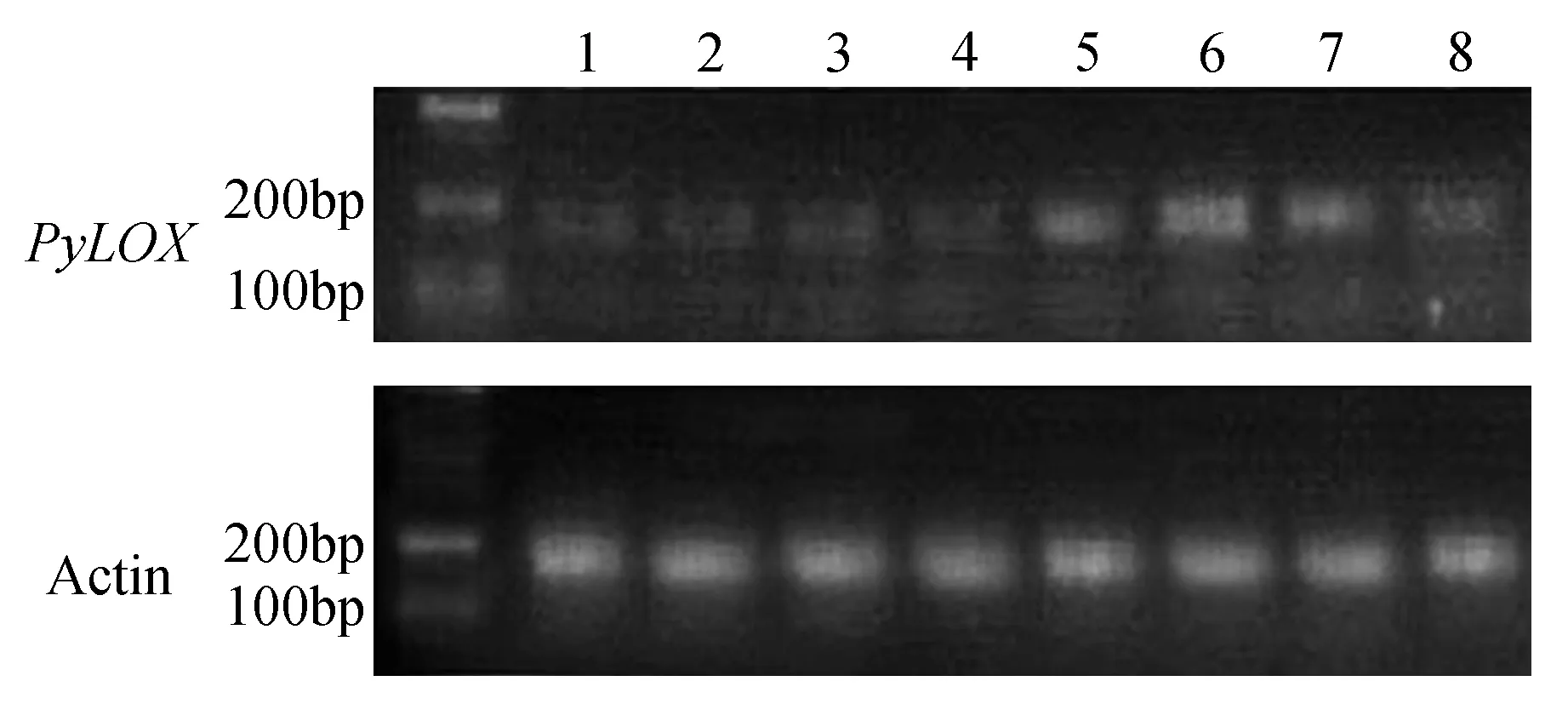
图13 PsLOX基因在不同时期的半定量RT-PCR表达 条带上方为试验材料序号:1.香梨果树嫩叶(4月中旬);2~8.香梨果实不同时期的果实(5月15日,6月16日,7月18日,8月20日,9月16日,10月17日,11月21日)Fig.13 Expression on semi-quantitative RT-PCR of PsLOX gene in different periods The test data number is above the strip: 1. Above as experimental materials for pear fruit leaves(mid April); 2-8. Fruit of Korla pear in different periods(May 15th,June 16th,July 18th,August 20th,September 16th,October 17th,November 21st)
3 讨论
提取得到纯度高、完整性好总RNA是进行RT-PCR分析和构建cDNA的决定性因素。目前,提取植物总RNA经典的方法有报道的多见于CTAB法、SDS-苯酚法、异硫氢酸胍法等,TRIzol法由于具有步骤简单、配制试剂少等优点[27]使用日益广泛,本文证明TRIzol法不适合提取库尔勒香梨材料总RNA,这与王其海[28]、尹珍珍[29]等对果树组织提取总RNA结论结果一致。
脂氧合酶(lipoxygenase,LOX)不但参与果实发育成熟,也是果实香气合成过程中关键酶,是果实香气(脂香型)代谢第一步,决定着果实香型和香气的质量。研究表明在草莓[30]、葡萄[31]、苹果[32]、桃[12]上都有LOX基因家族成员的报道,然而在中国特色水果库尔勒香梨果实上LOX基因却鲜有报道,只有李萌(音译)等[15]一篇关于白梨LOX基因家族与其他5种蔷薇科基因家族的对比,至于在庞大的LOX基因家族中哪个是和香气有直接关系的即主效基因未见报道。所以鉴于前人的研究成果,利用生物信息学对PyLOX对应的编码蛋白进行序列分析,与苹果、梨、桃等多个物种的LOX转录因子的保守结构域相似性较高,与已知功能的LOX转录因子聚在同类,据此推测它与该类基因可能具有相似的功能,即和果实香气代谢有关。
单核苷酸多态性(SNP)作为一种能够提供丰富信息的具有高稳定性的分子标记,已开始被应用于农作物育种领域研究[33]。通过PyLOX基因的同源性分析发现,在该基因的CDS区段,有5个SNP位点,这为以后利用分子手段鉴定库尔勒香梨的种资资源、性状关联等提供重要依据。
利用半定量RT-PCR技术分析基因的表达量,进而推断基因在不同生育时期的作用,已在很多研究上应用。由于不同组织或细胞等来源的目的基因表达量不同,在同一个PCR体系条件下扩增得到的产物(目的基因和管家基因)不同,而管家基因的表达则相同[33]。本试验采用Actin-7作为内参基因,研究香梨香气次生代谢物合成关键酶LOX基因,结果证明确实可行。同时,条带对比说明,LOX基因作为脂类次生代谢物的调控基因与库尔勒香梨香气代谢关系紧密。
1.段亮亮,郭玉蓉,池霞蔚,等.澳洲青苹果实不同部位香气成分差异分析[J].食品科学,2010,31(18):262-267.
Duan L L,Guo Y R,Chi X W,et al.Analysis of aromatic components in different tissue parts of granny smith apple[J].Food Science,2010,31(18):262-267.
2.El Hadi M A M,Zhang F J,Wu F F,et al.Advances in fruit aroma volatile research[J].Molecules,2013,18(7):8200-8229.
3.张军,刘建福,范勇,等.顶空固相微萃取—气相色谱—质谱联用分析库尔勒香梨花序香气成分[J].食品科学,2016,37(2):115-120.
Zhang J,Liu J F,Fan Y,et al.Analysis of aroma components ofPyrussinkiangensisYü.cv.‘Korla Xiangli’ inflorescence by headspace solid phase micro-extraction coupled to gas chromatography-mass spectrometry[J].Food Science,2016,37(2):115-120.
4.潘俨,孟新涛,车凤斌,等.库尔勒香梨果实发育成熟的糖代谢和呼吸代谢响应特征[J].中国农业科学,2016,49(17):3391-3412.
Pan Y,Meng X T,Che F B,et al.Metabolic profiles of sugar metabolism and respiratory metabolism of Korla pear(PyrussinkiangensisYu)throughout fruit development and ripening[J].Scientia Agricultura Sinica,2016,49(17):3391-3412.
5.关军锋.果品品质研究[M].石家庄:河北科学技术出版社,2001:7-8.
Guan J F.Fruit quality research[M].Shijiazhuang:Hebei Science and Technology Press,2001:7-8.
6.Zhang B,Xi W P,Wei W W,et al.Changes in aroma-related volatiles and gene expression during low temperature storage and subsequent shelf-life of peach fruit[J].Postharvest Biology and Technology,2011,60(1):7-16.
7.Fellman J K,Miller T W,Mattinson D S,et al.Factors that influence biosynthesis of volatile flavor compounds in apple fruits[J].HortScience,2000,35(6):1026-1033.
8.Dirinck P,De Pooter H,Schamp N.Aroma development in ripening fruits[M].//Teranishi R,Buttery R G,Shahidi F.Flavor chemistry.ACS symposium series.Washington:American Chemical Society,1989:23-34.
9.曹颖,胡尚连,张慧莹,等.番茄醇酰基转移酶基因SlAAT1克隆、序列分析和原核表达[J].植物研究,2012,32(6):731-736.
Cao Y,Hu S L,Zhang H Y,et al.Cloning,sequence analysis and prokaryotic expression of an alcohol acyltransferase(AAT) gene in tomato(Solanumlycopersicum)[J].Bulletin of Botanical Research,2012,32(6):731-736.
10.张涛,智军海,赵彩平,等.桃脂氧合酶基因(LOX)家族cDNA全长克隆与序列分析[J].农业生物技术学报,2009,17(6):1056-1061.
Zhang T,Zhi J H,Zhao C P,et al.Cloning and analysis of full-length lipoxygenase gene(LOX)family cDNA[J].Journal of Agricultural Biotechnology,2009,17(6):1056-1061.
11.田英.桃脂氧合酶(LOX-3)基因的表达分析[D].杨凌:西北农林科技大学,2011.
Tian Y.The expression analysis of geneLOX-3 in peach(PrunuspersicaL.)[D].Yangling:Northwest Agriculture and Forestry University,2011.
12.郭绍雷,张斌斌,马瑞娟,等.桃脂氧合酶基因家族生物信息学及其表达特性分析[J].果树学报,2016(3):257-267.
Guo S L,Zhang B B,Ma R J,et al.Bioinformatics analysis of the lipoxygenase gene family and their gene expression characteristics analysis in peaches[J].Journal of Fruit Science,2016(3):257-267.
13.胡钟东,章镇,乔玉山.砂梨LOX基因家族的克隆与序列分析[J].园艺学报,2009(S1):1886.
Hu Z D,Zhang Z,Qiao Y S.Cloning and sequence analysis ofLOXgene family in Pyrus[J].Acta Horticulturae Sinica,2009(S1):1886.
14.苏雅男.黄金梨脂氧合酶基因cDNA片段的克隆及表达分析[D].保定:河北农业大学,2012.
Su Y N.Molecular cloning and expression analysis of lipoxygenase gene form Whangkeumbae[D].Baoding:Agricultural University of Hebei,2012.
15.Li M,Li L T,Dunwell J M,et al.Characterization of the lipoxygenase(LOX) gene family in the Chinese white pear(Pyrusbretschneideri) and comparison with other members of the Rosaceae[J].BMC Genomics,2014,15(1):444.
16.陈计峦,周珊,王强,等.新疆库尔勒香梨的香气成分分析[J].食品科技,2007(6):95-98,103.
Chen J L,Zhou S,Wang Q,et al.Analysis of aroma components of Xinjiang Kuerle fragrant pear[J].Food Science and Technology,2007(6):95-98,103.
17.于建娜,刘文杰.库尔勒香梨果实香气成分的气相色谱—质谱分析[J].塔里木大学学报,2006,18(4):10-12,22.
Yu J N,Liu W J.Analysis in aroma components ofPyrussinkiangensisyü by gas chromatography-mass spectrometry[J].Journal of Tarim University,2006,18(4):10-12,22.
18.李芳芳,张虎平,何子顺,等.套袋对‘库尔勒香梨’果实糖酸组分与香气成分的影响[J].园艺学报,2014,41(7):1443-1450.
Li F F,Zhang H P,He Z S,et al.Effects of bagging on soluble sugars,organic acids,and aroma compounds inPyrussinkiangensis‘Korla Xiangli’ fruit[J].Acta Horticulturae Sinica,2014,41(7):1443-1450.
19.Japelaghi R H,Haddad R,Garoosi G.Rapid and efficient isolation of high quality nucleic acids from plant tissues rich in polyphenols and polysaccharides[J].Molecular Biotechnology,2011,49(2):129-137.
20.王艳,高疆生,金强,等.不同RNA提取方法在南疆特色果树上的应用[J].新疆农业科学,2015,52(3):461-466.
Wang Y,Gao J S,Jin Q,et al.Application of different methods for RNA extraction of five featured fruit trees in Southern Xinjiang[J].Xinjiang Agricultural Sciences,2015,52(3):461-466.
21.栾福磊,韩友生,邵毅,等.高效提取富含多糖多酚的果实组织中总RNA的方法[J].食品科学,2009,30(13):29-32.
Luan F L,Han Y S,Shao Y,et al.Effective isolation of high-quality total RNA from polysaccharide or polyphenol-rich fruits[J].Food Science,2009,30(13):29-32.
22.张富春,李江伟.分子生物学实验技术[M].乌鲁木齐:新疆大学出版社,2008.
Zhang F C,LI J W.Protocols in molecular biology[M].Urumqi:Xinjiang University Press,2008.
23.Geourjon C,Deléage G.SOPM:a self-optimized method for protein secondary structure prediction[J].Protein Engineering,1994,7(2):157-164.
24.Kyte J,Doolittle R F.A simple method for displaying the hydropathic character of a protein[J].Journal of Molecular Biology,1982,157(1):105-132.
25.Petersen T N,Brunak S,Von Heijne G,et al.SignalP 4.0:discriminating signal peptides from transmembrane regions[J].Nature Methods,2011,8(10):785-786.
26.Chou K C,Shen H B.Cell-PLoc:a package of web servers for predicting subcellular localization of proteins in various organisms[J].Nature Protocols,2008,3(2):153-162.
27.Simms D,Cizdziel P E,Chomczynski P.TrizolTM:a new reagent for optimal single-step isolation of RNA[J].Focus,1993:99-102.
28.王其海,王裔娜,刘晓华,等.桃不同发育时期叶片总RNA提取方法的比较[J].河南农业科学,2014,43(3):116-120.
Wang Q H,Wang Y N,Liu X H,et al.Comparative analysis of total RNA extraction methods for leaves of peach at different development stages[J].Journal of Henan Agricultural Sciences,2014,43(3):116-120.
29.尹珍珍,吴光斌,陈发河,等.莲雾果实高质量总RNA提取方法的建立[J].食品科学,2015,36(14):1-4.
Yin Z Z,Wu G B,Chen F H,et al.Comparison of extraction methods for high quality RNA from wax apple(SyzygiumsamarangenseMerr.et Perry)[J].Food Science,2015,36(14):1-4.
30.Pérez A G,Sanz C,Olías R,et al.Lipoxygenase and hydroperoxide lyase activities in ripening strawberry fruits[J].Journal of Agricultural and Food Chemistry,1999,47(1):249-253.
31.Podolyan A,White J,Jordan B,et al.Identification of the lipoxygenase gene family fromVitisviniferaand biochemical characterisation of two 13-lipoxygenases expressed in grape berries of Sauvignon Blanc[J].Functional Plant Biology,2010,37(8):767-784.
32.Vogt J,Schiller D,Ulrich D,et al.Identification of lipoxygenase(LOX) genes putatively involved in fruit flavour formation in apple(Malus×domestica)[J].Tree Genetics & Genomes,2013,9(6):1493-1511.
33.李兆波,吴禹,王岩,等.SNP标记技术及其在农作物育种中的应用[J].辽宁农业职业技术学院学报,2010,12(3):8-9.
Li Z B,Wu Y,Wang Y,et al.On the SNP technology and its application in crops breeding[J].Journal of Liaoning Agricultural College,2010,12(3):8-9.
Xinjiang Uygur Autonomous Region Natural Science Foundation(2016D01A053)
introduction:ZHANG Jun(1978—),Male,associate professor,Ph.D.Candidate,research on germplasm resources of fruit trees.
date:2017-05-08
CloningandBioinformaticsAnalysisofPsLOXGeneinPyrussinkiangensisYü. ‘Korlaxiangli’
ZHANG Jun1LIU Xu-Xin1XU Li-Sheng2Tuerxunayi1LI Jiang3*
(1.Garden Science & Technology College,Xinjiang Agricultural Vocational Technical College,Changji 831100; 2.Xinjiang Kashgar Supervisory Bureau for Quality and Technology,Kashgar 844000; 3.College of Forestry and Horticulture,Xinjiang Agricultural University,Urumqi 830088)
In order to provide a rationale and study for the aroma metabolism mechanism of pear fruit, which wasPsLOX, the lipoxygenase gene of Korla pear(PyrussinkiangensisYü. ‘Korla xiangli’) was cloned, the expression profiles ofPsLOXwere monitored in different periods of fruit development. As materials in leaves and fruit of Korla pear at different periods, total RNA was extracted by using the reagent kit, cDNA sequence of target genePsLOXwas obtained by RT-PCR. The function ofPsLOXwas predicted by bioinformatics, the expression characteristics and differences of this gene were analyzed using semi quantitative RT-PCR(SqRT-PCR) technology with leaves and peel of Korla pear in period of growth and shelf-life. The CDS sequence ofPsLOXgene was 912 bp length, coded a predicted protein of 303 amino acids. By bioinformatics,PsLOXgene belongs to the lipoxygenase family. We compared with other plant LOX genes, the amino acid sequence had high homology, and the highest homology with Nanguo pear reached 99%. There was significant difference in the expression ofPsLOXgene in development period of pear fruit, which was very low level in the expression quantity at growth development prophase of young fruit, and was the highest value in mature to ripe period, then expression quantity began to decrease. ThePsLOXgene was homologously cloned, which were speculated to play a crucial role in aroma metabolism process of Korla pear fruit.
PyrussinkiangensisYü. ‘Korla xiangli’;PsLOXgene;semi quantitative RT-PCR;aroma
新疆维吾尔自治区自然科学基金项目(2016D01A053)
张军(1978—),男,副教授,博士研究生,主要从事果树种质资源方面的研究。
* 通信作者:E-mail:lijiangxj@163.com
2017-05-08
* Corresponding author:E-mail:lijiangxj@163.com
S661.2
A
10.7525/j.issn.1673-5102.2017.06.011
