基于转录组数据的杜仲红叶性状SSR分子标记分析
2017-12-22苗作云刘攀峰郑新芳杜红岩朱景乐
苗作云 杨 赟 刘攀峰 郑新芳 杜红岩 朱景乐*
(1.中国林业科学研究院经济林研究开发中心,郑州 450003; 2.黄河科技学院,郑州 450063; 3.河南农业大学,郑州 450002; 4.华南农业大学,广州 510642)
基于转录组数据的杜仲红叶性状SSR分子标记分析
苗作云1,2杨 赟3刘攀峰1郑新芳4杜红岩1朱景乐1*
(1.中国林业科学研究院经济林研究开发中心,郑州 450003;2.黄河科技学院,郑州 450063;3.河南农业大学,郑州 450002;4.华南农业大学,广州 510642)
对‘华仲12号’杜仲的幼嫩叶片(绿色)和成熟叶片(红色)及‘华仲11号’杜仲成熟叶片(绿色)进行转录组测序,进行测序数据的拼接和组装,且对转录组获得的基因(Unigenes)进行SSR分析。研究得到54 517条平均长度为806.90 bp的Unigenes,其中25 993条Unigenes在Nr、Swiss-Prot、KEGG和COG蛋白数据库获得功能注释,占所有Unigenes的47.68%。参照KEGG数据库,可将注释到的6 910条Unigenes划分到122个代谢途径分支,其中花色苷代谢途径相关酶基因39个,类黄酮代谢途径38个,类胡萝卜素合成途径34个。54 517条Unigenes中共包含17 010个完整型SSR位点,占总SSR位点的96.28%。完整型SSR位点共包含67种重复基元,其中出现频率最高的重复基元类型为单核苷酸重复中的A/T(7 747个),其次是AG/CT(5 039个)和AT/AT(850个)从花色苷代谢途径、类黄酮代谢途径及类胡萝卜素代谢途径中共找到13个SSR位点。为今后杜仲遗传多样性分析、遗传图谱构建及杜仲红叶性状分子标记开发等方面奠定了分子基础。
杜仲;红叶;微卫星;转录组;高通量测序
杜仲(EucommiaulmoidesOliv.)属杜仲科(Eucommiaceae),单科单属单种,是中国重要的名贵经济树种,雌雄异株,风媒花。其在我国27个省(区、市)均有分布,不但是中国分布最广的胶源植物和名贵中药材[1],还是一种理想的城市绿化和庭院观赏树种[2]。‘华仲12号’杜仲是中国林科院经济林中心审定的观赏与药用价值兼备的杜仲良种,雄株,花量大,叶片呈紫红色[3~4]。除了‘华仲12号’杜仲外,尚有一些关于红(紫)叶性状的报道[5~6],且这些报道称发现红叶性状单株的位置距离很远,陕西和湖南都有发现[7~8],随着对杜仲红叶性状的关注,不断有新的具有该性状的杜仲单株被发现。但所报道的具备红叶性状的资源亲缘关系如何,其是在各自群体发生基因突变?亦或通过种子异地调运等方式将携带的遗传信息带到其他省份等原因尚未可知,目前尚无关于如何鉴定这些红叶单株亲缘关系的报道,亦未见关于红叶性状分子标记开发的报道。
微卫星分子标记又称 SSR 分子标记技术,因其具备多态性高、共显性和特异性强等优点,在遗传多样性分析、DNA指纹图谱的构建、基因定位、分子标记辅助育种等方面得到广泛应用[9~10]。随着转录组测序技术的成熟及成本降低,根据生物的转录组序列开发SSR标记日益受到研究者青睐,其不但具有时空特异性,且开发的分子标记与某一性状连锁性强,从而使其在用于植物遗传多样性和分子标记辅助育种研究的准确性更高[11]。
本研究拟在‘华仲12号’杜仲的幼嫩叶片和成熟叶片及‘华仲11号’杜仲成熟叶片6个样品转录组测序的基础上,采用生物信息学方法对拼接组装后的微卫星特征分析等,为开发杜仲叶片的红叶性状分子标记奠定理论基础。
1 材料与方法
1.1 研究材料
本研究以1年生‘华仲12号’杜仲和‘华仲11号’杜仲嫁接苗为对象,从中选择‘华仲12号’杜仲幼嫩叶片(呈绿色,编号H1),‘华仲12号’杜仲成熟叶片(呈红色,编号H3)及‘华仲11号’杜仲成熟叶片(呈绿色,编号L4),将3个叶片混合成一个样品,生物学重复2次,共计6个样品,样品编号方法为取样状态—重复号,如H1-1即为‘华仲12号’杜仲幼嫩叶片的第一个生物学重复,依次类推。
1.2 总RNA提取
6个样品分别提取总RNA,使用Trizol Kit(Promega,USA)试剂盒提取RNA,并对通过Agilent 2100 Bioanalyzer检测RNA浓度及峰值,判断所得RNA是否达到RNA-Seq的试验标准(总量≥20 ng、质量浓度≥400 ng·μL-1、OD260/280为1.8~2.2、RNA integrity number值≥8、28S/18S>1.0)。
1.3 cDNA文库的构建
各样品分别构建cDNA文库,通过Agilent 2100仪器检测文库的长度,并用荧光定量PCR仪测定文库的浓度,合格的文库用Illumina HiSeq2000分别进行测序。
1.4 原始数据处理及序列组装
对原始测序数据进行过滤后(去除含adaptor的reads、N的比例大于10%的reads及低质量reads(质量值Q≤5的碱基数占整个read的50%以上)),采用Trinity(20140717)软件对所得数据进行混合从头组装,得到杜仲叶片的Unigenes序列。
1.5 Unigenes的功能注释、功能分类和代谢通路分析
通过blastx(2.2.29+)将Unigenes序列比对到蛋白数据库Nr、Swiss-Prot、KEGG和COG(E-value<0.000 01),获得Unigenes在4大数据库中的功能注释。根据Nr注释信息,使用Blast2GO(2.3.5)软件得到Unigenes的GO注释信息[12]。然后用WEGO软件对所有Unigenes做GO功能分类统计[13]。根据Unigenes的kegg功能注释结果,对所有的Unigenes进行代谢通路分析[14]。
1.6 SSR位点分析
采用MISA(version 1.0)软件(Microsatellite identificantion tool,http://pgrc. ipk-gatersleben .de /misa)搜索杜仲叶片转录组中Unigenes的简单重复序列SSR[15],并对SSR重复基元类型进行特征分析。查找标准为单核苷酸基元最少重复10次,而二、三、四、五和六核苷酸基元则最少重复6、5、4、4和4次。
2 结果与分析
2.1 RNA质量检验
RNA浓度及峰值的Agilent 2100检测结果见图1,所提RNA条带清晰,浓度、RIN值及RNA总量均达到测序标准,具有明显的检测峰,符合RNA测序的需求,可直接进行下一步测序。
2.2 测序结果与组装
对杜仲叶片RNA测序后共得到331 016 720条clean reads,GC含量为50.68%,Q20值为96.03%,各项指标均符合转录组测序的要求。利用Trinity软件[16]对样品数据进行组装共得到54 517条Unigenes,组装的N50为1 377 nt,平均长度为806.90 nt,组装效果较好。其中GC含量为42.17%,长度为200~300 nt的序列最多,有15 902条,其次为300~400和400~500 nt的序列,具体的Unigenes的长度分布见图2。

图2 杜仲叶片的Unigene长度分布图Fig.2 Unigene length distribution of E.ulmoides ‘Hongye’

图3 四大数据库注释维恩图Fig.3 Four database notes Venn diagram
2.3 Unigenes功能注释
利用BLAST软件[17]将Unigenes序列分别与NR、GO、COG和KEGG数据库比对分析,后统计每个库上注释到的Unigenes数目。统计结果显示共有25 993条Unigenes得到注释,28 524条Unigenes未得到注释,注释比例仅为47.68%,其中Nr数据库中注释的有25 929条,在Swissprot数据库中注释到的有18 070,在COG数据库中注释到的有8 674条,在KEGG数据库中注释到的有6 910条。上述基因中有27 732条基因能编码蛋白序列,占总Unigenes数量的50.87%。
将所有Unigenes在Nr、Swiss-Prot、KEGG和COG四大数据库中的最佳比对结果的比对E值进行统计,将其分为6个范围,并统计每个范围内的基因个数比对E值(Unigenes与数据库中匹配序列为同源序列的假阳性概率),具体信息见图4,从中可知Nr数据库中信息更加可靠。

图4 Unigene注释结果分别在4大数据库中的E值分布图Fig.4 The distribution of the E values in the 4 databases

图5 物种分布统计图(只展示前十种)Fig.5 Species distribution map (showing only the first ten)
利用blastx将组装出来的Unigenes序列与Nr数据库进行比对后,取每个Unigenes在Nr库中比对结果最好(E值最低)的那一条序列为对应同源序列(如有并列,取第一条),确定同源序列所属物种,统计比对到各个物种的同源序列数量见图5,同源基因比对得到杜仲中有6 032条Unigenes与葡萄(Vitisvinifera)数据库中信息接近,其Unigenes数量占Nr数据库注释的总Unigenes数量的23.26%,说明与葡萄属的亲缘关系更近,其次是可可树(Theobromacacao)和番茄(Solanumlycopersicum),接近的Unigenes数量分别为4 535条和3 638条,所占的比例分别是17.49%和14.03%。在后期的研究中可优先参考这几个物种的遗传信息。
2.4 Unigenes功能分类
2.4.1 Unigenes的GO分类
为了进一步研究Unigenes的功能,将所得Unigenes向GO数据库的各个term进行映射,发现共有12 421个Unigenes在生物过程(biologicalprocess)、细胞组分(cellularcomponent)和分子功能(molecularfunction)分类中得到了GO注释。其中,在生物过程中,涉及代谢过程(metabolic process)、细胞过程(cellular process)和刺激响应(response to stimulus)的Unigenes数较多,分别占GO注释Unigenes数的52.20%(6484 Unigenes)、48.59%(6 035 Unigenes)和19.31%(2 399 Unigenes);在细胞组分中,涉及细胞(cell)、细胞部分(cell part)和细胞器(organelle)的Unigenes较多,分别占GO注释Unigenes的55.29%(6 868 Unigenes)、55.29%(6 868 Unigenes)和38.63%(4 799 Unigenes);在分子功能中,涉及催化活性(catalytic activity)和结合(binding)的Unigenes较多,分别占GO注释Unigenes的52.17% (6480 Unigenes)和47.37%(5 884 Unigenes)。
2.4.2 Unigenes的COG分类
利用COG数据库比对分析共得到13434条杜仲Unigenes的同源序列,它们分为25个类别(图7),其中复制、重组和修饰(1 342,9.99%)、转录(1 238,9.22%)和信号转导(1 077,8.02%)等与生物过程相关的Unigenes数量最多;其次是与功能预测类别相关的Unigenes基因(2 438,18.15%);此外,与代谢相关的Unigenes基因数量为2 931条,占总Unigenes数量的21.82%。
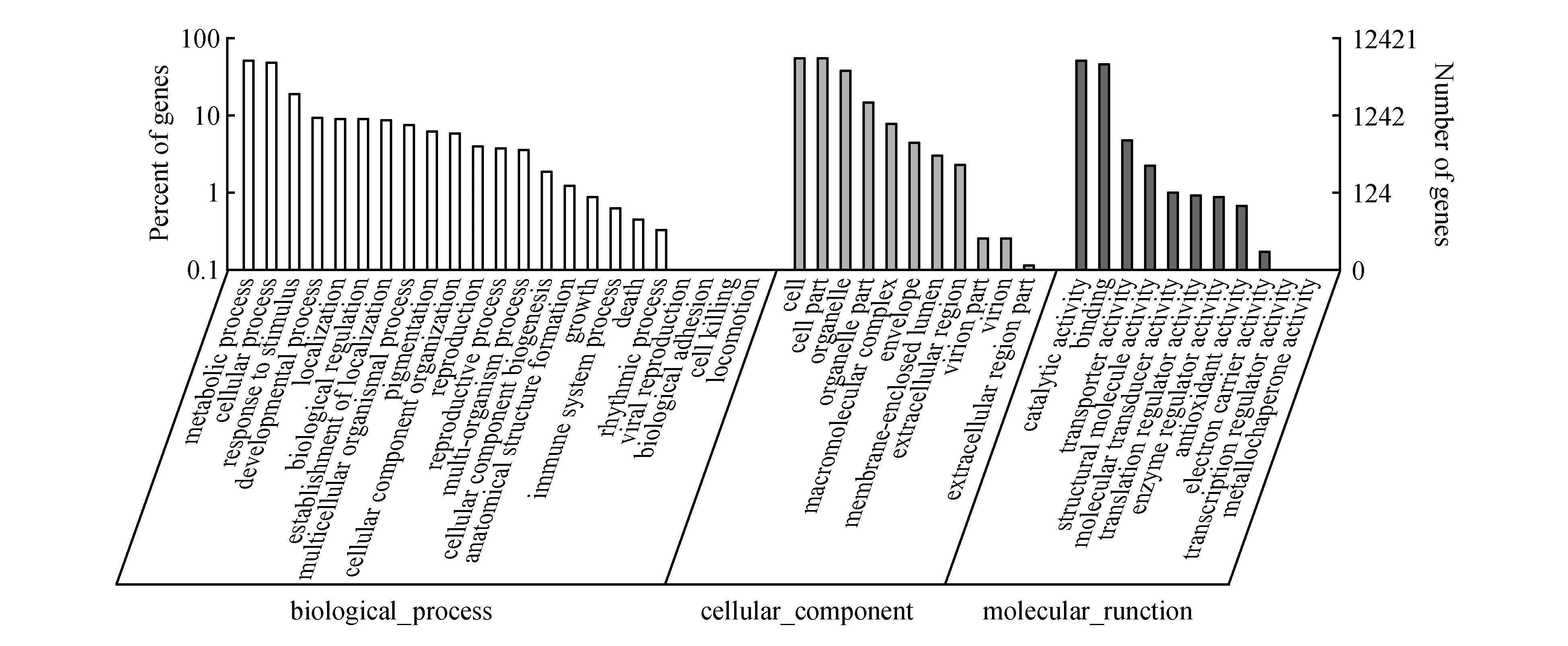
图6 GO功能显著性富集分析Fig.6 Enrichment analysis of GO function

图7 COG功能分类图Fig.7 COG function classification
2.4.3 KEGG pathways分析
根据KEGG数据库注释信息及对非冗余Unigenes的pathway进行富集性分析,共识别出6 910条Unigenes,可归为122个信号途径。与植物叶片颜色及叶片生长直接相关的代谢途径(注释的Unigenes数量)分别有类黄酮代谢途径(38条)、光合作用途径(97条)、光合生物中的碳固定途径(89条)、叶绿素代谢途径(53条)、光合作用—天线蛋白合成途径(43条)、花色苷代谢途径(39条)及类胡萝卜素的生物合成途径(34条)等。

表1 杜仲转录组的KEGG代谢途径分类图(部分)
2.5 微卫星特征分析
2.5.1 微卫星的种类和数量分布
利用MISA软件对杜仲转录组的54 517条Unigenes(总长度为43 989 941 bp)进行SSR搜索,在54 517条Unigenes中共发现17 667个SSR位点(其中包含657个混合型的SSR和17 010个完整型SSR位点),其中包含2个及以上SSR位点的Unigenes共有3 121条。其发生频率(即含SSR位点的Unigenes占总Unigenes的百分比)为23.64%,平均每2.5 kB就出现一个SSR位点,其出现频率(即检测出的SSR位点个数占总Unigenes的百分比)为32.41%。
完整型SSR位点共有17 010个,占总SSR位点的96.28%(图8)。完整型SSR位点共包含67种重复基元,二核苷酸至六核苷酸分别有4、10、14、16、21种(表2)。其中,完整型SSR重复基元的重复次数均在4~27次,其中以重复10次(3 685个)的最多,占总SSR的20.86%;其次为重复次数6、7、11、5和8次,其SSR位点个数分别为3 115、1 864、1 766、1 412和1 198。重复5~10次的SSR位点有12 144个,占总SSR的68.74%;重复11次以上的SSR位点有4 866个,占27.54%。
表2杜仲SSR中6种重复基元的类型及频率
Table2SixtypesofSSRrepeatmotifsandtheirfrequencyinE.ulmoides

重复基元类型Repeatmotif重复次数Repeatnumber567891011>11总计Total频率Frequency(%)A/T311116143022774745.54C/G6843711821.07AC/GT34518412281712938354.91AG/CT18271175908691369672503929.62AT/AT32321513895661218505CG/CG2194340.2AAC/GTT812810111210.71AAG/CTT42420897217324.3AAT/ATT83442531550.91ACC/GGT153844142821.66ACG/CGT561562790.46ACT/AGT3094430.25AGC/CTG1013315111510.89AGG/CCT1637941712911.71ATC/ATG100441841660.98CCG/CGG12233181731.02AAAC/GTTT82100.06AAAG/CTTT92110.06AAAT/ATTT214250.15AACC/GGTT220.01AAGG/CCTT1120.01AATC/ATTG2130.02AATG/ATTC110.01AATT/AATT110.01ACAT/ATGT92110.06ACCG/CGGT110.01ACTC/AGTG1120.01AGAT/ATCT741120.07AGCG/CGCT330.02AGGG/CCCT8190.05AAAAC/GTTTT110.01AAACC/GGTTT110.01AAAGG/CCTTT110.01AAAGT/ACTTT110.01AAATT/AATTT110.01AACCT/AGGTT110.01AAGAC/CTTGT110.01AAGAT/ATCTT110.01AAGTG/ACTTC110.01ACCCC/GGGGT110.01ACCCT/AGGGT110.01ACCTC/AGGTG110.01AGCAT/ATGCT110.01AGGAT/ATCCT110.01AGGCG/CCTCG110.01AGGGG/CCCCT110.01AAAAAC/GTTTTT110.01AAAACG/CGTTTT110.01AAAGTG/ACTTTC110.01AAATTG/AATTTC110.01AACAGC/CTGTTG110.01AACCCT/AGGGTT110.01AACTTC/AAGTTG110.01AAGCAC/CTTGTG110.01AAGGGC/CCCTTG110.01AAGTAG/ACTTCT110.01AAGTGG/ACTTCC110.01AATCAC/ATTGTG110.01ACACCG/CGGTGT110.01ACACGC/CGTGTG110.01ACCAGC/CTGGTG110.01ACCATC/ATGGTG110.01ACCTCC/AGGTGG110.01AGAGGC/CCTCTG110.01AGAGGG/CCCTCT110.01AGCATC/ATGCTG110.01AGGGCG/CCCTCG110.01总计Total141231151864119887036851766310017010100频率Frequency(%)8.318.3110.967.045.1121.6610.3818.22100
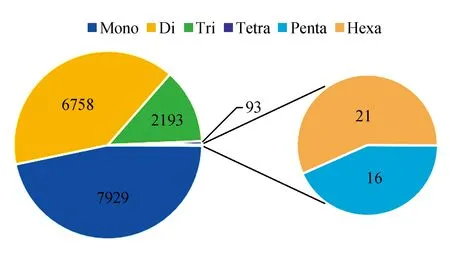
图8 杜仲完整型SSR种类和数量分布图Fig.8 Types and numbers of complete SSR in E.ulmoides
2.5.2 微卫星不同重复基序的频率
杜仲叶片转录组中完整型SSR种类丰富(图8),单核苷酸到六核苷酸的重复类型都存在,但其出现频率差异较大。其中以A/T类型为主的单核苷酸重复所占的比例最高(7 747个,45.54%);其次是以AG/CT类型为主的二核苷酸重复(5 039个,29.62%);再次是以AAG/CTT类型为主的三核苷酸重复(732个,4.3%),四核苷酸重复类型为93个,以AAAT/ATTT类型为主;五和六核苷酸重复基元的类型较多,但数量都很少,出现频率均较低。对检测到的SSR位点分析显示,出现频率最高的5类基序依次为:A/T(4 597个,47.78%)、AG/CT(5 039个,29.62%)、AT/AT(850个,5.00%)、AC/GT(835个,4.91%)和AAG/CTT(732个,4.30%)(表2)。
杜仲2种单碱基微卫星重复基元的统计结果显示:A/T为主要的重复基元,共有7 747个,占45.54%,C/G仅占1.07%(图9A)。二碱基重复基元有4种(AC/GT、AG/CT、AT/AT、CG/CG),其中AG/CT的重复基元数量最多(5 039个),占总体的29.62%;其次是AT/AT(850个)和AC/GT(835个),分别占5.00%和4.91%。最少的是CG/CG(34个,0.2%)(图9(B))。三碱基重复类型共有10个,AAG/CTT的数量最多,共732个,占总体的4.30%;其次是AGG/CTT(291个)和ACC/GGT(282个),分别占1.71%和1.66%;再次是CCG/CGG(173个,1.02%)、ATC/ATG(166个,0.98%),其他重复类型则相对较少(表2)。在14种四碱基重复类型中,AAAT/ATTT的数量最多(25个),占总体的0.15%;其次是AGAT/ATCT(12个,0.07%);再次是AAAG/CTTT、ACAT/ATGT,均占0.06%。五碱基和六碱基重复类型分别有16和21种,但每种类型均只有1个。
表3是杜仲叶片颜色形成相关基因的SSR重复基元信息,我们从所有的SSR重复信息中找出来与类黄酮代谢途径有关的2条,与花色苷合成途径有关的Unigenes 2条,与类胡萝卜素合成的Unigenes 8条,并从中找到13个SSR位点。利用找出来的这13个位点可以为进一步开发杜仲叶片红色性状的分子标记做准备。

表3 杜仲叶片颜色形成相关基因的SSR重复基元信息

图9 单碱基和二碱基微卫星的频率Fig.9 Frequency of mononucleotide and dinucleotide repeat SSRs
3 讨论
转录组测序技术(RNA-seq)是近期兴起的一种新技术,其灵敏度高,且利用其研究转录组时对材料的遗传背景要求低,较常规方法具有明显的优势[18]。由于杜仲是单科单属单种,关于杜仲转录组测序方面的研究报道较少。李铁柱、赵德刚、冯延芝等分别构建了杜仲叶片、果实、芽及树皮等器官在不同发育时期的转录组数据库,并对杜仲转录组数据中相关基因功能、代谢通路等进行了初步研究[19~22]。相比前人的研究,本次转录组测序得到54 517条Unigenes。由于本研究仅选用‘华仲12号’品种不同时期及‘华仲11号’的叶片,而前人的研究均为多器官的混合样品,所以得到的Unigenes数量虽少,但组装Unigenes的N50为1 377 nt,平均长度806.90 nt,碱基Q20均在95%以上,组装效果更好,后期的分析也更加可靠。
随着转录组测序技术的成熟,通过其开发分子标记及后期基因功能研究也成为一种主要的方式。黄海燕、吴敏等在转录组测序的基础上进行相关SSR分子标记的开发,并对其遗传多样性、亲缘关系、群体遗传学、性别鉴定等方面开展了系统研究[11,23~24]。林开勤、申响宝及冯延芝等通过MISA软件对杜仲转录组测序所得Unigenes进行微卫星特征分析,并开发系列引物,为杜仲不同种群间的遗传结构和遗传多样性分析研究提供了依据[22,25~26]。本研究共得到17 010个完整型SSR位点,占总SSR的96.28%,其出现频率为32.41%,高于红豆杉(2.24%)[27],楠木(13.97%)[28]〗和刺梨(20.37%)[29]等,低于芙蓉李(54.51%)等[30],其原因可能是基因库中不同植物的数据有所差异或者计算的方法有很大不同。此外,本研究发现杜仲SSR序列重复基元类型存在比较严重的偏倚性,这与冯延芝在杜仲果实转录组SSR分子标记研究中所得结论相同,原因可能是由于这些基元本身组成的SSR单位长度的限制。
叶片颜色是一个复合性状,其最终由叶绿素、类胡萝卜素及花色苷等植物色素的含量和比例所决定[31]。研究控制这些色素合成的关键基因及相关的SSR位点信息,可针对此开发相应引物,开展红叶性状的分子标记,不但可以进行种质资源的早期选择,还可以结合红叶性状是形态标记的特点,对开发的分子标记进行验证。本研究利用高通量测序技术建立了杜仲叶片红叶性状的转录组数据库,经代谢通路分析和SSR位点分析,找与叶片颜色形成相关的12个基因和13个SSR,为下一步开发杜仲红叶性状SSR分子标记提供参考,在分子基础上鉴定杜仲叶片的呈色性状上迈进一步。
致谢感谢广州基迪奥生物科技有限公司对文章的测序工作和生物信息分析工作的协助。
1.杜红岩.我国的杜仲胶资源及其开发潜力与产业发展思路[J].经济林研究,2010,28(3):1-6.
Du H Y.Discussion on industry development and exploration potential of gutta-percha resource in China[J].Nonwood Forest Research,2010,28(3):1-6.
2.杜红岩,胡文臻,俞锐.杜仲产业绿皮书:中国杜仲橡胶资源与产业发展报告[M].北京:社会科学文献出版社,2013.
Du H Y,Hu W Z,Yu R.Green book of Eucommia industry:The report on development of China’sEucommiarubber resources and industry[M].Beijing:Social Sciences Academic Press,2013.
3.杜红岩.中国杜仲图志[M].北京:中国林业出版社,2014:367.
Du H Y.ChinaEucommiapictorial[M].Beijing:China Forestry Publishing House,2014:367.
4.朱景乐,杜红岩,李芳东,等.3个杜仲品种叶片性状及活性成分质量分数[J].东北林业大学学报,2014,42(3):42-44.
Zhu J L,Du H Y,Li F D,et al.Leaf traits and active component content in three varieties ofEucommiaulmoides[J].Journal of Northeast Forestry University,2014,42(3):42-44.
5.彭少兵,张春贺,任利益,等.紫叶杜仲叶色变异研究[J].浙江林业科技,2013,33(3):61-66.
Peng S B,Zhang C H,Ren L Y,et al.Analysis on leaf color variation ofEucommiaulmoides[J].Journal of Zhejiang Forestry Science and Technology,2013,33(3):61-66.
6.董娟娥,杜红岩,张康健,等.观赏与药用杜仲无性系的选择[J].林业科学,2008,44(5):165-169.
Dong J E,Du H Y,Zhang K J,et al.Selection of ornamental and officinal clones ofEucommiaulmodies[J].Scientia Silvae Sinicae,2008,44(5):165-169.
7.胡笃敬.湖南的杜仲[J].植物生理学通讯,1956(2):26-27.
Hu D J.TheEucommiaulmodiesof Hunan[J].Plant Physiology Journal,1956(2):26-27.
8.周政贤,郭光典.我国杜仲类型、分布及引种[J].林业科学,1980,16(S1):84-91.
Zhou Z X,Guo G D.The form,distribution and introduction ofEucommiaulmoidesin China[J].Scientia Silvae Sinicae,1980,16(S1):84-91.
9.罗冉,吴委林,张旸,等.SSR分子标记在作物遗传育种中的应用[J].基因组学与应用生物学,2010,29(1):137-143.
Luo R,Wu W L,Zhang Y,et al.SSR marker and its application to crop genetics and breeding[J].Genomics and Applied Biology,2010,29(1):137-143.
10.Kalia R K,Rai M K,Kalia S,et al.Microsatellite markers:an overview of the recent progress in plants[J].Euphytica,2011,177(3):309-334.
11.黄海燕,杜红岩,乌云塔娜,等.基于杜仲转录组序列的SSR分子标记的开发[J].林业科学,2013,49(5):176-181.
Huang H Y,Du H Y,Wuyun T N,et al.Development of SSR molecular markers based on transcriptome sequencing ofEucommiaulmoides[J].Scientia Silvae Sinicae,2013,49(5):176-181.
12.Conesa A,Götz S,García-Gómez J M,et al.Blast2GO:a universal tool for annotation,visualization and analysis in functional genomics research[J].Bioinformatics,2005,21(18):3674-3676.
13.Ye J,Fang L,Zheng H K,et al.WEGO:a web tool for plotting GO annotations[J].Nucleic acids research,2006,34(S2):W293-W297.
14.安文燕,孙君灵,龚文芳,等.陆地棉矮化突变体Ari1327茎尖的转录组分析[J].植物遗传资源学报,2014,15(5):1046-1052.
An W Y,Sun J L,Gong W F,et al.Transcriptome analysis of stem apex of an upland cotton dwarf mutant Ari1327[J].Journal of Plant Genetic Resources,2014,15(5):1046-1052.
15.贾新平,叶晓青,梁丽建,等.基于高通量测序的海滨雀稗转录组学研究[J].草业学报,2014,23(6):242-252.
Jia X P,Ye X Q,Liang L J,et al.Transcriptome characteristics ofPaspalumvaginatumanalyzed withIlluminasequencingtechnology[J].Acta Prataculturae Sinica,2014,23(6):242-252.
16.Grabherr M G,Haas B J,Yassour M,et al.Full-length transcriptome assembly from RNA-Seq data without a reference genome[J].Nature Biotechnology,2011,29(7):644-652.
17.Altschul S F,Madden T L,Schäffer A A,et al.Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J].Nucleic Acids Research,1997,25(17):3389-3402.
18.周华,张新,刘腾云,等.高通量转录组测序的数据分析与基因发掘[J].江西科学,2012,30(5):607-611.
Zhou H,Zhang X,Liu T Y,et al.Data processing and gene discovery of high-throughput transcriptome sequencing[J].Jiangxi Science,2012,30(5):607-611.
19.李铁柱,杜红岩,刘慧敏,等.杜仲幼果和成熟果实转录组数据组装及基因功能注释[J].中南林业科技大学学报,2012,32(10):9-17.
Li T Z,Du H Y,Liu H M,et al.Transcriptome data assembly and gene function annotation ofEucommiamature fruits and young fruits[J].Journal of Central South University of Forestry & Technology,2012,32(10):9-17.
20.李铁柱,杜红岩,刘慧敏,等.杜仲果实和叶片转录组数据组装及基因功能注释[J].中南林业科技大学学报,2012,32(11):122-130.
Li T Z,Du H Y,Liu H M,et al.Transcriptome data assembly and gene function annotation ofEucommiafruits and leaves[J].Journal of Central South University of Forestry & Technology,2012,32(11):122-130.
21.赵德刚,李岩,赵懿琛,等.杜仲雌雄株转录组测序数据组装及基因功能注释[J].山地农业生物学报,2015,34(1):1-13.
Zhao D G,Li Y,Zhao Y C,et al.Transcriptome data assembly and gene function annotation of female and male plants inEucommiaulmoides[J].Journal of Mountain Agriculture and Biology,2015,34(1):1-13.
22.冯延芝,李芳东,魏琦琦,等.基于RNA-Seq的杜仲转录组微卫星特征分析[J].中国农业大学学报,2016,21(9):68-79.
Feng Y Z,Li F D,Wei Q Q,et al.Microsatellites characteristics of transcriptomic sequences fromEucommiaulmoidesOliv.based on RNA-Seq[J].Journal of China Agricultural University,2016,21(9):68-79.
23.吴敏,杜红岩,乌云塔娜,等.杜仲基因组微卫星特征及SSR标记开发[J].林业科学研究,2015,28(3):387-393.
Wu M,Du H Y,Wuyun T N,et al.Characterization of genomic microsatellites and development of SSR markers ofEucommiaulmoides[J].Forest Research,2015,28(3):387-393.
24.林开勤,赵德刚,李岩,等.杜仲性别相关EST-SSR标记的开发[J].林业科学,2016,52(10):146-152.
Lin K Q,Zhao D G,Li Y,et al.Development of gender-related EST-SSR markers inEucommiaulmoides[J].Scientia Silvae Sinicae,2016,52(10):146-152.
25.林开勤,李岩,赵德刚.杜仲转录组SSR发掘及标记开发[J].分子植物育种,2016,14(6):1548-1558.
Lin K Q,Li Y,Zhao D G.SSR mining and marker development inEucommiaulmoidesOliver transcriptome[J].Molecular Plant Breeding,2016,14(6):1548-1558.
26.申响保,闫文德,梁小翠,等.杜仲转录组微卫星特征分析[J].经济林研究,2016,34(4):47-51.
Shen X B,Yan W D,Liang X C,et al.Characteristic analysis of microsatellites inEucommiaulmoidestranscriptome[J].Nonwood Forest Research,2016,34(4):47-51.
27.李炎林,杨星星,张家银,等.南方红豆杉转录组SSR挖掘及分子标记的研究[J].园艺学报,2014,41(4):735-745.
Li Y L,Yang X X,Zhang J Y,et al.Studies on SSR molecular markers based on transcriptome ofTaxuschinensisvar.mairei[J].Acta Horticulturae Sinica,2014,41(4):735-745.
28.时小东,朱学慧,盛玉珍,等.基于转录组序列的楠木SSR分子标记开发[J].林业科学,2016,52(11):71-78.
Shi X D,Zhu X H,Sheng Y Z,et al.Development of SSR markers based on transcriptome sequence ofPhoebezhennan[J].Scientia Silvae Sinicae,2016,52(11):71-78.
29.鄢秀芹,鲁敏,安华明.刺梨转录组SSR信息分析及其分子标记开发[J].园艺学报,2015,42(2):341-349.
Yan X Q,Lu M,An H M.Analysis on SSR information in transcriptome and development of molecular markers inRosaroxburghii[J].Acta Horticulturae Sinica,2015,42(2):341-349.
30.方智振,叶新福,周丹蓉,等.‘芙蓉李’转录组SSR信息分析与分子标记开发[J].果树学报,2016,33(4):416-424.
Fang Z Z,Ye X F,Zhou D R,et al.Analysis on SSR information in‘Furongli’plum transcriptome and development of molecular markers inPrunussalicinaLindl[J].Journal of Fruit Science,2016,33(4):416-424.
31.朱书香,杨建民,王中华,等.4种李属彩叶植物色素含量与叶色参数的关系[J].西北植物学报,2009,29(8):1663-1669.
Zhu S X,Yang J M,Wang Z H,et al.Relationship between pigment contents and leafcolor parameters of four leaf-colored species ofPrunus[J].Acta Botanica Boreali-Occidentalia Sinica,2009,29(8):1663-1669.
State Key Laboratory of Forest Genetics and Breeding(Chinese Academy of Forestry)(TGB2015002);Special Fund for Basic Business Expenses of the Central Level Public Welfare Research Institutes of the Chinese Academy of forestry(CAFYBB2014QA037);The forest tree germplasm resources SURVEY of henan province
introduction:MIAO Zuo-Yun(1976—),female,Master,lecturer,mainly engaged in the breeding and cultivation ofEucommiaulmoides.
date:2017-04-02
AnalysisofRedLeafColorSSRMolecularMarkersbyTranscriptomeSequencingofEucommiaulmoides
MIAO Zuo-Yun1,2YANG Yun3LIU Pan-Feng1ZHENG Xin-Fang4DU Hong-Yan1ZHU Jing-Le1*
(1.Non-timber Forest Research and Development Center of Chinese Academy of Forestry,Zhengzhou 450003; 2.Huanghe Science and Technology College,Zhengzhou 450063; 3.Henan Agricultural University,Zhengzhou 450002; 4.South China Agricultural University,Guangzhou 510642)
The transcriptomes ofEucommiaulmoides‘Huazhong 12’ young and mature leaves and ‘Huazhong 11’ mature leaves were sequenced. The transcriptome data was assembled and classified by function, and microsatellites characteristics from obtained Unigenes and analyzed. The Unigenes were assembly generated a total of 54 517 Unigenes with an average length of 806.90 bp. Among them, 25 993 Unigenes accounted for 47.68% were annotated by comparing the assembled Unigenes with the Nr, Swiss-Prot, KEGG, and COG protein databases. KEGG pathway analysis presented that 6 910 annotated Unigenes divided into 122 classes according to its function. Among them, the Unigenes of ascorbate and aldarate metabolism was 39, flavonoid biosynthesis was 38, carotenoid biosynthesis was 34. There were 17 010 complete SSR located in 54 517 Unigenes accounted for 96.28% of the total SSR. The complete SSR included 67 frequent motifs, and the highest repeat of complete SSR type was A/T(7 747), following by AG/CT(5 039), AT/AT(850). There were 13 SSR located in ascorbate and aldarate metabolism, flavonoid biosynthesis and carotenoid biosynthesis. Our study would provide the molecular basis for genetic diversity analysis, genetic map construction and red leaf molecular marker development ofE.ulmoides.
Eucommiaulmoides;red leaf;microsatellites;transcriptome;high-throughput sequencing
林木遗传育种国家重点实验室(中国林业科学研究院)开放课题(TGB2015002);中国林科院中央级公益性科研院所基本业务费专项资金(CAFYBB2014QA037);河南省林木种质资源普查
苗作云(1976—),女,硕士,讲师,主要从事杜仲育种与栽培。
* 通信作者:E-mail:zhujingle1982@126.com
2017-04-02
* Corresponding author:E-mail:zhujingle1982@126.com
S727.3
A
10.7525/j.issn.1673-5102.2017.06.013
