基于CRISPR/Cas9系统定点编辑小鼠MAD2L1基因及其脱靶效应分析
2017-12-22陈丽香彭秀华谭丹韩铖潇赵锐王超李顺周晓辉
陈丽香,彭秀华,谭丹,韩铖潇,赵锐,王超,李顺,周晓辉
(1.上海市公共卫生临床中心,上海 201508; 2.上海交通大学农业与生物学院,上海 200240)
研究报告
基于CRISPR/Cas9系统定点编辑小鼠MAD2L1基因及其脱靶效应分析
陈丽香1*,彭秀华1*,谭丹1,韩铖潇2,赵锐1,王超1,李顺1*,周晓辉1*
(1.上海市公共卫生临床中心,上海 201508; 2.上海交通大学农业与生物学院,上海 200240)
目的建立利用CRISPR/Cas9系统对小鼠MAD2L1基因第二外显子基因编辑的方法体系,并分析CRISPR/Cas9对MAD2L1基因编辑的脱靶效应。方法通过CHOPCHOP网站设计位于小鼠MAD2L1基因第二外显子的靶点序列,并构建Cas9-MAD2L1载体,将该载体转染到NIH/3T3细胞中,通过嘌呤霉素筛选并结合荧光显微镜观察GFP后,提取细胞转染后的基因组DNA,利用PCR方法,结合T7E1分析和桑格尔测序,鉴定对小鼠MAD2L1基因编辑情况,并对转染后的NIH/3T3细胞进行CRISPR/Cas9脱靶效应分析。结果将Cas9-MAD2L1载体转染到NIH/3T3细胞并进行嘌呤霉素筛选后,荧光显微镜下观察到大量表达GFP的细胞,PCR结合T7E1结果显示,以转染后的细胞DNA为模板扩增出的228 bpMAD2L1 PCR产物,可被酶切成166 bp和62 bp片段,测序结果显示,成功对MAD2L1基因第二外显子靶点处进行基因编辑,脱靶效应分析未检测到CRISPR/Cas9有对脱靶位点进行基因编辑。结论成功建立对小鼠MAD2L1基因第二外显子进行基因编辑的方法体系,设计的对MAD2L1基因编辑靶点未检测到脱靶效应的发生。
CRISPR/Cas9;小鼠;MAD2L1;脱靶效应
MAD2L1(有丝分裂阻滞缺陷蛋白2)和MAD1L1(有丝分裂阻滞缺陷蛋白1)是有丝分裂检查点复合蛋白的重要组成部分,其中,MAD2L1基因在人中位于4号染色体,在小鼠中位于6号染色体上。Chung等[1]报道在有丝分裂前期,MAD1L1可直接招募MAD2L1与染色体着丝粒分离,该过程对实现MAD2L1与有丝分裂后期促进复合体(anaphase promoting complex)正调节因子CDC20相互作用是非常重要的。CDC20与MAD2L1和MAD1L1复合体直接作用可抑制有丝分裂后期促进复合体的活性,最终导致在中期—后期转换过程中姐妹染色单体失去粘着力[2,3]。目前研究表明,在哺乳动物细胞中MAD2L1功能的破坏可影响纺锤体检查点的功能,进而导致细胞非整倍性或肿瘤的发生。部分缺失MAD2L1的细胞(MAD2L1+/-)伴随着染色体不稳定(chromosome instability,CIN)现象的发生,在小鼠中最终导致肺癌的形成[4];在小鼠中过表达MAD2L1同样可导致染色体不稳定、细胞的非整倍性和肿瘤的发生[5],而染色体不稳定是肿瘤发生的主要驱动力之一[6]。
在过去的几十年中,基因打靶技术(gene targeting)和基因编辑工具得到了长足的发展(如:ZFN (zinc finger nucleases)[7]、TALENs (transcription activator-like effector nucleases)[8]和CRISPR/Cas9 (clustered regularly interspaced short palindromic repeats/CRISPR associated endonuclease cas9)[9])。其中,CRISPR/Cas9 是细菌和古细菌在长期选择压力中形成的一种适应性免疫防御,可用来有效抵御病毒及外源DNA的入侵。CRISPR的Type II已经被广泛用于基因工程,它包含Cas9、crRNA和tracrRNA,其中crRNA含有能够识别20 bp的靶向互补序列[10]。Cas9、crRNA和tracrRNA组成复合体,识别并结合crRNA互补的序列,然后解开DNA双链,Cas9中的HNH活性位点剪切crRNA的互补DNA链,RuvC活性位点剪切非互补链,最终导致DNA的双链断裂(DSB),进而实现基因的敲除和插入。目前,CRISPR/Cas9系统已经广泛用于对哺乳动物细胞、细菌、斑马鱼、小鼠、猪、猴的基因编辑[11-13]。
关于MAD2L1基因敲除,Floris等[14]近期结合Cre/loxP系统,利用基因打靶载体通过同源重组构建出MAD2L1f/f小鼠,该小鼠可与表达Cre小鼠进行杂交,实现条件性敲除MAD2L1基因。但目前尚未见利用CRISPR/Cas9技术对小鼠MAD2L1基因敲除的报道。本研究利用CRISPR/Cas9技术对小鼠MAD2L1基因第二外显子进行精确修饰,实现对小鼠MAD2L1基因敲除,以期为利用基因编辑工具CRISPR/Cas9建立MAD2L1基因敲除小鼠构建相应癌症模型奠定基础,最终推动利用基因编辑工具构建癌症模型研究领域的发展[15]。
1 材料与方法
1.1 材料
小鼠胚胎成纤维细胞系(NIH/3T3)购买自上海酶研生物科技有限公司,大肠杆菌感受态细胞DH5α菌株、质粒抽提试剂盒、基因组DNA提取试剂盒、转染试剂盒、DNA Marker均购买自北京全式金生物技术有限公司;限制性内切核酸酶和T7E1酶购买自NEB公司;胎牛血清购买自Gibco公司;pGEM-T载体、Wizard SV Gel和PCR Clean-Up System购买自Promega公司;引物合成、基因测序均由上海生物工程公司完成。
1.2 方法
1.2.1 小鼠MAD2L1基因序列分析和基因敲除靶点序列设计
NCBI查询小鼠MAD2L1基因组序列(NC_000072.6)(https://www.ncbi.nlm.nih.gov/nuccore/NC_000072.6?from=66535226&to=66541109 & report=genbank)和MAD2L1基因mRNA序列(NM_019499.4)(https://www.ncbi.nlm.nih.gov/nuccore/NM_019499.4)并进行序列比对,分析出MAD2L1基因含有5个外显子。利用CHOPCHOP软件(https://chopchop.rc.fas.harvard.edu/)设计位于MAD2L1基因第二外显子上20 bp的sgRNA靶点序列,靶点和PAM序列信息为5’GGGGTCAGTAGTTGTAAGCAAGG 3’。对选择的sgRNA靶点序列设计合成引物oligo, 序列为MAD2L1F1(5’AAACACCGGGGGTCAGTAGTTGTAAGCA 3’)和MAD2L1R1(5’ CTCTAAAACTGCTTACAACTACTGACCCC 3’)。
1.2.2 用于敲除小鼠MAD2L1的CRISPR/Cas9载体(Cas9-MAD2L1)构建
(1) Oligo二聚体的形成
oligo二聚体(oligo duplex)反应体系:将合成的oligo分别稀释成10 μmol/L后,按下列体系进行配置:上游引物1 μL;下游引物1 μL; Solution I 5 μL;补ddH2O至10 μL。反应条件:95℃ 3 min,95~25℃ (0.05℃/s),16℃ (5 min),-20℃保存备用。
(2) Oligo二聚体插入到Cas9/gRNA载体中
反应体系:Cas9/gRNA Vector 1 μL;oligo二聚体2 μL;补ddH2O至10 μL,25℃静置5 min后,取5 μL连接产物用于转化,挑取单菌落摇菌,提取质粒DNA进行测序,正确插入MAD2L1靶点序列的Cas9/gRNA载体(Cas9-MAD2L1)保存于-20℃备用。
1.2.3 Cas9-MAD2L1载体转染小鼠NIH/3T3细胞和嘌呤霉素筛选
NIH/3T3细胞转染前,将细胞铺板到12孔板中,待细胞生长至完全汇合时,用含有不同浓度的嘌呤霉素培养基(8,7,6,5,4,3,2和1 μg/mL)培养细胞,确定嘌呤霉素对NIH/3T3细胞最适筛选浓度。将NIH/3T3细胞接种到12孔板中,待细胞密度达到80%汇合度时进行细胞转染实验。用100 μL DMEM培养基稀释1.6 μg Cas9-MAD2L1质粒,为摸索最佳转染试剂量(TransIntroTMEL),取3.2 μL或4.8 μLTransIntroTMEL分别加入到稀释好的质粒DNA中,轻柔混匀,室温静置20 min后将质粒TransIntroTMEL复合物加入NIH/3T3细胞中,于37℃ CO2培养箱培养,转染4 h后更换培养基,48 h后用含有1 μg/mL嘌呤霉素的培养基进行细胞阳性筛选,荧光显微镜观察表达GFP的细胞数量比,待表达GFP的细胞占比较多时提取细胞基因组DNA进行后续分析(未转染的细胞基因组DNA用于对照)。
1.2.4 小鼠MAD2L1基因修饰分析
用位于MAD2L1基因靶点两侧的引物MAD2L1F2(5’TCAGCGTGGCATTTATCCGT 3’)和MAD2L1R2(5’TCCATTGCTTGCTTCCTCCC 3’),扩增转染后的细胞基因组DNA(未转染的细胞基因组DNA用于对照),PCR反应条件为95℃ 3 min;95℃ 1 min,60℃ 1 min, 72℃ 1 min,38 cycles;72℃ 7 min,2%琼脂糖凝胶电泳鉴定,如条带单一进行PCR产物直接纯化。分别对纯化后的PCR产物进行变性杂交处理:纯化后PCR产物8 μL,10× NE buffer2 2 μL,补ddH2O至19 μL,反应条件为95℃ 5 min,95℃以-2℃/s的速率降至85℃,再以-0.1℃/s的速率降至25℃,4℃保存。反应结束后,每组样品加入1 μL T7E1酶,37℃酶切1 h,2%琼脂糖凝胶电泳检测。PCR产物送样至上海生物工程公司测序,同时PCR产物与pGEM-T克隆载体连接,菌落PCR鉴定后将10个单菌落送至上海生物工程公司测序。
1.2.5 Cas9-MAD2L1脱靶效应分析
根据Jine 等[16]报道,鉴定CRISPR/Cas9系统潜在脱靶位点(potential off-target site)通常选取与20 bp靶点序列3’端有10~14 bp以上同源性的位点为潜在脱靶位点。通常,与20 bp靶点序列3’端同源性越高意味着该位点是潜在脱靶位点的可能性越大。在小鼠全基因组水平上进行Cas9-MAD2L1潜在脱靶位点,并用T7E1分析该脱靶位点的潜在脱靶效应,所使用的引物信息如表1。

表1 脱靶效应分析引物Tab.1 The primers for off-targeted analysis
2 结果
2.1 小鼠MAD2L1基因修饰靶点设计结果
NCBI查询小鼠MAD2L1基因组序列(NC_000072.6)和MAD2L1基因mRNA序列(NM_019499.4)并进行序列比对,分析出MAD2L1基因含有5个外显子。利用CHOPCHOP软件(https://chopchop.rc.fas.harvard.edu/)设计位于MAD2L1基因第二外显子上20 bp的sgRNA靶点序列 (图1)。

注:MAD2L1基因结构示意图(方框示意外显子,横线示意内含子)(上),MAD2L1基因修饰靶点(红线)和PAM区域(蓝线)。图1 小鼠MAD2L1基因修饰原理图Note.The box indicates the exon,the horizontal line indicates the intron (above),MAD2L1 gene editing site (red line) and PAM region (blue line).Fig.1 Gene editing strategy for the mouse MAD2L1
2.2 用于敲除小鼠MAD2L1的CRISPR/Cas9载体(Cas9-MAD2L1)构建结果
将连接了MAD2L1靶点序列的Cas9/gRNA质粒DNA进行测序分析,测序结果正确的质粒命名为Cas9-MAD2L1。质粒图谱如图2所示。

图2 Cas9-MAD2L1质粒图谱Fig.2 Map of Cas9-MAD2L1 plasmid
2.3 荧光显微镜观察嘌呤霉素筛选Cas9-MAD2L1载体转染后的NIH/3T3细胞
NIH/3T3细胞在不同浓度的嘌呤霉素培养基(8,7,6,5,4,3,2和1 μg/mL)中培养48 h后,对细胞生长状态进行观察,嘌呤霉素对NIH/3T3细胞最适筛选浓度为1 μg/mL。Cas9-MAD2L1质粒转染NIH/3T3细胞48 h后,用含有1 μg/mL嘌呤霉素培养基对细胞进行筛选,48 h后观察细胞如图3所示。
2.4 MAD2L1基因敲除结果分析
对加入4.8 μL或3.2 μL转染试剂(TransIntroTMEL)的Cas9-MAD2L1质粒转染NIH/3T3细胞进行嘌呤霉素筛选后,分别提取基因组DNA,命名为DNA- 4.8和DNA- 3.2。用位于MAD2L1基因修饰靶点两侧的引物扩增该两基因组DNA,电泳检测结果显示样品均扩增得到228 bp的目的片段(图4 A)。对扩增出的228 bp目的片段进行T7E1酶切分析(图4B),结果显示T7E1酶将228 bp片段切割成166 bp和62 bp,而对照样品228 bp片段没有被T7E1酶切(图4B)。DNA- 4.8样品的T7E1酶切效率约50%,DNA- 3.2样品的T7E1酶切效率约14%。以MAD2L1基因修饰的基因组DNA样品为模板扩增出的228 bp目的片段PCR产物测序结果发现在MAD2L1靶点序列后有双峰结构,而野生型基因组DNA样品为模板扩增出的228 bp目的片段PCR产物测序结果为单峰(图5)。228 bp PCR产物TA克隆后挑选10个单克隆测序,结果表明MAD2L1基因靶点处有发生缺失1个、17个、7个、13个碱基和增加1个碱基的情况。(图6)
2.5 脱靶效应分析
在小鼠全基因组水平上,对MAD2L1基因20 bp靶点序列(GGGGTCAGTAGTTGTAAGCAAGG)的脱靶位点进行比对分析,选取其中10个可能性相对较高的脱靶位点,如图7所示。随机选取该10个脱靶位点中的3个位点进行T7E1分析,PCR产物均未被T7E1酶切,结果表明Cas9-MAD2L1载体并未对所检测的潜在脱靶位点进行基因编辑(图8),证实Cas9-MAD2L1载体的靶点识别专一性相对较好。
3 讨论
大量研究表明,MAD2L1基因的缺失可引起染色体不稳定(CIN),进而驱动肿瘤的发生[17,18]。在小鼠肝脏中缺失MAD2L1基因可引起肝癌、肺癌的发生[4,14];对小鼠T细胞进行MAD2L1和p53基因敲除可导致T细胞急性淋巴母细胞性淋巴瘤(acute lymphoblastic lymphoma)的发生,最终小鼠因胸腺过大和呼吸困难于4~5月内死亡[14]。本研究首次利用CRISPR/Cas9工具对小鼠MAD2L1基因进行精确编辑,为后续利用该基因编辑工具建立MAD2L1基因敲除小鼠或直接编辑成年鼠特定组织敲除MAD2L1基因构建相应癌症模型奠定基础,在利用基因编辑工具建立癌症小鼠模型领域有重要意义。

图3 Cas9-MAD2L1质粒转染NIH/3T3细胞后嘌呤霉素筛选结果Fig.3 Puromycin screening NIH/3T3 cells after Cas9-MAD2L1 plasmid transfection

注:A:位于MAD2L1基因修饰靶点两侧的引物扩增得到228 bp的目的片段(M:100 bp DNA marker,1:DNA- 4.8样品,2:DNA- 3.2样品,H2O空白对照)。B:PCR产物T7E1酶切分析(M:100 bp DNA marker,1:DNA- 4.8样品,2: DNA- 3.2样品,3:野生型DNA对照)。图4 PCR和T7E1酶切结果分析Note.A.A pair of primer located in the two sides of MAD2L1 gene editing site was used to amplified a 228 bp fragment.(M: 100 bp DNA marker,1: DNA- 4.8 sample,2: DNA- 3.2 sample,H2O blank control).B.T7E1 analysis (M: 100 bp DNA marker,1: DNA- 4.8 sample,2: DNA- 3.2 sample,3: wild-type DNA control).Fig.4 Results of PCR and T7E1 analysis
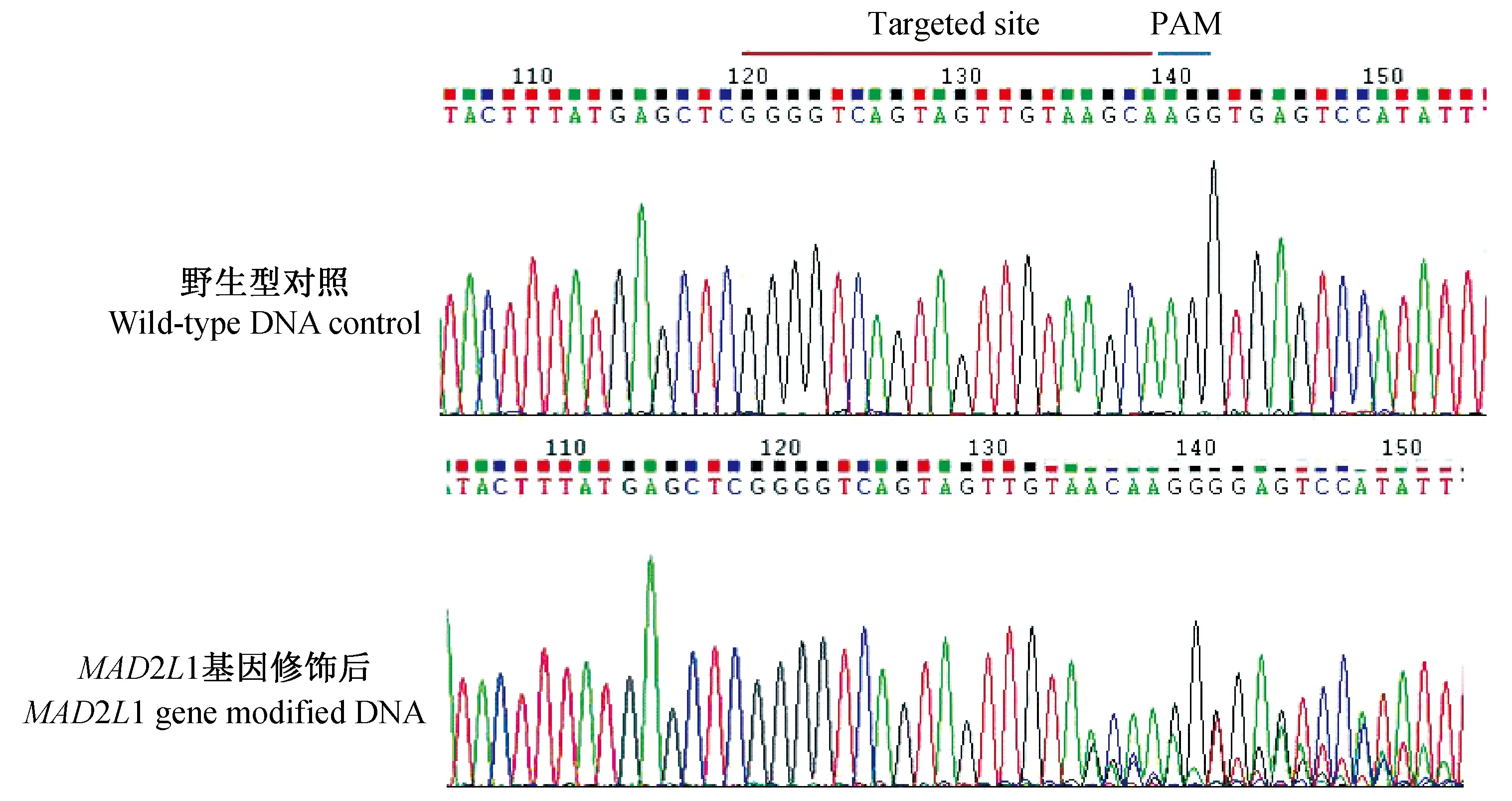
图5 野生型对照DNA和MAD2L1基因修饰DNA的PCR扩增产物桑格尔测序分析Fig.5 Sanger sequencing analysis of PCR amplification products of wild-type control DNA and MAD2L1 gene-modified DNA
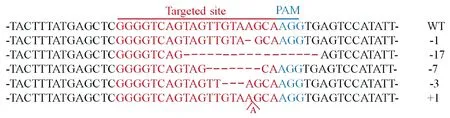
图6 MAD2L1基因修饰DNA的PCR扩增产物TA克隆后单克隆测序分析Fig.6 Monoclonal sequencing analysis of MAD2L1 gene-modified DNA PCR amplification products

图7 小鼠MAD2L1基因脱靶位点分析Fig.7 Off-target sites analysis for the mouse MAD2L1 gene
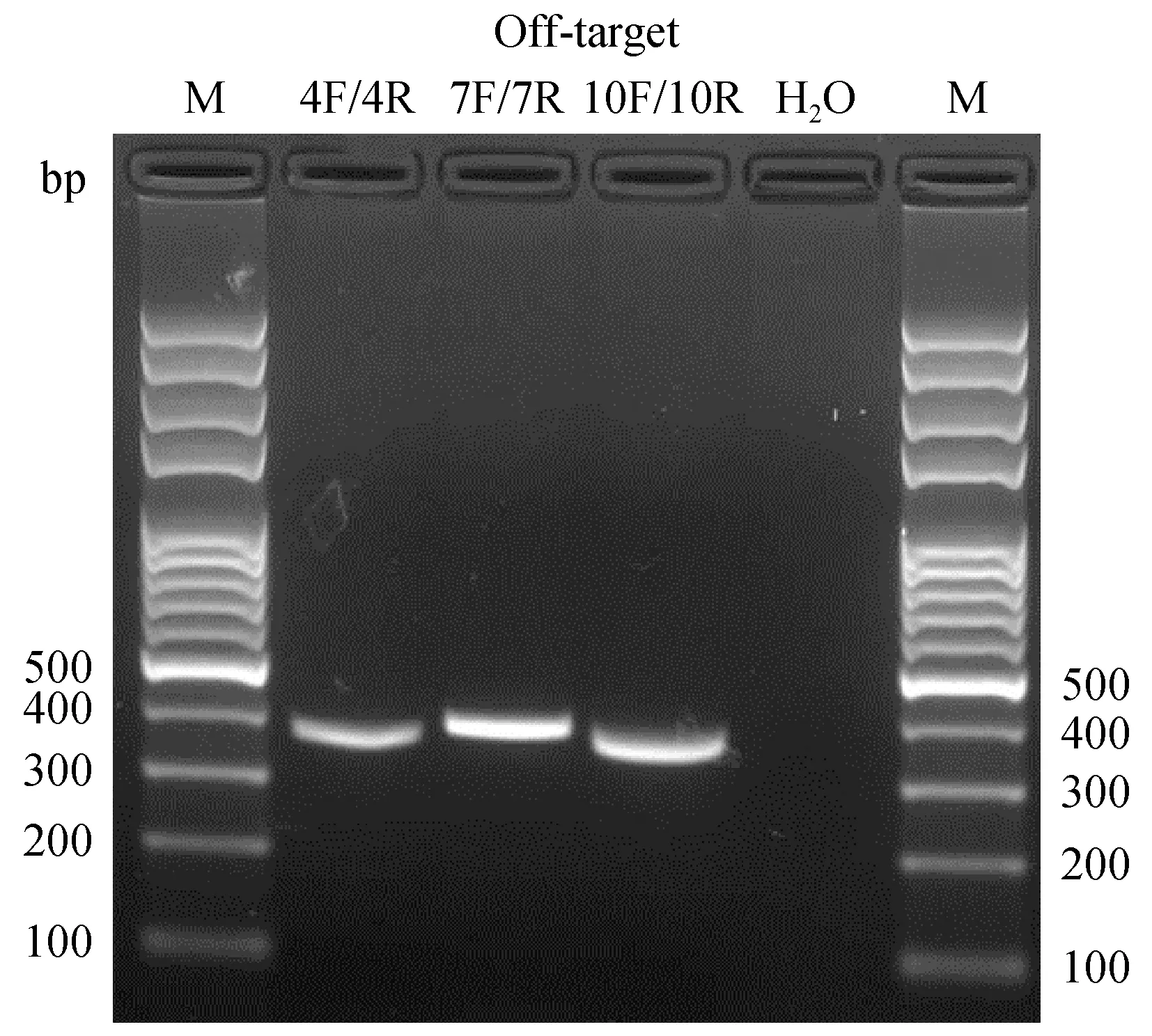
注:M.100 bp DNA marker,4F/4R:脱靶位点4 (361 bp),7F/7R:脱靶位点7 (378 bp),10F/10R:脱靶位点10 (361 bp)。图8 脱靶位点T7E1分析Note.M.100 bp DNA marker,4F/4R: off-target site 4 (361 bp),7F/7R: off-target site 7 (378 bp),10F/10R: off-target site 10 (361 bp).Fig.8 T7E1 analysis for off-target sites
CRISPR/Cas9工具相对于传统的基因打靶、ZFN和TALENs系统具有成本低、操作简单、基因编辑效率高、可同时编辑多个基因等优点,目前已广泛应用于编辑真菌、植物、动物等物种以及人类疾病治疗、新药开放等生物医学研究领域。本研究构建的Cas9-MAD2L1质粒携带有表达Cas9蛋白开放阅读框、小鼠MAD2L1 sgRNA、GFP基因和嘌呤霉素基因。利用该质粒进行细胞转染时,可通过嘌呤霉素抗生素进行细胞筛选,并结合荧光显微镜观察表达GFP的细胞,从而可直观初步判定发生基因编辑的细胞比例。该含有荧光标记和抗生素标记的基因编辑系统比不含荧光标记和抗生素的CRISPR/Cas9系统在筛选转染后的细胞方面较方便。本研究结合了T7E1酶切、PCR产物测序和单克隆测序,结果表明成功在NIH/3T3细胞中对MAD2L1基因进行了精确的编辑,为进一步利用CRISPR/Cas9工具构建MAD2L1小鼠奠定了基础。
目前已报道利用CRISPR/Cas9系统进行基因编辑有脱靶情况的发生[19,20],Gao等[21]报道利用Cas9 nikase (Cas9D10A)或Cas9-FokI[22]可降低CRISPR/Cas9系统脱靶效率的发生。此外,利用基因编辑靶点设计软件设计出低脱靶效率的靶点序列也是可行方法之一。本研究利用CHOPCHOP软件设计出小鼠MAD2L1基因修饰靶点,获得了较好的基因编辑效果。对该基因修饰靶点进行预测的脱靶位点分析,结果提示没有检测到该基因修饰靶点脱靶情况的发生,证实了对小鼠MAD2L1位点基因修饰的特异性。本研究结果为后续结合高压水动力法运送CRISPR/Cas9系统到成年小鼠体内敲除MAD2L1基因构建相关癌症小鼠模型奠定了基础,以期最终促进相关癌症发生的分子机制、癌症信号通路、新型生物标记物、靶向药物的开发和癌症研究成果的转化。
[1] Chung E,Chen RH.Spindle checkpoint requires Mad1-bound and Mad1-free Mad2 [J].Mol Biol Cell,2002,13(5): 1501-1511.
[2] Pfleger CM,Salic A,Lee E,et al.Inhibition of Cdh1-APC by the MAD2-related protein MAD2L2: a novel mechanism for regulating Cdh1 [J].Genes Dev,2001,15(14): 1759-1764.
[3] Zhang Y,Lees E.Identification of an overlapping binding domain on Cdc20 for Mad2 and anaphase-promoting complex: model for spindle checkpoint regulation [J].Mol Cell Biol,2001,21(15): 5190-5199.
[4] Michel L,Diaz-Rodriguez E,Narayan G,et al,Complete loss of the tumor suppressor MAD2 causes premature cyclin B degradation and mitotic failure in human somatic cells [J].Proc Natl Acad Sci U S A,2004,101(13): 4459-4464.
[5] Sotillo R,Hernando E,Diazrodriguez E,et al.Mad2 overexpression promotes aneuploidy and tumorigenesis in mice [J].Cancer Cell,2007,11(1): 9-23.
[6] Michor F.Chromosomal instability and human cancer [J].Philos Trans R Soc Lond B Biol Sci,2005,360(1455): 631-635.
[7] Yang S,Ding S,Xu Q,et al.Genetic manipulation by zinc-finger nucleases in rat-induced pluripotent stem cells [J].Cell Reprogram,2017,19(3):180-188.
[8] Yang L,Liu Y,Zhang N,et al.Novel impact of the DNMT3A R882H mutation on GSH metabolism in a K562 cell model established by TALENs [J].Oncotarget,2017,8(18): 30395-30409.
[9] Xue W,Chen S,Yin H,et al.CRISPR-mediated direct mutation of cancer genes in the mouse liver [J].Nature,2014,514 (7522): 380-384.
[10] Garneau JE,Dupuis ME,Villion M,et al.The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA [J].Nature,2010,468(7320): 67-71.
[11] Zetsche B,Heidenreich M,Mohanraju P,et al.Multiplex gene editing by CRISPR-Cpf1 using a single crRNA array [J].Nat Biotechnol,2017,35(1): 31-34.
[12] Li S,Edlinger M,Saalfrank A,et al.Viable pigs with a conditionally-activated oncogenic KRAS mutation [J].Transgenic Res,2015,24(3): 509-517.
[13] Li S,Flisikowska T,Kurome M,et al.Dual fluorescent reporter pig for Cre recombination: transgene placement at the ROSA26 locus [J].PLoS One,2014,9(7): e102455.
[14] Foijer F,Albacker LA,Bakker B,et al.Deletion of the MAD2L1 spindle assembly checkpoint gene is tolerated in mouse models of acute T-cell lymphoma and hepatocellular carcinoma [J].eLife,2017,6: e20873.
[15] Mou H,Kennedy Z,Anderson DG,et al.Precision cancer mouse models through genome editing with CRISPR-Cas9 [J].Genome Med,2015,7(1): 53.
[16] Jinek M,Chylinski K,Fonfara I,et al.A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity [J].Science,2012,337(6096): 816-821.
[17] Kastan MB,Bartek J.Cell-cycle checkpoints and cancer [J],Nature,2004,432 (7015): 316-323.
[18] Guo Y,Zhang X,Yang M,et al.Functional evaluation of missense variations in the human MAD1L1 and MAD2L1 genes and their impact on susceptibility to lung cancer [J].J Med Genet,2010,47(9): 616-622.
[19] Kim D,Bae S,Park J,et al.Digenome-seq: genome-wide profiling of CRISPR-Cas9 off-target effects in human cells [J].Nat Methods,2015,12(3): 237-243.
[20] Martin F,Snchez-Hernndez S,Gutiérrez-Guerrero A,et al.Biased and unbiased methods for the detection of off-target cleavage by CRISPR/Cas9: An overview[J].Int J Mol Sci,2016,17(9): 1507.
[21] Gao Y,Wu H,Wang Y,et al.Single Cas9 nickase induced generation of NRAMP1 knockin cattle with reduced off-target effects [J].Genome Biol.2017,18(1): 13.
[22] Sternberg SH,Doudna JA.Expanding the biologist’s toolkit with CRISPR-Cas9 [J].Mol Cell,2015,58(4): 568-574.
MouseMAD2L1geneeditingbyCRISPR/Cas9andanalysisofitsoff-targeteffect
CHEN Li-xiang1*,PENG Xiu-hua1*,TAN Dan1,HAN Cheng-xiao2,ZHAO Rui1,WANG Chao1,LI Shun1*,ZHOU Xiao-hui1*
(1.Shanghai Public Health Clinical Center,Shanghai 201508,China;2.School of Agricalture and Biology,Shanghai Jiao Tong University,Shanghai 200240,China)
ObjectiveTo establish a method for specific gene editing of the second exon of mouseMAD2L1 gene by CRISPR/Cas9,and analyze its off-target effect.MethodsThe gene editing site forMAD2L1 gene was designed by CHOPCHOP,and the Cas9-MAD2L1 vector was constructed based on the designed editing site.Cas9-MAD2L1 was then transfected into NIH/3T3 cells and screened with puromycin,followed by observing GFP expression using fluorescence microscopy.The genomic DNA from transfected cells was extracted and a partial fragment ofMAD2L1 gene was amplified by PCR.T7E1 analysis and Sangger sequencing were used for gene editing and off-target analysis.ResultsAfter Cas9-MAD2L1 transfection and puromycin screening,a large number of GFP-expressing cells were observed under the fluorescence microscope.Combined the PCR result with TE71 analysis,the amplified 228 bp PCR products can be digested into 166 bp and 62 bp fragments.The sequencing result showed that the second exon ofMAD2L1 gene was successfully edited,and the off-target effect was undetected in our system.ConclusionsThe method for specific gene editing of the second exon of mouseMAD2L1 gene by CRISPR/Cas9 is successfully established,and off-target effect ofMAD2L1 gene is not detected.
CRISPR/Cas9; Mice;MAD2L1; Off-target effect
LI Shun.E-mail:lishun86@126.com; ZHOU Xiao-hui.E-mail: zhouxiaohui@shaphc.org
国家自然科学基金青年基金项目(No.31601908);上海市科技发展基金实验动物研究项目(No.15140904000);上海市人才发展资金(No.2017043);上海市卫生和计划生育委员会科研课题项目(No.20154Y0075);上海市公共卫生临床中心院内项目(No.2016-02,KY-GW-2017-04)。
陈丽香(1988-),女,硕士在读,从事实验动物模型构建研究。E-mail:xiaripaomo@126.com;
彭秀华(1973-),女,高级兽医师,从事实验动物模型构建研究。E-mail:pengxh1973@sina.com
李顺(1986-),男,博士,助理研究员,E-mail:lishun86@126.com;
周晓辉(1973-),男,博士,研究员,E-mail:zhouxiaohui@shaphc.org
Q95-33
A
1005-4847(2017) 06-0587-07
10.3969/j.issn.1005-4847.2017.06.001
2017-05-22
