结构DNA纳米技术应用新进展
2017-12-18张洪芝张志庆王秀凤张国栋刘婷婷刘淑贞
张洪芝 张志庆 王 芳 周 亭 王秀凤 张国栋 刘婷婷 刘淑贞
结构DNA纳米技术应用新进展
张洪芝 张志庆*王 芳 周 亭 王秀凤 张国栋 刘婷婷 刘淑贞
(中国石油大学(华东)理学院,山东 青岛 266580)
DNA具有非凡的分子识别性能和显著的结构特征,这使得它在材料的纳米级调控方面具有独特的优越性,在许多领域也展现出广阔的应用前景。本文从模块化DNA自组装和DNA折纸术两个方面综述了近些年DNA纳米技术,包括近年来DNA纳米技术中比较新型的组装方法;并从DNA纳米结构作为模板定位纳米粒子和蛋白以及用于生物医药等方面介绍了DNA纳米技术的应用;同时,对DNA纳米技术发展及应用进行了展望。
DNA纳米技术;自组装;DNA模块;DNA折纸术;靶向给药
1 引言
DNA作为储存遗传信息的载体,是人类探寻生命密码的钥匙。但是长久以来结构模型的难以确定,一直困扰着人们。直到1953年,Watson和Crick1提出DNA是通过碱基互补配对形成的双螺旋结构,这一模型的提出,可谓是20世纪人类最伟大的发现之一,也为后来DNA纳米技术2−6的发展提供了可能。
2 DNA纳米技术
DNA纳米结构的构造主要通过DNA的自组装。自组装7是指通过弱的和较小方向性的非共价键,把原子、离子连接在一起构成纳米结构,这种方法是自然界组装高复杂度材料的主要方法。而在所有天然自组装分子之中,DNA自组装高复杂度的纳米材料效果最显著。利用氢键、堆叠、静电和疏水性相互协同作用,驱动DNA链按非常精确的碱基配对规则1,8与它的互补链组装成一个双螺旋结构,加上其良好的可设计性和可寻址性,使得DNA成为构建纳米结构9−11的良好原料。
2.1 DNA模块自组装
2.1.1 DNA自组装成独立纳米小结构
利用DNA的碱基互补配对可以制备出独立的纳米小结构。Chen和Seeman12利用DNA单链组装了一个正六面体(图1a),该正六面体由六条单链组成,将连接的DNA分子形成互连环以创建一个立方体状结构。该结构中的每个单链与其四个相邻的链连接两次,因此,每个边缘包含两个DNA双螺旋。Shih等13报道了一个包括六个柔性接头和12个连接支柱的截角八面体(图1b)。类似的,Goodman等14合成了一个具有刚性的手性DNA四面体(图1c),这是一个由两条63个碱基,一条73个碱基,一条53个碱基的DNA单链构成的四面体,并利用其结构特性在其内部包裹了蛋白质细胞色素。

(a−c) molecular models of a DNA cube12,21, DNA octahedron13, DNA tetrahedron14; (d) DNA hexagonal ring15; (e) DNA organize Au particles into triangles and squares16; (f) Several DNA Polyhedrons17; (g) self-assembly of DNA polyhedra18; (h) Self-assembly of DNA icosahedra19.
此后,研究者们为DNA纳米结构注入了新的元素,即有机骨架,DNA结合有机骨架便于组装构型的固定,且合成方法简单易行。Aldaye和Sleiman15利用六条两端连有DNA单链的有机骨架,通过端部DNA的碱基互补配对,合成了一个有机顶点的正六边环(图1d)。在此基础之上,二人16利用同样的原理设计了具有刚性的带有有机顶点的三角形和四边形,并用它们作为动态支架来组装金纳米颗粒;以这些刚性的环状物为模板,将连有金纳米颗粒的DNA单链,通过DNA配对连接,使金纳米颗粒按照预期的位置精密地排列;利用DNA的成链和解链特性,实现“写/擦”的结构化转变(图1e)。之后,该课题组17又利用不同形状的等边环状模板结合DNA链组装成三棱柱、立方体、六棱柱等(图1f),该办法还构建了第一个动态3D DNA胶囊,其大小可以在三个不同维度进行可逆的转换。Mao课题组18利用对称原则,通过使用同臂构建模块组装3D组装体。他们采用三种不同类型的DNA链,先分步组装成三角星形结构的单体,然后将单体通过一锅混合的简单程序制备出不同的3D纳米结构。通过可灵活控制的对称三臂结构,合成了四面体,十二面体和一个巴基球(图1g)。按照同样的思路,该课题组又采用五角星模块构造出二十面体,以及可以作为纳米容器的DNA笼子19(图1h)。
2.1.2 基于DNA瓦片的无限自组装
美国科学家Seeman20曾受生物重组过程的Holliday交叉结启发,利用四条单链DNA构建了一种四臂结构11,21,并形象的将此命名为DNA“瓦片”(DNA tile),图2(a, b)简单描述了Seeman最初的想法。这个DNA“瓦片”的四个臂均留有粘性末端,不同瓦片之间通过这种粘性末端相互连接,可构成无限延伸的二维平面结构;通过巧妙地设计带有六个粘性末端的DNA“瓦片”,构筑成一个三维立体的网络结构,进而将蛋白质分子固定到网络中,利用此网络结构便于蛋白质结构的解析。之后,Seeman与其同事22,23通过链的交换方式连接,设计出两个双螺旋并列DX模块(double-crossover)(图2c),DX模块的宽度可达到双螺旋宽度,通过设计合适的粘性末端,DX模块可成功组装成周期性的双链紧密排列的片状DNA无限结构24−26。DX模块较之普通的DNA双螺旋,其结构刚性大大增加,因此适合于构造具有空间复杂性的DNA纳米结构。在DX模块的基础上,研究者们进一步设计了含有三个27、四个28、六个29等不同DNA螺旋数的组装模块,以满足构建更为复杂多变的DNA纳米组装结构的需要。
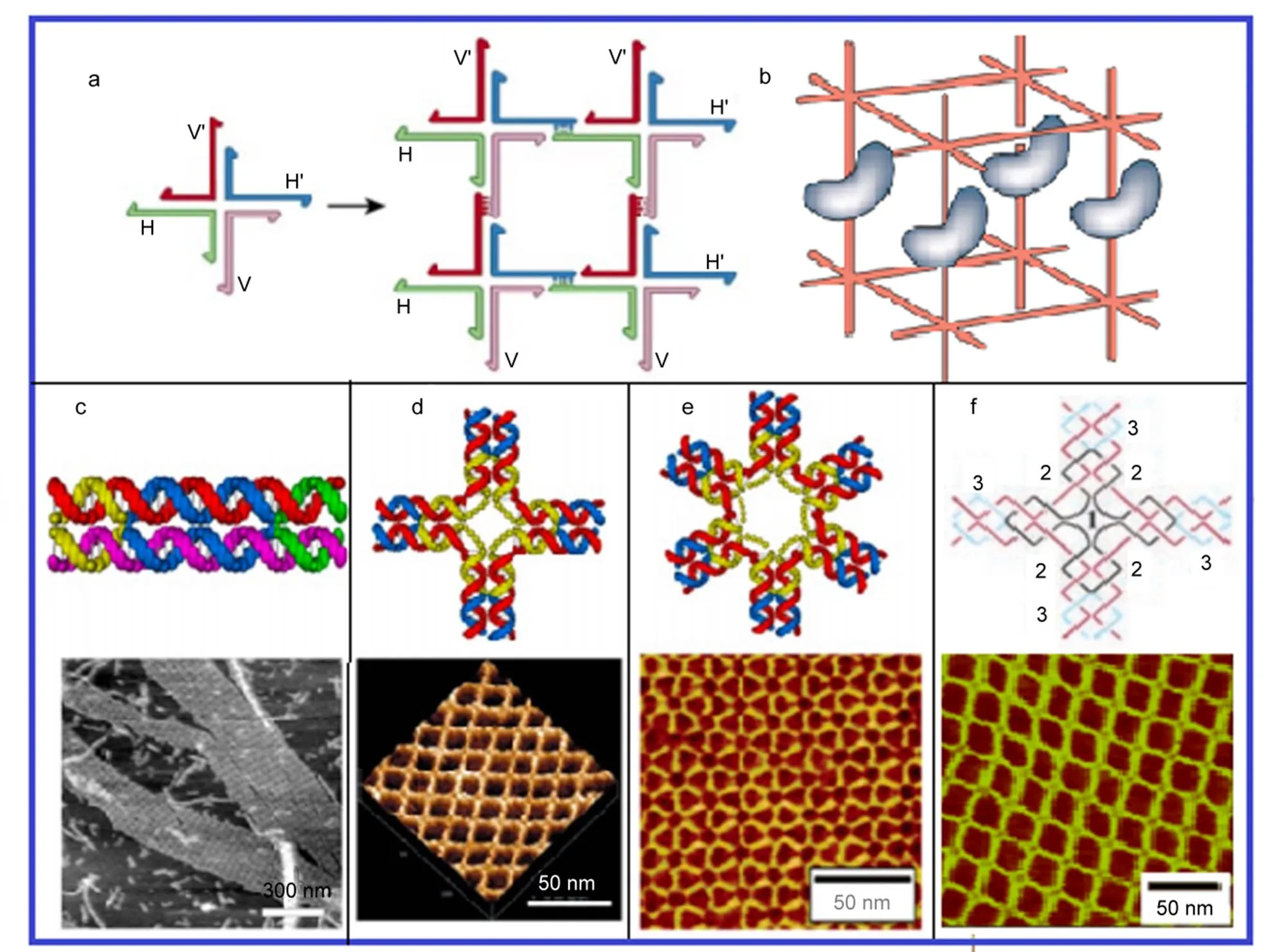
图2 典型的DNA模块及其组装成的纳米结构
(a, b) Seeman’s original proposals to use immobile DNA junctions to create self-assembling arrays and self-assembled 3D DNA lattices21; (c−f) top: DNA nanostructure motifs used to create periodic 2D arrays11,22,23,30,33,34, and their corresponding AFM images.
Yan等30从最初的DNA“瓦片”和DX模块出发,构建了十字形“瓦片”(Four-arm junction) (图2d),该十字单元共用了三种长度不等的九条DNA短链,并以此构建出结构十分牢固的DNA无限网络结构。之后研究者们又构建出三角形31、三角星形32、六角星形33(图2e)等DNA模块,并用这些基础单元构建了不同的无限网络格子。为了简化序列的设计,Mao课题组34利用十字形瓦片的四重对称性,仅使用三条不同DNA链就制备出对称的十字形“瓦片”(图2f)。Mao将原有的九条DNA链简化为三条,每种长度相同的链序列相同。这种中心对称的瓦片极大地减少了所需DNA链的种类,极大地降低了成本和实验误差。更重要的是,序列的高度对称性确保了组装结构的几何对称性,可以有效消除在自组装过程中产生的不可预测的扭曲,进一步保证了DNA阵列的生长。
2.2 DNA折纸术
自DNA模块的自组装技术发展以来,涌现出大量独具创意的模块,研究者们在这些不同的模块基础上,构造了丰富多彩的图形结构。他们共同的思路都是利用小的基本单元通过黏性末端的碱基互补拼接成大的图形。然而,基于模块的自组装也显现出共同的局限性,比如对大型结构的可控性差,很难构造出高复杂度和不规则的结构。直到折纸术的出现为传统的自组装带来了新的生机,从此自组装技术步入折纸的新篇章。
2.2.1 经典的DNA折纸术
2006年Rothemund35首次提出DNA折纸术,这是一种不同于传统自组装(自下而上的组装)方法36,37。设计思路是将一条长度为7429个(M13mp18噬菌体DNA)碱基的DNA作为脚手架链来回折叠,并利用200多条订书钉链将形状固定。成功得到了正方形、矩形、五角星、笑脸和三角形等结构(图3a)。实验过程免除了过度的纯化带来的繁琐,也不需要极其精确的浓度配比,使实验过程变得十分简便,而且产量有的高达90%,充分证明了折纸方案的可行性和优越性。随后形式多样的折纸结构相继出现,包括Qian等38制作的拟中国地图和Andersen等39设计的奥胡斯大学的校徽-海豚等等(图3b,c)。设计的多样性使得短链的设计变得困难,为此,研究者们开发了软件包SARSE、Tiamat和Cadnano等软件包,利用它可以设计出任何形状的二维DNA折纸结构。
随着二维结构的不断发展,三维结构也逐渐建立起来。在海豚图形之后,Andersen等人40又以M13mp18为脚手架链,将一条M13分成六个部分,每个部分单独折叠成一个矩形,然后将他们连接起来,设计出一个3D纳米盒子(42 nm × 36 nm × 36 nm)该纳米盒子的盖子可以在“DNA钥匙”的存在下可控的实现开启和关闭(图3d)。无独有偶,Ke与其合作者41采用类似的方法,并选择性的加入跨面订书钉,制备出一个立方体结构。Douglas等人42还制备了一个六螺旋DNA纳米管,这个纳米管长度约为410 nm,是由6个DNA螺旋结构通过交叉连接的方式连接起来。随着结构越来越多样化,制备具有空间曲率的结构是一项新的挑战。Han等43将研究聚焦在DNA的拓扑结构上,制备出三维莫比斯环应对了这个挑战(图3e),它是采用Routhemund的设计方法,将M13mp18结合165条短链,并通过使用链的置换得到了Kirigami-ring和Kirigami-catenane两种图形,这一发现推动了折纸技术进一步的发展,紧接着该课题组44又制备出空心球和花瓶折纸结构。除了制备单分散的折纸结构,Thomas等45还将立体的折纸结构通过粘性末端的连接,制备出折纸纤维(图3f),并首次建立了利用3D DNA折纸结构完成可切换纳米颗粒自组装的设计原则。这一策略的建立将促进这两个相对独立领域的合并,也为自组装胶体系统提供可供基础研究的单分散的构建模块。

图3 传统方法的二维和三维DNA折纸结构
(a) 2D smiley face and triangle shapes35; (b) Simulation of the Chinese map38; (c) The 2D origami of dolphin39; (d) The DNA box40; (e) The Kirigami-ring and Kirigami-catenane43; (f) 3D origami Cuboids and DNA origami fibrils45.
2.2.2 新型的DNA折纸术
经过近十年的发展,DNA折纸在二维三维结构上都取得了很大进展,得到的折纸结构丰富多样,精度控制也达到更高水平,但是由于所使用的脚手架链M13的碱基限制,折纸结构的尺寸受到制约。
杨百翰大学的Pound等46利用PCR技术,制备出不同长度的DNA链,通过筛选不同长度的DNA来用于构建折纸结构,折出的该校简称B、Y、U三个字母(图4a)是第一个细线分叉图形。Wei等47另辟蹊径,提出了一种新的不需要骨架长链的DNA折纸方案--单链瓦片(single-stranded tile,SST),它由42个碱基的DNA链组成,在自组装过程中与四个相邻的tile连接。这种完全由短链组装的折纸构思,在图形样式上操控更灵活易行,可以任意人为地剪切塑造自己想要的形状,利用该方法他们得到了各种各样的二维图形(图4b),包括0−9个阿拉伯数字、英文字母、金木水火土等汉字和其他图形。樊春海课题组48又将折纸技术发展到新的高度,他们结合滚环扩增技术49,50(Rolling Circle Amplification),利用滚环扩增产物的周期性特点,将此作为折纸的脚手架链DNA,结合几个订书钉链就可以完成折纸结构。如利用三个订书钉将一个具有96个碱基重复单元的滚环扩增单链折叠成一条纳米线(图4c),通过滚环扩增模板链的周期长度来控制纳米线的宽度,并使用预留的粘性末端将两层、三层纳米线连接起来,制成具有空间结构的纳米线。此外,我国南京大学肖守军51课题组使用环状DNA成功制备出DNA纳米管(图4d)。这种折纸方法相比于经典的折纸术来说,极大地较少了使用订书钉的数量,更简化了繁琐的序列设计。
3 DNA纳米结构的应用
随着DNA纳米技术的发展,DNA自组装体结构不断地精密化,人们能够制备出的组装体复杂度也越来越高,除了关注结构本身,人们对其应用价值的探索也一直在深入,DNA组装体也以其独特的性质在生物医药、复合纳米材料、电子芯片等领域展现着广阔的应用前景。
3.1 DNA纳米结构组装纳米粒子和蛋白
近年来,研究者对纳米材料的可控制备取得令人鼓舞的成果52,但如何将散乱的纳米粒子变成有序的纳米结构,以提供进一步的性能特点,仍然是一项挑战。DNA纳米结构的出现为此提供了机遇,使用DNA纳米结构为模板可以精确定位和组装纳米粒子,得到各种功能化的纳米器件。例如以DNA结构为模板组装金属纳米粒子,使得金属纳米粒子具有分子尺寸的间隙。表面增强拉曼光谱结果表明,具有分子尺寸间隙的金属粒子对耦联于粒子间的分子的拉曼散射具有极大的增强效应53−55,这种性质被认为是构建新型光子器件、分子标尺56及纳米催化器件的基础57,58。
Ding等59利用DNA折纸得到的三角形结构为模板,将不同尺寸的金纳米粒子精确地组装到DNA折纸结构表面,该结构局部场增强效应得到明显的提高。他们首先在折纸结构上预先设定好位点,然后将连有短链的不同尺寸的金纳米粒子加入,通过金纳米颗粒表面连有的短链和折纸位点碱基互补结合,从而将不同尺寸的纳米粒子精确定位到折纸表面,得到的纳米复合结构其紫外可见光谱从521 nm迁移到526 nm,说明了在组装过程中相邻金纳米之间发生了等离子体共振耦合。Pal等60还利用类似的方法组装单分散的银纳米粒子,得到具有可靠产率的银纳米粒子复合折纸结构,包括单分散、二聚体、三聚体银纳米粒子和金银杂交的复合折纸结构。类似的,Weller等61以DNA折纸结构为模板,将一个40 nm的金纳米颗粒和一个40 nm的银纳米颗粒固定在DNA折纸结构上(图5a),调控两个纳米粒子之间的间隙,有助于研究金银电子间的直接耦合作用。Shen等62在长方形折纸结构的特定位点延伸出捕捉链DNA来固定金纳米颗粒,并通过折叠链将长方形DNA折纸结构进行卷曲粘合形成DNA管状结构,金纳米粒子也随之变成具有螺旋结构的三维立体结构(图5b)。
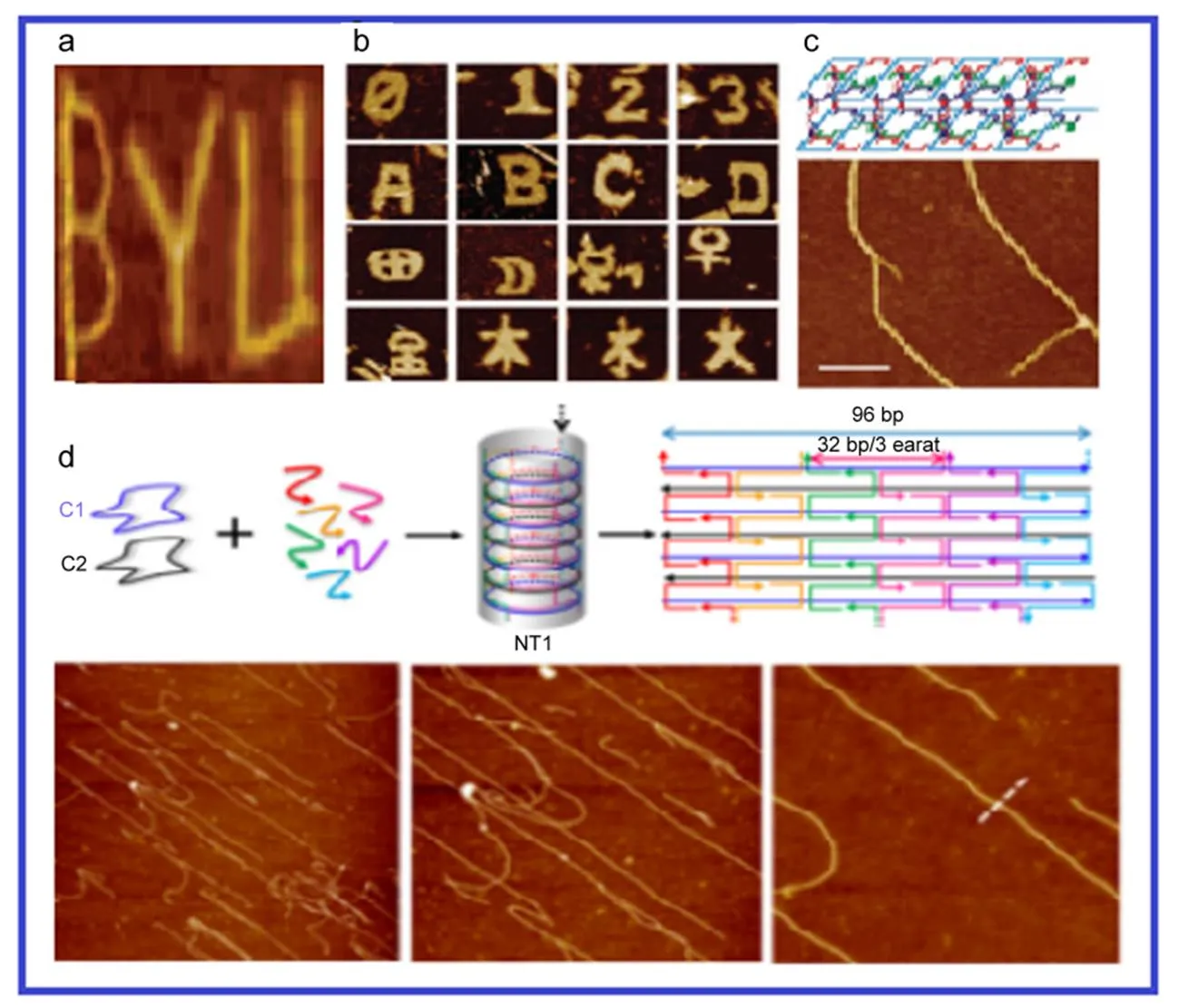
图4 新型DNA折纸术构造的纳米结构
(a) Nanoalphabet “BYU”46; (b) Complex shapes designed using a molecular canvas47; (c) The nanowires made by RCA product48; (d) The nanotube made by ring DNA51.
这种三维螺旋金纳米粒子组装结构在纳米金的等离子体共振波长处具有明显的圆二色信号。Urban等63还在直径为120 nm的环状的折纸结构上镶嵌上13 nm的金纳米粒子,制备出两个手性环(图5c),且这两个手性环状复合结构可产生相反的圆二色性光谱。这样的异二聚体结构Wang等64结合模块DNA组装和DNA折纸技术,设计出DNA折纸瓦片,并利用瓦片连接成有序的二维DNA阵列和DNA纳米管,在二维阵列和纳米管上巧妙地将金纳米粒子镶嵌在单体瓦片的中心(图5d),制备出具有光学性质的超级材料。DNA不仅能够有效地结合和固定无机离子,其高效性更令人为之一惊。Shi等65发明了一种极其高效的DNA与金纳米粒子连接方法,一定条件下,DNA与金纳米棒的有效连接可以在短短5 min内完成。
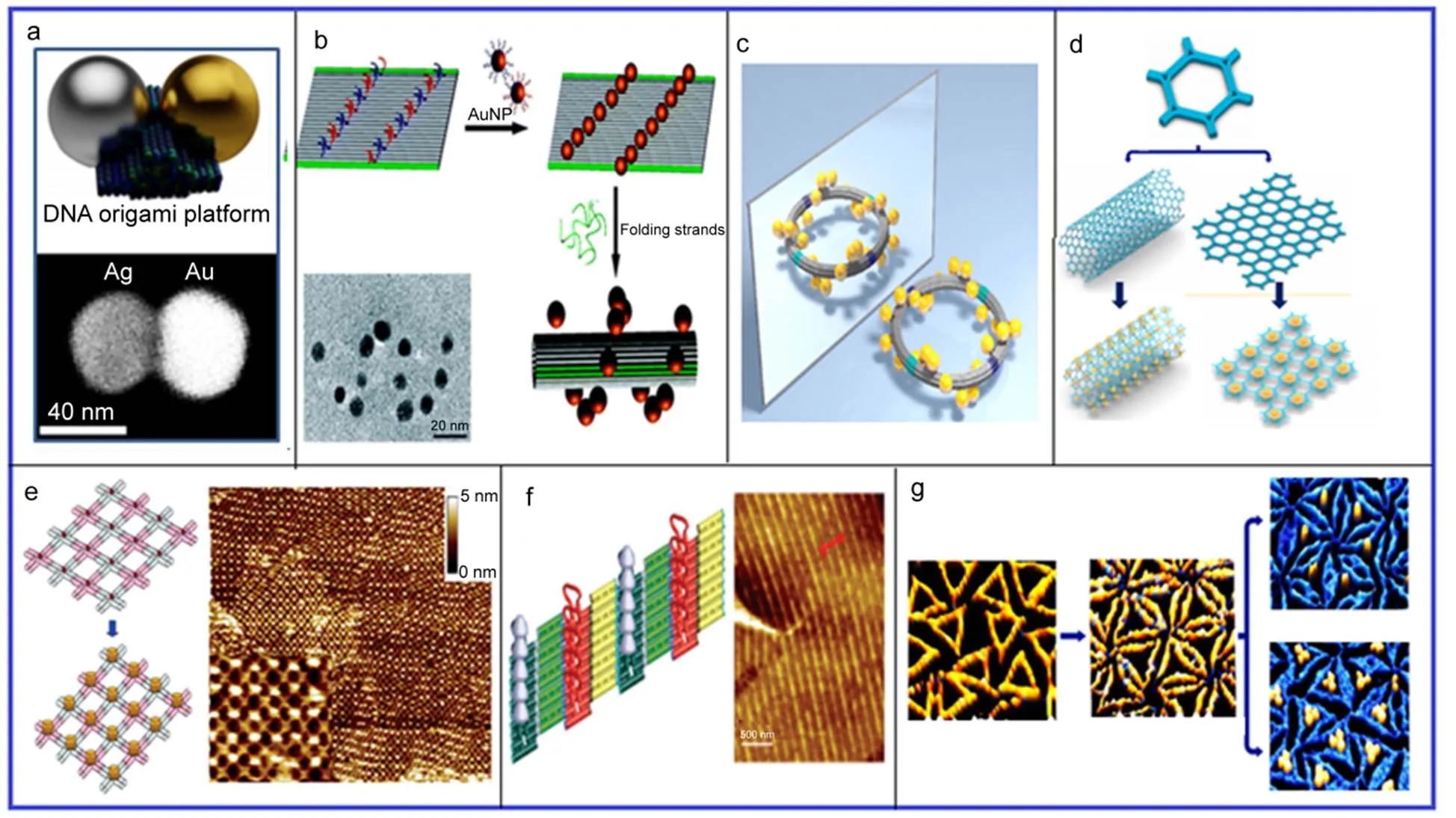
图5 DNA纳米结构组装纳米粒子
(a)DNA Origami-Based Ag−Au heterodimer61; (b) Rolling up gold nanoparticle-dressed DNA origami into chiral nanotube62; (c) Chiral DNA origami ring63; (d) programming self-assembly of DNA-origami honeycomb lattices and microscale AuNP superlattices fabricated from DNA-origami 2D lattices or tubes64; (e) Isstreptavidin bound to the bare DNA nanogrids66; (f) Periodic 2D multiprotein nanoarrays67; (g) The protein is localized in the origami structure and the mica gap68.
在纳米级别精确控制生物大分子位点,如精密控制蛋白的位置,有助于我们对蛋白质相互作用的认识以及酶反应动力学和机理的研究21。Park等66用组装的DNA网格作为模板,通过在瓦片上预先绑定的生物素来有效编排和定位链霉亲和素(图5e),使其有规律的在DNA组装体上均匀排布。随后,该课题组67还在DX模块组装体的二维阵列上通过血小板源性生长因子(PDGF)的适配体定位血小板源性生长因子(图5f)。Ramakrishnan等68使用单层有序排列的DNA折纸纳米结构与云母片表面形成的空腔作为分子光刻定位蛋白质的载体(图5g)。通过控制缓冲液和吸附条件,暴露区域的蛋白质覆盖度可以从单一蛋白质变化到紧密包裹的单层蛋白质。为了证明该方法的多功能性,研究者们尝试了四种不同蛋白质。他们进一步证明了在蛋白质定向吸附之后,DNA折纸结构可以脱附下来,这种性能能够有效实现不同种类的蛋白质组成分层结构。因为吸附的选择性是通过蛋白质和暴露的区域之间的静电作用实现,所以该方法还可以胜任其它带电分子甚至纳米颗粒的大规模图案化。通过对有序周期性阵列的调控,可极大推动分子生物学和细胞生物学的发展。对于深入了解生物分子间地相互作用、基本规律以及实现仿生合成都具有重要意义。
3.2 DNA纳米结构在生物医药领域的应用
人类医疗水平虽迅速提升,但对于癌症的检测和治疗仍不尽如人意。检测的时效性和灵敏性、治疗的高效性以及靶向性都迫切需要提升。因此,开发有效地检测和治疗方法是人类在生物医疗面临的重大挑战。目前,一些纳米颗粒69−72由于较强的渗透力和滞留效应已经被证实具有较好的抗癌和检测作用,但是其安全性仍存在质疑73。相比较而言,DNA作为天然分子,具有无法比拟的生物相容性,加上DNA纳米技术的迅速发展,自组装DNA纳米结构可为多种功能组件提供平台74−76,诸如可输送肿瘤靶向配体77,小干扰RNA(siRNA)78,小分子药物79,80,等等。因此,DNA是开发多功能型检测和载药工具的良好材料。
Bhatia及其同事81制备一个DNA二十面体,类似一个胶囊(图6a)。并利用其特有的空腔负载FITC-葡萄聚糖。相比于单纯的FITC-葡萄聚糖,该负载胶囊可以被细胞特异性的内吞,也就是说FITC-葡萄聚糖可以被这个DNA胶囊运送到特定的受体表达细胞。更为重要的是,这个负载胶囊可以被秀丽隐杆线虫细胞内吞,并能反应细胞内的pH环境,秀丽隐杆线虫胞内溶酶体的大量成熟会引起pH的变化,该负载胶囊从而检测秀丽隐杆线虫胞内溶酶体的成熟情况。Jiang等82合成了一种多臂DNA四面体纳米结构(TDNs) (图6b),并利用近红外(NIR)荧光和单光子发射计算机断层扫描(SPECT)将该四面体用于双模态体内成像。研究者们发现TDN结构中臂链的存在可以显着增强它们在体外的稳定性,使其在血清中可完整保持至少12 h。通过使用TDN上不同检测功能的多臂结构,可以精确地检测功能基团的数量,从而对癌细胞具有靶向成像能力,以此实现对癌细胞的靶向检测。
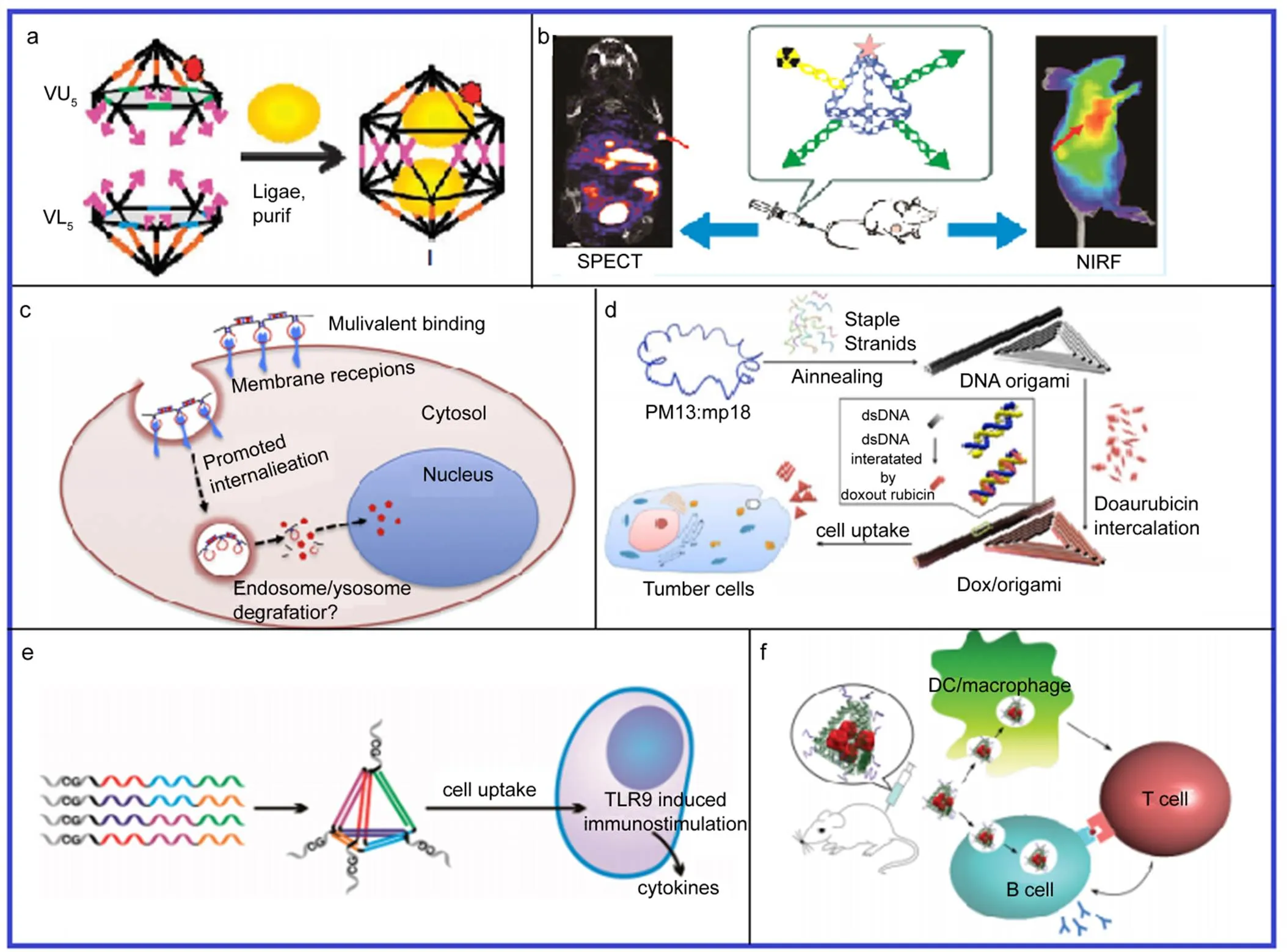
图6 DNA纳米结构在生物医药领域的应用
(a) Schematic illustration of the formation of FD10-loaded icosahedra81; (b) Schematic design of dual-modality imaging probe based on DNA nanostructures and it for tumor-targeting, dual-modalityimaging82; (c) Concept of making Poly-Aptamer-Drug and using it to target and kill cancer cells79; (d) DNA origami and doxorubicin origami delivery system assembly80; (e) The assembly of CpG bearing DNA tetrahedron and its immunostimulatory effect95; (f) Schematic design of the DNA scaffolded adjuvant-antigen vaccine complex96.
在癌症的治疗上,化疗仍然是主要手段之一,但化疗药物缺乏选择性,副作用大,因此,研究者们将焦点关注在靶向传递药物上83−88。在靶向性癌症治疗系统中,通常利用的是单价分子(例如抗体和寡核苷酸适配体)来识别靶向分子和癌症细胞的受体89−93。这种方法仍然存在定向性差、细胞内化的效率低等缺点。张志庆等79开发了一种新形式的多价适配体载药体系(Poly-Aptamer-Drug),通过滚环扩增方法扩增出具有多个适配体重复单元(图6c),并通过物理方法插入化学药物,利用白血病适配体和阿霉素作为一个模型系统,证明多价适配体载药体系相比于单价分子载药体系因其较强的亲和力,能够高效的被细胞内化可有效应用于靶向治疗癌症,能够减少化疗的副作用。随后,张志庆与其同事94又制备合成了一个多功能的多价适配体系统,该系统具有特异的调节细胞表面收集信息功能,更能显著的诱导目标癌细胞的凋亡。此外,Jiang及同事80通过非共价键作用将阿霉素固定到三角形和管状的DNA折纸结构上(图6d),得到了一个高效的载药体系,这种载药体系不仅能够有效地进入普通的人类乳腺癌细胞,还能够顺利进入对阿霉素产生抗药性的癌症细胞,更能够抵制产生抗药性的癌症细胞。
除了在癌症的诊断和治疗方面具有独特的应用之外,DNA纳米技术在免疫疗法方面的应用也逐渐引起人们的重视。上海应用物理研究所的樊春海课题组95构建了一种功能化的DNA四面体(图6e),并在上面加载了四个未甲基化CpG基元,这种纳米结构具有良好的机械稳定性和生物相容性,它可以有效地进入活的巨噬细胞样-RAW264.7细胞中去,有效地产生免疫活性。该课题组还利用滚环扩增技术制备出的长链DNA作为折纸结构的脚手架链制出纳米带,该纳米带不仅可以进入活细胞,带有CpG的纳米带还可以有效地被哺乳细胞内吞47,并产生免疫刺激。Liu等96通过将抗原嵌入组装的DNA四面体结构的空腔中(图6f),合成一种免疫疫苗,在外部CpG的辅助作用下,这种疫苗在测试小鼠体内表现出强烈并持久的抗体反应。
3.3 其他方面的应用
随着微电子技术的发展,对器件的要求也随之苛刻,DNA纳米技术作为新的制备纳米材料的方法,在纳米芯片制备方面具有巨大的潜在应用价值。通过将DNA折纸术功能化,使用金属包裹DNA,从而实现DNA的金属化,获得纳米级电路,制备出纳米级的金属导线97,98。Ke等99,100以长方形的DNA折纸结构为基底,制作出第一张具有探测RNA序列功能的折纸术芯片。
化学和生物反应过程复杂,倘若能够监测其单分子反应过程将会对人们推断反应机理带来极大的帮助。Voigt等101发现在DNA折纸结构上可检测出单个分子的化学反应。研究者们利用DNA折纸术的可寻址性与分子间化学键的合成与断裂,成功地使某些特殊结构能够按人为设计的轨道“行走”。用矩形的折纸结构作为基板,通过加入二硫苏糖醇、四溴荧光素和炔属烃、胺叠氮化物促进生物素-霉亲合素的断键与成键,通过原子力显微镜可观察到DNA折纸结构表面化合物的分解和合成。这一方法为探究单分子反应提供一大思路。
一直以来,表面化学反应吸引了研究者们广泛的关注。相比于同种体系来说,固体表面能为产物的反应途径产生诸多影响。Wang等102基于DNA纳米结构为模板来合成荧光银纳米簇,通过改变银纳米簇在DNA纳米结构上的附着位点不同,探究其对荧光性质的影响。这为合成形状控制的金属纳米结构提供了一种简单的方法,也探究表面化学反应的空间效应机制提供了新的思路。
4 结论与展望
DNA纳米技术自上个世纪九十年代发展以来,从最初的简单模块化组装到多样化的折纸结构,DNA纳米技术虽然取得了巨大进展,但仍有很多科学问题亟待解决。首先,构造出丰富多样的纳米结构虽已实现,但人们对于DNA的组装机理仍不十分明确,组装过程中的热力学和动力学问题仍不清晰。正因为组装机理的不明确,组装过程中的错配问题难以解决,也很难得到稳定的高产率。因此探索组装机理,获取高产率、稳定的组装体是目前仍需攻克的难题。其次,DNA纳米技术虽已应用到生物医药、电子科学等领域,但DNA纳米结构高昂的制备成本制约着DNA纳米技术的实际应用。因此,开发更加廉价便捷的DNA扩增手段是需要应对的又一挑战。再次,如何开发和构建出能够应对复杂的生物环境的DNA纳米器件,有效地完成装载、运输和卸载大分子,真正实现自然界中的生物马达,从而将DNA纳米器件真正被应用到临床医学上,这仍然需要研究者的进一步探索和解决。尽管如此,DNA纳米技术仍是极具潜力的发明,我们有理由相信,DNA纳米技术的发展将会为人类在DNA芯片、纳米器械、生物医药等方面带来新的光明,也必定推动相关领域学科的进步。
(1) Watson, J. D.; Crick, F. H. C.1953,(4361), 737. doi: 10.1038/171964b0
(2) Zadegan, R. M.; Norton, M. L.2012,(6), 7149. doi: 10.3390/ijms13067149
(3) Seeman, N. C.. 2010,(79), 65. doi: 10.1146/annurev-biochem-060308-102244
(4) Yang, D. Y.; Hartman, M. R.; Derrien, T. L.; Hamada, S.; An, D.; Yancey, K. G.; Cheng, R.; Ma, M, L.; Luo, D. R.2014,(6), 1902. doi: 10.1021/ar5001082
(5) Lu, C. H.; Willner, B.; Willner, I.2013,(10), 8320. doi: 10.1021/nn404613v
(6) Suzuki, Y.; Endo, M.; Sugiyama, H.2015, 9 (4), 3418. doi: 10.1021/acsnano.5b01723
(7) Aldaye, F. A.; Palmer, A. L.; Sleiman, H. F.2008,(5897), 1795. doi: 10.1126/science.1154533
(8) Harriman, A.; Kubo, Y. J.; Sessler, L.1992,(1), 388. doi: 10.1021/ja00027a074
(9) Tan, S. J.; Campolongo, M. J.; Luo, D.; Cheng, W. L.2011,(5), 268. doi: 10.1038/nnano.2011.49.
(10) Zenk, J.; Tuntivate, C.; Schulman, R. J.2016, 138 (10), 3346. doi:10.1021/jacs.5b10502
(11) Zhang, F.; Nangreave, J.; Liu, Y.; Yan, H.2014,(32),11198. doi: 10.1021/ja505101a
(12) Chen, J. H.; Seeman, N. C.1991,(6319), 631. doi: 10.1038/350631a0
(13) Shih, W. M.; Quispe, J. D.; Joyce, G. F.2004,(6975), 618. doi: 10.1038/nature02307
(14) Goodman, R. P.; Heilemann, M.; Doose, S.; Erben, C. M.; Kapanidis, A. N.; Turberfield, A. J.2008,(2), 93. doi: 10.1038/nnano.2008.3
(15) Aldaye, F. A.; Sleiman, H. F.. 2006,(14), 2204. doi: 10.1002/anie.200502481
(16) Aldaye, F. A.; Sleiman, H. F.2007,(14), 4130. doi: 10.1021/ja070017i
(17) Aldaye, F. A.; Sleiman, H. F.2007,(44), 13376. doi: 10.1021/ja075966q
(18) He, Y.; Ye, T.; Su, M.; Zhang, C.; Ribbe, A. E.; Jiang, W.; Mao, C. D.2008,(7184), 198. doi: 10.1038/nature06597
(19) Zhang, C.; Su, M.; He, Y.; Zhao, X.; Fang, P. A.; Ribbe.; A. E.; Jiang, W.; Mao, C. D.2008,(31), 10665. doi: 10.1073/pnas.0803841105
(20) Seeman, N. C.. 1982, 99 (2), 237. doi:10.1016/0022-5193(82)90002-9
(21) Seeman, N. C.2003,(6921), 427. doi: 10.1038/nature01406
(22) Fu, T. J.; Seeman, N. C.1993,(13), 3211. doi: 10.1021/bi00064a003
(23) Li, X. J.; Yang, X. P.; Qi, J.; Seeman, N. C.. 1996,(26), 6131. doi: 10.1021/ja960162o
(24) Winfree, E.; Liu, F.; Wenzler, L. A.; Seeman, N. C.1998,(6693), 539. doi: 10.1038/28998
(25) Liu, F.; Ruojie Sha, A.; Seeman, N. C.1999,(5), 917. doi: 10.1021/ja982824a
(26) Lin, C. X.; Liu, Y.; Rinker, S.; Yan, H.. 2006,(8), 1641. doi: 10.1002/cphc.200600260
(27) Labean, T. H.; Yan, H.; Kopatsch, J.; Liu, F. R.; Winfree, E.; Reif, J. H.; Seeman, N. C.. 2000,(9), 1848. doi: 10.1021/ja993393e
(28) Reishus, D.; Shaw, B.; Brun, Y.; Chelyapov, N.; Adleman, L... 2005,(50), 17590. doi: 10.1021/ja0557177
(29) Mathieu, F.; Liao, S.; Kopatsch, J.; Wang, T.; Mao, C. D.; Seeman, N. C.. 2005,(4), 661. doi: 10.1021/nl050084f
(30) Yan, H.; Park, S. H.; Finkelstein, G.; Reif, J. H.; LaBean, T. H.2003,(5641), 1882. doi: 10.1126/science.1089389
(31) Liu, D. G.; Wang, M. S.; Deng, Z. X.; Walulu, R.; Mao, C. D.2004,(8), 2324. doi: 10.1021/ja031754r
(32) He, Y.; Chen, Y.; Liu, H. P.; Ribbe, A. E.; Mao, C. D.. 2005,(35), 12202. doi: 10.1021/ja0541938
(33) He, Y.; Tian, Y.; Ribbe, A. E.; Mao, C. D.. 2006,(50), 15978. doi: 10.1021/ja0665141
(34) He, Y.; Tian, Y.; Chen, Y.; Deng, Z.; Ribbe, A. E.; Mao, C. D.2005,(41),6694. doi: 10.1002/anie.200502193
(35) Rothemund, P. W.2006,(7082), 297. doi :10.1038/nature04586
(36) Castro, C. E.; Kilchherr, F.; Kim, D. N.; Shiao, E. L.; Wauer, T.; Wortmann, P.; Bathe, M.; Dietz, H.2011,(3), 221. doi: 10.1038/nmeth.1570.
(37) Lin, C. X.; Liu, Y.; Yan, H.2009,(8), 1663. doi: 10.1021/bi802324w
(38) Qian, L. L.; Wang, Y.; Zang, Z.; Zhao, J.; Pan, D.; Zhang, Y.; Liu, Q.; Fan, C. H.; Hu, J.; He, L. 2006,(24), 2973. doi: 10.1007/s11434-006-2223-9
(39) Andersen, E. S.; Dong, M.; Nielsen, M. M.; Jahn, K.; Lind-Thomsen, A.; Mamdouh, W.; Gothelf, K. V.; Besenbacher, F.; Kjems, J.. 2008,(6), 1213. doi: 10.1021/nn800215j
(40) Andersen, E. S.; Dong, M.; Nielsen, M. M.; Jahn, K.; Subramani, R.; Mamdouh, W.; Golas, M. M. Sander, B.; Stark, H.; Oliveira, C. L.; Pedersen, J. S.; Birkedal, V.; Besenbacher, F.; Gothelf, K. V.; Kjems, J.2009,(7243), 73. doi: 10.1038/nature07971
(41) Ke, Y.; Sharma, J.; Liu, M.; Jahn, K.; Liu, Y.; Yan, H.2009,(6), 2445. doi: 10.1021/nl901165f
(42) Douglas, S. M.; Dietz, H.; Liedl, T.; Hogberg, B.; Graf, F.; Shih, W, M.2009,(7245), 414. doi: 10.1038/nature08016
(43) Han, D. R.; Pal, S.; Liu, Y.; Yan, H.2010,(10), 712. doi: 10.1038/nnano.2010.193
(44) Han, D.; Pal, S.; Nangreave, J.; Deng, Z.; Liu, Y.; Yan, H.2011,(6027), 342. doi: 10.1126/science.1202998
(45) Tigges, T.; Heuser, T.; Tiwari, R.; Walther, A.2016,(12), 7870. doi: 10.1021/acs.nanolett.6b04146
(46) Pound, E.; Ashton, J. R.; Becerril, H. A.; Woolley, A. T.2009,(12), 4302. doi: 10.1021/nl902535q
(47) Wei, B.; Dai, M.; Yin, P.2012,(7400), 623. doi: 10.1038/nature11075
(48) Ouyang, X. Y.; Li, J.; Liu, H. J.; Zhao, B.; Yan, J.; Ma, Y. Z.; Xiao, S. J.; Song, S. P.; Huang, Q.; Chao, J.; Fan, C. H.2013,(18), 3082. doi: 10.1002/smll.201300458
(49) Dean, F. B.; Nelson, J. R.; Giesler, T. L.; Lasken, R. S.2012,(1), 146. doi: 10.1021/ja209861x
(50) Schweitzer, B.; Roberts, S.; Grimwade, B.; Shao, W. P.; Wang, M. J.; Fu, Q.; Shu, Q. P.; Laroche, I.; Zhou, Z. M.; Tchernev, V. T.; Christiansen, J.; Velleca, M.; Kingsmore, S. F.. 2002,(4), 359. doi: 10.1038/nbt0402-359
(51) Zheng, H.; Xiao, M.; Yan, Q.; Ma, Y.; Xiao, S. J.2014,(29), 10194. doi: 10.1021/ja504050r
(52) Wang, X.; Zhuang, J.; Peng, Q.; Li, Y. D.2005,(7055), 121. doi: 10.1038/nature03968
(53) Lim, D. K.; Jeon, K. S.; Hwang, J. H.; Kim, H.; Kwon, S.; Suh, Y. D.; Nam, J. M.2011,(7), 452. doi: 10.1038/nnano.2011.79.
(54) Lim, D. K.; Jeon, K. S.; Kim, H.; Nam, J. M.; Suh, Y. D.2010,(1), 60. doi: 10.1038/nmat2596
(55) Sawai, Y.; Takimoto, B.; Nabika, H.; Ajito, K.; Murakoshi, K.. 2007,(6), 1658. doi: 10.1021/ja067034c
(56) Sönnichsen, C.; Reinhard, B. M.; Liphardt, J.; Alivisatos, A. P.2005,(6), 741. doi: 10.1038/nbt1100
(57) Luk'yanchuk, B.; Zheludev, N. I.; Maier, S. A.; Halas N. J.; Nordlander, P.; Giessen, H.; Chong, C. T.2010,(9), 707. doi:10.1038/nmat2810
(58) Mayer, K. M.; Hafner, J. H.2011,(6), 3828. doi: 10.1021/cr100313v
(59) Ding, B. Q.; Deng, Z. T.; Yan, H.; Cabrini, S.; Zuckermann, R. N.; Bokor, J.2010,(10),3248. doi: 10.1021/ja9101198
(60) Pal, S.; Deng, Z.; Ding, B.; Yan, H.; Liu, Y.2010,(15), 2700. doi: 10.1002/anie.201000330
(61) Weller, L.; Thacker, V.; Herrmann, L.; Hemmig, E.; Lombardi, A.; Keyser, U.; Baumberg, J.2016,(9), 1589. doi: 10.1021/acsphotonics.6b00062
(62) Shen, X.; Song, C.; Wang, J. Y.; Shi, D. W.; Wang, Z. G.; Liu, N.; Ding, B. Q.2012,(1), 146. doi: 10.1021/ja209861x
(63) Urban, M. J.; Dutta, P. K.; Wang, P. F.; Duan X. Y,.; Shen X. B.; Ding, B. Q.; Ke, Y. G.; Liu N.. 2016,(17), 5495. doi: 10.1021/jacs.6b00958
(64) Wang, P. F.; Gaitanaros, S.; Lee, S.; Bathe, M.; Shih, W. M.; Ke, Y. G.2016,(24),7733. doi: 10.1021/jacs.6b03966.
(65) Shi, D. W.; Song, C.; Jiang, Q.; Wang, Z. G.; Ding, B. Q.2013,(25), 2533. doi: 10.1039/c3cc39093d
(66) Park, S. H.; Yin, P.; Liu, Y.; Reif, J. H.; LaBean, T. H.; Yan, H.2005,(4), 729. doi: 10.1021/nl050175c
(67) Chhabra, R.; Sharma, J.; Ke, Y.; Liu, Y.; Rinker, S.; Lindsay, S.; Yan, H.. 2007,(34), 10304. doi: 10.1021/ja072410u
(68) Ramakrishnan, S.; Subramaniam, S.; Stewart, A. F.; Grundmeier, G.; Keller, A.2016,, (45), 31239. doi: 10.1021/acsami.6b10535
(69) Maeda, H.; Wu, J.; Sawa, T.; Matsumura, Y.; Hori, K.2000,(1−2), 271. doi: 10.1016/S0162-3109(99)00104-6
(70) Albanese, A.; Tang, P. S.; Chan, W. C.. 2012,(14), 1. doi: 10.1146/annurev-bioeng-071811-150124
(71) Farokhzad, O. C.; Langer, R.2009,(1), 16. doi: 10.1021/nn900002m
(72) Kim, C. K.; Ghosh, P.; Rotello, V. M.2009,(1), 61. doi: 10.1039/b9nr00112c
(73) Zhan, P.; Jiang, Q.; Wang, Z. G.; Li, N.; Yu, H.; Ding, B. Q.2014,(9), 2013. doi: 10.1002/cmdc.201402137
(74) Voigt, N. V.; Tørring, T.; Rotaru, A.; Jacobsen, M. F.; Ravnsbaek, J. B.; Subramani, R.; Mamdouh, W.; Kjems, J.; Mokhir, A.; Besenbacher, F.; Gothelf, K. V.2010,(3), 200. doi: 10.1038/nnano.2010.5
(75) Rusling, D. A.; Chandrasekaran, A. R.; Ohayon, Y. P.; Brown, T.; Fox, K. R.; Sha, R.; Mao ,C. D.; Seeman, N. C.. 2014,(15), 3979. doi: 10.1002/anie.201309914
(76) Wei, B. Y.; Dai, M. J.; Yin, P.2012,(7400), 623. doi: 10.1038/nature11075
(77) Chang, M.; Yang, C. S.; Huang, D. M.2011,(8), 6156. doi: 10.1021/nn200693a
(78) Lee, H.; Lytton-Jean, A. K.; Chen, Y.; Love, K. T.; Park, A. I.; Karagiannis, E. D.; Sehgal, A.; Querbes, W.; Zurenko, C. S.; Jayaraman, M.; Peng, C. G.; Charisse, K.; Borodovsky, A.; Manoharan, M.; Donahoe, J. S.; Truelove, J.; Nahrendorf, M.; Langer, R.; Anderson, D. G.2012,(6), 389. doi: 10.1038/nnano.2012.73
(79) Zhang, Z. Q.; Ali, M. M.; Eckert, M. A.; Kang, D. K.; Chen, Y. Y.; Sender, L. S.; Fruman, D. A.; Zhao, W. A.2013,(37), 9728. doi: 10.1016/j.biomaterials.2013.08.079
(80) Jiang, Q.; Song, C.; Nangreave, J.; Liu, X. W.; Lin, L.; Qiu, D. L.; Wang, Z. G.; Zou, G. Z.; Liang, X. J.; Yan, H.; Ding, B. Q.2012,(32), 13396. doi: 10.1021/ja304263n
(81) Bhatia, D.; Surana, S.; Chakraborty, S.; Koushika, S. P.; Krishnan, Y.2011,(2), 339. doi: 10.1038/ncomms1337
(82) Jiang, D. W.; Sun, Y. H.; Li, J.; Li Q.; Lv, M.; Zhu, B.; Tian, T.; Cheng, D. F.; Xia, J, Y.; Zhang, L.; Wang L. H.; Huang, Q.; Shi, J. Y.; Fan, C. H. ACS.2016,(7), 4378. doi: 10.1021/acsami.5b10792
(83) Mallikaratchy, P. R.; Ruggiero, A.; Gardner, J. R.; Kuryavyi, V.; Maguire, W. F.; Heaney, M. L.; McDevitt, M. R.; Patel, D. J.; Scheinberg, D. A.2011,(6), 2458. doi: 10.1093/nar/gkq996
(84) Yang, L.; Meng, L.; Zhang, X.; Chen, Y.; Zhu, G.; Liu, H.; Xiong, X.; Sefah, K.; Tan, W.2011,(34), 13380. doi: 10.1021/ja201285y
(85) Peer, D.; Karp, J. M.; Hong, S.; Farokhzad, O. C.; Margalit, R.; Langer, R.2007,(12), 751. doi: 10.1038/nnano.2007.387
(86) Liu, Z.; Duan, J. H. Song, Y. M.; Ma, J.; Wang, F. D.; Lu, X.; Yang, X. D.2012,(1), 1. doi: 10.1186/1479-5876-10-148
(87) Xu, W.; Siddiqui, I. A.; Nihal, M.; Pilla, S.; Rosenthal, K.; Mukhtar, H.; Gong, S.2013,(21), 5244. doi: 10.1016/j.biomaterials.2013.03.006
(88) Hubbell, J. A.; Chilkoti, A.2012,(6092), 303. doi: 10.1016/B978-0-12-407796-6.00007-5
(89) Keefe, A. D.; Pai, S.; Ellington, A.. 2010,(7), 537. doi: 10.1038/nrd3141
(90) Bagalkot, V.; Farokhzad, O. C.; Langer, R.; Jon, S.2006,(48), 8149. doi: 10.1002/anie.200602251
(91) Huang, Y. F.; Shangguan, D.; Liu, H.; Phillips, J. A.; Zhang, X.; Chen, Y.; Tan, W.2009,(5), 862. doi: 10.1002/cbic.200800805
(92) Ray, P.; White, R. R.2010,(6), 1761.
doi: 10.3390/ph3061761
(93) Thiel, K.. 2004,(6), 649. doi: 10.1038/nbt0604-649
(94) Zhang, Z. Q.; Eckert, M. A.; Ali, M. M.; Liu, L.; Kang, D. K.; Chang, E.; Pone, E. J.; Sender, L. S.; Fruman, D. A.; Zhao, W. A.2014,(9), 1268. doi: 10.1002/cbic.201402100
(95) Li, J.; Pei, H.; Zhu, B.; Liang, L.; Wei, M.; He, Y.; Chen, N.; Li, D.; Huang, Q.; Fan, C. H.2011,(11), 8783. doi: 10.1021/nn202774x
(96) Liu, X. W.; Xu, Y.; Yu, T.; Clifford, C.; Liu, Y.; Yan, H.; Chang, Y.2012,(8), 4254. doi: 10.1021/nl301877k
(97) Yan, H.; Park, S. H.; Finkelstein, G.; Reif, J. H.; LaBean, T. H.2003,(5641), 1882. doi: 10.1126/science.1089389
(98) Liu, D.; Park, S. H.; Reif, J. H.; LaBean, T. H.2004,(3), 717. doi: 10.1073/pnas.0305860101
(99) Ke, Y. G.; Lindsay, S.; Chang, Y.; Liu, Y.; Yan, H.2008,(5860), 180. doi: 10.1126/science.1150082
(100) Ke, Y. G.; Nangreave, J.; Yan, H.; Lindsay, S.; Liu, Y.2008,(43), 5622. doi: 10.1039/b811332g
(101) Voigt, N. V.; Tørring, T.; Rotaru, A.; Jacobsen, M. F.; Ravnsbaek, J. B.; Subramani, R.; Mamdouh, W.; Kjems, J.; Mokhir, A.; Besenbacher, F.; Gothelf, K. V.2010,(3), 200. doi: 10.1038/nnano
(102) Wang, Z. G.; Liu, Q.; Li, N.; Ding, B. Q.. 2016,(23), 8834. doi: 10.1021/acs.chemmater.6b04150
Application of Structural DNA Nanotechnology
ZHANG Hong-Zhi ZHANG Zhi-Qing*WANG Fang ZHOU Ting WANG Xiu-Feng ZHANG Guo-Dong LIU Ting-Ting LIU Shu-Zhen
()
DNA possesses extraordinary molecular recognition properties and remarkable structural features in the nano-level material regulation fields, which have shown enormous applications in many areas. In this review, we focus on DNA nanotechnology, including aspects ranging from modular DNA self-assembly to DNA origami, in addition to the recently reported novel assembly methods. Moreover, we summarize some applications of DNA nanotechnology, such as DNA-directed nanoparticle spatial positioning and orientation, and well-defined assembly of proteins on the DNA structure, as well as its uses, such as in the biomedical field, etc. The development and potential applications of DNA nanotechnology are also discussed.
DNA nanotechnology; Self-assembly; DNA module; DNA origami; Targeted drug delivery
February 16, 2017;
April 7, 2017;
April 27, 2017.
Corresponding author. Email: zhangzq@upc.edu.cn; Tel: +86-532-86984568.
10.3866/PKU.WHXB201704271
O648
The project was supported by the Scientific Research Foundation for Returned Overseas Chinese Scholars, China (2014010615), National Natural Science Foundation of China (21603276, 21303267), Natural Science Foundation of Shandong Province, China (ZR2016BL14) and Fundamental Research Funds for the Central Universities, China (15CX05027A, 16CX05013A).
教育部留学回国人员科研启动基金(2014010615), 国家自然科学青年基金(21603276, 21303267), 山东省自然科学基金(ZR2016BL14), 中央高校基本科研业务费项目(15CX05027A, 16CX05013A)资助
