胸腺上皮肿瘤中CDH13基因启动子区甲基化检测的临床意义
2017-12-18陈晨陆璨陆前进
陈晨,陆璨,陆前进
(1.中南大学湘雅二医院 胸外科,湖南 长沙 410011;2.中南大学湘雅医院 口腔医学中心,湖南 长沙 410008;3.医学表观基因组学湖南省重点实验室,湖南 长沙 410011)
·论 著·
胸腺上皮肿瘤中CDH13基因启动子区甲基化检测的临床意义
陈晨1,陆璨2,陆前进3
(1.中南大学湘雅二医院 胸外科,湖南 长沙 410011;2.中南大学湘雅医院 口腔医学中心,湖南 长沙 410008;3.医学表观基因组学湖南省重点实验室,湖南 长沙 410011)
目的观察胸腺上皮肿瘤(TETs)组织中CDH13基因启动子区甲基化情况,探索CDH13基因启动子甲基化与TETs肿瘤发生进展之间的关系。方法收集胸腺上皮肿瘤组织,采用巢式甲基化特异性聚合酶链式反应(MSP)技术检测CDH13基因启动子区甲基化状态,并分析其与临床病理之间的联系。结果CDH13基因启动子区甲基化总检出率为37.8%;CDH13基因启动子区甲基化情况与患者性别、年龄以及是否合并重症肌无力无关(P >0.05);按照世界卫生组织(WHO)组织学分型,分化越差的TETs组织中CDH13基因启动子区甲基化发生率越高,大多数甲基化阳性结果均来自于B2/B3/C组,A/AB/B1组仅有少量甲基化阳性结果(P =0.002);按照Masaoka病理分期,I+II期肿瘤中CDH13基因启动子甲基化率明显低于III+IV 期(P =0.001)。结论CDH13基因启动子区的异常甲基化与TETs肿瘤的病理分期和组织学分型密切相关。
胸腺上皮肿瘤;甲基化;DNA;CDH13
胸腺上皮肿瘤(thymic epithelial tumors,TETs)是前纵隔常见的肿瘤之一,其在生物学和组织学上特性极为复杂。Masaoka等[1]首先提出按照肿瘤侵犯周围组织程度将TETs分为4期,1999年世界卫生组织(World Health Organization,WHO)组织提出TETs组织学分型,根据TETs内上皮细胞与淋巴细胞的数量相对比值,以及上皮细胞的异形性,将TETs分成6种亚型,这种分型方法很好的从组织学角度阐述了该疾病的特点。近年来的研究证实,Masaoka病理分期和WHO组织学分型均可以对疾病的预后判断提供帮助[2-3]。但也有学者研究发现某些良性TETs患者远期出现肿瘤转移,预后不佳,因而提出寻找有效的分子生物学标记物的必要性。
目前已有研究证明,肝癌、结肠癌、肺癌等多种肿瘤的发生发展与p16INK4a、CDH1、FHIT、RASSF1A和MGMT等抑癌基因启动子区异常甲基化有关,抑癌基因启动子区异常甲基化可以作为肿瘤发生进展的标志物[4-7],但在胸腺肿瘤中相关研究并不多见。CDH13基因编码的H-钙粘蛋白是一种重要的细胞间粘附分子,在生理条件下维持细胞与细胞之间、细胞与基质之间的粘附,使细胞维持极性,从而维持组织结构的形态和完整性。近期研究表明,CDH13在多种肿瘤转移时常表现为表达降低或失活,如乳腺癌、宫颈癌、膀胱癌、结肠癌、肺癌和前列腺癌等,且其失活的主要机制之一就是启动子的高甲基化[4,6,8-10]。这提示了CDH13失活在肿瘤侵袭过程中发挥的关键作用。本研究通过检测TETs组织中CDH13基因启动子区甲基化情况,探索CDH13基因启动子甲基化与TETs肿瘤发生进展之间的关系,对寻找TETs肿瘤诊断和预后分子标记物具有重要意义。
1 资料与方法
1.1 一般资料
选取2009年5月‐2014年6月本院手术切除的45例TETs标本为研究对象,所有标本均为石蜡标本。全部肿瘤组织均经病理诊断为胸腺上皮肿瘤,按照WHO胸腺肿瘤分类标准进行组织病理学分型,同时根据术中情况和影像学材料进行Masaoka病理分期,并记录病例一般资料。所有病例中,A型10例、AB型6例、B1型9例、B2型4例、B3型6例和C型10例;按Masaoka病理分期标准分为I期16例、II期12例、III期11例和IV期6例。所有肿瘤患者,男27例,女18例;年龄16~65岁,平均49.2岁。合并重症肌无力的患者术前均服用溴吡斯的明片或接受免疫抑制治疗,症状控制后接受手术治疗;所有患者术前均未接受化疗或放疗;本研究经过本院伦理委员会批准后实施,研究开始前受试者或其委托人已签署相关知情同意书。
1.2 检测方法
1.2.1 DNA样本提取和亚硫酸氢钠修饰 采用Promega 公司的MagneSil®Genomic,Fixed Tissue System试剂盒提取石蜡标本基因组DNA,步骤简述如下:石蜡标本连续切片6张,第一张和最后一张为4 μm,行苏木精-伊红染色法(hematoxylineosin staining,HE)染色,并作为模板;其余切片均为10 μm。显微镜下观察HE染色切片,确定肿瘤组织范围,收集该区域内的肿瘤细胞(每个病例收集4张病理切片);加入Incubation Buffer和Proteinase K溶液,56℃孵育过夜;加入磁珠,按照说明书抽提DNA;采用Qiagen公司的EpiTect bisulfite kit试剂盒对组织DNA进行亚硫酸氢钠修饰,获得可用于后续多聚酶链反应技术(polymerase chain reaction,PCR)扩增的DNA。

表1 CDH13巢式MSP引物序列
1.2.2 巢式甲基化特异性聚合酶链式反应(methylation specific PCR,MSP)分析 采用巢式MSP技术检测石蜡标本中CDH13基因甲基化情况,CDH13基因扩增引物由上海Invitrogen公司合成,具体引物序列见表1[7]。巢式PCR反应分为两轮,第一轮采用针对修饰后单链DNA设计的外侧引物,其产物大小相对较大,且第二轮PCR反应的DNA片段正好包含于其中;第二轮以第一轮PCR反应的产物为DNA模板,分别采用甲基化特异性和未甲基化特异性的引物进行扩增。第一轮PCR反应体系为25 μl,包括重亚硫酸氢钠修饰后的DNA模 板(50~100 ng)2 μl,10×Ex Taq Buffer(Mg2+Plus)2.5 μl,2.5 mmol/L dNTP Mixture 2 μl,TaKaRa Ex TaqTM HS(5 u/μl)0.5 μl,上下游引物各1 μl(100 ng),去离子水补齐至25 μl。反应条件:95℃变性5 min,94℃变性30 s,TM退火20 s,72℃延伸20 s,共循环40个周期,72℃延伸10 min,4℃时结束反应(Tm:退火温度)。
第一轮产物经1∶50稀释后作为第二轮PCR反应的DNA模板,进行甲基化特异性和未甲基化特异性扩增,PCR反应体系如下:包括稀释后的第一轮产物2 μl作为DNA模板,10×Ex Taq Buffer(Mg2+Plus)2.5 μl,2.5 mmol/L dNTP Mixture 2 μl,TaKaRa Ex TaqTM HS(5 u/μl)0.5 μl,特异性上下游引物各 1 μl(100 ng),去离子水补齐至25 μl。反应条件:95℃变性5 min,94℃变性30 s,Tm℃退火30 s,72℃延伸45 s,共循环35个周期,72℃延伸10 min,4℃时结束反应。
1.3 统计学方法
应用SPSS 20.0软件对数据进行统计学分析。两组间均数比较采用独立样本t检验;CDH13启动子区甲基化状态与临床病理参数的关系采用Wilcoxon秩和检验、χ2检验或Fisher′s确切概率法。P <0.05为差异有统计学意义。
2 结果
2.1 MSP结果分析
本研究采用巢式MSP技术,第一轮PCR反应外侧引物产物序列较大,第二轮PCR产物刚刚好包含于第一轮产物之中;第二轮PCR反应根据修饰后基因序列中甲基化与非甲基化的不同设计两对不同的引物,以第一轮PCR产物为样本DNA,分别进行甲基化特异性和未甲基化特异性扩增,进而判断DNA甲基化情况[11]。由于肿瘤组织中可能会带有一些正常细胞,因而巢式MSP结果中,同时具有甲基化引物扩增产物(M条带)和非甲基化引物扩增产物(U条带)者为启动子区甲基化的组织,而只有非甲基化引物扩增产物者(U条带)为启动子区未甲基化的组织,见图1。

图1 巢式MSP
在所有45例TETs中,CDH13基因启动子区甲基化总的检出率为37.8%,其中A型病例中无1例样本检测到CDH13基因启动子区甲基化;在AB型、B1型、B2、B3和C型病例检测到CDH13基因启动子区存在甲基化的检出率分别为16.67%(1/6)、33.33%(3/9)、50.00%(2/4)、66.67%(4/6)和70.00%(7/10)。
2.2 CDH13启动子区甲基化与临床病理特征之间的关系
如表2示,CDH13基因启动子区甲基化情况与患者性别、年龄以及是否合并重症肌无力(myasthenia gravis,MG)无关(P >0.05);按照WHO组织学分型,分化越差的TETs组织中CDH13基因甲基化发生率越高,大多数甲基化阳性结果均来自于B2/B3/C组,A/AB/B1组仅有少量甲基化阳性结果(P <0.05) ;按照 Masaoka病理分期,肿瘤分期越晚,CDH13基因甲基化发生率越频繁,Masaoka分期I+II期肿瘤中CDH13基因启动子甲基化率明显低于III+IV期(P <0.05)。
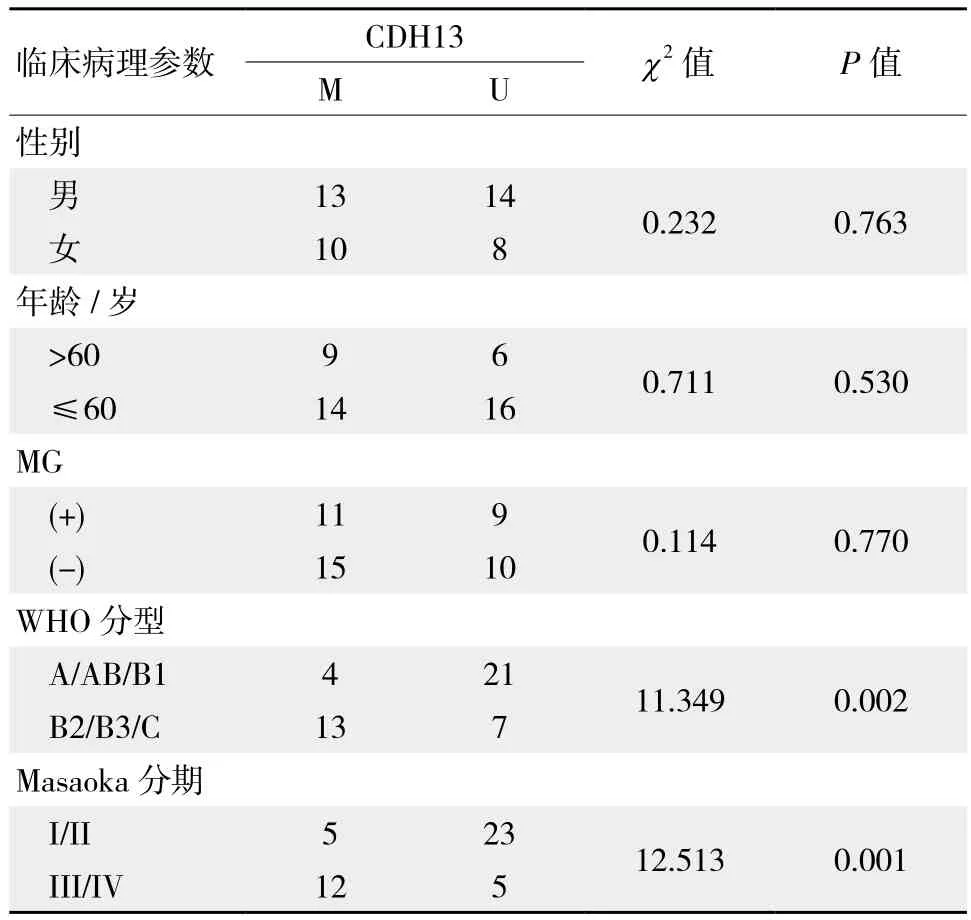
表2 CDH13启动子区甲基化与临床病理特征之间的关系 例
3 讨论
目前,对TETs的预后判断因素还存在着争议。Masaoka分期在肿瘤侵犯周围组织程度的基础上对TETs进行分期,可以较好的对预后做出判断,但该分期方法未考虑组织学因素,存在明显的局限性。WHO组织学分型按照肿瘤上皮细胞和淋巴细胞的比例及细胞异型性的大小,将TETs分成6种亚型,这种分型方法从组织病理学角度为TETs的分型及对预后的判断提供了良好的标准[2-3],但该方法过于复杂。有学者提出WHO分型中的A型和AB型可归属于良性TETs;B1型、B2型和B3型则归属为低度恶性TETs;C型为恶性TETs,也就是胸腺癌。但也有学者研究发现,部分B1型患者预后明显高于B2、B3型患者,提示B1型TETs相对于其他B亚型来说更倾向于良性TETs[12]。从组织病理学角度看,B1型TETs在结构上与正常胸腺组织相似,有类似正常的胸腺结构,细胞异型性小;而B2型则缺乏正常胸腺组织结构,细胞出现异型性;在B3型当中淋巴细胞成分极少,新生的异形上皮细胞成分占主导成分,通常也可以认为属于分化较好的胸腺癌。因而有人提出,将恶性程度较低的A型、AB型和B1型TETs病例分为一组,而将相对恶性程度较高的B2型、B3型和C型病例分为另一组,这种分型方法简化了WHO分型,为临床和病理学研究TETs提供了新的思路[13]。本研究中将病例分为A/AB/B1组和B2/B3/C两组,结果显示大多数CDH13甲基化阳性结果均来自于B2/B3/C组,A/AB/B1组仅有少量甲基化阳性结果,提示分化越差的TETs组织中CDH13基因甲基化发生率越高(P =0.002);按照Masaoka病理分期,I+II期TETs肿瘤中CDH13基因启动子甲基化率明显低于III+IV期TETs,提示肿瘤分期越晚CDH13基因甲基化发生越频繁(P =0.001)。
目前认为,肿瘤发生进展的分子生物学本质是细胞内遗传信息和表观遗传调控的紊乱[14-15]。近来研究发现,DNA甲基化与肿瘤的发生及发展密切相关,尤其是肿瘤抑癌基因启动子区域CpG岛的高甲基化,可以明显抑制基因的表达,导致基因失活。目前已证实抑癌基因启动子区高甲基化是多种肿瘤发生进展当中的重要事件,且抑癌基因启动子区高甲基化可以作为肿瘤早期诊断和复发检测的有效标记物[11,15]。
CDH13基因又称为上皮钙粘蛋白基因,定位于16q23.3,是一种重要的细胞间粘附分子。在生理条件下CDH13编码的H-钙粘蛋白可以调节细胞与细胞之间、细胞与基质之间的粘附反应,并维持细胞极性和参与分化调节,可以维持组织结构形态和完整性[8-9]。越来越多的研究表明,CDH13在肿瘤淋巴结转移时常表现为表达降低或失活,H-钙粘蛋白表达下降,且其失活的主要机制之一就是启动子的高甲基化。在关于乳腺癌、宫颈癌、膀胱癌、结肠癌和肺癌等肿瘤转移的研究中发现,有淋巴结转移的肿瘤与无淋巴结转移的肿瘤中CDH13表达有显著差异,伴有淋巴结转移者多表达降低,而在CDH13表达降低的肿瘤中,骨和肺转移率也较高,提示CDH13与肿瘤的转移密切相关[4,6-10]。TETs是一种侵袭力很强的肿瘤,本研究中发现在分化较差B2、B3和C型与分期较晚III和IV期的TETs中CDH13的启动子区甲基化率明显上升,这提示了CDH13基因启动子区甲基化在TETs肿瘤侵袭过程中发挥的关键作用。
甲基化特异性PCR(MSP)是目前公认的检测DNA甲基化的有效方法,该方法利用亚硫酸盐处理DNA后序列产生差异的特点,设计两对引物,即甲基化特异性PCR引物和未甲基化特异性PCR引物,分别对不同甲基化状态下的DNA序列进行扩增,从而对甲基化进行检测。MSP方法特异性较高,其缺点是亚硫酸盐处理DNA时会有大量DNA发生降解,对于石蜡切片中的小片段、微量DNA检测灵敏度欠佳。本研究参考国外文献设计了巢式MSP检测办法,采用两轮PCR,极大的提高了检测的灵敏度,即使仅存在微量DNA也可良好的进行扩增反应[5]。
综上所述,本研究提示CDH13启动子区甲基化在TETs肿瘤发生进展过程中扮演重要作用,采用巢式MSP技术检测TETs石蜡组织样本中CDH13启动子区甲基化具有高特异性、高灵敏度,且对临床有重要的应用价值。
[1]Okumura M,Ohta M,Tateyama H,et al.The World Health Organization histologic classification system reflects the oncologic behavior of thymoma:a clinical study of 273 patients[J].Cancer,2002,94(3):624-632.
[2]Kondo K,Monden Y.Therapy for thymic epithelial tumors:a clinical study of 1,320 patients from Japan[J].The Annals of Thoracic Surgery,2003,76(3):878-884,discussion 884-875.
[3]Kim BK,Cho BC,Choi HJ,et al.A single institutional experience of surgically resected thymic epithelial tumors over 10 years:clinical outcomes and clinicopathologic features[J].Oncology Reports,2008,19(6):1525-1531.
[4]Abudukadeer A,Bakry R,Goebel G,et al.Clinical relevance of CDH1 and CDH13 DNA-methylation in serum of cervical cancer patients[J].International Journal of Molecular Sciences,2012,13(7):8353-8363.
[5]Brock MV,Hooker CM,Ota-Machida E,et al.DNA methylation markers and early recurrence in stage I lung cancer[J].The New England Journal of Medicine,2008,358(11):1118-1128.
[6]Guo Q,Wang HB,Li YH,et al.Correlations of promoter methylation in WIF-1,RASSF1A,and CDH13 genes with the risk and prognosis of esophageal cancer[J].Medical Science Monitor International Medical Journal of Experimental and Clinical Research,2016,22:2816-2824.
[7]Kim DS,Kim MJ,Lee JY,et al.Aberrant methylation of E-cadherin and H-cadherin genes in nonsmall cell lung cancer and its relation to clinicopathologic features[J].Cancer,2007,110(12):2785-2792.
[8]Pu W,Geng X,Chen S,et al.Aberrant methylation of CDH13 can be a diagnostic biomarker for lung adenocarcinoma[J].Journal of Cancer,2016,7(15):2280-2289.
[9]Yang J,Niu H,Huang Y,et al.A systematic analysis of the relationship of CDH13 promoter methylation and breast cancer risk and prognosis[J].PloS one,2016,11(5):e0149185.
[10]Ye M,Huang T,Li J,et al.Role of CDH13 promoter methylation in the carcinogenesis,progression,and prognosis of colorectal cancer:a systematic meta-analysis under PRISMA guidelines[J].Medicine,2017,96(4):e5956.
[11]Chen C,Yin B,Wei Q,et al.Aberrant DNA methylation in thymic epithelial tumors[J].Cancer Investigation,2009,27(5):582-591.
[12]Nakagawa K,Asamura H,Matsuno Y,et al.Thymoma:a clinicopathologic study based on the new World Health Organization classification[J].The Journal of Thoracic and Cardiovascular Surgery,2003,126(4):1134-1140.
[13]Chen G,Marx A,Chen WH,et al.New WHO histologic classification predicts prognosis of thymic epithelial tumors:a clinicopathologic study of 200 thymoma cases from China[J].Cancer,2002,95(2):420-429.
[14]Feinberg AP.A genetic approach to cancer epigenetics[J].Cold Spring Harbor Symposia on Quantitative Biology,2005,70:335- 341.
[15]Laird PW.Cancer epigenetics[J].Human Molecular Genetics,2005,14(suppl1):65-76.
Clinical significance of CDH13 gene promoter methylation in thymic epithelial tumors
CHEN Chen1,LU Can2,LU Qianjin3
(1.Department of Thoracic Surgery,the Second Xiangya Hospital of Central South University,Changsha,Hunan 410011,China; 2.Department of Stomatology,Xiangya Hospital of Central South University,Changsha,Hunan 410008,China; 3.Hunan Key Laboratory of Medical Epigenomics,Changsha,Hunan 410011,China)
【Objective】To investigate the promoter methylation of CDH13 status in patients with thymic epithelial tumors(TETs).【Methods】Nested methylation-specific polymerase chain reaction (nMSP) was performed to evaluate the promoter region methylation patterns of CDH13 in TETs.The correlation between CDH13 promoter methylation and clinicopathological data were analyzed.【Results】CDH13 promoter hypermethylation was detected in 37.8% of the patients with TETs.The methylation rate of CDH13 was significantly higher in B2/B3/C group and stage III+IV group,compared to A/AB/B1 group and stage I+II group,respectively.No correlations were found between CDH13 promoter hypermethylation and age,gender,or the occurrence of myasthenia gravis (MG) symptoms (P>0.05).【Conclusion】CDH13 promoter methylation plays an important role in the development of TETs,and is closely related to the Masaoka staging and World Health Organization criteria.
thymic epithelial tumors; methylation; DNA; CDH13
R736.3
A
10.19338/j.issn.1672-2019.2017.09.001
2017-08-13
(刘东京 编辑)
