OsNAC2转录因子介导生长素代谢通路调节水稻早期根的发育
2017-12-14何建美奚丹丹邓其明
何建美,吕 波,奚丹丹,邓其明,明 凤
(1. 复旦大学 生命科学学院 植物科学研究所,上海 200433;2. 复旦大学 遗传工程国家重点实验室,上海 200433;3. 四川农业大学 水稻研究所,成都 611130)
OsNAC2转录因子介导生长素代谢通路调节水稻早期根的发育
何建美1,2,3,吕 波1,2,奚丹丹1,2,邓其明3,明 凤1,2
(1. 复旦大学 生命科学学院 植物科学研究所,上海 200433;2. 复旦大学 遗传工程国家重点实验室,上海 200433;3. 四川农业大学 水稻研究所,成都 611130)
NAC家族是植物特有的一类转录因子,在植物的生长发育和逆境胁迫等方面具有重要的功能.研究发现,OsNAC2基因的过表达(ON11)和RNAi(RNAi31)转基因株系,早期主根的长度有显著变化的特征.与野生型日本晴水稻(WT)相比,ON11植株的主根变短,而RNAi31的主根变长.通过对早期OsNAC2pro∶∶GUS株系根尖染色结果表明OsNAC2在根尖具有时序性表达.不同激素诱导OsNAC2表达谱和OsNAC2pro∶∶GUS株系对激素响应结果显示OsNAC2受生长素显著性诱导.结合芯片数据和qRT-PCR分析,OsNAC2转基因株系中生长素合成代谢及信号通路相关基因表达量变化较为明显.同时,ON11根尖淀粉粒发育及其向重力性受到抑制.因此,推测OsNAC2可能通过抑制生长素的合成代谢及信号通路相关基因的表达,降低生长素含量,并参与生长素响应通路,最终影响水稻根的生长.
OsNAC2; 生长素; 水稻; 根
根系是一株植物的全部根的总称,它在植物生长发育过程中起着极其重要的作用,是植物吸收水分及养分、固定和支撑植株的器官[1],也是植物激素、有机酸和氨基酸等物质合成与转化的重要场所.水稻属于禾本科单子叶作物,其根系由种子根、不定根和侧根组成[2].水稻根系的生长情况和活力对其生长发育,以及产量水平有直接的影响.
近年来,与水稻根系发育相关的基因已有很多报导.如水稻根系发育相关的调控基因OsRAA1过表达,会导致主根变短,不定根数目增加[3].而水稻中QHB基因过表达,引起不定根原基缺少,不定根减少[4].WOX11能直接抑制在冠状根原基表达的A型细胞分裂素响应调节因子RR2的表达,与RR2共同在冠状根发育中调节细胞的增殖[5].另外,某些基因的突变体,也会呈现出水稻根系发育受影响的特征.如碱性/中性转化酶基因OsCyt-inv1的突变体,其根系伸长区细胞萎缩,细胞纵向长度显著变短[6].类谷氨酸受体基因OsGLR3.1突变体的主根、不定根和侧根都变短,主根顶端直径变小,根分生组织活性丧失,还伴随着细胞程序性死亡的提高[7].水稻的葡聚糖内切酶基因OsGLU3突变后,会影响根细胞的分裂及细胞的伸长[8].
水稻根系的发育受很多因素的影响,如植物激素.其中,生长素(Indole-3-Acetic Acid, IAA)和细胞分裂素在根系中的研究相对较多.在水稻中,生长素的代谢、转运和信号转导紧密地影响着根系的生长发育.研究发现有些基因与生长素共同在水稻根系发育中起作用.在生长素合成过程中,YUCCA蛋白能催化色胺的氧化,是主要的限速步骤.当OsYUCCA1过表达后,植株体内IAA含量上升,不定根的数目增多[9].突变OsCOW1基因会导致不定根减少,根冠比降低[10].生长素的极性运输是依赖于它的输入和输出蛋白,如AUX1和PIN蛋白家族.在水稻中,抑制OsPIN1的表达时,水稻不定根的产生和发育受抑制[11].SCF复合体、AUX/IAAs和ARFs是生长素信号通路中的3类关键作用的蛋白.水稻中AUX/IAA成员有31个,作为AUX/IAA家族成员的OsIAA11,其获得性突变可抑制水稻侧根发育[12].CRL1/ARL1是一个编码含有植物特有AS2/LOB结构域蛋白的基因,它受外源生长素的诱导和AUX-ARF通路的调节,其突变会抑制不定根发育[13].此外发现,OsCAND1与拟南芥中的CAND1同源,参与了冠状根原基生长素信号维持的G2/M细胞周期的转化,影响冠状根的伸出[14].
NAC家族转录因子功能广泛,主要参与非生物胁迫[15-16]、抗病反应[17]以及植物的生长发育过程[18]等方面.但在植物根系发育中,有关NAC家族成员研究还较少.在拟南芥中,NAC1可在根尖和侧根起始部位表达,并且受生长素的诱导,过表达该基因可促进侧根形成[19].而SINAT5具有泛素化蛋白连接酶活性,能够泛素化NAC1,过表达SINAT5的拟南芥会形成较少的侧根[19-20].ANAC092(AtNAC2)也参与侧根的发育,过表达株系的侧根明显变多[21].
此外,参与胁迫诱导反应的NAC家族基因在根系发育方面也有研究.拟南芥中NTL8(NTM1like8)受到非生物胁迫的诱导,可促进侧根的形成[22].水稻NAC基因SNAC1通过增强转基因棉花根系的发育和降低蒸发率,提高其对干旱和盐胁迫的耐受性[15].过表达OsNAC5能增大水稻根系的直径,对干旱的耐受性有所提高,提高产量[23].另外,OsNAC9和OsNAC10也可通过影响根系的结构来提高其对干旱的耐受性[16,24].
本研究旨在通过研究转基因株系与野生型存在主根长度差异的分子机制,揭示OsNAC2对水稻根的发育的调控机制和代谢通路,为更深层次了解OsNAC2对水稻根的调控机制奠定基础.
1 材料和方法
1.1 材料与试剂
粳稻品种日本晴、OsNAC2过表达株系、RNAi株系及OsNAC2pro∶∶GUS株系为本实验室保存.KOD Plus高保真聚合酶购自ToYoBo公司;E.coliDH5α、RNA提取试剂盒、逆转录试剂盒、限制性内切酶及T4DNA连接酶购自TaKaRa公司;质粒提取试剂盒和胶回收试剂盒购自Axygen;PCR等相关引物购自上海生物工程有限公司.水稻转基因的化学试剂购自上海鼎国生物技术有限责任公司.其他化学试剂为国产或进口分析纯.
1.2 方 法
1.2.1 qRT-PCR分析
用TRIzol法提取一周龄的水稻根的总RNA,并用TaKaRa Prime Script RT reagent Kit with gDNA Eraser逆转录试剂盒进行基因组的清除和逆转录.用SYBR Premix Ex Taq TMII(Perfect Real Time)进行qRT-PCR反应检测目的基因的mRNA水平,至少3次生物性重复.qRT-PCR程序: 95℃(3min);94℃(10s),55℃(20s),40个循环.反应完成后,用2-ΔΔCT计算目的基因与内参基因的相对表达量.所用到目的基因和内参基因引物详见表1.

表1 qRT-PCR引物表Tab.1 Primers used for qRT-PCR
1.2.2 GUS组织化学染色
将一周龄的水稻根浸染在GUS染色液中,抽真空使材料沉至15mL的离心管底部,37℃过夜.吸去染色液,用70%乙醇除尽叶绿素,观察,拍照.GUS染色液的配制: 200mmol/L磷酸钠缓冲液(pH7.0),100mmol/L磷酸钠缓冲液(pH7.0),10mmol/L Na2EDTA,1mmol/L K3[Fe(CN)6],1mmol/L K4[Fe(CN)6],体积比5% Triton-100,20%甲醇,0.5mg/mL X-Gluc(DMSO助溶).
1.2.3 野生型水稻根系激素处理
将水稻日本晴种子室温浸种1d后移至37℃暗培养.催芽2d,置于水稻培养箱(16h光照/8h黑暗,28℃),营养液培养7d,移至分别含有10μmol/L IAA、10μmol/L 2,4-二氖苯氧乙酸(2,4-D)、5μmol/L赤霉素(GA3)、10μmol/L 乙烯(ACC)和未添加激素的营养液中处理8h.
1.2.4 转基因株系根长表型分析
OsNAC2过表达和RNAi转基因株系与野生型种子按1.2.3的方法进行培养.在2d~7d期间,每天取20株水稻进行主根长度的统计.
1.2.5OsNAC2在早期根尖发育中的表达和对IAA的响应
按1.2.3方法培养OsNAC2pro∶∶GUS株系,取第2~7d水稻的根进行GUS染色观察,每天取10株进行实验.GUS染色方法按1.2.2.
将OsNAC2pro∶∶GUS株系种子按1.2.3的方法培养,将一周龄的水稻移至不含激素,及含0.1,1,10,100μmol/L IAA的营养液中处理8h.取10株水稻的根进行GUS染色.GUS染色方法按1.2.2.
1.2.6 根尖淀粉粒染色
切取水稻1cm根尖浸泡在4% I2-KI染色液(8g KI,4g I2,ddH2O定容至400mL)中,染色5min后取出,加入水合氯醛(4g水合氯醛,1mL甘油,2mL ddH2O)透明,压片观察,拍照.
1.2.7 向重力性实验
将水稻种子经过次氯酸钠20~30min消毒后,灭菌水清洗3~5次,至无气泡为止,种子播于MS培养基上.3d后,转移至大培养皿,垂直培养至第7d后,旋转90°培养2h,再用ImageJ软件测量向重力弯曲角度.
2 结果与分析
2.1 OsNAC2在早期发育的根中时序表达
为了分析OsNAC2在水稻根中的表达情况,用含潮霉素的MS培养基,筛选出OsNAC2pro∶∶GUS纯合体株系.为进一步研究OsNAC2在根中的表达模式,选取第2天至一周龄OsNAC2pro∶∶GUS株系的根进行GUS染色观察,每天取10株进行染色观察.第3天开始,GUS在根尖成熟区的中柱鞘有表达,至第5天表达明显.在第6天和第7天时,发现GUS在整个根尖均有表达(图1(a)1~6).且发现OsNAC2在水稻根系的侧根原基及侧根顶端和基部均有表达(图1(b)7~9).以上结果说明OsNAC2可能时序性地参与根的发育过程.
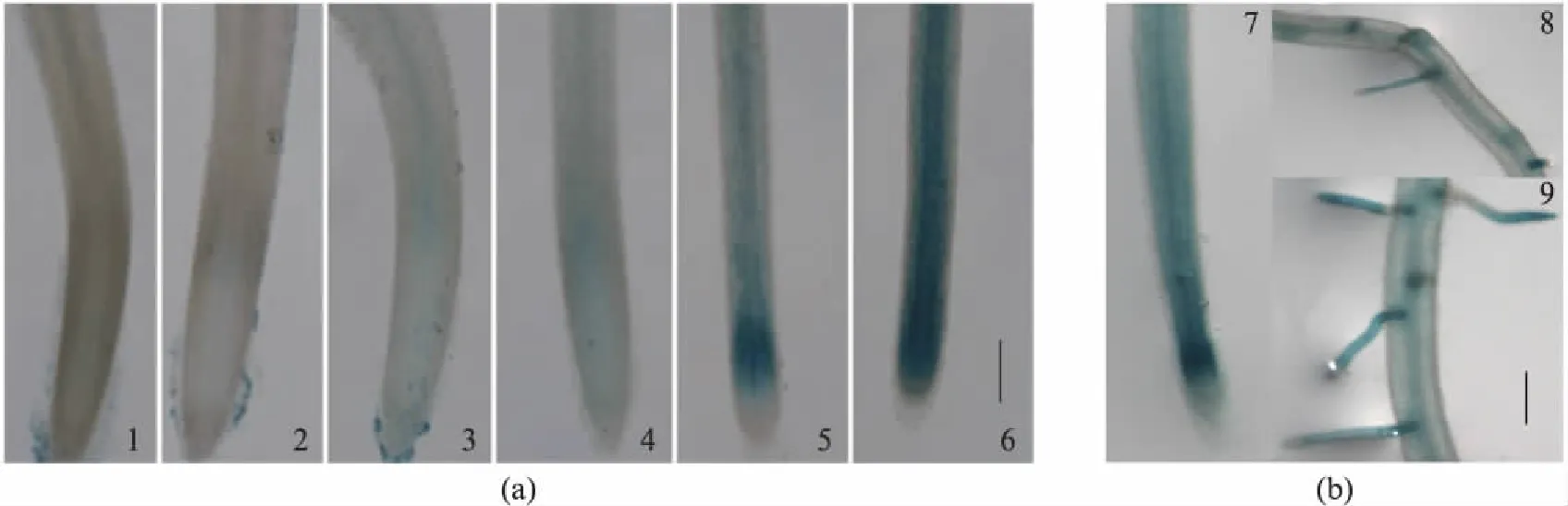
图1 OsNAC2在(a)水稻根部的表达,(b)侧根的表达Fig.1 OsNAC2 expression (a) in root, (b) in lateral root1~6: 2d~7d根尖GUS染色,n≥10,bar=0.2mm;7~9: 侧根、侧根原基及其顶部和基部,bar=0.5mm.
2.2 根中OsNAC2基因对不同激素的响应
为研究根中OsNAC2对不同激素的响应情况,将正常水培一周龄的野生型幼苗移到分别含10μmol/L IAA、10μmol/L 2,4-D、5μmol/L GA3、10μmol/L ACC和未添加激素的营养液中处理8h.通过qRT-PCR分析发现IAA和2,4-D可显著诱导OsNAC2的表达,表明OsNAC2可能参与生长素调节的水稻根发育;OsNAC2也受GA3的诱导,说明OsNAC2也可能参与了GA3调节根的发育;但OsNAC2对ACC的响应不明显(图2).
在以上激素表达谱实验中,生长素能显著诱导OsNAC2的表达.为了研究根尖中OsNAC2对生长素浓度的响应,用OsNAC2pro∶∶GUS株系进行实验.将正常水培一周龄的OsNAC2pro∶∶GUS株系移到分别含有0,0.1,1,10,100μmol/L IAA的水稻营养液中处理8h,通过对其根尖的GUS染色发现,在0.1μmol/L至10μmol/L IAA浓度的处理下,OsNAC2在根尖的诱导表达量随IAA浓度的增加而升高;但在100μmol/L IAA浓度下,OsNAC2的诱导量降低(图3(a)~(e)).说明在生长素调节根发育的过程中,OsNAC2可能起到了一定的作用,并具有反馈抑制响应.

图2 OsNAC2对不同激素的响应Fig.2 The OsNAC2 expression pattern under different hormonesMock为对照组,未添加激素.Student’s t-test,*P lt; 0.05,***P lt; 0.001. n≥3.

图3 OsNAC2pro∶∶GUS株系对不同浓度生长素的响应Fig.3 The GUS analysis of OsNAC2pro∶∶GUS lines treated with different concentrations of auxin(a)~(e)一周龄OsNAC2pro∶∶GUS株系在0,0.1,1,10,100μmol/L IAA处理下根尖GUS染色,bar=1mm.
2.3 OsNAC2过表达导致水稻主根变短
为研究OsNAC2的功能,对一周龄的转基因株系进行了OsNAC2的表达量分析.筛选出纯合体OsNAC2过表达转基因株系ON11和RNAi株系RNAi31(图4(a),(b)),作为后续研究.由于OsNAC2pro∶∶GUS株系在根系上的时序性表达,推测此基因在水稻根的发育上起作用.因此,我们测量了ON11和RNAi31转基因株系在不同天数下(2~7d)的主根长度(图4(c),(d)).结果显示,一周龄的RNAi31比WT的主根显著增长;而ON11比WT的主根显著变短.结果表明,OsNAC2在水稻主根发育上起一定的抑制作用.
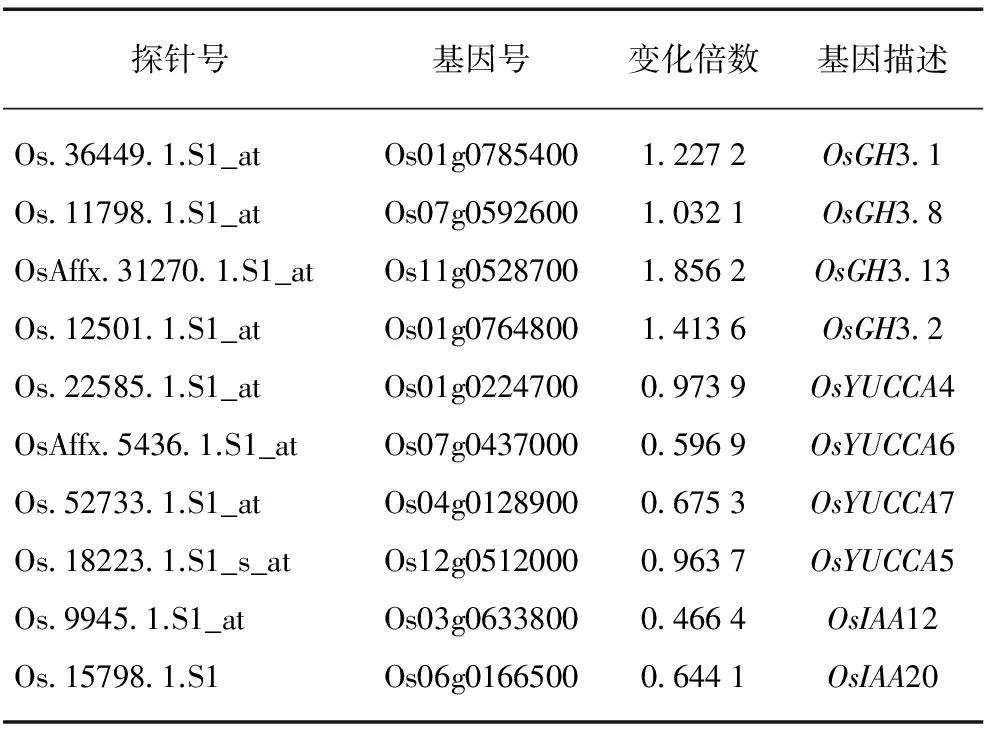
表2 ON11中IAA相关表达差异基因列表Tab.2 IAA related genes altered in ON11
2.4 OsNAC2参与生长素的合成代谢及信号通路
基于转基因株系表现出不同的主根长度的表型,便深入探究其内在分子机制.结合实验室已有的ON11与WT植株中根的基因芯片数据结果,发现众多生长素代谢相关基因在二者间具有显著的表达变化(表2).通过qRT-PCR验证(图5,见第416页),发现在ON11中: 与生长素响应相关的基因OsIAA9、OsIAA12和OsIAA20明显下调;GH3家族中编码吲哚乙酸氨基化合成酶基因OsGH3-1和OsGH3-8明显上调;与生长素合成相关的YUCCA-likegene家族中OsYUCCA2、OsYUCCA4、OsYUCCA6和OsYUCCA7下调,以及生长素运输基因OsPIN1b、OsPIN1c下调(图5).RNAi株系中这些基因表达量的趋势与过表达株系相反.依据基因的功能,可推测: 过表达株系中的OsGH3-1和OsGH3-8表达上升,导致植株体内IAA与氨基酸结合,从而减少游离的IAA;并通过OsYUCCA2、OsYUCCA4、OsYUCCA6和OsYUCCA7的表达下降,导致水稻根中生长素的合成减少.同时,OsNAC2通过抑制AUX/IAA家族基因OsIAA9、OsIAA12和OsIAA20的表达,影响生长素的信号通路.因此,生长素含量降低且信号通路受影响,最终造成过表达株系的主根变短.
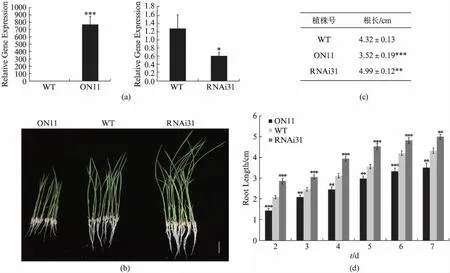
图4 OsNAC2转基因株系表达量检测及主根长度的测量Fig.4 The expression of OsNAC2 in transgenic lines and the measurement of primary root length(a) ON11和RNAi31株系根中OsNAC2表达量的鉴定,n=3;(b) 一周龄OsNAC2转基因株系根的表型,bar=2cm;(c) 一周龄OsNAC2转基因株系主根长度测定;(d) 不同天数的OsNAC2转基因株系的主根长度变化,Student’s t-test,*表示Plt;0.05,**表示Plt;0.01,***表示Plt;0.001.n≥10.
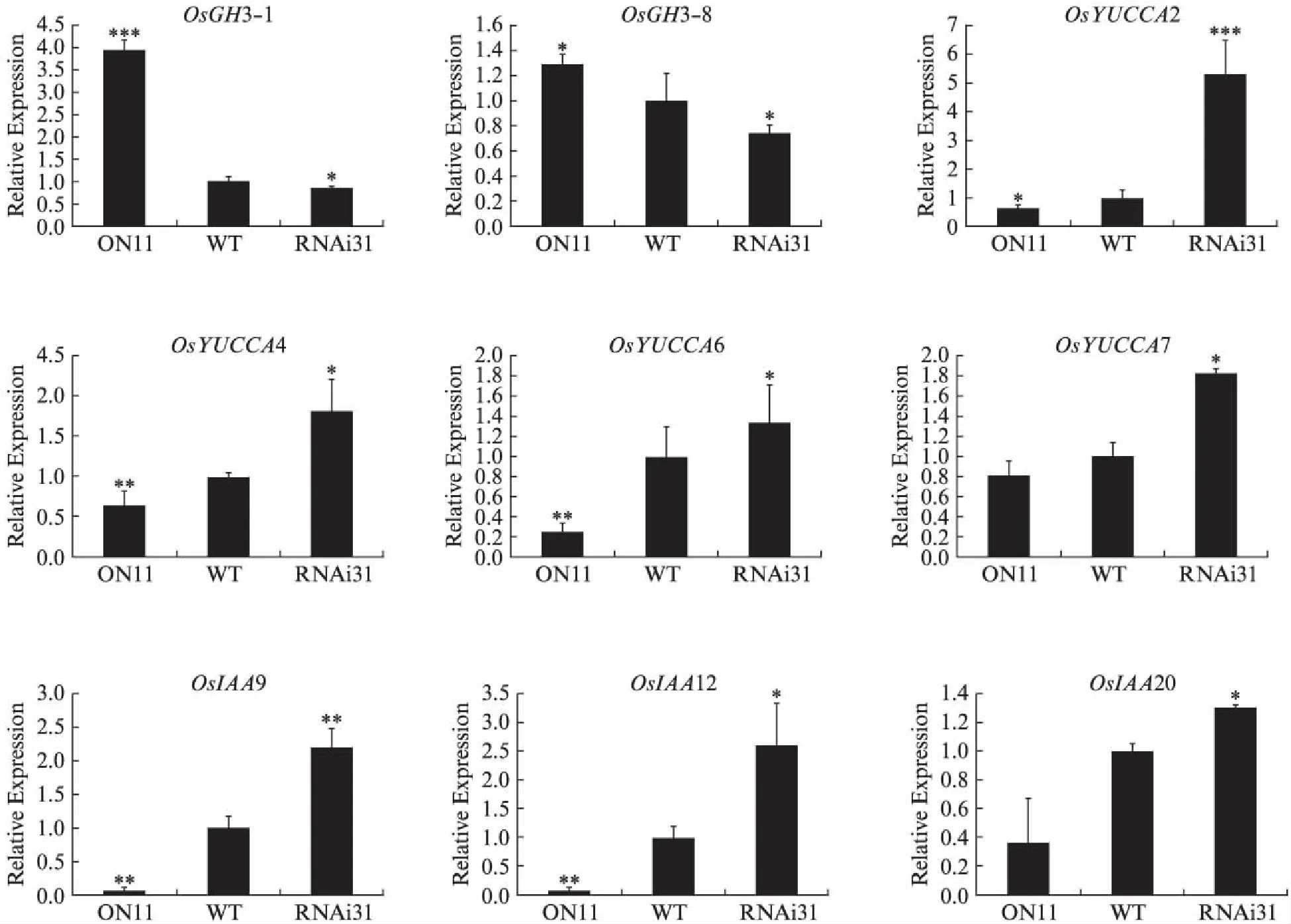
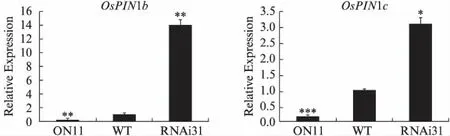
图5 OsNAC2野生型和转基因株系中IAA相关基因的表达分析Fig.5 IAA pathway related genes expression in OsNAC2 transgenic lines and WT Student’s t-test,*表示Plt;0.05,**表示Plt;0.01,***表示Plt;0.001,n≥3.
2.5 OsNAC2过表达影响根尖淀粉粒发育及其向重力性
通过上述的基因芯片数据和qRT-PCR发现,与生长素信号通路相关的基因在过表达中受到抑制.已有报道,生长素信号通路异常会影响水稻根尖淀粉粒的发育[25].因此,对一周龄的ON11和RNAi31的根尖进行淀粉粒染色观察.与WT根尖中淀粉粒比较,ON11根尖中淀粉粒显著减少,而RNAi31中显著增多(图6).
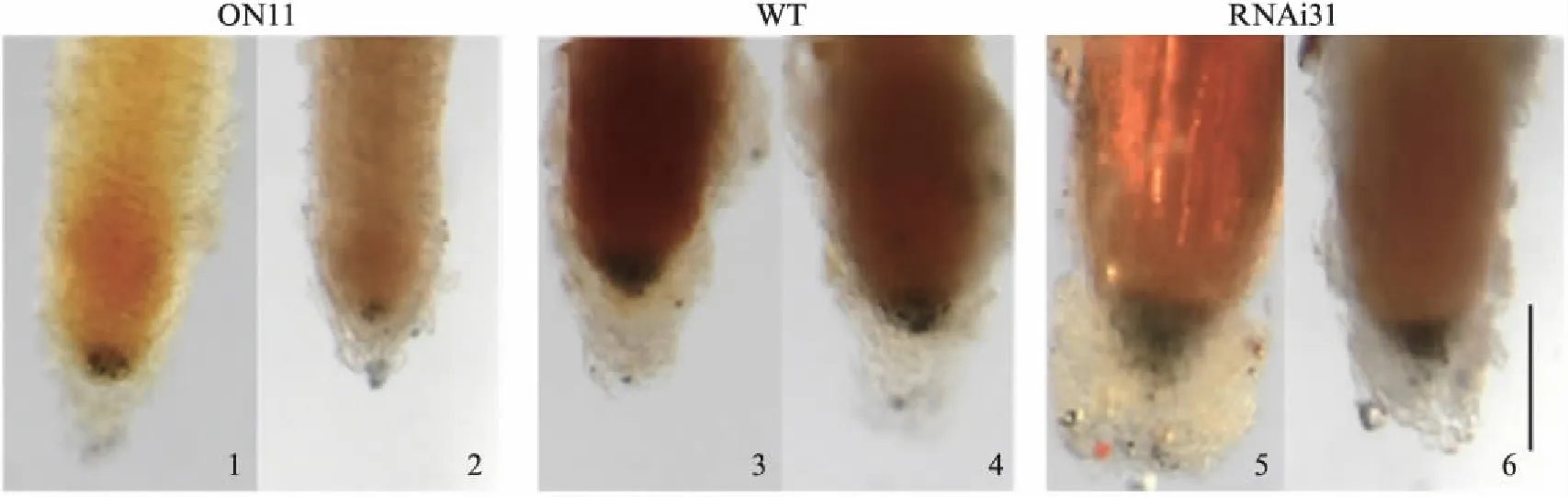
图6 OsNAC2转基因株系淀粉粒染色Fig.6 The starch grains dyeing of OsNAC2 transgenic lines根尖淀粉粒染色(1~2: ON11; 3~4: WT;5~6: RNAi31;bar=0.2mm)
研究表明,植物能否正确的响应重力信号的刺激,与生长素的生物合成、极性运输和信号传导联系紧密.生长素响应因子AUXIN RESPONSE FACTOR7(ARF7)和ARF19对向重力性的响应有着关键作用[26-28].拟南芥的淀粉体部分缺失突变体acg20和acg27表现出对重力响应减弱[29].由于根尖淀粉粒的发育与根的向重力性有关,因此进行了OsNAC2转基因株系与野生型株系的向重力性反应实验.实验结果发现,ON11株系向重力弯曲角度小于WT,而RNAi31株系的弯曲角度大于WT(图7).说明OsNAC2会减弱水稻根尖的向重力感应,即参与了水稻根的重力感应过程.推测OsNAC2可能通过生长素信号通路来调节水稻根的向重力感应.

图7 不同株系的向重力性反应及弯曲角度测量Fig.7 The gravitropism experiments and curvature measurement of OsNAC2 transgenic lines红色箭头指向为测量的角度,Student’s t-test,*表示Plt;0.05,**表示Plt;0.01,***表示Plt;0.001. n≥6.
3 讨 论
3.1 OsNAC2可能参与IAA信号通路
生长素的代谢、转运、信号转导与根系发育息息相关.在生长素信号通路过程中,AUX/IAA-ARF家族在生长素信号通路中起到负调节作用.ARF蛋白在生长素的浓度较低时会受到抑制,下游生长素响应基因无法表达;而ARF蛋白在生长素浓度较高时,解除抑制,下游生长素响应基因开始表达[30-31].
在激素表达谱和OsNAC2pro∶∶GUS株系对不同浓度生长素响应实验中,发现当生长素在一定浓度范围内时,OsNAC2可受到IAA显著诱导;但在100μmol/L生长素处理下时,其表达受到抑制(图3).结合芯片数据分析及生长素代谢相关基因qRT-PCR结果发现,与生长素相关基因的表达在过表达株系ON11中受到抑制(图5).说明OsNAC2可能通过抑制AUX/IAA家族基因的表达,调节生长素通路信号转导,但其具体的影响机制仍需进一步分析.
3.2 OsNAC2过表达可导致水稻主根变短
通过对转基因株系表型的鉴定,发现OsNAC2过表达会导致水稻主根变短(图4).有研究发现,过表达OsYUCCA1的转基因植株IAA水平升高,表现出过量生产生长素的表型特征,而反义表达OsYUCCA1 cDNA的植株则表现出与生长素不敏感突变体类似的表型[9].在水稻的GH3家族中,OsGH3.1的组成性表达降低了水稻中生长素的含量[32],催化过量的IAA与多种氨基酸结合来维持生长素的体内平衡[33].OsGH3-8编码一个IAA酰胺合成酶,可将多余的游离IAA与氨基酸结合,维持游离IAA含量的动态平衡;过量表达GH3-8可抑制伸展蛋白的表达及生长素的信号传导,造成植株形态的异常,如生长发育停滞[34].通过qRT-PCR结果发现(图5),与生长素合成相关的YUCCA-likegene家族中的OsYUCCAA2、OsYUCCA4、OsYUCCA6 和OsYUCCA7的表达量在过表达株系ON11中下调,GH3家族中的吲哚乙酸氨基化合成酶OsGH3-1和OsGH3-8上调.这可以推测出OsNAC2是通过减少植株体内生长素的合成及降低体内游离的IAA含量,从而导致过表达株系的主根变短.
3.3 OsNAC2可影响根尖淀粉粒发育并降低向重力感应
生长素的合成、极性运输和信号通路与水稻向重力性息息相关[26].OsNAC2过表达株系中,根尖淀粉粒及其向重力性受抑制(图6,图7),且过表达株系中AUX/IAA家族和YUCCA-likegene家族中一些基因的表达量明显受到抑制(图5).因此,推测OsNAC2可能通过抑制生长素信号通路和生长素的合成相关基因的表达,从而影响水稻根尖淀粉粒发育和向重力性.
3.4 OsNAC2也可能参与GA代谢通路影响水稻根的生长
由于影响水稻根发育的因素众多,本研究主要在生长素对根发育方面进行初步探究.OsNAC2除了可受IAA显著性诱导,也受GA3的诱导(图2).研究表明生长素能促进赤霉素降解DELLA蛋白,促进根的生长,而生长素运输或信号通路减弱会导致DELLA蛋白降解减慢[35].拟南芥中赤霉素缺失突变体gal-3的根变短,是由于赤霉素能促进伸长区细胞的伸长[36]和根系分生组织的细胞增殖[37].因此,OsNAC2也有可能参与GA代谢通路影响水稻根的生长.这也说明植物激素间通过相互作用来调节植物根的生长.
综上所述,OsNAC2可能是通过对生长素合成代谢及其信号通路的影响,从而调控水稻根的发育.但OsNAC2直接或者间接作用通路中相关因子的研究仍需后续研究加以揭示.
[1] HOCHHOLDINGER F, PARK W J, SAUER M,etal. From weeds to crops: Genetic analysis of root development in cereals [J].TrendsPlantSci, 2004,9(1): 42-48.
[2] 石庆华.大穗型水稻根系生长特性与产量形成的研究 [J].江西农业大学学报,1988,10(1): 52-62.
[3] GE L, CHEN H, JIANG J F,etal. Overexpression of OsRAA1 causes pleiotropic phenotypes in transgenic rice plants, including altered leaf, flower, and root development and root response to gravity [J].PlantPhysiol, 2004,135(3): 1502-1513.
[4] KAMIYA N, NAGASAKI H, MORIKAMI A,etal. Isolation and characterization of a rice WUSCHEL-type homeobox gene that is specifically expressed in the central cells of a quiescent center in the root apical meristem [J].ThePlantJournal, 2003,35(4): 429-441.
[5] ZHAO Y, HU Y F, DAI M G,etal. The WUSCHEL-related homeobox gene WOX11 is required to activate shoot-borne crown root development in rice [J].PlantCell, 2009,21(3), 736-748.
[6] JIA L Q, ZHANG B T, MAO C Z,etal. OsCYT-INV1 for alkaline/neutral invertase is involved in root cell development and reproductivity in rice(OryzasativaL) [J].Planta, 2008,228(1): 51-59.
[7] LI J, ZHU S H, SONG X,etal. A rice glutamate receptor-like gene is critical for the division and survival of individual cells in the root apical meristem [J].PlantCell, 2006,18(2): 340-349.
[8] ZHANG J W, XU L, WU Y R,etal. OsGLU3, a putative membrane-bound endo-1,4-beta-glucanase, is required for root cell elongation and division in rice(OryzasativaL) [J].MolecularPlant, 2012,5(1): 176-186.
[9] YUKO Y, NORIKO K, YOICHI M,etal. Auxin biosynthesis by the YUCCA genes in rice [J].PlantPhysiology, 2007,143(3): 1362-1371.
[10] WOO Y M, PARK H J, SUDI M,etal. Constitutively wilted 1, a member of the rice YUCCA gene family, is required for maintaining water homeostasis and an appropriate root to shoot ratio [J].PlantMolecularBiology, 2007,65(1/2): 125-136.
[11] XU M, ZHU L, SHOU H X,etal. A PIN1 family gene, OsPIN1, involved in auxin-dependent adventitious root emergence and tillering in rice [J].PlantandCellPhysiology, 2005,46(10): 1674-1681.
[12] ZHU Z X, LIU Y, LIU S J,etal. A gain-of-function mutation in OsIAA11 affects lateral root development in rice [J].MolecularPlant, 2012,5(1): 154-161.
[13] KITOMI Y, ITO H, HOBO T,etal. The auxin responsive AP2/ERF transcription factor CROWN ROOTLESS5 is involved in crown root initiation in rice through the induction of OsRR1, a type A response regulator of cytokinin signaling [J].ThePlantJournal, 2011,67(3): 472-484.
[14] WANG X F, HE F F, MA X X,etal. OsCAND1 is required for crown root emergence in rice [J].MolecularPlant, 2011,4(2): 289-299.
[15] LIU G, LI X L, JIN S X,etal. Overexpression of rice NAC gene SNAC1 improves drought and salt tolerance by enhancing root development and reducing transpiration rate in transgenic cotton [J].PLoSOne, 2014,9(1): e86895.
[16] REDILLAS M CFR, JEONG J S, KIM Y S,etal. The overexpression of OsNAC9 alters the root architecture of rice plants enhancing drought resistance and grain yield under field conditions [J].PlantBiotechnologyJournal, 2012,10(7): 792-805.
[17] SUN L J, ZHANG H J, LI D Y,etal. Functions of rice NAC transcriptional factors, ONAC122 and ONAC131, in defense responses againstMagnaporthegrisea[J].PlantMolecularBiology, 2013,81(1/2): 41-56.
[18] KUSANO H, ASANO T, SHIMADA H,etal. Molecular characterization of ONAC300, a novel NAC gene specifically expressed at early stages in various developing tissues of rice [J].MolecularGeneticsandGenomics, 2005,272(6): 616-626.
[19] XIE Q, FRUGIS G, COLGAN D,etal.ArabidopsisNAC1 transduces auxin signal downstream of TIR1 to promote lateral root development [J].GenesDev, 2000,14(23): 3024-3036.
[20] XIE Q, GUO H S, DALLMAN G,etal. SINAT5 promotes ubiquitin related degradation of NAC1 to attenuate auxin signals [J].Nature, 2002,419(6903): 167-170.
[21] HE X J, MU R L, CAO W H,etal. AtNAC2, a transcription factor downstream of ethylene and auxin signaling pathways, is involved in salt stress response and lateral root development [J].ThePlantJournal, 2005,44: 903-916.
[22] KIM S G, LEE A K, YOON H K,etal. A membrane-bound NAC transcription factor NTL8 regulates gibberellic acid mediated salt signaling inArabidopsisseed germination [J].ThePlantJournal, 2008,55: 77-88.
[23] JEONG S J, KIM Y S, REDILLAS M C F R,etal. OsNAC5 overexpression enlarges root diameter in rice plants leading to enhanced drought tolerance and increased grain yield in the field [J].PlantBiotechnolJ, 2013,11(1): 101-114.
[24] JEONG S J, KIM Y S, BAEK K H,etal. Root-specific expression of OsNAC10 improves drought tolerance and grain yield in rice under field drought conditions [J].PlantPhysiol, 2010,153(1): 185-197.
[25] OTTENSCHLAGER I, WOLFF P, WOLVERTON C,etal. Gravity-regulated differential auxin transport from columella to lateral root cap cells [J].ProcNatiAcadSciUSA, 2003,100(5): 2987-2991.
[26] LI J, DAI X, ZHAO Y. A role for auxin response factor 19 in auxin and ethylene signaling inArabidopsis[J].PlantPhysiol, 2006,140: 899-908.
[27] OKUSHIMA Y, OVERVOORDE P J, ARIMA K,etal. Functional genomic analysis of the AUXIN RESPONSE FACTOR gene family members inArabidopsisthaliana: Unique and overlapping functions of ARF7 and ARF19 [J].PlantCell, 2005,17: 444-463.
[28] WILMOTH J C, WANG S, TIWARI S B,etal. NPH4/ARF7 and ARF19 promote leaf expansion and auxin-induced lateral root formation [J].ThePlantJournal, 2005,43(1): 118-30.
[29] KISS J Z, WRIGHT J B, CASPAR T. Gravitropism in roots of intermediate-starch mutants ofArabidopsis[J].PlantPhysiol, 1996,97(2): 237-244.
[30] MOCKAITIS K, ESTELLE M. Auxin receptors and plant development: A new signaling paradigm [J].AnnualReviewofCellandDevelopmentalBiology, 2008,24: 55-80.
[31] JAIN M, KAUR N, GARG R,etal. Structure and expression analysis of early auxin-responsive Aux/IAA gene family in rice(Oryzasativa) [J].Functionalamp;IntegrativeGenomics, 2006,6(1): 47-59.
[32] DOMINGO C, ANDRÉS F, THARREAU D,etal. Constitutive expression of OsGH3.1 reduces auxin content and enhances defense response and resistance to a fungal pathogen in rice [J].MolecularPlant-MicrobeInteractions, 2009,22(2): 201-210.
[33] ZHAO S Q, XIANG J J, XUE H W,etal. Studies on the rice LEAF INCLINATION1(LC1), an IAA-amido synthetase, reveal the effects of auxin in leaf inclination control [J].MolecularPlant, 2013,6(1): 174-187.
[34] DING X H, CAO Y L, HUANG L L,etal. Activation of the indole-3-acetic acid-amido synthetase GH3-8 suppresses expansin expression and promotes salicylate and jasmonate independent basal immunity in rice [J].ThePlantCell, 2008,20(1): 228-240.
[35] FU X, HARBERD NP. Auxin promotesArabidopsisroot growth by modulating gibberellin response [J].Nature, 2003,421(6924): 740-743.
[37] ACHARD P, GUSTI A, CHEMINANT S,etal. Gibberellin signaling controls cell proliferation rate inArabidopsis[J].CurrBiol, 2009,19(14): 1188-1193.
OsNAC2TranscriptionFactorRegulatesEarlyRootDevelopmentbyMediatingAuxinMetabolicPathwaysinRice
HEJianmei1,2,3,LÜBo1,2,XIDandan1,2,DENGQiming3,MINGFeng1,2
(1.InstituteofPlantBiology,SchoolofLifeSciences,FudanUniversity,Shanghai200438,China;2.StateKeyLaboratoryofGeneticEngineering,FudanUniversity,Shanghai200438,China;3.InstituteofRiceResearch,SichuanAgriculturalUniversity,Chengdu611130,China)
The NAC family is one of the largest families of specific transcription factors in plant, which played a crucial role in plant development and environment stress. In this study, we found that constitutive overexpression ofOsNAC2 in rice(ON11) reduced primary root length during early growth period, meanwhile RNAi transgenic line(RNAi31) has longer primary root length compared with WT. GUS staining showed thatOsNAC2 temporal expressed in root tip. In addition,OsNAC2 was significantly induced by auxin after different hormone treatments. Furthermore, microarray analysis and qRT-PCR revealed that genes related to biosynthesis, metabolism and signaling pathway of auxin varied significantly among transgenic lines ON11, RNAi31 and WT. The starch grain development and gravitropism of root were also restrained in ON11. Taken together, OsNAC2 might regulate the expression of genes in auxin synthetic metabolism and signaling pathways, reduce the auxin concentration, involved in the auxin response pathways, and finally affect the rice root development.
OsNAC2; Indole-3-Acetic Acid; rice; root
0427-7104(2017)04-0411-09
2016-12-07
转基因生物新品种培育重大专项(2016ZX08009-001-008)和国家自然科学基金(31471152)
何建美(1991—),女,硕士研究生;明 凤(通信联系人),女,副教授,博士生导师,E-mail: fming@fudan.edu.cn;邓其明(通信联系人),男,副研究员,硕士生导师,E-mail: dengqmsc@163.com.
Q943.3
A
