沙门菌属特异性检测靶点碱基差异位点的识别及其血清型决定力
2017-12-11石秀清周秀娟施春雷史贤明
石秀清,周秀娟,施春雷,张 芬,史贤明*
沙门菌属特异性检测靶点碱基差异位点的识别及其血清型决定力
石秀清,周秀娟,施春雷,张 芬,史贤明*
(上海交通大学农业与生物学院,中美食品安全联合研究中心,微生物代谢国家重点实验室,上海 200240)
以美国国家生物技术信息中心(NCBI)已公布的28 株沙门菌(14 个血清型)全基因组序列为研究对象,对前期研究获得的7 个沙门菌属特异性检测靶点在不同血清型间的单核苷酸多态性进行比较分析,结果表明,S9和S69为碱基差异位点最多的两个靶点,具有沙门菌分子血清分型的潜在能力。随后,通过分析S9和S69在沙门菌不同血清型菌株中的差异位点,分别绘制两个血清分型靶点的差异位点表,从而获得了这两个靶点同时用于14 个沙门菌血清型的快速分子血清分型的差异位点组合。在这些组合中,同一血清型具有相同的差异位点,不同血清型可以通过差异位点得以区分。最后,采集食品样品192 份,用国标方法获得疑似沙门菌21 株;利用血清分子分型靶点S9和S69进行快速分型,并同时用传统玻片凝集反应鉴定方法进行验证,两种方法鉴定结果的符合率为100%。鉴定结果为:21 株沙门菌共包括4 个不同血清型,其中肠炎沙门菌10株、鼠伤寒沙门菌7 株、鸡沙门菌2 株、纽波特沙门菌2 株。基因组序列分析结果和分离株分型结果均表明,沙门菌特异分子检测靶点S9和S69相结合具备沙门菌的分子血清分型能力,以差异位点表为基础建立的血清分型方法有望替代传统的玻片凝集血清分型这一繁杂步骤,从而降低沙门菌血清鉴定的时间与成本,为聚合酶链式反应技术应用于沙门菌分子血清分型提供新思路。
沙门菌;分子血清分型;特异性检测靶点;食品样品;血清鉴定
沙门菌是引发食物中毒最主要的食源性致病菌之一,由其引发的食物中毒病例在世界范围内居首位,每年全球约有1 600万沙门菌感染病例出现,其中60万 例死亡[1-2]。近年来,我国细菌性食物中毒暴发事件中,由沙门菌引起的约占70%~80%[3]。据统计,我国每年约有40 000 例沙门菌感染病例,但实际的感染人数可能要达20 倍以上[1],食物传播是导致人类感染沙门菌的最主要途经。沙门菌属的菌株型别繁多,抗原复杂[4],目前全世界已分离出沙门菌的血清型多达2 600 种,46 个血清组,而我国约有300 种血清型,37 个血清组[5],这使得沙门菌溯源与污染特征的研究更加复杂。值得注意的是,沙门菌血清型存在明显的宿主特异性[6],特定的血清型常与食品类型相关联[7-9]。因此,建立快速、灵敏的分子血清分型技术对食品污染源的追踪、溯源,暴发疫情的鉴定以及最终有效地控制疫情意义重大。
当前世界各国家标准和ISO标准均采用传统的细菌学方法从食品中分离检测沙门菌[10],并根据抗原-抗体反应免疫学原理(血清玻片凝集)鉴定其血清型。但此方法对于血清型的判读流程繁琐,操作人员的主观判断所占比例较大,误差较大,重复性较差[11-12]。为了克服传统血清玻片凝集分型技术的缺点,许多现代的分子生物学方法被应用于开发新型的分子血清分型技术中[13-17],如:脉冲场凝胶电泳(pulsed field gel electrophoresis,PFGE)、多位点序列分型(multilocus sequence typing,MLST)和CRISPR(clustered regularly interspaced short palindromic repeats)等。而且随着测序技术的不断革新与进步,越来越多的沙门菌基因组序列已经测定完成并收录在GenBank、Sanger等数据库中[18]。它们与聚合酶链式反应(polymerase chain reaction,PCR)技术相结合极大地促进了沙门菌的快速检测与分子血清分型方法的发展[19-24]。
到目前为止,还没有PCR技术同时用于沙门菌属的检测和血清分型的实例。2016年,Zhou Xiujuan等[25]基于美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)公布的26 株沙门菌和2 180 株非沙门菌的全基因组序列,采用比较基因组的方法,从生物信息学和生物再验证两个角度,最终获得15 个性能优异的沙门菌属特异性检测靶点。在对这些检测靶点进一步分析中,Zhou Xiujuan等[25]还发现其中7 个沙门菌检测靶点与MLST中看家基因具有相似的保守性和血清型差异性,推测这些特异检测靶点将在沙门菌的检测和血清分型上具备巨大潜力。因此,这7 个沙门菌属特异性检测靶点不仅可以鉴定出未知菌株中的沙门菌,同时还具有进一步判定其血清型的潜力。
本实验比较Zhou Xiujuan等[25]发现的7 个沙门菌属特异性检测靶点在不同血清型间存在的碱基差异位点,并评价这些差异位点对血清型的决定力。通过整理靶点在沙门菌不同血清型菌株中的差异位点,绘制出血清分型靶点的差异位点表,并将此表格应用于食品样品中沙门菌的快速血清分型。最后将分子血清分型结果再与传统玻片凝集反应实验相比较,从而验证靶点在实际应用中的血清分型效果。
1 材料与方法
1.1 菌株基因组序列
从NCBI(http://www.ncbi.nlm.nih.gov/)上获取并筛选得到2016年6月12日已公布的常见沙门菌菌株的全基因组,总共14 种血清型的28 株沙门菌菌株基因组,见表1。1.2 试剂与仪器
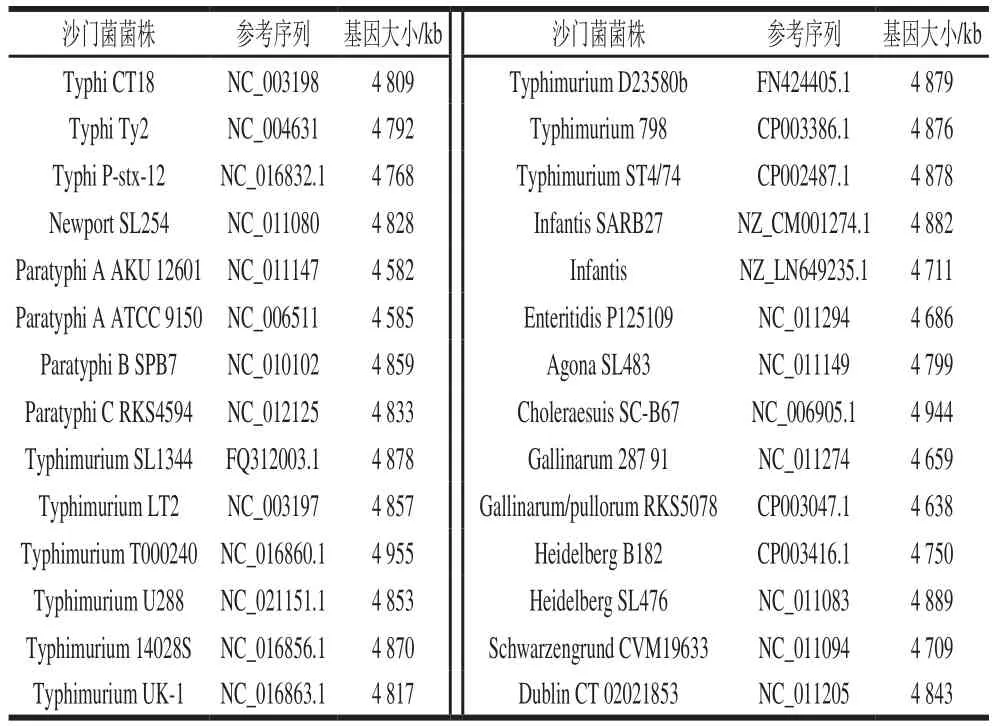
表1 用于血清分子分型靶点筛选的沙门菌基因组Table 1 Genome sequences of Salmonella strains used as molecular targets for serotyping
Taq DNA聚合酶、10×PCR buffer、Mg2+(MBI Fermentas)、dNTPs、DNA Marker 北京天根生化科技有限公司。
PCR扩增仪、5415D台式微量离心机 德国Eppendorf公司;PS2A200核酸电泳仪 美国Hoefer公司;GIS2020凝胶成像仪 上海天能公司;NanoDrop 2000c超微量分光光度计 美国Thermo Forma公司;EQR/GL-41超净工作台 新加坡Esco公司;血清鉴定试剂盒(20160101)宁波天润生物药业有限公司。
1.3 方法
1.3.1 食品样品采集
分别从上海市闵行区东川路A超市,春申路B超市,沧源路C菜市场,沧源路D超市和吴泾E超市5 个地点采集食品样品192 份,用于沙门菌菌株的分离。样品类型分别为肉制品类(55 份)、果蔬类(45 份)、速冻食品类(45 份)、即食食品类(47 份)。
1.3.2 MEGA 6.0软件与分子血清分型靶点SNP差异位点表的构建
从NCBI数据库中下载已完成测序的沙门菌全基因组序列。将NCBI中下载的序列,以相同靶点为单位,按照不同血清型排列并合为一个*.txt文本。将*.txt文本转化为fasta格式后,使用MEGA 6.0软件打开,以S. Typhi CT18对应的靶点序列为参比序列(从5’到3’依次编号)进行对齐处理,查找不同血清型中对照参比序列的单核苷酸多态性(single nucleotide polymorphism,SNP)差异位点。将所有SNP差异位点按照血清型的顺序归入Excel表格中。
1.3.3 沙门菌疑似菌株分离
采用GB 4789.4—2010《食品微生物学检验 总则》[11]分离获得沙门菌疑似菌株。具体步骤包括:1)每份样品取25 g或25 mL,移入225 mL缓冲蛋白胨水液体培养基中,在37 ℃的摇床(150 r/min)上增菌培养10 h;2)取1 mL增菌液转种与10 mL四硫磺酸钠煌绿增菌内,于42 ℃培养18~24 h,同时另取1 mL增菌液转种与10 mL亚硒酸盐胱氨酸增菌液内,于37 ℃培养18~24 h;3)分别用接种环取增菌液1环,划线接种于一个亚硫酸铋琼脂平板(于37 ℃培养40~48 h)和一个沙门菌属显色培养基平板(于37 ℃培养18~24 h),观察各个平板上生长的菌落,若菌落为黑色有金属光泽,棕褐色或灰色,则初步判定为沙门菌。
1.3.4 DNA的提取
参见文献[15],使用CTAB法提取沙门菌基因组DNA,取1 μL提取好的基因组DNA溶液,用超微量分光光度计NanoDrop 2000c在波长260 nm和280 nm条件下测定其纯度与浓度,剩余DNA原液置于-20 ℃保存[3]。
1.3.5 PCR反应体系与参数
1.3.5.1 PCR反应体系
本实验中PCR反应体系参照体系如下:(NH4)2SO4缓冲液(10×)2.5 μL、MgCl2溶液(50 mmol/L)1.5 μL、dNTP(2.5 mmol/L)1.5 μL、上、下游引物(各10 μmol/L)(0.5+0.5)μL;TaqDNA聚合酶(1 U/μL)1.0 μL、模板2.5 μL;无菌去离子水15 μL总体积25 μL[3,25]。
1.3.5.2 PCR反应参数
检测S9所用PCR的扩增条件为:94 ℃预变性5 min;接着进行35 个循环,每个循环包括94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸40 s;循环结束后72 ℃延伸10 min;降温至12 ℃,反应结束[25]。
检测S69所用PCR的扩增条件为:95 ℃预变性10 min;接着进行35 个循环,每个循环包括94 ℃变性30 s,58 ℃退火45 s,72 ℃延伸45 s;循环结束后72 ℃延伸10 min;降温至12 ℃,反应结束[3]。
1.3.5.3 所用引物序列
本实验S9对应的引物为:S9-F:5’-GAGGTCACGCACCATCACAAT-3’和S9-R:5’-ATACGGCACCACCGCATAG-3ʹ[25]。
S69对应的引物为:S69-F:5’-CACAGGAGGAGCAGGATAA-3’和S69-R:5’-CCAGTACAGCAAGGGACAT-3’[3]。
引物由生工生物工程(上海)股份有限公司合成,PCR产物序列测定由上海美吉生物医药科技有限公司完成。
1.3.6 凝胶电泳
本实验凝胶电泳步骤中,取5 μL PCR产物,与1 μL 6×加样缓冲液混匀,混合物加到含有稀释10 000 倍的核酸染料的1.5%琼脂糖凝胶点样孔中,同时在第1个点样孔中加入5 μL 100 bp DNA ladder Marker,接通电源,凝胶电泳电压控制为160~170 V,时间20~30 min。
1.3.7 沙门菌血清玻片凝集
依据抗原-抗体凝集原理,同时参照血清鉴定试剂盒产品说明书,根据不同的抗体凝集反应区分或分类鉴定沙门菌的血清型[26-27]。其中:H抗血清直接滴于干净的载玻片上,加入等体积的菌悬液混合,若凝集即为阳性,反之为阴性;而O抗血清需要与高温处理(100 ℃加热15 min)的菌悬液混合,短时间内凝集为阳性,否则为阴性。根据凝集结果,对照血清型的抗原式表[26]来判断沙门菌的血清型。
2 结果与分析
2.1 沙门菌属特异性检测靶点中SNP的识别
通过靶点引物,从NCBI上获取的14 个血清型的28 株肠道沙门菌菌株基因组上寻找到该靶点的碱基序列。通过各个靶点片段序列之间的比对发现:同一血清型菌株对应靶点序列完全一致,而不同血清型沙门菌株间存在碱基差异,因此,这些特异性检测靶点具备区分沙门菌血清型的潜力。
通过各靶点在不同血清型代表性菌株上对应基因片段的比较,以S. Typhi CT18对应的靶点序列为参比序列(从5’到3’依次编号),找出各靶点对应血清型之间的SNP位点个数,结果如图1所示。结果显示,在7 对沙门菌特异性靶点中,靶点S9对应片段在各血清型之间存在的SNP位点个数最多,其次是S69。王中强等[28]研究表明,在基因分型方法建立时,如果选择很少位点进行测定便增加了由于等位基因联合作用而造成混淆的概率,选择位点数量的增加可以提高分辨率,但是当选择的位点达到一定的数目,便只会增加人力、物力的消耗,而不能够获取更多的信息。实验结果表明:S9能区分14 个血清型中的11 个,S69能区分14 个血清型中的9 个。所以本实验如果仅以S9或者S69单个靶点对沙门菌进行血清分型,能够区分的血清型种类比较有限。但如果将二者组合起来,能够完全区分14 种沙门菌血清型。所以最终在7 个沙门菌属特异性靶点中筛选出2 个沙门菌血清分型的靶点:S9和S69,作为沙门菌分子血清分型候选靶点。
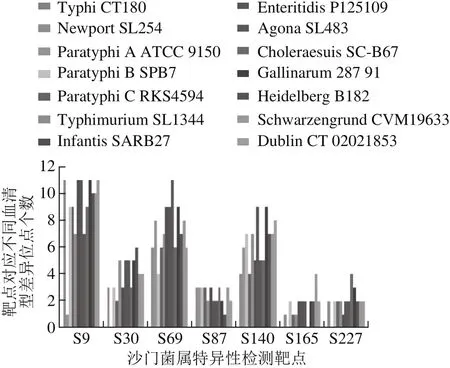
图1 沙门菌属特异性检测靶点在不同血清型沙门菌株上对应的SNP位点个数Fig. 1 Analysis of the serotyping ability of different Salmonella-specific targets by the number of SNPs
2.2 S9和S69碱基差异位点表的构建
采用S9和S69引物,对14 个血清型代表菌株基因组进行扩增,获取两靶点的基因片段序列。以S. Typhi CT18对应的S9序列为参比序列(从5’到3’依次编号),分析S9对应的14 个片段之间的碱基差异,对各位点进行编号和碱基变化统计,如表2所示。结果表明,靶点S9对应的14 个血清型菌株基因片段中,Newport、Gallinarum和Dublin这3 个血清型菌株对应的基因片段之间不存在碱基间的差异,即靶点S9无法区分这3 个血清型菌株。但是选取靶点S69,按照相同方法,对这3 个血清型菌株对应靶点碱基序列进行分析。结果显示,靶点S69对应此3 个血清型菌株基因片段存在明显碱基差异(差异位点如表2所示),能够将这3 个血清型菌株完全区分。最终得到如表2所示的靶点S9和S69对14 个沙门血清型碱基差异位点表。

表2 靶点对应不同血清型的差异位点Table 2 Distinct mutation sites of different serotypes of Salmonella strains
2.3 碱基差异位点表应用于沙门菌食品分离株的血清分型
采集192 份食品样品,按照GB 4789.4—2010[11]分离鉴定出21 份阳性样本,从每份阳性样本中选取分离株1 株,总共得到21 株疑似沙门菌菌株。经靶点S9 PCR扩增并进行凝胶电泳,此21 株菌均产生相应的明亮条带,如图2所示。结果表明,此21 株可疑沙门菌菌株全部为沙门菌。将S9扩增的PCR产物进行测序,测序结果对照表2,鉴定出这21 株沙门菌菌株中的17 株菌株的血清型:其中肠炎沙门菌10 株,鼠伤寒沙门菌7 株。余下4 株经S9扩增得到的序列完全一致,相互之间没有碱基差异,无法用靶点S9区分。然后,再使用S69对这4 株菌进行DNA的PCR扩增并测序,测序结果与表2进行比对,鉴定出其中2 株为鸡沙门菌,2 株为纽波特沙门菌,结果如表3所示。
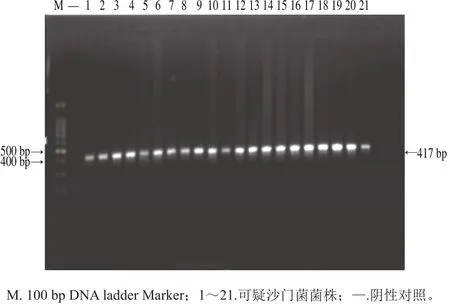
图2 沙门菌特异性靶点S9 PCR扩增Fig. 2 PCR amplification of Salmonella specific targets S9
2.4 传统血清鉴定方法对食品分离菌株血清型的验证
通过沙门菌H抗血清和O抗血清,对S9和S69鉴定的沙门菌菌株的血清型,即2.3节中21 株疑似沙门菌菌株对应的血清型进行验证。传统血清鉴定过程中,血清型菌株与对应血清型的H抗血清和O抗血清均发生阳性的凝集反应,其中肠炎沙门菌(9,12:g,m:-)10 株,鼠伤寒沙门菌(4,5,12:i:1,2)7 株,鸡沙门菌(9,12:-:-)2 株,纽波特沙门菌(6,8:e,h:1,2)2 株。验证结果表明:用靶点S9和S69所确定的沙门菌血清型与传统血清鉴定方法验证的结果符合率达到100%。证明沙门菌特异性分子检测靶点S9和S69能够快速准确地判定沙门菌血清型。最终统计结果如表3所示。
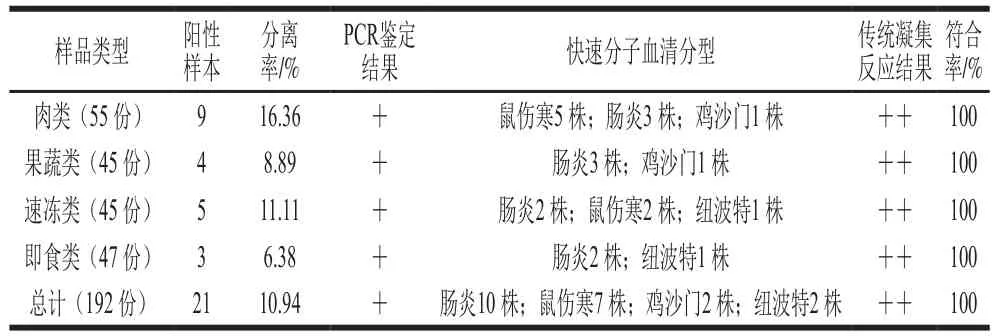
表3 食品样品分菌与血清分型结果Table 3 Iolation, identification and serotyping of Salmonella isolates from food samples
3 结论与讨论
对沙门菌进行分子血清分型时,选择分散于基因组内多个位点进行测定,大大增加了血清区分能力,而且能够建立起密切相关菌株的进化途径。但是当选择的位点达到一定的数目,便只会增加人力、物力的消耗,而不能够获取更多的信息[28]。因此,本实验从7 个沙门菌属特异性靶点中筛选得到碱基差异位点最多的两个靶点——S9和S69。然后通过比较和整理S9与S69在沙门菌不同血清型菌株中的差异位点,绘制了血清分型靶点的差异位点表,从而获得这两个靶点同时用于14 个沙门菌血清型的快速分子血清分型的差异位点组合,最后将此组合应用于食品样品分离株的血清分型。血清分型结果与传统血清玻片凝集方法得到的结果进行比较,相符率达到100%。
在对比优势上,传统方法对食品中分离的疑似沙门菌进行鉴定时,需要经过生物化学鉴定和血清学分型验证[29-30]。生物化学鉴定程序繁琐,且需要用到生化鉴定试剂盒或全自动微生物生化鉴定系统[11]。传统血清分型则更需要经过多重的反复验证才能确定沙门菌血清型,其重复确认操作加重了鉴定工作量,增加了时间和成本。且检测过程中,操作人员的主观判断所占比例较大,误差较大,重复性也差[29]。而用分子靶点对疑似沙门菌进行鉴定,只需要经过PCR扩增和凝胶电泳就可以判断疑似菌株是否为沙门菌,然后将PCR产物测序,得到的序列与构建的靶点对应不同血清型差异位点组合进行比对,24 h内就能确定沙门菌的血清型。这一程序相对简单,工作量少,成本低廉,操作人员的主观判断所占比例很小,重复性好。且比对现有的分子分型方法,如PFGE、MLST和CRISPR等,本研究开发的方法也具备两大优点:其一,由于选用独特的沙门菌属特异性检测靶点,所以它不仅能判断疑似菌株是否为沙门菌,还能同步判断该沙门菌的血清型。涵括了疑似菌株的生化鉴定和血清分型两个过程;其二,分型靶点个数少,在成本和时间上比其他分子分型方法更具时间和成本优势。
基于本实验的研究结果,S9和S69两个靶点能够解决一定范围内沙门菌血清型的判别,将特异性靶点不仅用于沙门菌的检测更同步用于其血清分型,开创了沙门菌检测和分型同步的先例,且能够从分子层面上为沙门菌血清分型提供新思路。其快速有效的方式,对于及时有效控制沙门菌流行病学疾病的传播与暴发起到至关重要的决定性作用。
[1] 刘雯静, 邱少富, 刘雪林, 等. 沙门氏菌的分子分型方法[J]. 现代生物医学进展, 2010, 10(20): 3948-3950.
[2] KING A. From the Centers for Disease Control and Prevention.Outbreak of Salmonella serotype Muenchen infections associated with unpasteurized orange juice--United States and Canada, June 1999[J].Jama the Journal of the American Medical Association, 1999, 282(8)∶726-728. DOI∶10.1016/j.jmb.2008.07.004.
[3] YANG B W, QU D, ZHANG X L, et al. Prevalence and characterization of Salmonella serovars in retail meats of marketplace in Shaanxi, China[J]. International Journal of Food Microbiology,2010, 141(1/2): 63-72. DOI:10.1016/j.ijfoodmicro.2010.04.015.
[4] 张河战. 沙门氏菌的分类、命名及中国沙门氏菌菌型分布[J].微生物学免疫学进展, 2002, 30(2): 74-76. DOI:10.3969/j.issn.1005-5673.2002.02.018.
[5] 庄孝飞. 基于CRISPR序列的沙门氏菌分子分型方法的建立[D].上海: 上海交通大学, 2015: 1-20.
[6] UZZAU S, BROWN D J, WALLIS T, et al. Host adapted serotypes of Salmonella enterica[J]. Epidemiology and Infection, 2000, 125(2):229-255. DOI:10.1017/S0950268899004379.
[7] CHIU C H, SU L C. Salmonella enterica serotype Choleraesuis∶epidemiology, pathogenesis, clinical disease, and treatment[J]. Clinical Microbiology Reviews, 2004, 17(2)∶ 311-322. DOI∶10.1002/ps.857.
[8] FINN S, HINTON J C, MCCLURE P, et al. Phenotypic characterization of Salmonella isolated from food production environments associated with low-water activity foods[J]. Journal of Food Protection, 2013,76(9)∶ 1488-1499. DOI∶10.4315/0362-028X.JFP-13-088.
[9] LITRUP E, TORPDAHL M, MALORNY B, et al. Association between phylogeny, virulence potential and serovars of Salmonella enterica[J]. Infection Genetics & Evolution, 2010, 10(7)∶ 1132-1139.DOI∶10.1016/j.meegid.2010.07.015.
[10] LAW J W, AB MUTALIB N S, CHAN K G, et al. Rapid methods for the detection of foodborne bacterial pathogens: principles, applications,advantages and limitations[J]. Frontiers in Microbiology, 2015, 5: 770.DOI:10.3389/fmicb.2014.00770.
[11] 卫生部. 食品微生物学检验总则: GB/T 4789.4—2010[S]. 北京:中国标准出版社, 2010: 1-9.
[12] AOAC/BAM(Bacteriological Analytical Manual). 伯杰细菌鉴定手册[M].8版. 北京: 科学出版社, 1984: 386-389.
[13] SUKHNANAND S, ALCAINEL S, WARNICK L D, et al. DNA sequence-based subtyping and evolutionary analysis of selected Salmonella enterica serotypes[J]. Journal of Clinical Microbiology,2015, 43(8): 3688-3698. DOI:10.1128/JCM.43.8.3688-3698.2005.
[14] TANKOUOSANDJONG B, SESSITSCH A, LIEBANA E, et al.MLST-v, multilocus sequence typing based on virulence genes, for molecular typing of Salmonella enterica subsp. enterica serovars[J].Journal of Microbiological Methods, 2007, 69(1): 23-36. DOI:10.1016/j.mimet.2006.11.013.
[15] SHI C, SINGH P, RANIER M L, et al. Molecular methods for serovar determination of Salmonella[J]. Critical Reviews in Microbiology,2015, 41(3): 309-325. DOI:10.3109/1040841X.2013.837862.
[16] BARROW P A. ELISAs and the serological analysis of Salmonella infections in poultry: a review[J]. Epidemiology and Infection, 1992,109(3): 361-369. DOI:10.1017/S0950268800050354.
[17] 宋启发, 王爱红, 杨元斌, 等. 分子生物学技术在一起食物中毒检测中的应用[J]. 中国食品卫生杂志, 2014, 26(3): 254-258.DOI:10.13590/j.cjfh.2014.03.011.
[18] 邓子新, 喻子牛. 微生物基因组学及合成生物学进展[M]. 北京: 科学出版社, 2014: 115-117.
[19] RAHN K, DE GRANDIS S A, CLARKE R C, et al. Amplification of an invA gene sequence of Salmonella typhimurium by polymerase chain reaction as a specific method of detection of Salmonella[J]. Molecular and Cellular Probes, 1992, 6(4): 271-279.DOI:10.1016/0890-8508(92)90002-F.
[20] LIN C K, HUNG C L, HSU S C, et al. An improved PCR primer pair based on 16S rDNA for the specific detection of Salmonella serovars in food samples[J]. Journal of Food Protection, 2004, 67(7): 1335-1343. DOI:10.4315/0362-028X-67.7.1335.
[21] KWANG J, LITTLEDIKE E T, KEEN J E. Use of the polymerase chain reaction for Salmonella detection[J]. Letters in Applied Microbiology, 1996, 22(1): 46-51. DOI:10.1111/j.1472-765X.1996.tb01106.x.
[22] 裴晓方, 文华, 占利, 等. 12小时PCR技术快速检测沙门菌[J]. 中国卫生检验杂志, 2002, 12(5): 526-527. DOI:10.3969/j.issn.1004-8685.2002.05.006.
[23] AABO S, RASMUSSEN O F, ROSEEN L, et al. Salmonella identification by the polymerase chain reaction[J]. Molecular and Cellular Probes, 1993, 7(3): 171-178. DOI:10.1006/mcpr.1993.1026.
[24] HALATSI K, OIKONOMOU I, LAMBIRI M, et al. PCR detection of Salmonella spp. using primers targeting the quorum sensing gene sdiA[J]. Fems Microbiology Letters, 2006, 259(2): 201-207.DOI:10.1111/j.1574-6968.2006.00266.x.
[25] ZHOU X J, ZHANG L D, SHI C L, et al. Genome-scale screening and validation of targets for identification of Salmonella enterica and serovar prediction[J]. Journal of Food Protection, 2016, 79(3): 376-383. DOI:10.4315/0362-028X.JFP-15-286.
[26] 朱超, 许学斌. 沙门菌属血清型诊断[M]. 上海: 同济大学出版社,2009: 68-77.
[27] 周秀娟. 沙门氏菌C1血清组特异基因功能分析[D]. 上海: 上海交通大学, 2015: 44-45.
[28] 王中强, 邱少富, 王勇, 等. 多位点序列分型技术及其研究进展[J]. 军事医学, 2010, 34(1): 76-79. DOI:10.3969/j.issn.1674-9960.2010.01.021.
[29] 但现龙. 沙门氏菌属特异分子检测靶点[D]. 上海: 上海交通大学,2011: 1-8.
[30] 向雪菲. 食品中沙门氏菌分子检测靶点的筛选与评价[D]. 上海:上海交通大学, 2008: 5-16.
Identification of Single Nucleotide Polymorphisms (SNPs) in Different Serotypes of Salmonella-Specific Targets and Their Potential for Molecular Serotyping
SHI Xiuqing, ZHOU Xiujuan, SHI Chunlei, ZHANG Fen, SHI Xianming*
(MOST-USDA Joint Research Center for Food Safety, State Key Laboratory of Microbial Metabolism,School of Agriculture and Biology, Shanghai Jiao Tong University, Shanghai 200240, China)
The single nucleotide polymorphisms (SNPs) in different serotypes of 7 specific targets for Salmonella detection were analyzed based on the whole genome sequences of 28 Salmonella strains (14 serotypes) published on NCBI. The results showed that S9 and S69 had more SNPs than any other target, making them a potential tool for Salmonella serotyping.Subsequently, the distinct nucleotide mutation sites of the two candidate molecular serotyping targets in different serotypes of Salmonella were analyzed. As a result, various combinations of the mutation sites were obtained for rapid molecular serotyping of 14 Salmonella serotypes, and the results showed that identical mutation sites existed in the same serovar,while different mutation sites were found in diverse serovars. Finally, 21 suspected Salmonella isolates were obtained by the national standard method from 192 food samples collected from supermarkets and open-air markets in Shanghai. The results of molecular serotyping by targets S9 and S69 were 100% consistent with the results of the traditional slide agglutination test; the Salmonella isolates were assigned to 4 different serovars, 10 strains of Salmonella Enteritidis, 7 strains of Salmonella Typhimurium, 2 strains of Salmonella Gallinarum and 2 strains of Salmonella Newport. The results of genome sequence analysis and strain serotyping showed that the combination of Salmonella-specific targets S9 and S69 had the potential for molecular serotyping of Salmonella, which is expected to be a time-saving and cheaper alternative to the traditional slide agglutination test and can provide a new idea for the application of PCR technology in Salmonella molecular serotyping.
Salmonella; molecular serotyping; Salmonella-specific targets; food samples; serum identification
2016-12-20
政府间国际科技创新合作重点专项(2016YFE0106100)
石秀清(1991—),男,硕士研究生,研究方向为食品安全与微生物。E-mail:249597976@qq.com
*通信作者:史贤明(1961—),男,教授,博士,研究方向为食品安全与微生物。E-mail:xmshi@sjtu.edu.cn
DOI∶10.7506/spkx1002-6630-201724008
Q781
A
1002-6630(2017)24-0047-06
石秀清, 周秀娟, 施春雷, 等. 沙门菌属特异性检测靶点碱基差异位点的识别及其血清型决定力[J]. 食品科学, 2017,38(24): 47-52.
10.7506/spkx1002-6630-201724008. http://www.spkx.net.cn
SHI Xiuqing, ZHOU Xiujuan, SHI Chunlei, et al. Identification of single nucleotide polymorphisms (SNPs) in different serotypes of Salmonella-specific targets and their potential for molecular serotyping[J]. Food Science, 2017, 38(24)∶ 47-52.(in Chinese with English abstract) DOI∶10.7506/spkx1002-6630-201724008. http∶//www.spkx.net.cn
