不同基因型谷子的RAPD分析
2017-12-02衡水学院生命科学学院河北衡水053000河北省农林科学院旱作农业研究所河北衡水053000
, , , (.衡水学院生命科学学院, 河北 衡水 053000; .河北省农林科学院旱作农业研究所, 河北 衡水 053000)
不同基因型谷子的RAPD分析
郭晓丽1,时丽冉1,吕亚慈1,李明哲2
(1.衡水学院生命科学学院, 河北 衡水 053000; 2.河北省农林科学院旱作农业研究所, 河北 衡水 053000)
利用随机多态性DNA (RAPD)分子标记技术分析了9种不同基因型谷子的遗传差异。采用20个随机引物对谷子基因组DNA进行RAPD-PCR扩增,共得到616条带,其中多态性条带有389条,多态性比例达到了63.2%。聚类分析表明,以遗传相似系数为0.68 处可将9个谷子品种划分为3类。
谷子; RAPD; 聚类分析
谷子,又名粟或者小米,现主要分布于我国东北、西北、华北等地区。谷子是北方人民的主食之一,谷粒有很高的价值,含有丰富的蛋白质和维生素等营养物质,同时谷子拥有生长周期短、耐瘠薄、耐旱、抗逆性强、籽粒易储藏等特性,是干旱地区的重要粮食作物[1-2]。但由于谷子的品种太过繁多,而且易与野生狗尾巴草杂交不断产生中间种,想要通过传统的形态标记等方法来完成品种鉴定、良种选育及谷子近缘种分析并不容易。
注:从左至右依次为品种1至品种9。下同。图1 9个谷子品种的DNA电泳检测
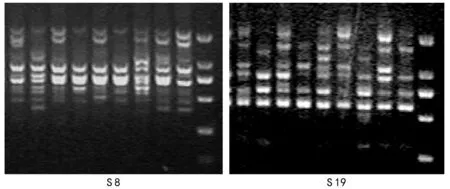
图2 引物S 8、S 19对9个谷子品种的RAPD-PCR扩增结果
随机扩增多态性DNA(RAPD)技术是1990年发明的一种建立于PCR基础之上的可以对未知序列的基因组进行多态性分析的分子标记技术[3-4]。RAPD技术快捷便利,不需要经过分子杂交这一操作,并且随着反应体系的不断优化和检测手段的相应进步,加之目前RAPD方法可大规模自动化分析样品,RAPD辅助选择育种和其他相关应用研究更为深入彻底,现已广泛应用于小麦[5]、玉米[6]、水稻[7]等作物的系谱分析、品种鉴定及亲缘关系分析。
本研究以9种不同基因型谷子为材料,采用20种随机引物(S 1~S 20)分别对不同谷子基因组DNA进行RAPD分析,进而探讨谷子样品间的遗传多样性和亲缘关系,从而为谷子资源的利用和开发奠定基础,同时为谷子育种的亲本选配工作提供一定的理论依据。
1 材料与方法
1.1 材 料
9个谷子品种:冀谷11、龙谷27、首农35、M 0902、晋谷21、衡谷10、200151、龙谷25及200152(依次标号为1~9),均由河北省农林科学院旱作农业研究所提供。
1.2 方 法
1.2.1 谷子的培养
选取优质饱满、无病虫害的谷子各100粒,用0.1%氯化汞消毒10 min,经去离子水冲洗干净后,放于铺有2层滤纸的培养皿中加入适量蒸馏水浸种发芽。选取发芽良好的植株置于25 ℃的培养箱中培养,待长至2叶1心时选取幼嫩叶片进行基因组DNA提取。
1.2.2 基因组DNA的提取
9个谷子品种的基因组DNA采用改良的CTAB法[8]提取。
1.2.3 DNA的浓度测定
在紫外分光光度计上检测样品在波长为260 nm处的光密度值,计算出DNA的浓度,并最终将所有样品的浓度稀释至15 ng/μL,备用。
1.2.4 RAPD-PCR扩增
RAPD引物是由生工生物工程(上海)股份有限公司合成的随机引物S 1~S 20。10μL反应体系包括:10×PCR Buffer 1μL,dNTP 1μL,RAPD引物0.5μL,DNA 2μL,DNA聚合酶0.5μL,ddH2O补足至10μL。反应程序为:94 ℃预变性5 min,94 ℃变性1 min,37 ℃退火1 min,72 ℃延伸2 min,共设45个循环,最后72 ℃延伸10 min,4 ℃保存。取RAPD扩增产物在浓度1.5%的琼脂糖凝胶上电泳,用荧光染料溴化乙锭进行染色,最后在紫外凝胶成像仪上扫描观察。
1.2.5 数据分析
采用Excel软件整理记录原始数据,统计出每个引物所扩增出的总带数和其中的多态性条带数,得出多态性位点的百分率。将每一条带视为一个性状,按凝胶同一位置上DNA带的有无进行统计,有带的记作“1”,无带的则记作“0”,构建出一个0-1矩阵,最后应用NTSYSv 2.10 e软件计算谷子的遗传相似系数,再用UPUMA方法进行谷子的聚类分析。
2 结果与分析
2.1 不同品种谷子基因组DNA检测
通过琼脂糖凝胶电泳对9种不同谷子的基因组DNA完整性进行检测(图1),所提取的基因组DNA条带清晰,降解程度较低,虽然混有少量的RNA,但不影响后续PCR扩增结果。
2.2 不同品种谷子RAPD-PCR电泳图分析
将RAPD-PCR扩增后的产物进行琼脂糖凝胶电泳,再用紫外凝胶成像系统进行扫描观察。每个引物重复扩增3次,选取清晰准确的图片进行扩增分析(图2)。

注:1为冀谷11;2为龙谷27;3为首农35;4为M 0902;5为晋谷21;6为衡谷10;7为200151;8为龙谷25;9为200152。图3 9个谷子品种的聚类分析
将扫描后的电泳图进行统计分析,结果如表1所示。在所有引物扩增出的特异性条带数中,引物S 4、S 15、S 16、S 19所占比例最高,达到70%以上。最低的为引物S 5,没有扩增出特异条带。随机引物S 1~S 20共计扩增出条带616条,其中多态性带有389条,多态性比例达到了63.2%。
2.3 不同品种谷子的聚类分析
以9个谷子品种的扩增图谱为基础,进一步采用NTSYS软件进行处理,应用UPGMA法进行不同谷子的聚类分析。由图3可以看出,9份谷子样本在遗传相似系数为0.68 处可划分为3类。第1类为:冀谷11,首农35,晋谷21,200151,龙谷25和200152。其中,龙谷25和200152谷子品质间的亲缘关系最为接近,遗传相似系数为0.851;其次为首农35和晋谷21。在本组中,各品种之间的遗传相似系数均在0.711~0.851之间,亲缘关系比较接近。第2类为:M 0902和衡谷10,它们之间的遗传相似系数为0.754。第3类为龙谷27,它与其他品种的遗传距离相对均较远。
表1 20种随机引物的扩增结果
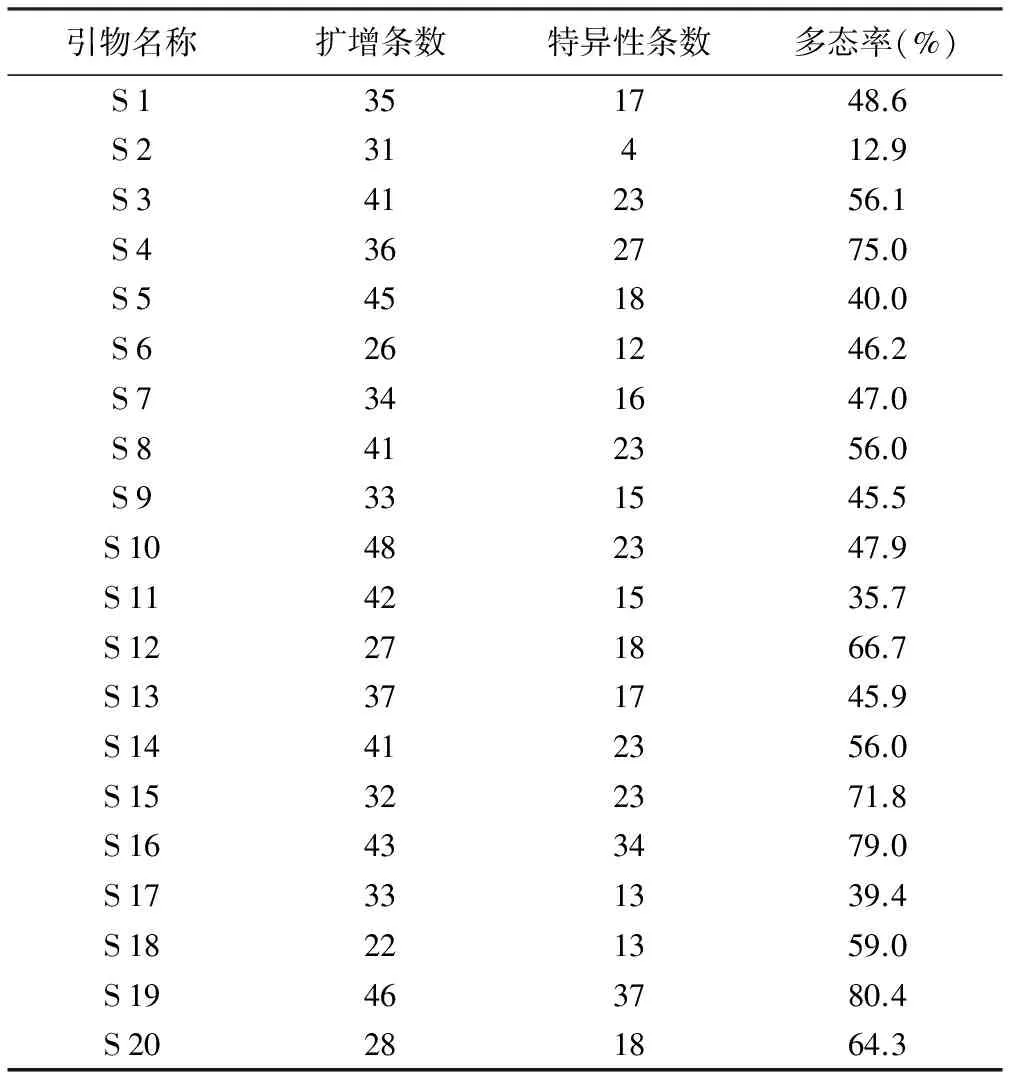
引物名称扩增条数特异性条数多态率(%)S1351748.6S231412.9S3412356.1S4362775.0S5451840.0S6261246.2S7341647.0S8412356.0S9331545.5S10482347.9S11421535.7S12271866.7S13371745.9S14412356.0S15322371.8S16433479.0S17331339.4S18221359.0S19463780.4S20281864.3
3 讨 论
种质资源是遗传育种的物质基础,作物的遗传多样性研究历来都是遗传育种工作的重要方面。随着分子生物学的理论基础及相关技术的迅猛发展,在农作物遗传育种的研究中越来越得到广泛的应用。近年来,随着RAPD、SSR、RFLP等分子标记技术的快速发展,分子标记越来越多的被应用于谷子研究中[9]。
本研究通过采用RAPD技术对不同品种谷子进行分析发现,9个谷子品种之间的遗传相似系数为0.526~0.851,表明不同品种间存在一定的遗传差异。同时谷子品种龙谷27和200151之间遗传距离最远(0.526),龙谷25和200152之间遗传距离最近(0.851)。同时,从电泳图中也发现,龙谷27在不同引物扩增结果中均出现了与其他品种差异较大的扩增条带(图2),充分说明了它与其他品种间的基因组差异较大,可在杂交育种中优先考虑作为候选品种。
[1]张雪峰.中国谷子产业发展问题研究[D].东北农业大学,2013:18-19.
[2]张海金.谷子在旱作农业中的地位和作用[J].安徽农学通报,2007,13(10):169-170.
[3]Williams,J.G.K.DNA polymorphisms amplified by arbitrary primers are useful as genetic markers[J].Nucleic Research,1990,18(22):6 531-6 535.
[4]杨天育,窦全文,沈裕琥,等.应用RAPD标记研究不同生态区谷子品种的遗传差异[J].西北植物学报,2003,23(5):765-770.
[5]郑爱泉,张宝林.DNA分子标记技术及其在小麦遗传多样性研究中的应用[J].安徽农业科学,2015(17):40-42.
[6]李树国,隋昌海,王琳.玉米种质资源RAPD分子标记优化体系的建立[J].吉林农业,2012(10):50-56.
[7]庞瑞华,王玲,李小宁,等.信阳地区水稻资源的遗传多样性分析[J].江苏农业科学,2016,44(7):113-116.
[8]王英,邱海燕,高和琼,等.甘蔗基因组DNA提取方法的研究[J].中国农学通报,2008,24(12):44-49.
[9]麻慧芳,杨成元,史关燕,等.我国谷子种质资源保存研究综述[J].甘肃农业科技,2015(3):57-60.
RAPD Analysis of Different Millet Genotypes
GUOXiaoli1,SHILiran1,LÜYaci1,LIMingzhe2
(1.Department of Life Science of Hengshui University,Hengshui Hebei 053000,China;2.Dry Land Farming Institute,Hebei Academy of Agricultural and Forestry Science,Hengshui Hebei 053000,China)
s:In this paper,RAPD molecular-marker technique was used to detect the genetic difference among 9 Millets.Using 20 primers for genomic DNA,we obtained 616 bands.389 polymorphic bands were detected,representing of 63.2%.UPGMA cluster analysis showed that these accessions could be divided into three groups.
millet; RAPD; cluster analysis
2017-01-13
河北省高等学校科学研究计划项目(Z 2014014)。
郭晓丽(1977—),女,河北邯郸人;博士,副教授,主要从事植物分子遗传学和基因工程方向研究;E-mail:gxllgf@163.com。
李明哲(1976—),男,河北饶阳人;硕士,副研究员,主要从事作物育种研究;E-mail:limingzhe1976@yahoo.com.cn。
10.16590/j.cnki.1001-4705.2017.07.029
S 515
A
1001-4705(2017)07-0029-03
