多油辣木转录组高通量测序及分析
2017-11-30欧奇李鑫田洋曾千春
欧奇+李鑫+田洋+曾千春


摘要:为了研究多油辣木(Moringa oleifera Lam.)中黄酮类化合物生物合成的分子基础,通过Illumina HiSeq 2000高通量测序技术对辣木茎、叶进行转录组测序,利用Trinity软件将数据组装成Unigene,基于BLAST对所有Unigene进行功能注释,共获得49 365个Unigene,平均长度为903 bp。通过GO分类,15 959个Unigene被分成生物学过程、细胞组分和分子功能3个主要类别。通过KOG分类,8 713个Unigene被分为26个种类。通过KEGG分类,8 397个Unigene分属于130个代谢途径,其中代谢所含Unigene最多,共3 620个。在代谢途径中,找到参与黄酮类化合物合成相关的Unigene 45个,其中包括查尔酮合酶、查尔酮异构酶、黄烷酮-3-羟化酶、黄酮醇合成酶和二氢黄酮醇还原酶等。研究结果为挖掘多油辣木黄酮类化合物生物合成关键基因提供了基础数据,并为下一步的资源开发和利用奠定了基础。
关键词:多油辣木;转录组;黄酮类化合物;高通量测序
中图分类号: Q522+.6 文献标志码: A 文章编号:1002-1302(2017)20-0071-05
多油辣木(Moringa oleifera Lam.)属辣木科(Moringaceae)辣木属(M. Adans),是一种具有独特经济价值的多年生热带植物,现广泛种植于亚洲、非洲和中美洲[1]。多油辣木中含有丰富的脂肪酸、蛋白质、多糖、维生素和黄酮类化合物[2]。相关研究表明,多油辣木提取液富含黄酮类化合物,具有显著的抗炎[3]、降血脂[4]、抗肿瘤[5]和抗氧化[6-7]等药用效果。黄酮类化合物包括黄酮、异黄酮、黄酮醇、查尔酮等,是植物在长期进化过程中产生的一类次生代谢产物[8],黄酮类化合物广泛存在于高等植物中,具有抗氧化、抗炎、降血脂、抗肿瘤和扩张血管等药理活性[9]。黄酮类化合物合成的一般途径已经明确,首先,丙二酰辅酶A(malonyl-CoA)和4-香豆酰辅酶A(4-coumaroyl-CoA)在查尔酮合酶(chalcone synthase,简称CHS)[10]催化下合成查尔酮;接着,查尔酮在查尔酮异构酶(chalcone isomerase,简称CHI)[11]的催化下合成黄烷酮;然后,黄烷酮能被黄酮合成酶(flavone synthase,简称FNS)[12]催化合成黄酮,可被异黄酮合成酶(isoflavone synthase,简称IFS)[13]催化合成异黄酮,也能被黄烷酮-3-羟化酶(flavanone-3-hydroxyalse,简称F3H)[14]催化合成二氢黄酮醇;最后,二氢黄酮醇在黄酮醇合成酶(flavonol synthase,简称FLS)[15]的催化下合成黄酮醇。黄酮类化合物合成关键酶还包括二氢黄酮醇还原酶(dihydroflavonol 4-reductase,简称DFR)[16]、类黄酮3-O-葡萄糖基转移酶(flavonol 3-O-glucosyltransferase,简称3GT)[17]等。
转录组(transcriptome)是指细胞或组织内全部RNA转录本的集合,代表基因在不同生命阶段、不同生理状态、不同组织类型以及不同环境条件下的表达水平,包括基因表达的所有转录产物(mRNA、Non-coding RNA)的总和。转录组测序(RNA sequencing)是利用Illumina HiSeq 2000高通量测序技术进行cDNA测序,能更全面快速地揭示生物个体特定组织和特定时期的基因表达情况[18]。与基因组学研究相比,转录组学更偏重基因编码区域,它能进一步呈现细胞内生命活动,能为诠释细胞的功能提供具有价值的参考信息[19]。由于转录组学在基因功能研究中的重要作用,近年来,转录组的研究对象逐渐从模式生物和重要农作物向其他植物延伸。本研究首次对多油辣木进行转录组测序并加以分析,以期从中挖掘具有重要功能的基因。
1 材料与方法
1.1 转录组测序
多油辣木样品采自云南昆明,选取同一生活环境下的一年生辣木茎、叶作为混合研究材料,于-80 ℃超低温冰冻保存备用。用TRIzol法[20]提取材料总RNA,构建测序文库,使用Illumina HiSeq 2000进行测序。
1.2 测序序列拼装
通过Base Calling将测序得到的原始图像转化为序列数据,这些数据称为原始测序序列(Raw Reads)。将获得的原始测序序列進行过滤得到处理序列(Clean Reads)。然后利用Trinity软件[21]对处理序列进行拼接。取每条基因中最长的转录本(transcript)作为Unigene,以此进行后续分析。
1.3 Unigene功能注释、GO分类和代谢路径分析
通过BLAST将Unigene与Nr、Nt、Pfam、KOG、Swiss-Prot、KEGG、GO七大数据库进行比对(E值<0.000 01),获得序列相似性最高的蛋白,从而将此蛋白信息用作该Unigene的蛋白功能注释信息。GO是一套国际标准化的基因功能描述的分类系统,分为生物过程(biological process)、分子功能(molecular function)和细胞组分(cellular component)三大类。它们分别用来描述基因编码的产物所参与的生物过程、所具有的分子功能及所处的细胞环境。通过Blast2GO软件[22]对Unigene进行GO注释,然后用WEGO软件[23]对其进行GO功能分类统计,从宏观上了解辣木基因功能分布特征。KEGG是系统分析基因产物和化合物在细胞中的代谢途径以及这些基因产物功能的数据库[24]。根据KEGG注释进一步得到Unigene的代谢路径。
2 结果与分析endprint
2.1 转录组序列的产出、组装和基因表达
通过Illumina Hiseq 2000高通量测序共获得了 57 818 454 条高质量短读序片段,总长度为9 309 341 614 bp,Q20值为95.89%,GC含量占比为46.92%。由Trinity软件组装共获得72 527个Contig,总长度为88 344 692 bp,平均长度为1 218 bp,N50长度为2 144 bp。共组装49 365个Unigene,总长度为44 565 055 bp,平均长度为903 bp,N50长度为 1 771 bp(表1)。对Contig、Unigene的长度分布特征进行分析可知,在总Contig中,0~300 bp区段所含的Contig比例最高,为23.34%,共16 931个;1 001~2 000 bp区段的Contig占21.47%;>2 000 bp區段的Contig占20.73%。在Unigene中,同样以0~300 bp区段所含Unigene比例最高,为 32.38%,共15 983个;1 001~2 000 bp区段的Unigene占 15.14%;大于2 000 bp区段的Unigene占12.75%(表2)。
2.2 功能注释
使用BLAST将所有Unigene与7大数据库进行比对,由表3可以看出,在49 365个Unigene中,共有23 810个Unigene获得注释信息,占总Unigene数的48.23%(表3)。在Nr数据库中注释Unigene数量最多,为21 442个,占总数的43.43%。在KEGG、KOG数据库中注释的Unigene数量都在10 000个以下,分别为8 397个(17.01%)、8 713个(17.65%);其他数据库中注释的Unigene数量都在10 000~20 000个之间,但仍有25 555个(51.77%)功能未知的Unigene。
2.3 GO注释
通过GO分析,15 959个Unigene被分成了生物学过程、细胞组分和分子功能3个主要类别(图1)。在“生物学过程”类别中,细胞过程(9 229个Unigene)功能组所含Unigene数量最多,细胞聚集(1个Unigene)所含Unigene数量最少。在“细胞组分”类别中,细胞(5 075个Unigene)和细胞部分(5 075 个Unigene)功能组所含Unigene数量最多,共质体(1个Unigene)所含Unigene数量最少。在“分子功能”类别中,结合功能(9 009个Unigene)所含Unigene数量最多,金属伴侣(3个Unigene)所含Unigene数量最少。
2.4 KOG注释
为了进一步评估Unigene的完整性和注释的有效性,对 8 713 个Unigene进行KOG注释和分类,共获得26个种类(图2)。在26个KOG种类中,一般功能预测(R)为最大类,共有1 492个Unigene;然后是翻译后修饰、蛋白转换和分子伴侣(O),有1 175个Unigene;未知功能蛋白(X)、细胞运动(N)为最小类,分别只有1、3个Unigene。
2.5 KEGG注释
为了识别辣木中活性高的代谢通路,对8 397个Unigene进行KEGG代谢通路分析,将其分为代谢(3 620个Unigene)、遗传信息调控(1 796个Unigene)、环境信息调控(337个Unigene)、有机系统(346个Unigene)和细胞过程(382个Unigene)5大类130个代谢途径(图3)。其中包含Unigene数量最多的是遗传信息调控类中的翻译,共有749个;代谢类中的糖代谢有732个Unigene;细胞过程类中的运输和代谢有382个Unigene;有机系统类中的环境适应性有346个Unigene;环境信息调控类中的膜运输包含Unigene数量最少,仅有55个。
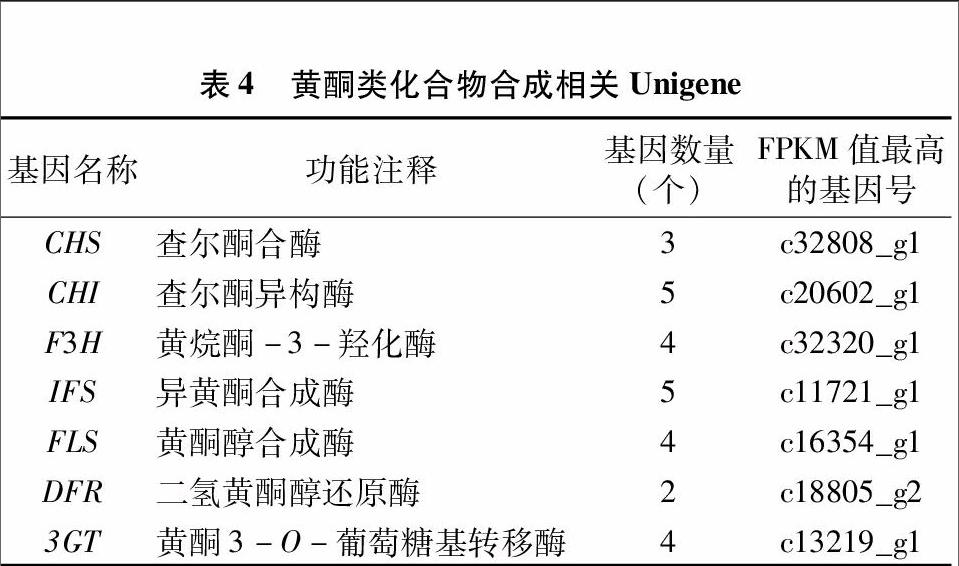
2.6 黄酮类化合物合成相关Unigene
基于数据库中已知的基因信息,找到与黄酮类化合物合成相关的Unigene 45个,其中与查尔酮合酶有关的Unigene有3个,每100万片段中来自某一基因每1 000碱基长度的片段数(expected number of fragments per kilobase of transcript sequence per millions base pairs sequenced,简称FPKM,FPKM同时考虑了测序深度、基因长度对片段计数的影响,是目前最为常用的基因表达水平估算方法)[25]最高的Unigene基因号为c32808_g1;与查尔酮异构酶有关的Unigene有5个,FPKM值最高的Unigene基因号为c20602_g1;与黄烷酮-3-羟化酶有关的Unigene有4个, FPKM值最高的Unigene基因号为c32320_g1;与异黄酮合成酶有关的Unigene有5个,FPKM值最高的Unigene基因号为c11721_g1;与黄酮醇合成酶有关的Unigene有4个,FPKM值最高的Unigene基因号为c16354_g1;与二氢黄酮醇还原酶有关的Unigene有2个,FPKM值最高的Unigene基因号为c18805_g2;与黄酮3-O-葡萄糖基转移酶有关的Unigene有4个,FPKM值最高的Unigene基因号为c13219_g1(表4)。
3 讨论与结论
本研究首次采用Illumina HiSeq 2000高通量测序技术对多油辣木进行转录组测序,共获得57 818 454条高质量短读序,经拼装后得到Unigene 49 365个,平均长度为903 bp,N50长度为1 771 bp。在Unigene中,0~300 bp区段所含比例最高,为32.38%;301~1 000 bp区段占39.74%;1 001~2 000 bp 区段占15.14%,>2 000 bp区段占12.75%。由此可见Unigene的整体长度分布均匀,组装的质量和长度都能满足转录组分析的基本要求。测序结果的好坏除了与组装质量和长度有关外,还与测序数据量密切相关,数据量越大得到的基因序列,越长越准确。相关研究表明,辣木基因组总大小为289 Mb[26],因此本研究8Gb的测序量足以保证获得足够长的组装序列和足够多的Unigene。在与7大数据库比对时,发现有25 555(51.77%)个功能未知的Unigene。对这些Unigene进行深入研究,极有可能获得与多油辣木特性有关的新基因。endprint
黄酮类化合物广泛存在于高等植物中[27-28],本研究找到多油辣木中与黄酮类化合物合成相关的Unigene 45个,包括CHS、CHI、F3H、FLS和DFR等关键合成基因,与其他植物中参与生物合成的关键基因基本一致[29]。多油辣木中与黄酮、黄酮醇合成的相关Unigene有8个,与异黄酮生物合成相关的Unigene有6个,与花青素生物合成有关的Unigene有5个,与其他黄酮类化合物合成有关的Unigene有26个。这些基因与淫羊藿属(Epimedium L.)[30]植物存在一定差异,造成差异的原因可能是不同物种所含的黄酮类化合物存在差异、特定时间和特定组织内基因表达存在差异、转录组测序过程中提取和拼接存在误差。
田洋等完成了多油辣木基因组草图,测序结果表明,辣木基因組289 Mb,共注释出19 465个基因,每个基因的平均长度为3 354.22 bp。这19 465个基因在GO数据库中共注释基因有10 476个,占53.82%;在KEGG中注释了10 936个基因,占56.18%;未知功能的基因有1 166个,占5.99%[26]。该团队还挖掘了热休克蛋白(HSP)基因、BAK1(BRI1 associated receptor kinase 1)基因、γ-氨基丁酸和谷甾醇的合成途径相关基因。通过比较可知,本研究获得的Unigene (49 365个)和在GO数据库中注释的Unigene(15 959个)均较多,但在KEGG数据库中注释的Unigene(8 397个)较少。本研究弥补了多油辣木转录组数据的缺陷,获得了多油辣木黄酮类化合物生物合成相关的候选基因,为多油辣木资源开发和利用提供了重要的理论基础。
参考文献:
[1]刘昌芬,李国华. 辣木的研究现状及其开发前景[J]. 云南热作科技,2002,25(3):20-24.
[2]刘凤霞,王苗苗,赵有为,等. 辣木中功能性成分提取及产品开发的研究进展[J]. 食品科学,2015,36(19):282-286.
[3]Waterman C,Cheng D M,Rojas-Silva P,et al. Stable,water extractable isothiocyanates from Moringa oleifera leaves attenuate inflammation in vitro[J]. Phytochemistry,2014,103(10):114-122.
[4]Ghasi S,Nwobodo E,Ofili J O. Hypocholesterolemic effects of crude extract of leaf of Moringa oleifera Lam in high-fat diet fed wistar rats[J]. Journal of Ethnopharmacology,2000,69(1):21-25.
[5]Jung I L. Soluble extract from Moringa oleifera leaves with a new anticancer activity[J]. PLoS One,2014,9(4):e95492.
[6]Vongsak B,Sithisarn P,Gritsanapan W. Bioactive contents and free radical scavenging activity of Moringa oleifera leaf extract under different storage conditions[J]. Industrial Crops and Products,2013,49(4):419-421.
[7]Verma A R,Vijayakumar M,Mathela C S,et al. In vitro and in vivo antioxidant properties of different fractions of Moringa oleifera leaves[J]. Food and Chemical Toxicology,2009,47(9):2196-2201.
[8]诸 姮,胡宏友,卢昌义,等. 植物体内的黄酮类化合物代谢及其调控研究进展[J]. 厦门大学学报(自然科学版),2007,46(增刊1):136-143.
[9]Chen Z,Hu Y,Wu H,et al. Synthesis and biological evaluation of flavonoids as vasorelaxant agents[J]. Bioorganic & Medicinal Chemistry Letters,2004,14(15):3949-3952.
[10]Koes R E,Quattrocchio F,Mol J N M. The flavonoid biosynthetic pathway in plants:function and evolution[J]. Bioessays,1994,16(2):123-132.
[11]Jez J M,Bowman M E,Dixon R A,et al. Structure and mechanism of the evolutionarily unique plant enzyme chalcone isomerase[J]. Nature Structural Biology,2000,7(9):786-791.
[12]Martens S,Forkmann G,Matern U,et al. Cloning of parsley flavone synthase I[J]. Phytochemistry,2001,58(1):43-46.endprint
[13]Jung W,Yu O,Lau S M,et al. Identification and expression of isoflavone synthase,the key enzyme for biosynthesis of isoflavones in legumes[J]. Nature Biotechnology,2000,18(2):208-212.
[14]Jin Z,Grotewold E,Qu W,et al. Cloning and characterization of a flavanone 3-hydroxylase gene from Saussurea medusa[J]. DNA Sequence,2005,16(2):121-129.
[15]Forkmann G,Martens S. Metabolic engineering and applications of flavonoids[J]. Current Opinion in Biotechnology,2001,12(2):155-160.
[16]Dick C A,Buenrostro J,Butler T,et al. Arctic mustard flower color polymorphism controlled by petal-specific downregulation at the threshold of the anthocyanin biosynthetic pathway[J]. PLoS One,2011,6(4):e18230.
[17]Owens D K,Mcintosh C A. Identification,recombinant expression,and biochemical characterization of a flavonol 3-O-glucosyltransferase clone from Citrus paradisi[J]. Phytochemistry,2009,70(11/12):1382-1391.
[18]Ajay S S,Parker S C,Abaan H O,et al. Accurate and comprehensive sequencing of personal genomes[J]. Genome Research,2011,21(9):1498-1505.
[19]吴 琼,孙 超,陈士林,等. 转录组学在药用植物研究中的应用[J]. 世界科学技术:中医药现代化,2010,12(3):457-462.
[20]姚宁涛,祝建波,邓福军. 改良Trizol法快速提取棉叶片总RNA[J]. 生物技术通报,2010(7):125-127.
[21]Grabherr M G,Haas B J,Yassour M,et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nature Biotechnology,2011,29(7):644-652.
[22]Conesa A,Gtz S,García-Gómez J M,et al. Blast2GO:a universal tool for annotation,visualization and analysis in functional genomics research[J]. Bioinformatics,2005,21(18):3674-3676.
[23]Ye J,Fang L,Zheng H,et al. WEGO:a web tool for plotting GO annotations[J]. Nucleic Acids Research,2006,34(Web Server issue):W293-W297.
[24]Kanehisa M,Araki M,Goto S,et al. KEGG for linking genomes to life and the environment[J]. Nucleic Acids Research,2008,36(Database issue):D480-D484.
[25]Trapnell C,Williams B A,Pertea G,et al. Transcript assembly and abundance estimation from RNA-Seq reveals thousands of new transcripts and switching among isoforms[J]. Nature Biotechnology,2010,28(5):511-515.
[26]田 洋,曾 严,张 静,等. 辣木(Moringa oleifera Lam.)的高质量参考基因组[J]. 中国科学:生命科学,2015(5):488-497.
[27]王兴娜,汪 晶,黄午阳,等. 青梅果实不同极性组分的抗氧化活性[J]. 江苏农业学报,2016,32(1):211-215.
[28]朱海军,生静雅,张普娟,等. 贮藏温度对薄壳山核桃抗氧化功能及品质的影响[J]. 江苏农业学报,2015,31(2):449-453.
[29]康亚兰,裴 瑾,蔡文龙,等. 药用植物黄酮类化合物代谢合成途径及相關功能基因的研究进展[J]. 中草药,2014,45(9):1336-1341.
[30]张华峰,王 瑛,黄宏文.endprint
