基于分子动力学模拟研究定点突变对葡聚糖酶热稳定性的影响
2017-11-30韦阳道易弋石征宇李家杰苏家敏伍时华黎娅
韦阳道+易弋+石征宇+李家杰+苏家敏+伍时华+黎娅

摘要:为提高葡聚糖酶的热稳定性,对葡聚糖酶的三维结构进行模拟分析,获得2个突变体R226A、G224A,用分子动力学的方法从原子水平上研究突变体与野生型的耐热性。结果显示,突变体R226A、G224A的均方根偏差,回旋半径和静电势能等参数值要低于野生型蛋白,表明突变体的热稳定性较野生型明显上升,更有利于在高温环境下发挥其生物活性。通过计算机模拟分析的方法研究定点突变对野生型酶的热稳定性的影响,可大大节省试验时间,同时可为阐明葡聚糖酶耐热性机制提供理论依据。
关键词:分子动力学;定点突变;葡聚糖酶;热稳定性
中图分类号: S188 文献标志码: A 文章编号:1002-1302(2017)20-0054-05
β-葡聚糖酶是专一作用于β-葡聚糖的1,3-及1,4-糖苷键,可产生3~5个葡聚糖单位的低聚糖及葡萄糖[1]。由于β-葡聚糖广泛存在于植物性饲料中而不能被动物所利用,进而造成饲料的饲用价值降低。此外,含有较多β-葡聚糖的饲料被动物食用后,会影响动物内源性消化酶与营养物质的接触,导致动物的消化率下降[2]。因此,在饲料的加工过程中添加β-葡聚糖酶将有效降低β-葡聚糖的不良影响,提高动物的消化率、饲料的利用率[3]。近年来,由于饲料加工过程中须经历高温高压的剧烈条件等原因,人们对嗜热 β-葡聚糖酶的研究越来越关注。
计算机分子模拟是以计算机及计算技术为工具和手段,运用计算数学的方法,解决复杂物理、化学、生物等问题的应用科学[4],特别是随着近年来计算机技术的飞速发展,该手段发展越来越完善,其模拟试验所得结果与真实试验结果极为相似,因此可以辅助真实试验[5],这样可以大大节省试验成本和人力。由于这些优点,计算机分子模拟技术被广泛应用,在生物学体系的研究中,该手段被用于解释核酸与蛋白质之间的相互作用[6]、蛋白质与蛋白质之间相互作用[7]、药物与抗原的作用机制[8]等,已经成为世界研究前沿的领域。
本研究在已筛选出产嗜热葡聚糖酶的嗜热脱氮芽孢杆菌并获得该酶基因序列的基础上,进一步利用分子动力学模拟的方法从原子水平上研究定点突变对蛋白质耐热性的影响,以期获得耐热性更高的突变体。但目前由于尚未有对该酶结构分子的研究,本研究中的部分氨基酸残基在该酶耐热性中的作用机制,可以为以后对该酶进行技术改造奠定理论基础。
1 材料与方法
1.1 研究对象
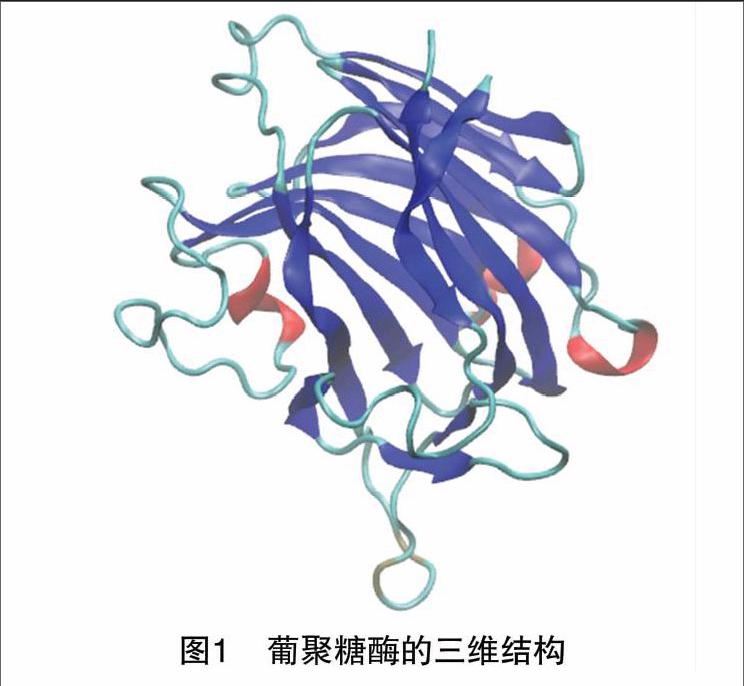
使用引自地衣芽孢杆菌(Bacillus licheniformis)SR01的葡聚糖酶基因序列(登录号:YP_006712752.1),在前期研究中,该基因表达的葡聚糖酶具有较高的热稳定性,其最适温度为55 ℃,并在90 ℃下2 h仍保持80%左右的生物活性[9];通过I-TASSER(http://zhanglab.ccmb.med.umich.edu/I-TASSER/)网站同源建模预测其三维结构,建模所得模型C端结构缺少22个氨基酸残基,但是由于它距离该酶的活性部位较远,故本研究将所得该结构模型直接作为试验模型,详见图1。该模型包含223个残基,1 808个原子,15个β折叠,3个α螺旋,其结构类似于三明治结构。同源结构的模板为1oq1.C,分辨率为1.70,序列相似性为6544%。
1.2 分子动力学模拟
通过同源序列分析,以及氨基酸疏水性(http://pic.mbu.iisc.ernet.in/index.html)、氨基酸深度和活性位点(http://mspc.bii.a-star.edu.sg/tankp/intro.html)预测分析,笔者得出R226、G224氨基酸残基位于该结构模型的内部,且2个氨基酸周围存在大量的疏水性氨基酸,因此笔者可以认为,R226、G224可能不利于该酶的热稳定性。为了验证分析结果,本试验使用Deepview软件对建模结构进行定点突变,得到R226A、G224A突变体模型;采用Groamcs软件[10]进行分子动力学模拟,力场选择OPLS-AA/L全原子力场[11],水分子选用SPCE模型[12],为了模拟真实环境,将蛋白质置于立方体盒子中,蛋白质距离盒子边缘1.0 nm,采用周期边界条件,盒子周围被相同的盒子包围,以消除边界效应,盒子内填充spc216.gro模型水分子,添加离子使得整个系统的电荷为0,处于中性的环境中。在能量优化后,使用约束动力学的方法,进行800 ps的约束动力学模拟,通过逐步升温的方法将体系的温度从50 K升到模拟需要的温度(324、344、364 K)。之后,采用弱耦合的方法保持體系中温度、压强(100 kPa)的稳定,温度和压强的耦合时间常数均为0.1 ps[13],范德华和库伦作用的截断半径均为1.4 nm[14],静电相互作用使用PME方法计算[15],模拟步长为2 fs,对3个体系进行20 ns的自由动力学模拟,每2 ps采集1次构象。
2 结果与分析
2.1 葡聚糖酶结构的总体特征
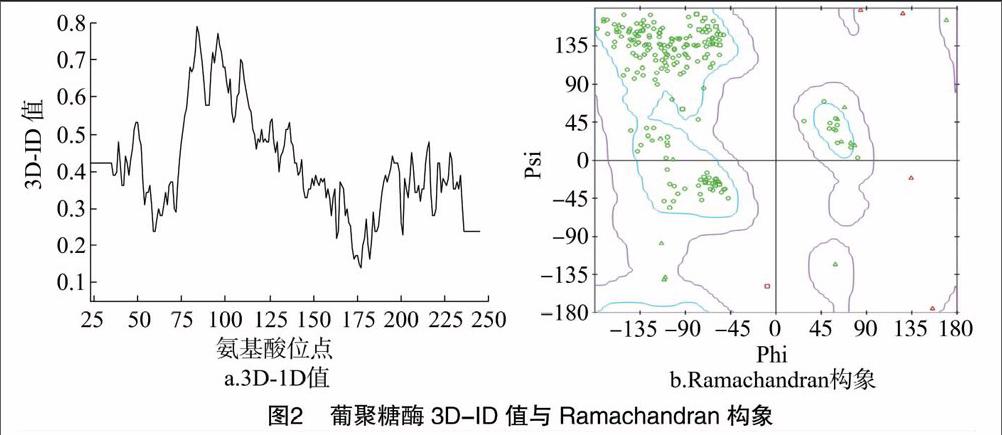
根据建模所得的模型,笔者分析了该模型的结构合理性,通过网络服务器(http://services.mbi.ucla.edu/SAVES/)进行分析。如图2所示,该模型的各个氨基酸残基的平均 3D-ID 值都在0以上,并且有96.86%氨基酸残基的3D-ID值≥0.2(蛋白质的总氨基酸至少有80%≥0.2),这说明其三维结构建模所用的模板序列兼容性较好。此外,葡聚糖酶主链二面角的99.4%Φ-Ψ角落在Ramachandran图的核心区和允许区,这也表明蛋白质主链结构具有合理性。
2.2 均方根偏差分析
采用计算蛋白质分子均方根偏差(root mean squared deviation,简称RMSD)的方法来分析模拟过程中3个体系偏离初始位置的大小[16]。在整个模拟过程中,初始结构的蛋白质骨架RMSD可以作为衡量蛋白质结构稳定的标准。在不同的温度下,野生蛋白与突变体R226A、G224A的RMSD变化如图3所示。可以看出,在324 K的温度下,3个体系均较快达到平衡,在10 ns后,突变体R226A的RMSD明显低于野生型蛋白,而G224A的RMSD整体略低于野生型蛋白。在 344 K 的温度下,起始在0~5 ns之间,3个体系的RMSD呈上升趋势,在10~20 ns之间呈平衡的趋势,这说明温度的增加对体系的影响较大。R226A突变体的RMSD变化较小,约为 0.16 nm,G224A与野生型蛋白的RMSD相差无几。在 364 K 的温度下,突变体体系在后10 ns内,其RMSD都低于野生蛋白。整体来看,突变体R226A在324、344 K温度下,其结构偏离较小,而在364 K温度下,相比初始结构,其偏离较大,约为0.2 nm;突变体G224A整体相比初始位置偏离较小,约为0.18 nm。这说明与野生型蛋白相比,突变体R226A、G224A的热稳定性得以提高,更有利于适应高温环境。endprint
2.3 蛋白质的均方根涨落(root mean square fluctuation,简称RMSF)分析
随着温度的升高,蛋白质的空间结构也会有相应的变化,
在较高的温度下,会使蛋白质失去一些力的作用,例如氢键,通过RMSF值主要是分析蛋白质构象的柔性[17]。由图4可以看出,温度对不同位置的残基影响不一样,其中在β折叠(73~82、86~96、99~109、184~199aa)内的氨基酸残基受温度影响波动较小,其RMSF在0.15 nm以下波动,在loop环(61~72、110~126、209~220aa)和α螺旋(148~152aa)内的氨基酸残基受温度影响较大,最大RMSF可达到0.4 nm;在324、344 K温度下,突变体G224A与野生型蛋白的RMSF较突变体R226A大,说明G224A与野生型的柔性高于R226A,在364K温度下,G224A整体柔性低于R226A和野生型,这与上文中蛋白质分子均方根偏差的研究结果一致。此外,在突变位点处的RMSF与野生型相差不大,这也说明本研究所选用的位点残基突变不会影响蛋白质的三级结构,且该突变点附近构象的异质性较小。
2.4 回旋半径分析
体系的回旋半径(Rg)是指体系中每个粒子离体系质心距离的几何平均值。其计算公式如下:Rg=1n∑ni=1r2i。式中:n表示体系中粒子数;ri表示体系内粒子离体系质量中心的距离。对于相同粒子数的体系,Rg反映了体系的紧密程度:Rg越大,说明体系越膨胀,结构越松散[18]。为了考察3个体系在不同温度下的形态变化,本研究分别计算了其回旋半径径向分布函数。由图5可知,在324 K下,突变体R226A的回旋半径分布在1.675~1.720 nm范围内,其构象集中在1.690 nm附近;野生型蛋白回旋半径分布在1.690~1.730 nm 范围内,其构象集中在1.710 nm附近;G224A的回旋半径分布在1.690~1.720 nm范围内,其构象集中在 1.705 nm 附近;在344 K下,野生型蛋白构象集中在 1.715 nm 附近,R226A构象集中在1.700 nm附近,G224A构象集中在1.710 nm附近;在364K温度下,野生型蛋白构象集中在 1.720 nm 附近,R226A构象集中在1.705 nm附近,而G224A构象集中在1.685 nm附近。整体来看,R226A、G224A的回旋半径都低于野生型蛋白,这说明突变体体系比野生型更加紧密。在不同温度下,随着温度的增加,R226A与野生型蛋白的Rg值呈现上升的趋势,这说明温度的增加破坏了部分相互作用,使得整个体系变得膨胀,而G224A在 364 K 下比 324 K 的Rg值小,这可能是由于温度的增加破坏了一部分互相作用,同时也形成了新的相互作用,使得整个体系变得紧密,更能抵抗高温环境。
2.5 疏水性分析
疏水相互作用是指非极性基团即疏水基团为了避开水相而聚集在一起的作用力。一般来说,疏水性残基被包埋在蛋白质结构的内部,对蛋白质结构稳定起到重要的作用[19]。本试验通过研究不同温度下的疏水溶剂可及表面的大小来衡量蛋白质解折叠的程度,进而判断蛋白质结构的稳定性[20],模拟试验结果如图6所示。可以看出,在324 K下,野生型蛋白质的疏水溶剂可及表面的分布范围为53~61 nm2,其构象集中在58 nm2附近;R226A的疏水溶剂可及表面的主要分布范围为52~62 nm2,其构象集中在57 nm2附近;G224A的疏水溶剂可及表面主要分布在53~61 nm2,构象集中在55~57 nm2 附近;在344、364 K下,野生型蛋白的疏水溶剂可及表面变化较小,主要分布在53~62 nm2;R226A体系疏水溶剂可及表面呈随温度增加而增加的趋势,主要分布在53~63 nm2,其构象分别集中于57、58 nm2附近;而G224A体系随着温度的增加,其疏水溶剂可及表面呈下降趋势,其构象分别主要集中在59、56 nm2 附近,这说明温度的增加使得该体系的結构变得更紧密,以抵抗高温环境,与以上研究中G224A回旋半径变小一致,进一步可以说明,疏水性大小对蛋白质的热稳性起着重要作用。整体来看,R226A、G224A在324、364 K 下,其疏水溶剂可及表面都低于野生型,这可能是由突变后的氨基酸造成的。Arg226、Gly224氨基酸残基是极性氨基酸,具有亲水性,并位于蛋白质结构的疏水内核,不利于蛋白质结构的稳定,而突变为Ala226、Ala224氨基酸残基后,由于它实际上并非极性氨基酸,具有疏水性,有利于蛋白质疏水内核更好地折叠,因此可以推测,突变体系的热稳定性要高于野生型。
2.6 其他参数分析
影响蛋白质结构稳定性的因素有很多,例如氢键、盐键和分子静电势能等[21],本研究分别考察了突变体与野生型蛋白的氢键数、盐键数和分子静电势能,进一步研究温度对突变体系、野生型体系的影响。由表1 可以看出,随着温度升高,突变体与野生型蛋白形成的盐桥数量增加,在相同的温度下,突变体的盐桥数略高于野生型。由于盐桥可以增强蛋白质分子的刚性,维持蛋白质高级结构的稳定性,因此认为,在高温环境下,嗜热酶可能会形成更多的盐桥来维持自身结构的稳定[22]。在不同的温度下,突变体和野生型分子间、蛋白质与水分子间的氢键数随温度上升有下降趋势,因此可认为,温度升高破坏了部分氢键,使得蛋白质分子结构间的作用力下降,造成结构松散,抵抗高温环境能力变差;在相同的温度下,野生型蛋白与突变体蛋白相比氢键数相差不大,而野生型蛋白质与水分子之间的氢键数高于突变体系,这也可能与野生型可及溶剂表面高于突变体系,可以与水分子形成更多的氢键有关。蛋白质分子静电势能是衡量蛋白质结构分子是否稳定的重要参数,其值越小,说明该结构越稳定。在相同温度下,突变体体系与野生型分子静电势能相差较大,突变体体系之间静电势能相差无几;而随着温度的升高,野生型与突变体体系的分子静电势能出现明显增加的趋势,这说明温度的升高使3个体系的蛋白质结构变得不稳定。整体来看,温度破坏了分子结构的各种作用力,并且重新形成了新的作用力,因此评价蛋白质结构是否稳定,需要综合各种参数作出正确的判断。endprint
3 结论与讨论
目前,嗜热蛋白以其自身独特的優越性被广泛关注,已成为研究的热门。有研究表明,增加蛋白质中的氢键[23]、盐键[24]和二硫键[25]有利于提高酶的稳定性。也有研究表明,增加精氨酸(Arg)、脯氨酸(Pro)、丙氨酸(Ala)的含量,降低谷氨酰胺(Gln)、天冬酰胺(Asn)的含量有助于提高酶的热稳定性,主要的原因是Ala利于螺旋结构的形成[26];Pro由于其结构熵相比其他氨基酸小而更易于发生折叠,折叠后的结构需要更高的能量才能使其去折叠,这样在不影响酶的高级结构的前提下,引入Pro有助于提高酶的热稳定性[27];Arg由于具有胍基和较大的侧链, 可提高与其他氨基酸残基之间的疏水作用和离子间的作用力,有助于提高蛋白质的热稳定性[28];而Asn和Gln由于在高温条件下易发生脱氨作用,造成酶部分失活,因此不利于提高酶的热稳定性[29]。
为了研究葡聚糖酶的热稳定性机制,本研究通过使用分子模拟的方法,从原子水平分析了突变体R226A、G224A和野生型蛋白的热动力学特性。相关试验数据显示,在相同温度下,突变体R226A、G224A的RMSD要低于野生型,在不同温度下,突变体与野生型蛋白的RMSD均升高,这表明突变体由于一级结构发生变化进而使得高级结构发生变化,使其与野生型相比,热稳定性提高;同时也可以得出,温度可以破坏蛋白质结构内部的作用力,造成结构的不稳定;通过对突变体与野生型蛋白的均方根涨落分析可以得出,氨基酸片段突变体较野生型的无规则卷曲和α螺旋部分波动大,而β折叠部分波动较小,且突变体R226A的柔性总体要低于野生型,α螺旋柔性较大可能与其所处的位置及氨基酸的组成、数量有关。在不同温度下,随着温度的升高,部分氨基酸残基的柔性有增加的趋势,然而也有部分氨基酸残基的柔性降低,这可能是与其他氨基酸残基形成新的作用力造成的。回旋半径表示的一个体系的膨胀程度,回旋半径越小,说明该体系结合越紧密,分子间的作用力越强,蛋白质分子的耐热性越好。在相同的温度下,可以得出,突变体R226A、G224A的回旋半径比野生型小,这说明突变体的热稳定性较野生型强,可能是与突变后的氨基酸有关;然而在不同的温度下,突变体与野生型的回旋半径变化略微增大,这可能与本研究所使用的温度不足以使得蛋白质发生去折叠有关。疏水作用在蛋白质结构与功能方面有着重要的作用,本研究通过分析突变体与野生型蛋白的疏水溶剂可及表面得出,突变体R226A的疏水溶剂可及表面积比野生型蛋白小,由此可以推测,突变体R226A主要是由于突变后的A226是疏水氨基酸,改变了蛋白质的疏水环境,导致蛋白质结构发生变化,使得内部疏水内核进一步折叠,增强了该突变体的热稳定性。本研究同时分析了不同温度下3个体系的其他参数,试验数据表明:随着温度的升高,突变体与野生型蛋白的分子势能明显升高,蛋白质间与蛋白质与水之间的氢键数下降,而盐桥数有上升的趋势,这与Thomas等研究盐键在100 ℃下比25 ℃下的作用力大幅度增强,并对于体系温度的增加具有一定弹性一致[22]。由此,笔者通过结合以上研究结果,认为R226A、G224A有助于提高葡聚糖酶的热稳定性。本研究首次从原子水平上探讨葡聚糖酶的耐热机制,对今后提高葡聚糖酶热稳定性提供了理论指导意义。
参考文献:
[1]Gohl B,Alden S,Elwinger K,et al. Influence of β-glucanase on feeding value of barley for poultry and moisture content of excreta[J]. British Poultry Science,1978,19(1):41-47.
[2]张 洁,蔡敬民,吴 克,等. β-葡聚糖酶的研究与应用前景[J]. 安徽农业科学,2003,31(5):895-896.
[3]郭小权,胡国良,刘 妹. β-葡聚糖的抗营养作用及β-葡聚糖酶在饲料中的应用[J]. 江西饲料,2001,2(2):11-13.
[4]唐 赟,李卫华,盛亚运. 计算机分子模拟——2013年诺贝尔化学奖简介[J]. 自然杂志,2013,35(6):408-415.
[5]詹冬玲,高 楠,韩葳葳,等. 分子对接和动力学模拟提高嗜热蛋白酶PhpI的活力[J]. 高等学校化学学报,2013,34(3):628-633.
[6]Matsushita Y,Murakawa T,Shimamura K,et al. Change in specific interactions between lactose repressor protein and DNA induced by ligand binding:molecular dynamics and molecular orbital calculations[J]. Molecular Simulation,2016,42(3):242-256.
[7]Blaszczyk M,Kurcinski M,Kouza M,et al. Modeling of protein-peptide interactions using the CABS-dock web server for binding site search and flexible docking[J]. Methods,2016,93:72-83.
[8]Cole D J,Tirado-Rives J,Jorgensen W L. Molecular dynamics and Monte Carlo simulations for protein-ligand binding and inhibitor design[J]. Biochimica et Biophysica Acta,2015,1850(5):966-971.endprint
[9]邓 春,易 弋,韦阳道,等. 嗜热地衣芽孢杆菌SR01葡聚糖酶基因的原核表達及酶学性质研究[J]. 粮食与饲料工业,2016,2(2):60-64.
[10]Berendsen H J C,van der Spoel D,van Drunen R,et al. GROMACS:a message-passing parallel molecular dynamics implementation[J]. Computer Physics Communications,1995,91(1/2/3):43-56.
[11]Kaminski G A,Friesner R A,Tirado-Rives J,et al. Evaluation and reparametrization of the OPLS-AA force field for proteins via comparison with accurate quantum chemical calculations on peptides[J]. The Journal of Physical Chemistry. B,2001,105(28):6474-6487.
[12]Zielkiewicz J. Structural properties of water:comparison of the SPC,SPCE,TIP4P,and TIP5P models of water[J]. The Journal of Chemical Physics,2005,123(10):104501.
[13]Berendsen H C,Postma J M,van Gunsteren W F,et al. Molecular dynamics with coupling to an external Bath[J]. The Journal of Chemical Physics,1984,81(8):3684-3690.
[14]Darden T,York D,Pedersen L,et al. Particle mesh Ewald:an N·log(N) method for Ewald sums in large systems[J]. The Journal of Chemical Physics,1993,98(12):10089-10092.
[15]Wennberg C L,Murtola T,Páll S,et al. Direct-Space corrections enable fast and accurate Lorentz-Berthelot combination rule Lennard-Jones lattice summation[J]. Journal of Chemical Theory and Computation,2015,11(12):5737-5746.
[16]Maiorov V N,Crippen G M. Significance of root-mean-square deviation in comparing three-dimensional structures of globular proteins[J]. Journal of Molecular Biology,1994,235(2):625-634.
[17]Bahar I,Atilgan A R,Erman B. Direct evaluation of thermal fluctuations in proteins using a single-parameter harmonic potential[J]. Folding & Design,1997,2(3):173-181.
[18]Lobanov M Y,Bogatyreva N S,Galzitskaya O V. Radius of gyration as an indicator of protein structure compactness[J]. Molecular Biology,2008,42(4):623-628.
[19]Pace C N,Fu H,Fryar K L,et al. Contribution of hydrophobic interactions to protein stability[J]. Journal of Molecular Biology,2011,408(3):514-528.
[20]Gilis D,Rooman M. Predicting protein stability changes upon mutation using database-derived potentials:solvent accessibility determines the importance of local versus non-local interactions along the sequence[J]. Journal of Molecular Biology,1997,272(2):276-290.
[21]Vogt G,Woell S,Argos P. Protein thermal stability,hydrogen bonds,and ion pairs[J]. Journal of Molecular Biology,1997,269(4):631-643.
[22]Thomas A S,Elcock A H. Molecular simulations suggest protein salt bridges are uniquely suited to life at high temperatures[J]. Journal of the American Chemical Society,2004,126(7):2208-2214.endprint
[23]Kumar S,Tsai C J,Nussinov R. Factors enhancing protein thermostability[J]. Protein Engineering,2000,13(3):179-191.
[24]Kumar S,Ma B,Tsai C J,et al. Electrostatic strengths of salt bridges in thermophilic and mesophilic glutamate dehydrogenase monomers[J]. Proteins-Structure Function and Bioinformatics,2000,38(4):368-383.
[25]Beeby M,OConnor B D,Ryttersgaard C,et al. The genomics of disulfide bonding and protein stabilization in thermophiles[J]. PLoS Biology,2005,3(9):e309.
[26]Argos P,Rossmann M G,Grau U M,et al. Thermal stability and protein structure[J]. Biochemistry,1979,18(25):5698-5703.
[27]Sriprapundh D,Vieille C,Zeikus J G. Molecular determinants of xylose isomerase thermal stability and activity:analysis of thermozymes by site-directed mutagenesis[J]. Protein Engineering,2000,13(4):259-265.
[28]盧柏松,王国力,黄培堂. 嗜热与嗜常温微生物的蛋白质氨基酸组成比较[J]. 微生物学报,1998,38(1):20-25.
[29]Ichikawa J K,Clarke S. A highly active protein repair enzyme from an extreme thermophile:the L-isoaspartyl methyltransferase from Thermotoga maritima[J]. Archives of Biochemistry and Biophysics,1998,358(2):222-231.endprint
