桃蛀螟CpunPBP2基因的全长序列克隆、分子结构及系统发育分析
2017-11-30朱秀云徐鹿王志强陈耕李进步张亚楠
朱秀云+徐鹿+王志强+陈耕+李进步+张亚楠

摘要:气味结合蛋白(odorant binding protein,简称OBP)能结合进入到触角内的脂溶性气味物质进行并运送到感受神经元受体部位,被认为是昆虫嗅觉的第1个生化步骤,其中性信息素结合蛋白(pheromone binding protein,简称PBP)属于OBP家族。利用cDNA末端快速扩增技术(rapid-amplification of cDNA ends,简称RACE)在已有报道桃蛀螟CpunPBP2片段的基础上,设计特异性扩增引物获得该基因的全长cDNA序列。序列分析表明,CpunPBP2基因具有昆虫OBP典型特征,即6个保守的半胱氨酸位点和信号肽序列。同源比对结果发现,CpunPBP2的氨基酸序列与其他鳞翅目昆虫PBP2均具有较高的相似性。进一步进化分析结果表明,CpunPBP2能聚在鳞翅目昆虫PBP/GOBP超家族内。本研究结果为进一步分析桃蛀螟PBP2的生理功能奠定了分子基础。
关键词:桃蛀螟;基因;气味结合蛋白;性信息素结合蛋白;cDNA末端快速扩增技术;全长克隆;系统发育分析
中图分类号: S433.4 文献标志码: A 文章编号:1002-1302(2017)20-0050-04
昆虫作为历史上最长的动物类群之一,长期进化使它们形成了以触角为主的发达的嗅觉感受系统,借此识别环境中大量的化学信息作出准确判断,并产生一系列生理和行为反应,如寻找栖息、产卵场所等[1]。自1981年Vogt等用标记性信息素方法发现了第1个昆虫气味结合蛋白(OBP)——多音大蚕蛾雄蛾(Antheraea polyphemus)触角中的性信息素结合蛋白(pheromone binding protein,简称PBP)[2]以来,随着基因组和转录组测序技术快速发展及成本大幅度降低,已在不同昆虫中发现有超过400个OBP,它们分属于不同的目和科[3]。
根据保守半胱氨酸的数量以及氨基酸序列,昆虫OBP可分为4类:(1)经典OBP(classic OBP),特征为6个保守的半胱氨酸结构,鳞翅目昆虫的性信息素结合蛋白、普通气味结合蛋白(general odorant binding protein,简称GOBP)以及触角结合蛋白(antennal binding protein,简称ABP)均属于此类;(2)多C OBP(plus-C OBP),特征为超过6个以上的保守半胱氨酸结构,还常会在保守位置具有一个脯氨酸;(3)少C OBP(minus-C OBP),特征为少于6个半胱氨酸结构,仅在4个保守位置处有半胱氨酸;(4)典型OBP(atypical OBP),特征为拥有一段较长的C端序列[4-6]。在鳞翅目昆蟲中,OBP的保守性较高,分类清晰,可以明显地分为3类:性信息素结合蛋白(PBP)、普通气味结合蛋白、触角特异性蛋白(antennal specific protein,简称ASP)或触角结合蛋白。鳞翅目雄蛾的触角高表达或专一表达PBP,且PBP可以结合由雌虫释放的性信息素[7-8]。但雌雄触角感器均有表达的PBP并不一定是对性信息素特异结合的,也能结合其他非性信息素物质[9-10]。
桃蛀螟[Conogethes punctiferalis(Guenée)]别称桃斑螟、桃蛀心虫、桃蛀野螟,隶属于鳞翅目(Lepidoptera)螟蛾科(Pyralidae)。桃蛀螟食性杂、分布广,其幼虫可危害板栗、桃、李、杏、玉米、蓖麻、向日葵等40多种果树、油料和粮食作物。近年来,陆续有报道发现,桃蛀螟在我国的长江流域、中部和北部地区对梨、板栗、向日葵、高粱、玉米的产量造成了重大危害[11-12]。桃蛀螟在幼虫期取食的部位以致于传统的化学农药很难对它们产生直接有效的作用。桃蛀螟性引诱剂的开发已成功应用于种群的预测预报,但直接用于害虫防治,同该技术应用于其他重要蛾类害虫的情况相似,无论是用交配干扰法还是大量诱捕法,都存在成本偏高、效果不理想等问题。况且现在使用的化学农药不仅仅会使害虫对杀虫剂产生抗药性,而且还会大大加剧环境污染和对非靶标生物的杀伤作用[13]。因此,生产上亟须更为高效的行为调控技术来防治这类害虫。桃蛀螟性信息素所含有的2种主要醛类组分(Z10-16:Ald、E10-16:Ald)在化学结构上也与其他螟蛾科害虫相同或相似。另外,桃蛀螟所危害的果树和农作物同样也是鳞翅目其他科害虫的取食对象,因此以桃蛀螟为研究材料的研究成果,也将对其他昆虫的相关研究具有很好的借鉴意义和应用价值。
1 材料与方法
1.1 供试昆虫
于2014年7—9月利用灯诱的方式,在淮北师范大学相山校区校园内采集试虫。随后将收取的成虫(约60头)触角按照雌雄1 ∶ 1的比例放于-80 ℃冰箱备用。
1.2 总RNA提取及RACE-cDNA的合成
总RNA提取试剂Trizol购自Invitrogen公司,并按操作说明书进行提取。5′/3′ -RACE-ready cDNA模板合成使用SMARTTM RACE cDNA Amplication Kit(634925,TaKaRa & Clontech),并存放于-20 ℃冰箱中。
1.3 PCR引物设计、反应体系及程序
1.3.1 PCR引物设计 按照SMARTerTM PCR cDNA Synthesis Kit试剂盒使用说明中关于引物设计的要求,利用Premier 5.0软件设计RACE引物(gene specific primer,简称GSP)(表1)。
1.3.2 PCR反应体系及程序 利用特异引物(表1)和RACE分别扩增桃蛀螟PBP2基因的5′、3′序列,所采用的RACE-PCR条件按照操作试剂盒(SMART Kit,TaKaRa)说明书进行。
1.4 PCR产物纯化、克隆、测序和序列分析endprint
PCR产物纯化:采用AxyPrep DNA凝胶回收试剂盒(Axygen)对PCR产物进行回收纯化,操作按照试剂盒说明书进行。
连接反应:经胶回收后的PCR扩增产物连接于pEASY-T3载体上(全式金生物,北京),操作按照试剂盒说明书进行。
转化反应:上步连接成功的载体和目的基因转化至 Trans-T1感受态细胞(全式金生物,北京)中,操作按照试剂盒说明书进行。
1.5 序列鉴定及系统发育分析
通过GeneDoc软件进行序列比对分析,并利用NCBI-Blast(http://blast.ncbi.nlm.nih.gov/)及与NCBI-GenBank已经登录的基因序列进行同源搜索比对,信号肽预测由网络服务器SignalP V4.1(http://www.cbs.dtu.dk/services/SignalP/)完成,蛋白分子量和等电点由http://www.expasy.org/网站预测,双螺旋结构和3D同源建模由http://espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgi、http://swissmodel.expasy.org预测分析。
利用ClustalW 2.0及Mega 6.0采用邻接法(neighbor-joining method)构建系统发育树(抽样分析为1 000次),对桃蛀螟等昆虫的PBP基因进行系统分析,选用的其他昆虫序列参考[14]。
2 结果与分析
2.1 全长克隆与分析
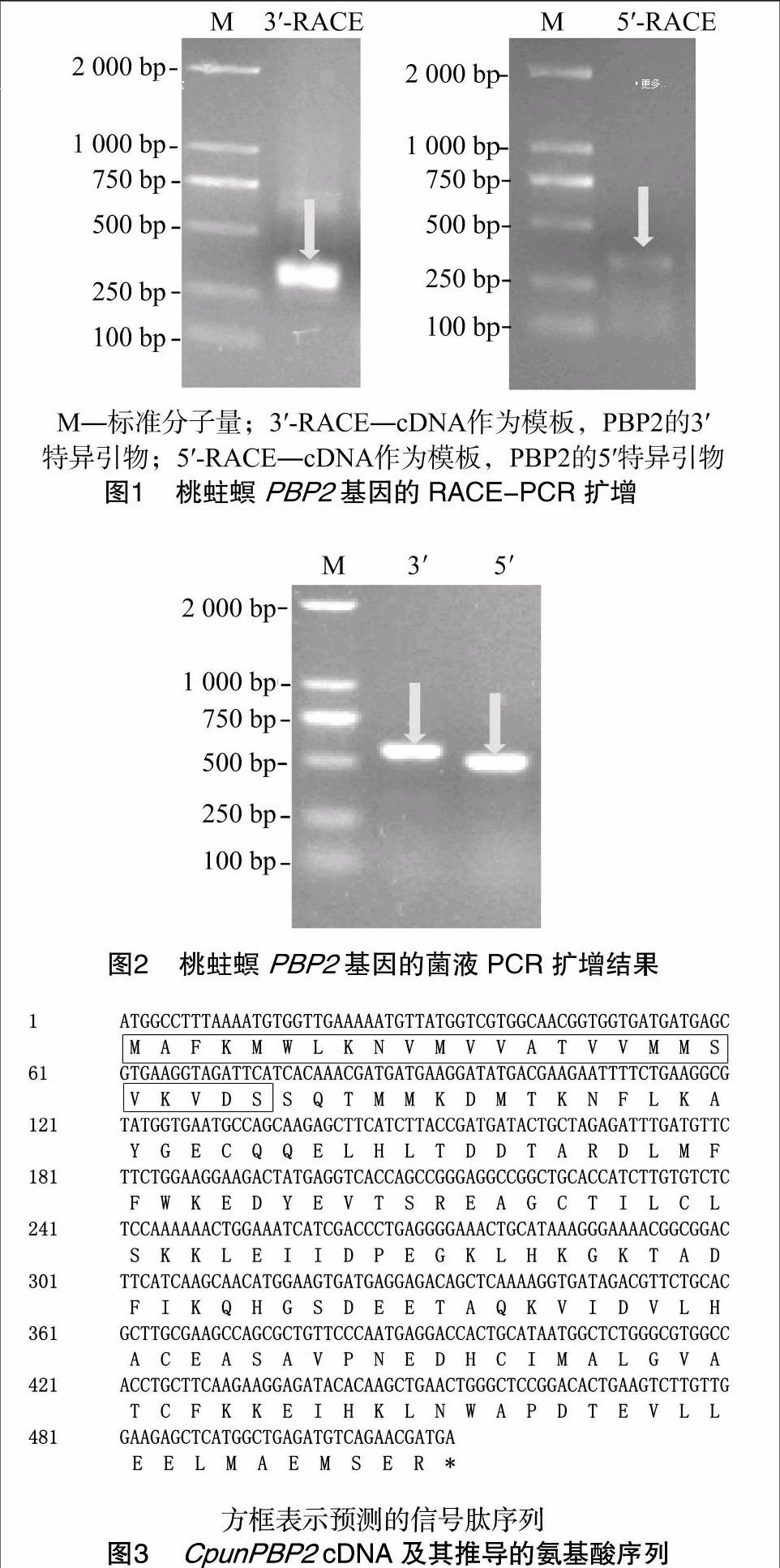
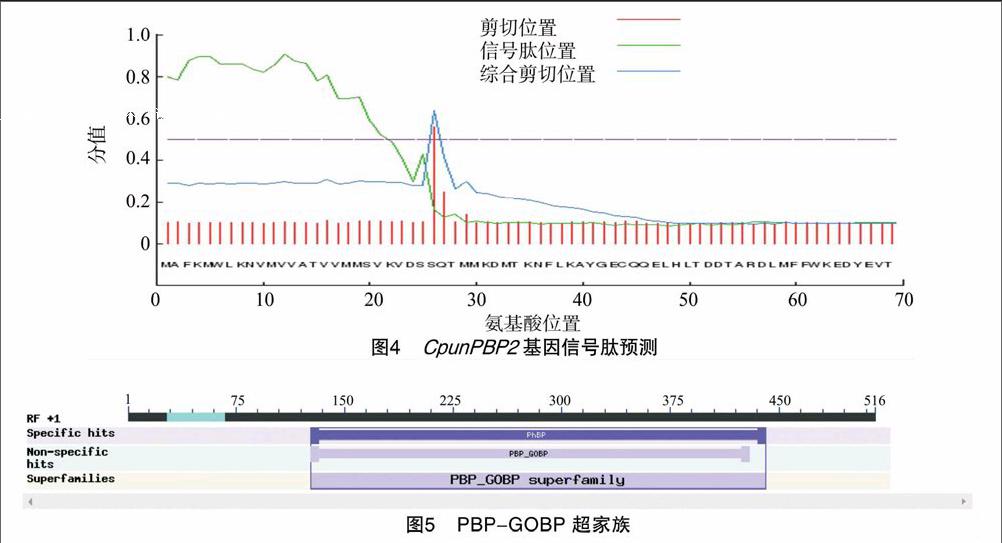
在原有CpunPBP2片段的基础上[15],设计特异引物并利用RACE-PCR扩增出300 bp左右的条带(图1),与预测大小相符。纯化后的PCR产物克隆到pEASY-T3 easy載体中,随后经菌液PCR扩增出500 bp左右条带(图2),选取5个阳性克隆进行测序。通过分析5′、3′RACE的测序结果,得到开放阅读框为513 bp,编码170个氨基酸的CpunPBP2序列(图3)。经软件预测,CpunPBP2的N-端最初25个氨基酸为信号肽(图4),成熟蛋白的分子量为16.51 ku,等电点为4.93。
2.2 序列同源分析
利用NCBI-BLAST网络服务器(http://blast.ncbi.nlm.nih.gov/)及NCBI-GenBank序列库中已登录序列进行家族预测分析,结果(图5)表明,CpunPBP2基因属于昆虫 PBP-GOBP超家族。
将CpunPBP2 cDNA所推导的氨基酸序列,与已知其他鳞翅目昆虫PBP2进行同源比对分析(图6),结果表明,CpunPBP2的氨基酸序列与其他昆虫PBP2均(MsexPBP2、SinfPBP2、SexigPBP2、HvirPBP2)具有较高的相似性,分别为62%、69%、67%、62%。从图6还可看出,CpunPBP2具有典型的昆虫OBP结构:6个保守的半光氨酸位点,且第2个和第3个半胱氨酸间隔3个氨基酸、 第5个和第 6 个半胱氨酸间隔8个氨基酸[16]。利用CpunPBP2同已有的家蚕BmorPBP晶体结构的同源预测发现,CpunPBP2同BmorPBP类似,即含有6个α-螺旋和1个额外α-螺旋(图7),这些螺旋机构可能会参与到桃蛀螟性信息素的识别过程中。
2.3 系统发育分析
选取已报道的鳞翅目蛾类昆虫:家蚕(Bombyx mori)、大螟(Sesamia inferens)、海灰翅夜蛾(Spodoptera littoralis)和烟草天蛾(Manduca sexta)[17-18]与桃蛀螟的PBP2进行进化树构建,结果表明,CpunPBP2能聚在鳞翅目昆虫PBP/GOBP超家族内(图8)。
3 结论与讨论
本研究基于桃蛀螟CpunPBP2的片段信息,利用RACE技术成功克隆该基因的全长cDNA序列。通过序列分析比对发现,CpunPBP2具有OBP基因的典型序列特征,即分子量约15~17 ku、N末端具1条20个氨基酸左右的信号肽、序列中具有6个保守的半胱氨酸位点[19],并且与其他昆虫PBP表现出较高的相似性等。CpunPBP2基因全长序列的成功获得,为进一步研究其生理功能以及筛选高效的桃蛀螟行为控制剂提供了基础[20]。
另外,张亚楠对大螟所有鉴定到OBP的组织表达分析发现,大多数OBP在触角中高表达,并且半数左右的OBP在非触角组织中(胸和腹部)也检测到微弱的条带,这其中就包括PBP和GOBP,这暗示昆虫PBP和GOBP可能在大螟的嗅觉行为中起到重要的作用[21]。值得一提的是,该研究在大螟的幼虫中也检测到性信息素结合蛋白PBP1、PBP3的微弱表达信号,并且该结果与之前报道的海灰翅夜蛾(S. littoralis)幼虫类似,Poivet等的解释是海灰翅夜蛾幼虫中的PBP能感受吸附在或是雌蛾产卵时残留在绿叶上的性信息素,所以可利用这种行为帮助幼虫寻找食物[22]。这些研究表明,鳞翅目昆虫PBP功能可能不仅仅只在成虫感受性信息素途径中起到作用,可能在幼虫取食或其他的生理活动中担当重要的角色。
结果表明,蛾类昆虫PBP具有以下典型特征:(1)同科雄蛾触角中具有多个PBP基因和雌蛾腺体中具有多种性信息素组分,且大部分物种性信息素的数量多于PBP的数量;(2)同一PBP在雌雄触角间差异表达,不同PBP在雌虫或雄虫触角中差异表达;(3)PBP在触角感器分布具有多样性,而对CpunPBP2同其他昆虫PBP的进化分析结果表明,该基因属于昆虫PBP超家族,因此CpunPBP2很有可能也具有以上特征。
参考文献:
[1]Vogt R G. Molecular basis of pheromone detection in insects. In comprehensive insect physiology,biochemistry,pharmacology and molecular biology[M]. London:Elsevier,2005,753-804.endprint
[2]Vogt R G,Riddiford L M. Pheromone binding and inactivation by moth antennae[J]. Nature,1981,293(5828):161-163.
[3]Pelosi P,Maida R. Odorant-binding proteins in insects[J]. Comparative Biochemistry and Physiology Part B(Biochemistry and Molecular Biology),1995,111(3):503-514.
[4]Hekmat-Scafe D S,Scafe C R,McKinney A J,et al. Genome-wide analysis of the odorant-binding protein gene family in Drosophila melanogaster[J]. Genome Research,2002,12(9):1357-1369.
[5]Lagarde A,Spinelli S,Qiao H,et al. Crystal structure of a novel type of odorant-binding protein from Anopheles gambiae,belonging to the C-plus class[J]. Biochemical Journal,2011,437(3):423-430.
[6]Spinelli S,Lagarde A,Iovinella I,et al. Crystal structure of Apis mellifera OBP14,a C-minus odorant-binding protein,and its complexes with odorant molecules[J]. Insect Biochemistry and Molecular Biology,2012,42(1):41-50.
[7]Sun M,Liu Y,Wang G. Expression patterns and binding properties of three pheromone binding proteins in the diamondback moth,Plutella xyllotella[J]. Journal of Insect Physiology,2013,59(1):46-55.
[8]Liu N Y,Liu C C,Dong S L. Functional differentiation of pheromone-binding proteins in the common cutworm Spodoptera litura[J]. Comparative Biochemistry and Physiology Part A(Molecular and Integrative Physiology),2013,165(2):254-262.
[9]Liu N Y,He P,Dong S L. Binding properties of pheromone-binding protein 1 from the common cutworm Spodoptera litura[J]. Comparative Biochemistry and Physiology B(Biochemistry and Molecular Biology),2012,161(4):295-302.
[10]Picimbon J F,Gadenne C. Evolution of noctuid pheromone binding proteins:identification of PBP in the black cutworm moth,Agrotis ipsilon[J]. Insect Biochemistry and Molecular Biology,2002,32(8):839-846.
[11]鹿金秋,王振營,何康来,等. 桃蛀螟研究的历史、现状与展望[J]. 植物保护,2010,36(2):31-38.
[12]徐丽荣. 桃蛀螟人工饲养和滞育诱导特性及抗寒性研究[D].北京:中国农业科学院,2011.
[13]冯成玉. 我国稻田大螟发生规律与防治技术研究进展[J]. 农业工程,2012,2(10):62-65,35.
[14]Zhang Y N,Jin J Y,Jin R,et al. Differential expression patterns in chemosensory and non-chemosensory tissues of putative chemosensory genes identified by transcriptome analysis of insect pest the purple stem borer Sesamia inferens(Walker)[J]. PLoS One,2013,8(7):e69715.
[15]葛 星. 桃蛀螟嗅觉相关基因的筛选与表达谱分析[D]. 保定:河北农业大学,2013.
[16]Pelosi P,Zhou J J,Ban L P,et al. Soluble proteins in insect chemical communication[J]. Cellular and Molecular Life Sciences,2006,63(14):1658-1676.
[17]Forstner M,Gohl T,Breer H,et al. Candidate pheromone binding proteins of the silkmoth Bombyx mori[J]. Invertebrate Neuroscience,2006,6(4):177-187.
[18]Legeai F,Malpel S,Montagné N,et al. An expressed sequence tag collection from the male antennae of the Noctuid moth Spodoptera littoralis:a resource for olfactory and pheromone detection research[J]. BMC Genomics,2011,12(1):86.
[19]Vogt R G,Prestwich G D,Lerner M R,et al. Odorant-binding-protein subfamilies associate with distinct classes of olfactory receptor neurons in Insects[J]. Developmental Neurobiology,1991,22(1):74-84.
[20]金俊彦. 大螟气味结合蛋白基因的克隆和功能分析[D]. 南京:南京农业大学,2013.
[21]张亚楠. 大螟性信息素合成和感受相关基因的克隆和功能分析[D]. 南京:南京农业大学,2014.
[22]Poivet E,Rharrabe K,Monsempes C,et al. The use of the sex pheromone as an evolutionary solution to food source selection in caterpillars[J]. Nature Communications,2012,3(3):1047.endprint
