芒果ANS基因的鉴定及与其他植物的比较分析
2017-11-30盖江涛黄建峰党志国朱敏陈华蕊王鹏陈业渊
盖江涛+黄建峰+党志国+朱敏+陈华蕊+王鹏+陈业渊

摘要:花青素合成酶(anthocyanidin synthase,ANS,EC:1.14.11.19)是花青素合成中的一个关键酶。为了鉴定和分析芒果ANS基因,以芒果ANS基因作为研究目标,以拟南芥的ANS基因为参考,通过BLAST方法获得了番木瓜、金钱橘、甜橙、苹果、小果野蕉、桃子、葡萄、龙眼、甜樱桃、枣、石榴、西洋梨、中华猕猴桃、荔枝、无油樟的ANS基因序列,对其进行了系统进化分析、理化性质分析、结构分析等。系统进化分析结果显示,双子叶植物与单子叶植物的ANS基因进化树没有单独形成分支,推测在进化过程中ANS基因的分化在双子叶植物和单子叶植物形成之后;理化性质分析结果显示,酸性蛋白占97.2%、稳定性蛋白占13.9%,有导肽的蛋白占8.3%,所有蛋白均为无信号肽的亲水性蛋白;蛋白二级结构分析结果显示,α-螺旋、无规则卷曲是主要结构元件。说明芒果ANS基因具有稳定的花青素合成酶结构,并与本试验中的绝大多数植物ANS基因具有相似的性质。这为进一步研究芒果ANS基因在花青素合成途径中的作用提供了理论支持。
关键词:芒果;花青素;花青素合成酶;系统进化;同源性;蛋白二级结构
中图分类号: S667.701 文献标志码: A 文章编号:1002-1302(2017)20-0043-07
芒果(Mangifera indica Linnaeus)俗称芒果,是漆树科(Anacardiaceae)芒果属(Mangifera Linnaeus)的著名热带果树,因其果肉细腻、风味独特、营养价值丰富,且具有理气、健脾、益胃、降低胆固醇、防治心血管疾病、预防乳腺癌等多种功效,深受人们欢迎[1]。芒果果实色泽作为人们挑选芒果的一个重要参考指标,它的形成是一个非常复杂的代谢过程,主要与类胡萝卜素和花青素代谢途径有关。花青素(anthocyanin)又称花色素,是一类决定颜色的重要的天然水溶性色素,属于黄酮类化合物,广泛分布于多种植物组织中,是形成植物花、果实等组织颜色的主要色素[2]。花青素的含量在决定植物的花色、叶色、果色和其他经济器官的色泽及其营养品质方面起着重要作用,还可以起到吸引昆虫和植食性动物以达到传播花粉和种子的目的[3-4]。
花青素合成酶(无色花色素双加氧酶,anthocyanidin synthase,ANS/leucoanthocyanidin dioxygenase,LDOX,EC:114.11.19)是花青素合成途径末端的一个关键酶,通过Fe2+和 2-酮戊二酸离子将无色花青素氧化生成花青素,属于双加氧酶家族[5]。因此,可通过上调或抑制ANS基因的表达来改变花青素的含量或成分,从而引起植物花色的变化。ANS (LDOX)最初是从玉米的A2突变体中克隆得到[6]。目前,ANS基因已在拟南芥[7]、荔枝[8]、芒果[9]、龙眼[10]、葡萄[11]、苹果[12]、越橘[13]、茶树[14]、棕色棉[15]等多种植物中克隆。
ANS基因的表达水平与花青素累积之间的关系也在多种植物中被研究。游向荣等研究结果表明,龙眼芽轴颜色和ANS基因的表达差异有关[10]。龙眼正常成花的顶芽芽轴萌动时,芽轴颜色变红,ANS基因正常表达;而发生成花逆转时,芽轴颜色保持浅黄色,ANS基因下调表达。Honda等在苹果中发现,5个花青素合成相关基因在果皮着色过程中协同表达,并且其表达水平和花青素含量呈正相关[16]。Salvatierra等研究表明,在草莓果实成熟过程中,花青素合成相关基因的转录水平明显增加,并且其在红色果实中的表达水平明显高于白色果實[17]。Li等研究发现,ANS基因表达活性上调,使得桑树的果实变为红色[18]。李晓艳等研究发现,越橘从开花到果实成熟的整个发育过程中都能检测到ANS基因的表达,在成熟果的果皮中,ANS基因的表达量最高;而花色素苷的含量随着果实的成熟而增高,在越橘成熟果的果皮中,花色素苷的含量最高,与ANS基因表达量的变化一致[13]。研究说明,ANS基因在越橘果实花色素苷形成过程中可能起到调控作用,并具有一定的组织特异性。
本试验以芒果ANS基因作为研究目标,以已知ANS基因功能的双子叶植物纲(Dicotyledoneae)十字花科(Brassicaceae)拟南芥[Arabidopsis thaliana (Linnaeus) Heynhold]的ANS基因为参考,依托全基因组数据库,通过BLAST的方法获得了双子叶植物(Dicotyledons)番木瓜科(Caricaceae)番木瓜(Carica papaya Linnaeus),芸香科(Rutaceae)金钱橘(Citrus clementina Hortulanorum ex Tanaka)、甜橙[Citrus sinensis = C. ×sinensis (Linnaeus) Osbeck],蔷薇科(Rosaceae)苹果(Malus domestica Borkhausen=Malus pumila Miller)、桃子[Prunus persica (Linnaeus) Batsch= Amygdalus persica Linnaeus]、甜樱桃[Prunus avium (Linnaeus) Linnaeus = Cerasus avium (Linnaeus) Moench]、西洋梨(Pyrus communis Linnaeus),葡萄科(Vitaceae)葡萄(Vitis vinifera Linnaeus),鼠李科(Rhamnaceae)枣(Ziziphus jujuba Miller),石榴科(Punicaceae)石榴(Punica granatum Linnaeus),猕猴桃科(Actinidiaceae)中华猕猴桃(Actinidia chinensis Planchon),漆树科芒果,无患子科(Sapindaceae)龙眼(Dimocarpus longan Loureiro)、荔枝(Litchi chinensis Sonnerat),无油樟科(Amborellaceae)无油樟(Amborella trichopoda Baillon),单子叶植物(Monocotyledons)芭蕉科(Musaceae)小果野蕉(Musa acuminata Colla)的ANS基因序列,通过系统进化分析、理化性质分析、结构分析等分析这17种植物ANS基因序列,比较果树中ANS基因的特性,为分析ANS基因在芒果中的表达特性及其在花青素合成途径中的作用机制奠定基础,为推动芒果ANS基因功能研究提供理论支持。endprint
1 材料与方法
1.1 同源性搜索和数据提取
从NCBI (http://www.ncbi.nlm.nih.gov/)和phytozome V9.1[19](http://www.phytozome.net)中搜索得到拟南芥含有花青素合成酶结构域(pfam:cl21496、pfam:cl21672)的序列;从赵志常等文章中获得芒果ANS蛋白序列[9]。将得到的序列合并,删除重复序列。以得到的无重复序列集作为探针,通过BLASTP进行同源性搜索,为了防止错误丢失与探针相似度低但含有花青素合成酶结构域的序列,搜索的E值设为10,对应的检索阈值为Pfam数据库提供的“cut-off ”值。从Kiwifruit Genome Database[20] (http://bioinfo.bti.cornell.edu/cgi-bin/kiwi/download.cgi)中搜索得到中华猕猴桃ANS基因序列,从赵志常等文章中获得荔枝ANS蛋白序列[8]。合并检索得到的所有序列,删除冗余序列和结构域较短的序列,得到芒果、拟南芥、番木瓜、金钱橘、甜橙、苹果、桃子、甜樱桃、西洋梨、葡萄、枣、石榴、中华猕猴桃、龙眼、荔枝、小果野蕉、无油樟的ANS蛋白序列,共36条序列,均含有完整的花青素合成酶结构域。
1.2 多序列比对、系统发育分析及物种进化分析
通过MEGA 6.0[21]软件中的MUSCLE[22]工具进行蛋白质的多序列比对,参数设置为默认值。将比对结果导入Gblocks[23-24]在线服务器中,选择保守区域进行后续研究,为了获得更多保守区域位点,选中3个较为宽松的选项(option for a less stringent selection)。调整后的序列使用MrBayes 322[25]进行基于贝叶斯法的系统发育分析,共进行2次独立运算,每次运行设置2 000 000代,且每次运算使用4条蒙特卡洛-马尔科夫链(Markov chain Monte Carlo,MCMC)进行随机取样,冷链温度设置为0.1,在取样中每1 000代保存1棵树,共保存2 000棵树。前20的树作为burn-in舍弃,其余80的树用来构建贝叶斯一致树和后验概率。生成的贝叶斯一致树保存为nexus格式,并在Figtree v1.4.0软件中进行编辑。物种系统进化分析通过在线工具(http://www.ncbi.nlm.nih.gov/Taxonomy/CommonTree/wwwcmt.cgi)完成。
1.3 蛋白质生物信息学分析
利用CD search (http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)[26]进行保守结构域分析,采用ProtParam tool (http://web.expasy.org/protparam/)[27]在线工具预测分析蛋白质的理化性质,应用TMpred程序(http://www.ch.embnet.org/software/TMpred-form.html)[28]在线分析来预测蛋白质跨膜区和跨膜方向。用TargetP 1.1 Server (http://www.cbs.dtu.dk/services/TargetP/)[29]在線预测氨基酸序列导肽,应用signalP 4.1 server (http://www.cbs.dtu.dk/services/SignalP/)[30]中完成蛋白质信号肽的预测。亚细胞定位应用Cell-PLoc 2.0 package软件(http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)[31]进行在线分析。二级结构的预测和分析用SOPMA (http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)[32]完成。利用NetPhos 2.0 Server (http://www.cbs.dtu.dk/services/NetPhos/)[33]分析编码蛋白的磷酸化位点。
1.4 拟南芥ANS基因生育期芯片数据表达模式的分析
从Plant Expression Database (http://www.plexdb.org/)[34]的数据库中下载拟南芥的RMA文件,提取拟南芥的5条ANS基因AT4G22880.1、AT4G16330.2、AT5G05600.1、AT2G38240.1、AT3G11180.2分别在各生育期组织[下胚轴(7 d)、子叶(7 d)、幼叶(10 d)、成熟叶(17 d)、老叶(35 d)、叶柄(17 d)、种子(8周)、根(7 d)、根(15 d)、根(21 d)、芽(7 d)、茎尖(14 d)、花萼(>21 d)、花瓣(>21 d)、雄蕊(>21 d)、雌蕊(>21 d)、花粉(6周)、花梗(>21 d)]的表达情况,结果用gplots软件包中的heatmap.2软件绘制热图。
2 结果与分析
2.1 芒果与其他16种植物ANS蛋白的保守结构域分析
利用CD search对36条ANS氨基酸序列进行保守结构域分析,结果显示所有的ANS蛋白序列包含1个DIOX-N[35](non-haem dioxygenase in morphine synthesis N-terminal,吗啡合成N-端的非血红素加氧酶)亚家族结构域和1个2OG-FeII_Oxy[36](2-oxoglutarate Fe(II) oxygenase,2-酮戊二酸-Fe2+氧化酶)亚家族结构域,具有花色素合成酶家族特征多肽序列,ANS酶通过Fe2+和(2-oxoglutarate)离子将无色花青素氧化生成花青素,属于双加氧酶家族。芒果ANS蛋白序列的结构域如图1,与其他16种植物ANS蛋白的保守结构域一致。endprint
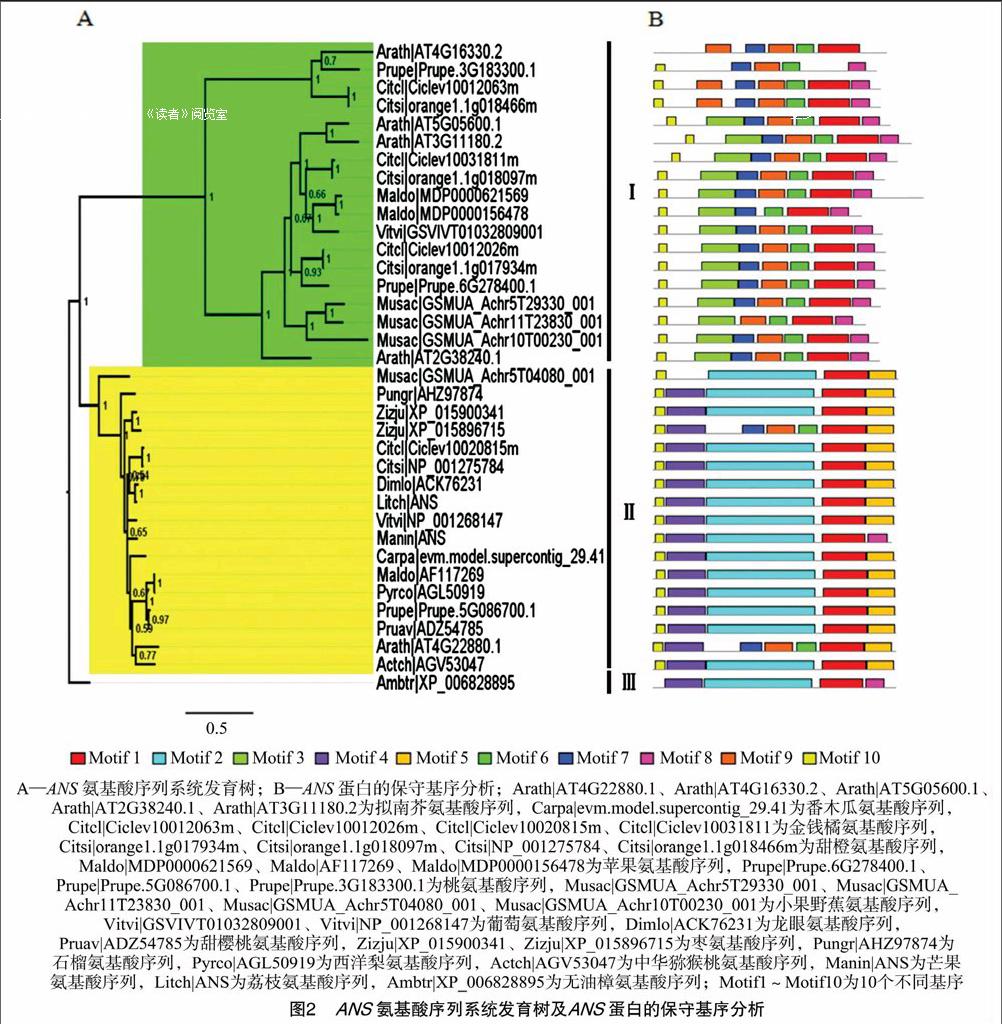
2.2 芒果与其他16种植物ANS蛋白序列的系统进化分析及ANS蛋白质氨基酸基序分析
本试验得到的36条氨基酸序列中,包含拟南芥5条、番木瓜1条、金钱橘4条、甜橙4条、苹果3条、小果野蕉4条、桃子3条、葡萄2条、龙眼1条、甜樱桃1条、枣2条、石榴1条、西洋梨1条、中华猕猴桃1条、芒果1条、荔枝1条、无油樟1条。对36条氨基酸序列进行系统发育分析,选自成一目、一科、一属的孑遗植物无油樟为外类群,结果(图2-A)显示,进化树分为3支,其中被选为外类群的无油樟科无油樟的ANS基因为单独一支,其余16种植物的ANS基因分为2支,双子叶植物与单子叶植物的ANS基因进化树没有单独形成分支,而是互相掺杂在一起,推测在进化过程中ANS基因的分化在双子叶植物和单子叶植物形成之后。
通过MEME程序在ANS蛋白中识别出10个潜在的保守基序(图2-B),不同的基序用不同颜色的方框表示。根据这10个基序的分布,可以将36条ANS蛋白分为与进化结果相似的3个亚家族。由图2-B可以看出,除Prupe|Prupe.3G183300.1外,都有Motif1基序;除Arath|AT4g16330.2与Ambtr|XP_006828895外,都有Motif10。在3个亚家族中,基序的排列顺序都高度保守。第Ⅰ亚家族中基序的分布情况:(1)都有Motif6基序;(2)除Musac|GSMUA_Achr11T23830_001外,都有Motif7;(3)除Maldo|MDP0000156478外,都有Motif9;(4)除Arath|AT4G16330.2外,都有Motif8;(5)除Arath|AT4G16330.2、Prupe|Prupe.3G183300.1、Citcl|Ciclev10012063m、Citsi|orange1.1g018466m外,都有Motif3。第Ⅱ亚家族中分布情况:(1)除Musac|GSMUA_Achr5T04080_001外,都有Motif4;(2)除Zizju|XP_015896715、Arath|AT4G22880.1外,都有Motif2;(3)除Manin|ANS外,都有Motif5。在第Ⅲ亚家族中,Ambtr|XP_006828895基序的特征:(1)有绝大多数氨基酸都有的Motif1基序,却没有绝大多数氨基酸都有的Motif10;(2)有第Ⅰ亚家族的保守基序Motif8,有第Ⅱ亚家族的保守基序Motif2、Motif4。
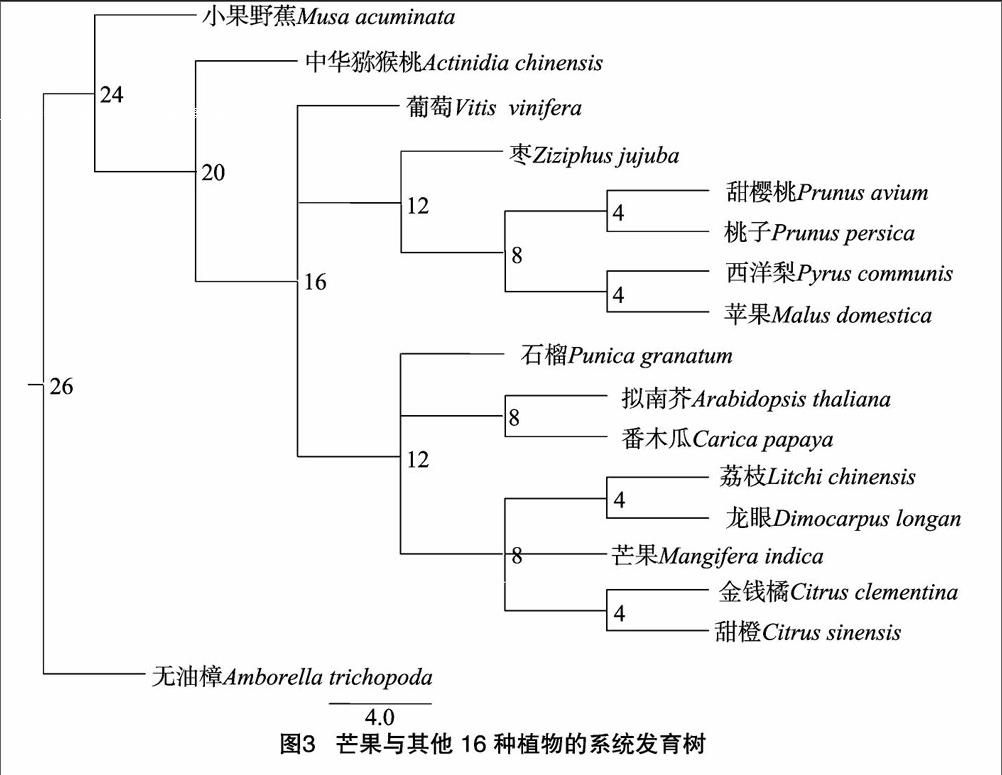
2.3 芒果与其他16种植物ANS基因的物种进化分析
将本试验中选定的17种植物做物种进化分析,结果(图3)显示,双子叶植物纲的植物聚为一大分支,单子叶植物纲的植物为一支,外类群植物为一支。在本研究选取的15种果树中,同是蔷薇科的苹果、西洋梨、桃子、甜樱桃聚为一支,同是芸香科金钱橘和甜橙聚为一支,同是无患子科的龙眼、荔枝聚为一支,说明在物种进化过程中,同纲植物亲缘关系更近,不同纲的植物亲缘关系较远。同时,芒果处于金钱橘、甜橙与荔枝、龙眼之间,说明在物种进化过程中,芒果与金钱橘、甜橙、荔枝、龙眼的亲缘关系更近,其次是番木瓜,而与苹果、西洋梨、桃子、甜樱桃、枣、葡萄、中华猕猴桃亲缘关系相对较远,与小果野蕉的亲缘关系最远。
2.4 芒果与其他16种植物ANS蛋白的理化性质分析
对36条ANS氨基酸序列进行理化性质分析,结果(表1)显示,除Arath|AT2G38240.1为碱性蛋白(理论pI>7)外,其余蛋白均为酸性蛋白质(理论pI<7),酸性蛋白质占972%;根据Guruprasad方法[37],除Arath|AT5G05600.1、Citcl|Ciclev10031811m、Maldo|MDP0000156478、Musac|GSMUA_Achr10T00230_001、Ambtr|XP_006828895为稳定性蛋白(不稳定系数<40)外,其余蛋白均为不稳定性蛋白(不稳定系数>40),稳定性蛋白占13.9%;相对分子量除Maldo|MDP0000621569为52841.2外,其余位于36.21~46.16 ku;仅金钱橘Citcl|Ciclev10012063m与甜橙Citsi|NP_001275784、Citsi|orange1.1g018466m基因具有线粒体靶向肽M (mitochondrial targeting peptide,mTP),其余氨基酸序列均没有导肽,有导肽的蛋白占8.3%;所有蛋白均为亲水性蛋白(GRAVY<0)、无信号肽;所有基因亚细胞定位于细胞质中。说明芒果与其他绝大多数植物ANS蛋白具有相似的性质,为非分泌性蛋白,在细胞内进行代谢活动。
2.5 芒果ANS蛋白二级结构分析
蛋白质二级结构是指多肽链中有规则重复的构象,限于主链原子的局部空间排列,不包括与肽链其他区段的相互关系及侧链构象。常见的二级结构元件有α-螺旋、β-折叠、β-转角、无规则卷曲4种形式,α-螺旋是蛋白质中最常见的二级结构元件。α-螺旋靠氢键维持稳定,在DNA结合基序中有非常重要的作用;β-折叠是由伸展的肽链组成,通过位于同一个肽链或相邻肽链的另一个酰胺氢和一个肽键的羰基氧之间的氢键维持;β-转角连接蛋白质分子中的二级结构(α-螺旋和β-折叠),是肽链走向改变的一种非重复多肽区,蛋白质的抗体识别、磷酸化、糖基化和羟基化位点经常出现在转角;无规则卷曲主要指那些不能被归入明确的二级结构如折叠片或螺旋的多肽区段,也是明确而稳定的结构,它们受侧链相互作用的影响很大,经常构成酶活性部位和其他蛋白质特异的功能部位。
应用在线工具SOPMA预测17种植物的ANS氨基酸序列的二级结构(表1),除Arath|AT5G05600.1、Arath|AT3G11180.2、Maldo|MDP0000621569、Maldo|MDP0000156478的主要結构元件是无规则卷曲、β-折叠外,其余蛋白均以α-螺旋、无规则卷曲为主要结构元件。芒果ANS氨基酸序列的二级结构,结果(图4)显示,α-螺旋是芒果ANS最大量的结构元件,占43.3%;其余由31.91%的无规则卷曲、15.38%的β-折叠、9.4%的β-转角组成。说明芒果ANS与本试验中的绝大多数植物ANS蛋白一样,具有稳定的花青素合成酶结构,进一步推断芒果ANS基因可能在花青素代谢途径中具有重要作用。endprint
2.6 芒果ANS蛋白的磷酸化位点分析
蛋白磷酸化是调节和控制蛋白质活力和功能的基本机制,一般发生在翻译后修饰;由于氨基酸侧链加入了1个带强负电荷的磷酸基团,发生酯化作用,从而改变了蛋白质的构象、活性。因此,蛋白质的磷酸化位点是判断其功能的特征之一。蛋白质磷酸化作为普遍存在于生物体内的调节方式,其主要发生在丝氨酸(Ser)、苏氨酸(Thr)和酪氨酸(Tyr)残基侧链的羟基上。磷酸化位点分析结果表明,芒果ANS基因所编码的蛋白质含有15个磷酸化位点(图5),其中Ser 11个、Thr 1个和Tyr 3个。推测芒果ANS蛋白的磷酸化可能与膜融合蛋白通过改变自身构象来转运金属离子并转运至外模蛋白有关,也可能与细胞信号转导有关。
2.7 拟南芥ANS基因生育期芯片中的表达模式
由于芒果ANS基因的研究目前只限于克隆,在进一步的试验研究过程中,以期在模式植物的研究成果中得到借鉴作用。现以拟南芥ANS基因生育芯片的表达模式为例,绘制出拟南芥ANS基因家族的表达模式图,如图6所示。拟南芥5条ANS基因的表达均表现出明显的组织特异性:AT4G16330.2基因除在花粉(6周)表达较弱外,其余组织均表达较强;AT5G05600.1基因在成熟叶(17 d)、花萼(>21 d)中表达最强,在花粉(6周)、茎尖(14 d)、幼叶(10 d)、芽(7 d)、下胚轴(7 d)、花梗(>21 d)表达较弱,其余组织均表达较强;AT2G38240.1基因在雄蕊(>21 d)表达较强,在花瓣(>21 d)、成熟叶 (17 d)、叶柄(17 d)表达较弱,其余组织均无表达;AT4G22880.1在老叶(35 d)中表达最强,在种子(8周)、老叶(35 d)、芽(7 d)、下胚轴(7 d)、子叶(7 d)、成熟叶(17 d)中表达较弱,其余组织均无表达;AT3G11180.2除在花粉(6周)、种子(8周)中有较强表达外,其余组织均无表达。
3 讨论
ANS基因作为花青素合成途径中的关键基因,广泛用于植物基因工程研究。本试验以芒果ANS基因作为研究目标,以拟南芥ANS基因为参考,对芒果、拟南芥、番木瓜、金钱橘、甜橙、苹果、桃子、甜樱桃、西洋梨、葡萄、枣、石榴、中华猕猴桃、龙眼、荔枝、小果野蕉和无油樟17种植物共36条ANS基因编码蛋白进行了系统发育分析、理化性质分析、结构分析等。
系统发育分析结果显示,系统进化树中被选为外类群的双子叶植物纲无油樟科无油樟为单独一支,双子叶植物与单子叶植物的ANS基因互相掺杂在一起,ANS基因进化树没有将双子叶植物与单子叶植物分开,与植物物种之间的亲缘关系及进化关系不一致,推测在进化过程中ANS基因的分化在双子叶植物和单子叶植物形成之后。
理化性质分析结果显示,所有蛋白中,酸性蛋白占972%、稳定性蛋白占13.9%,有导肽的蛋白占8.3%,所有蛋白均为无信号肽的亲水性蛋白。通过蛋白质导肽分析可知,仅金钱橘基因Citcl|Ciclev10012063m与甜橙基因 Citsi|NP_001275784、Citsi|orange1.1g018466m具有线粒体靶向肽M (mTP)外,其余氨基酸序列均没有导肽。芒果ANS蛋白与其他植物ANS蛋白表现出相似的性质,为酸性、无信号肽、无导肽的不稳定亲水性蛋白,是一种非分泌性蛋白,由游离核糖体合成,参与细胞内生化反应,在细胞内代谢过程中发挥重要作用。这与亚细胞定位于细胞质中的结果一致,也与花色素苷的合成主要在细胞质中完成一致。
结构分析结果显示,芒果ANS蛋白与其他蛋白序列都包含1个DIOX-N亚家族结构域和1个2OG-FeII_Oxy亚家族结构域,具有花色素合成酶家族特征多肽序列,属于双加氧酶家族,具有ANS基因家族的特征结构;芒果蛋白二级结构与大多数蛋白一样,以α-螺旋、无规则卷曲为主要结构元件,具有稳定的花青素合成酶结构,进一步推断芒果ANS基因可能在花青素代谢途径中具有重要作用。
蛋白磷酸化位点分析表明,芒果ANS蛋白存在15个磷酸化位点。一般来说,多肽链中的氨基酸潜在的磷酸化位点越多,发挥更多功能的可能性就越大。芒果ANS蛋白的磷酸化可能与膜融合蛋白通过改变自身构象来转运金属离子并转运至外模蛋白有关,也可能与细胞信号转导有关,磷酸化在该蛋白上的具体功能还有待进一步验证。
模式植物拟南芥ANS基因生育期芯片表达模式分析结果表明,5条基因的表达差异较大,表现出明显的组织特异性。以拟南芥的ANS基因为参考,以期对研究芒果ANS基因的表达、功能具有借鉴作用。
目前,虽有关于芒果中ANS基因的研究,但主要是对单个基因的克隆及生化特性的研究[9]。本研究以不同植物ANS基因具有较高的同源性为依据,选取17个物种(包括15种果树、1种模式植物、1种系统发育树选为外类群的植物),比较了芒果与其他植物中ANS基因的特性,具有更强的针对性和借鉴性。研究结果得出,芒果ANS蛋白性质与其他植物ANS蛋白表现出高度相似性,芒果ANS基因具有ANS基因家族特征,推测芒果ANS基因与其他植物起着相同或相似的生物学功能,但还需进一步的实验验证。本试验对进一步研究芒果ANS基因的时空表达特性及在花青素合成途径中的作用机制提供理论支持。
参考文献:
[1]羅学兵. 芒果的营养价值、保健功能及食用方法[J]. 中国食物与营养,2011,17(7):77-79.
[2]Andersen M,Jordheim M. The anthocyanins[M]//Andersen M,Markham K R. Flavonoids:chemistry,biochemistry and applications. Boca Raton,Florida:CRC Press,2006:471-551.endprint
[3]Ravindra P V,Narayan M S. Antioxidant activity of the anthocyanin from carrot (Daucus carota) callus culture[J]. International Journal of Food Sciences & Nutrition,2003,54(5):349-355.
[4]Lev-Yadun S,Gould K S. Role of anthocyanins in plant defence[M]//Gould K,Davies K,Winefield C. Anthocyanins:biosynthesis,functions,and applications. New York:Springer,2009:22-28.
[5]Xie D Y,Jackson L A,Cooper J D,et al. Molecular and biochemical analysis of two cDNA clones encoding dihydroflavonol-4-reductase from Medicago truncatula[J]. Plant Physiology,2004,134(3):979-994.
[6]Menssen A,Hhmann S,Martin W,et al. The En/Spm transposable element of Zea mays contains splice sites at the termini generating a novel intron from a dSpm element in the A2 gene[J]. The EMBO Journal,1990,9(10):3051-3057.
[7]Wilmouth R C,Turnbull J J,Welford R W,et al. Structure and mechanism of anthocyanidin synthase from Arabidopsis thaliana [J]. Structure,2002,10(1):93-103.
[8]趙志常,胡福初,黄建峰,等. 荔枝ANS基因的克隆及其序列分析[J]. 北方园艺,2012 (19):118-121.
[9]赵志常,陈业渊,高爱平,等. 芒果ANS基因的克隆及其序列分析[J]. 华北农学报,2014,29 (增刊1):6-9.
[10]游向荣,许鸿川,梁文裕,等. 龙眼花芽ANS基因的克隆与原核表达[J]. 热带亚热带植物学报,2010,18(3):288-292.
[11]Wang H,Wang W,Zhang P,et al. Gene transcript accumulation,tissue and subcellular localization of anthocyanidin synthase (ANS) in developing grape berries[J]. Plant Science,2010,179(1):103-113.
[12]Kim S H,Lee J R,Hong S T,et al. Molecular cloning and analysis of anthocyanin biosynthesis genes preferentially expressed in apple skin[J]. Plant Science,2003,165(2):403-413.
[13]李晓艳,裴嘉博,张志东,等. 越橘VcANS基因的克隆及表达分析[J]. 西北农林科技大学学报(自然科学版),2012(6):201-209.
[14]金琦芳,陈志丹,孙威江,等. 茶树CsANS基因及其启动子的克隆与生物信息学分析[J]. 茶叶科学,2016,36(2):219-228.
[15]孙宏伟,李艳军,王雅琴,等. 棕色棉GhANS基因的克隆与表达分析[J]. 核农学报,2014,28(2):200-207.
[16]Honda C,Kotoda N,Wada M,et al. Anthocyanin biosynthetic genes are coordinately expressed during red coloration in apple skin[J]. Plant Physiology & Biochemistry,2002,40(11):955-962.
[17]Salvatierra A,Pimentel P,Moya-Leon M A,et al. Comparison of transcriptional profiles of flavonoid genes and anthocyanin contents during fruit development of two botanical forms of Fragaria chiloensis ssp. chiloensis[J]. Phytochemistry,2010,71(16):1839-1847.
[18]Li J,Lü R H,Zhao A C,et al. Isolation and expression analysis of anthocyanin biosynthetic genes in Morus alba L.[J]. Biologia Plantarum,2014,58(4):618-626.
[19]Goodstein D M,Shu S,Howson R,et al. Phytozome:a comparative platform for green plant genomics[J]. Nucleic Acids Research,2012,40(D1):D1178-D1186.endprint
[20]Huang S,Ding J,Deng D,et al. Draft genome of the kiwifruit Actinidia chinensis [J]. Nature Communications,2013,4:2640.
[21]Tamura K,Stecher G,Peterson D,et al. MEGA6:molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology & Evolution,2013,30(12):2725-2729.
[22]Edgar R C. MUSCLE:multiple sequence alignment with high accuracy and high throughput[J]. Nucleic Acids Research,2004,32(5):1792-1797.
[23]Castresana J. Selection of conserved blocks from multiple alignments for their use in phylogenetic analysis[J]. Molecular Biology & Evolution 2000,17(4):540-552.
[24]Talavera G,Castresana J. Improvement of phylogenies after removing divergent and ambiguously aligned blocks from protein sequence alignments[J]. Systematic Biology,2007,56(4):564-577.
[25]Ronquist F,Teslenko M,van der Mark P,et al. MrBayes 3.2:efficient Bayesian phylogenetic inference and model choice across a large model space[J]. Systematic Biology,2012,61(3):539-542.
[26]Marchler-Bauer A,Derbyshire M K,Gonzales N R,et al. CDD:NCBIs conserved domain database[J]. Nucleic Acids Research,2015,43(Database issue):222-226.
[27]Gasteiger E,Hoogland C,Gattiker A,et al. Protein identification and analysis tools on the ExPASy server[M]//Walker J M. The Proteomics protocols handbook. Totowa,New Jersey,US:Humana Press,2005:571-607.
[28]Hofmann K,Stoffel W. Tmbase—A database of membrane spanning protein segments[J]. Biological Chemistry Hoppe-Seyler,1993,374:166.
[29]Emanuelsson O,Brunak S,von Heijne G,et al. Locating proteins in the cell using TargetP,SignalP and related tools[J]. Nature Protocols,2007,2(4):953-971.
[30]Petersen T N,Brunak S,von Heijne G,et al. SignalP 4.0:discriminating signal peptides from transmembrane regions[J]. Nature Methods,2011,8(10):785-786.
[31]Chou K C,Shen H B. Plant-mPLoc:a top-down strategy to augment the power for predicting plant protein subcellular localization[J]. PLoS One,2010,5(6):e11335.
[32]Sapay N,Guermeur Y,Deléage G. Prediction of amphipathic inplane membrane anchors in monotopic proteins using a SVM classifier[J]. BMC Bioinformatics,2006,7:255.
[33]Blom N,Gammeltoft S,Brunak S. Sequence and structure-based prediction of eukaryotic protein phosphorylation sites[J]. Journal of Molecular Biology,1999,294(5):1351-1362.
[34]Dash S,Van Hemert J,Hong L,et al. PLEXdb:gene expression resources for plants and plant pathogens[J]. Nucleic Acids Research,2012,40(D1):D1194-D1201.
[35]Hagel J M,Facchini P J. Dioxygenases catalyze the O-demethylation steps of morphine biosynthesis in opium poppy[J]. Nature Chemical Biology,2010,6(4):273-275.
[36]Aravind L,Koonin E V. The DNA-repair protein AlkB,EGL-9,and leprecan define new families of 2-oxoglutarate-and iron-dependent dioxygenases[J]. Genome Biology,2001,2(3):181-200.
[37]Guruprasad K,Reddy B V B,Pandit M W. Correlation between stability of a protein and its dipeptide composition:a novel approach for predicting in vivo stability of a protein from its primary sequence[J]. Protein Engineering,1990,4(2):155-161.endprint
