二倍体棉花热激转录因子HSFs家族全基因组生物信息学分析
2017-11-30张华崇闫振华赵树琪黄晓莉戴宝生李蔚
张华崇+闫振华+赵树琪+黄晓莉+戴宝生+李蔚


摘要:热激因子(heat shock factors,HSFs)是生物体内调节热激应答的一类转录因子,在热胁迫信号转导和耐热性的产生过程中发挥了重要的作用。从已释放的二倍体亚洲棉和雷蒙德氏棉全基因组测序结果中分别鉴定到42个和40个HSFs蛋白家族成员,运用生物信息学分析其序列特征、亚细胞定位、聚类分析和保守基序等。结果表明,亚洲棉HSFs蛋白长度116~526个氨基酸,分子量13 748.4~60 028.2 u,等电点4.40~8.75;雷蒙德氏棉HSFs蛋白长度为190~528个氨基酸,分子量21 993.0~60 413.6 u,等电点4.36~9.50。染色体定位显示二倍体棉花中的HSFs基因不均匀分布在13条染色体上;聚类分析表明,HSFs蛋白序列分为A、B、C 3类,每一亚类中二倍体棉花与拟南芥的蛋白具有较高的同源性,趋向于聚集在一起,且每个亚组具有相同或相似的保守基序类型及排列顺序。
关键词:二倍体棉花;热激转录因子;全基因组;生物信息学
中图分类号: Q755 文献标志码: A 文章编号:1002-1302(2017)20-0035-08
热激因子(heat shock factors,HSFs)是生物体内调节热激应答的一类转录因子,在热胁迫信号转导和耐热性的产生过程中发挥了重要的作用[1-2]。1988年,HSFs基因首先在酵母中发现[3],植物中的HSFs基因最先在番茄中克隆获得[4]。典型的热激转录因子一般包括4个部分:N端的DNA结合域(DNA binding domain,DBD)、寡聚化结构域(HR-A/B)、细胞核定位信号(Nuclear localization signal,NLS)、细胞核输出信号(Nuclear export signal,NES),少数还具有一个C端激活域(Cterminal activation domain,CTAD)[5]。位于HSFs基因N端的DBD中含有一个螺旋-转角-螺旋的疏水结构,能特异地结合热激元件(heat stress element,HSE),诱导下游热激蛋白表达[6-7]。HR-A/B含有2个疏水七肽重复区域A和B,通过形成卷曲螺旋结构促使HSFs形成同源三聚体,根据保守DBD和HR-A/B区的结构特点,热激转录因子又可以分为A、B、C 3类[8-9],在A 类、C类基因中,HR-A/B中A、B结构域之间分别有21个和7个氨基酸插入;在B类基因中,无氨基酸残基插入[6,10]。NLS和NES含有一簇碱性氨基酸残基,与HSFs蛋白家族的亚细胞定位相关[11-12]。CTAD以芳族的、大的疏水的及酸性的氨基酸(AHA)基元为特征,在A类基因中含特有激活HSFs功能的AHA基元[6]。
棉花是喜温植物,适当高温可加快棉株生长发育,但长时间的高温严重影响花粉发育、授粉和受精,对棉花产量和品质产生不利的影响。随着全球气候的变暖,棉花的耐高温育种显得尤其重要。本研究利用已释放的二倍体亚洲棉和雷蒙德氏棉全基因组测序结果[13-14],通过生物信息学技术扫描全基因组HSFs,进一步分析其进化关系、亚细胞定位、理化性质预测和保守基序等,为下一步的选育耐高温新品种提供理论基础。
1 材料与方法
1.1 全基因组检索HSF基因
HSF基因的结构域信息(PF00447)下载自Pfam数据库[15],利用软件HMMER3.1b2[16]在Cottongen(https://www.cottongen.org/)数据库中检索二倍体亚洲棉和雷蒙德氏棉HSFs基因家族氨基酸序列,经Smart网站[17](http://smart.embl-heidelberg.de/smart/set_mode.cgi?NORMAL=1)进一步结构域预测确认。
拟南芥HSF基因家族氨基酸序列来自PlantTFDB(http://planttfdb.cbi.pku.edu.cn/)数据库[18]。
1.2 理化性质预测
通过ExPASy网站中的程序Protparam(http://web.expasy.org/protparam/)对HSFs蛋白家族蛋白分子量、等电点等基本理化性质进行预测分析。
1.3 亚细胞定位
利用在线软件WOLF PSORT(http://www.genscript.com/wolf-psort.html),以及在线网站Soft Berry(http://linux1.softberry.com/berry.phtml)中的PROTCOMP 程序共同对HSFs蛋白家族进行亚细胞定位预测分析。
1.4 聚类分析
利用生物信息学软件MEGA5.2对鉴定的二倍体棉花中的HSFs蛋白家族和拟南芥中的HSFs蛋白家族进行多重序列比对,采用邻接法构建系统发育树,进行1 000次重复。
1.5 基序分析
利用MEME软件分析HSFs蛋白家族的motif类型和顺序。具体参数设置如下:预测数10,其他参数为默认。
2 结果与分析
2.1 棉花HSF基因家族鉴定
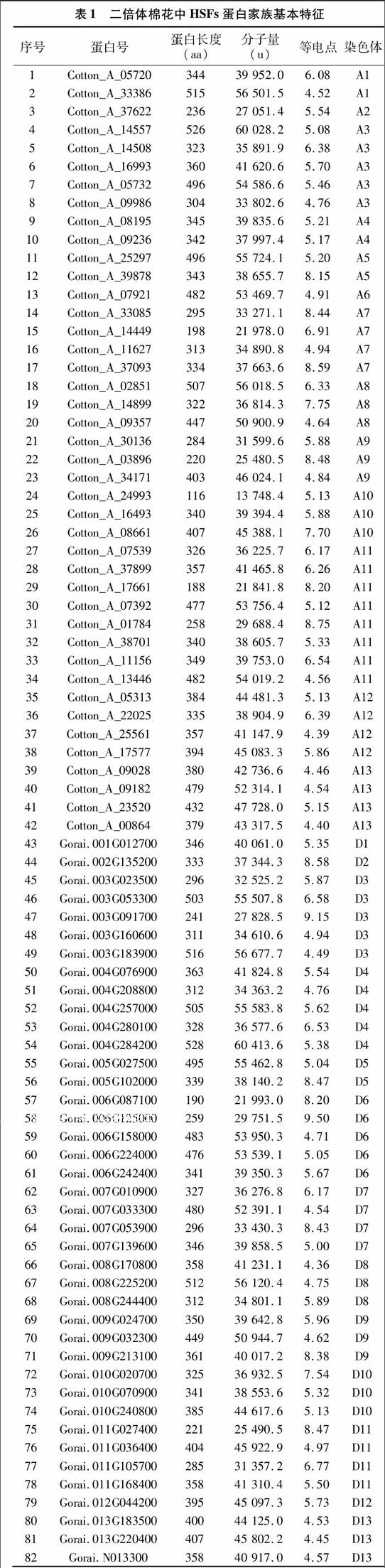
利用结构域预测得到的结构域信息(PF00447),通过对二倍体棉花全基因组搜索,发现亚洲棉和雷蒙德氏棉分别有42条和40条具有典型HSF结构域蛋白,并统计了其蛋白号、蛋白长度及对应基因所在的染色体(表1),结果显示,亚洲棉HSFs蛋白长度为116~526个氨基酸不等;雷蒙德氏棉HSFs蛋白长度为190~528个氨基酸不等。亚洲棉基因组中的42个HSFs基因家族成员不均匀分布在13条染色体上,其中,A11号染色体分布最多,为8个;A6号染色体分布最少,为1个。雷蒙德氏棉基因组中的40个HSFs基因家族成员也不均匀分布在13条染色体上,其中,D3、D4和D6号染色体分布最多,均为5个;D1、D2和D12號染色体分布最少,均为1个。endprint
2.2 棉花HSFs蛋白家族理化性質预测
利用在线网站对二倍体棉花HSFs蛋白家族理化性质进行了预测(表1),预测结果,二倍体棉花中HSFs蛋白分子量存在较大差异。其中,亚洲棉中分子量最大的是Cotton_A_14557,达到了60 028.2 u,分子量最小的是Cotton_A_24993,为13 748.4 u;雷蒙德氏棉中分子量最大的是Gorai.004G284200,为60 413.6 u,分子量最小的是Gorai.006G087100,为21 993.0 u。等电点变化范围也比较大,其中,亚洲棉中Cotton_A_01784等电点最大,为8.75,Cotton_A_00864最小,为4.40;雷蒙德氏棉中Gorai.006G125000等电点最大,为9.50,Gorai.008G170800最小,为4.36,表明HSFs蛋白家族从酸性到碱性都有分布。
2.3 棉花HSFs蛋白家族亚细胞定位
为了了解HSFs蛋白家族发挥功能的区域,利用2种亚细胞定位软件预测了二倍体棉花HSFs蛋白家族亚细胞定位(表2),结果表明,2种软件WoLF PSORT和Protcomp预测结果类似,大部分HSFs蛋白家族定位于细胞核。根据软件WoLF PSORT预测发现,HSFs蛋白家族定位于1~4个位置不等,但除亚洲棉中Cotton_A_24993和除了雷蒙德氏棉中Gorai.006G087100分别定位于叶绿体和细胞质外,其余HSFs蛋白家族定位于细胞核分值最高;根据Protcomp预测发现,除了雷蒙德氏棉中Gorai.004G284200定位于细胞质中,其余HSFs蛋白家族均定位于细胞核。综上所述,二倍体棉花的HSFs蛋白家族中至少有一种预测定位于细胞核,这符合转录因子在细胞核中调控基因表达的作用特点。
2.4 棉花HSFs蛋白家族聚类分析
为了揭示二倍体棉花HSFs蛋白家族的进化关系,将亚洲棉和雷蒙德氏棉的42条和40条HSFs蛋白,与下载的21条拟南芥HSFs蛋白家族进行聚类分析(图1)。结果显示,103条HSFs蛋白序列分为了A、B、C 3类,其中A类含有9个亚类(A1、A2、A3、A4、A5、A6、A7、A8、A9),二倍体棉花都不含有A7亚类,每一亚类中二倍体棉花与拟南芥的蛋白具有较高的同源性,趋向于聚集在一起,例如,雷蒙德氏棉 Gorai.008G170800、Gorai.N013300和亚洲棉Cotton_A_25561、Cotton_A_00864、Cotton_A_24993与拟南芥中的HSF蛋白AT1G67970(AtHsfA8a)互为同源蛋白而聚集在一起。B类含有5个亚类(B1、B2、B3、B4、B5),且二倍体棉花中完整地含有5个亚类,而拟南芥不含有亚类B5。C类中只有1个亚类C1,且二倍体棉花和拟南芥中都存在。此外,不同亚类HSF蛋白虽聚集在一起,但二倍体棉花与拟南芥却趋向于形成不同的分枝,且亚洲棉中存在与雷蒙德氏棉同源的HSF蛋白(除Cotton_A_24993和Cotton_A_22025)。
2.5 棉花HSFs蛋白家族基序分析
对二倍体棉花82个HSFs蛋白家族保守基序的分析结果见图2,82个HSFs蛋白家族含有10个保守基序,长度为11~50个氨基酸。进一步分析10个保守基序在不同亚组HSFs蛋白中的分布(图3), 结果表明, HSFs蛋白家族含有保守基序个数从2~8个不等,每个亚组具有相同或相似的保守基序类型及排列顺序。例如,A1亚类Gorai.003G053300、Gorai.003G183900、Gorai.007G033300、Gorai.004G257000、Gorai.008G225200、Gorai.013G183500、Cotton_A_02851、Cotton_A_05732、Cotton_A_23520、Cotton_A_07921、Cotton_A_33386和Cotton_A_09182具有相似的基序(7~8个),也具有相似的排列顺序。
3 讨论
在全球气候变暖的环境下, 高温对棉花的正常生长发育产生了不利的影响,常常诱使棉叶凋萎,花粉失水干缩,蕾铃大量脱落[19]。HSFs是植物响应热激胁迫保护自身免受高温伤害的一类转录因子,研究发现拟南芥、水稻、大豆中分别有21、25、52个HSFs[20-22]。本研究基于二倍体棉花雷蒙德氏棉和亚洲棉测序的完成,通过全基因组进行搜索分析,亚洲棉和雷蒙德氏棉分别鉴定出42个和40个具有典型HSF结构域蛋白,Wang等利用表达序列标签组装和全基因组分析技术在陆地棉D亚组中也鉴定得到40个HSFs基因[9]。本研究中HSFs蛋白家族分为A、B、C 3类,每类含有的HSF数目不同,理化性质也存在较大差异,且不均匀地分布在不同染色体上,表明二倍体棉花HSFs蛋白家族存在广泛的多样性,这也为功能的多样性奠定了基础。
HSFs作为一类转录因子,要通过调控相关基因的表达行使功能,其表达模式有组成型表达和诱导型表达2类,广泛参与植物响应高温、干旱和氧化等多种逆境胁迫[23-25]。虽然在模式植物拟南芥和番茄中的研究较为详尽,拟南芥AtHSFA2基因过表达显著提高了转基因拟南芥耐热性[26-27],而敲除AtHSFA2基因抑制了下游热激蛋白基因的表达,明显提高了拟南芥对高温敏感性[28-29]。棉花中关于HSFs研究的报道还很少,相关研究表明,陆地棉中HSFs基因在响应高温和纤维发育过程中发挥重要作用[9]。但HSFs表达调控机制尤其是上游调控机制,不同HSFs基因在响应同一外界刺激和同一HSFs基因在不同逆境胁迫中是否存在保守的信号转导途径等等,还有待进一步研究阐明。
参考文献:
[1]Morimoto R I. Regulation of the heat shock transcriptional response:cross talk between a family of heat shock factors,molecular chaperones,and negative regulators[J]. Genes & Development,1998,12(24):3788-3796.endprint
[2]Baniwal S K,Bharti K,Chan K Y,et al. Heat stress response in plants:a complex game with chaperones and more than twenty heat stress transcription factors[J]. Journal of Biosciences,2004,29(4):471-487.
[3]Wiederrecht G,Seto D,Parker C S. Isolation of the gene encoding the S. cerevisiae heat shock transcription factor[J]. Cell,1988,54(6):841-853.
[4]Scharf K D,Rose S,Zott W,et al. Three tomato genes code for heat stress transcription factors with a region of remarkable homology to the DNA-binding domain of the yeast HSF[J]. Embo Journal,1990,9(13):4495-4501.
[5]Hübel A,Schffl F. Arabidopsis heat shock factor:isolation and characterization of the gene and the recombinant protein[J]. Plant Molecular Biology,1994,26(1):353-362.
[6]Scharf K D,Berberich T,Ebersberger I,et al. The plant heat stress transcription factor (Hsf) family:structure,function and evolution[J]. Biochimica Et Biophysica Acta,2012,1819(2):104-119.
[7]Sakurai H,Enoki Y. Novel aspects of heat shock factors:DNA recognition,chromatin modulation and gene expression[J]. FEBS Journal,2010,277(20):4140-4149.
[8]Kotak S,Port M,Ganguli A,et al. Characterization of C-terminal domains of Arabidopsis heat stress transcription factors (Hsfs) and identification of a new signature combination of plant class A Hsfs with AHA and NES motifs essential for activator function and intracellular localization[J]. The Plant Journal,2004,39(1):98-112.
[9]Wang J,Sun N,Deng T,et al. Genome-wide cloning,identification,classification and functional analysis of cotton heat shock transcription factors in cotton (Gossypium hirsutum)[J]. BMC Genomics,2014,15(1):961.
[10]Harrison C J,Bohm A A,Nelson H C. Crystal structure of the DNA binding domain of the heat shock transcription factor[J]. Science,1994,263(5144):224-227.
[11]Grlich D,Kutay U. Transport between the cell nucleus and the cytoplasm[J]. Annual Review of Cell and Developmental Biology,1999,15:607-660.
[12]Heerklotz D,Dring P,Bonzelius F,et al. The balance of nuclear import and export determines the intracellular distribution and function of tomato heat stress transcription factor HsfA2[J]. Molecular and Cellular Biology,2001,21(5):1759-1768.
[13]Li F,Fan G,Wang K,et al. Genome sequence of the cultivated cotton Gossypium arboreum[J]. Nature Genetics,2014,46(6):567-572.
[14]Wang K,Wang Z,Li F,et al. The draft genome of a diploid cotton Gossypium raimondii[J]. Nature Genetics,2012,44(10):1098-1103.endprint
[15]Finn R D,Coggill P,Eberhardt R Y,et al. The Pfam protein families database:towards a more sustainable future[J]. Nucleic Acids Research,2016,44(D1):279-285.
[16]Finn R D,Clements J,Eddy S R. HMMER web server:interactive sequence similarity searching[J]. Nucleic Acids Research,2011,39:29-37.
[17]Schultz J,Milpetz F,Bork P,et al. SMART,a simple modular architecture research tool:identification of signaling domains[J]. Proceedings of the National Academy of Sciences of the United States of America,1998,95(11):5857-5864.
[18]Jin J,He K,Tang X,et al. An Arabidopsis transcriptional regulatory map reveals distinct functional and evolutionary features of novel transcription factors[J]. Molecular Biology and Evolution,2015,32(7):1767-1773.
[19]阿布力孜,開赛尔,阿吉古丽,等. 干热风对棉花生长发育的危害及对策建议[J]. 农业科技通讯,2009(10):68-69.
[20]Nover L,Bharti K,Dring P,et al. Arabidopsis and the heat stress transcription factor world:how many heat stress transcription factors do we need?[J]. Cell Stress & Chaperones,2001,6(3):177-189.
[21]Guo J,Wu J,Ji Q,et al. Genome-wide analysis of heat shock transcription factor families in rice and Arabidopsis[J]. Journal of Genetics and Genomics,2008,35(2):105-118.
[22]Chung E,Kim K M,Lee J H. Genome-wide analysis and molecular characterization of heat shock transcription factor family in Glycine max[J]. Journal of Genetics and Genomics,2013,40(3):127-135.
[23]Storozhenko S,De Pauw P,Van Montagu M,et al. The heat-shock element is a functional component of the Arabidopsis APX1 gene promoter[J]. Plant Physiology,1998,118(3):1005-1014.
[24]Panchuk I I,Volkov R A,Schffl F. Heat stress-and heat shock transcription factor-dependent expression and activity of ascorbate peroxidase in Arabidopsis[J]. Plant Physiology,2002,129(2):838-853.
[25]Panikulangara T J,Eggers-Schumacher G,Wunderlich M,et al. Galactinol synthase 1. A novel heat shock factor target gene responsible for heat-induced synthesis of raffinose family oligosaccharides in Arabidopsis[J]. Plant Physiology,2004,136(2):3148-3158.
[26]Nishizawa A,Yabuta Y,Yoshida E,et al. Arabidopsis heat shock transcription factor A2 as a key regulator in response to several types of environmental stress[J]. The Plant Journal,2006,48(4):535-547.
[27]Ogawa D,Yamaguchi K,Nishiuchi T. High-level overexpression of the Arabidopsis HsfA2 gene confers not only increased themotolerance but also salt/osmotic stress tolerance and enhanced callus growth[J]. Journal of Experimental Botany,2007,58(12):3373-3383.
[28]Schramm F,Ganguli A,Kiehlmann E,et al. The heat stress transcription factor HsfA2 serves as a regulatory amplifier of a subset of genes in the heat stress response in Arabidopsis[J]. Plant Molecular Biology,2006,60(5):759-772.
[29]Charng Y Y,Liu H C,Liu N Y,et al. A heat-inducible transcription factor,HsfA2,is required for extension of acquired thermotolerance in Arabidopsis[J]. Plant Physiology,2007,143(1):251-262.endprint
