中国花生品种更替引发的SSR位点遗传多样性变化趋势分析
2017-11-10李玉荣程增书陈四龙宋亚辉张朋娟
王 瑾,李玉荣,程增书,陈四龙,宋亚辉,张朋娟
(河北省农林科学院粮油作物研究所河北省作物遗传育种实验室,石家庄 050035)
中国花生品种更替引发的SSR位点遗传多样性变化趋势分析
王 瑾,李玉荣*,程增书,陈四龙,宋亚辉,张朋娟
(河北省农林科学院粮油作物研究所河北省作物遗传育种实验室,石家庄 050035)
【目的】旨在了解自20世纪50年代以来我国花生品种更替所引发的SSR位点遗传多样性变化,以期为花生育种提供参考。【方法】选用154对SSR引物对68个大面积推广种植品种进行检测。【结果】共获得173个位点,检测到872个等位变异,平均为5.04个等位变异/位点,遗传多样性指数的变化范围为0.014~0.881,平均0.477。20条染色体中,b07遗传多样性最高,染色体a04最低。品种更替过程中,20世纪80年代品种更替对SSR位点遗传多样性影响最为显著,与20世纪70年代及以前品种相比,表现为等位基因遗传丰富度增加、多样性指数增加、品种间遗传距离略有降低。20世纪80年代以后,SSR位点的等位基因丰富度增加、多样性指数和品种间遗传距离均无明显变化。检测到5个等位变异数随年代增加减少的SSR位点。聚类分析结果显示类群分布与系谱及地理来源相关,与品种更替年代无关。【结论】本研究结果表明,在花生品种更替过程中,主栽品种等位基因丰富度增加,而等位基因分布均匀度尚未产生显著性改变。
花生;品种更替;主栽品种;SSR;遗传多样性
花生(Arachishypogaea L.)脂肪含量38%~60%,蛋白质含量24%~36%,糖分含量20%左右,含有丰富的维生素 B2、PP、A、D、E,钙和铁等,是深受中国人喜爱的油料作物。目前,中国花生年均总产1342万t,占世界花生总产的39.47%,居世界首位。种植面积为505.6万公顷,占世界花生面积的19.12%,仅次于印度居世界第二位[1]。花生种植效益高,是促进农民增收的重要作物,在农业供给侧结构改革中占有重要地位。
自20世纪50年代以来,我国花生经历了5次品种更替,每次更替均显著提高了单位面积的产量,使花生生产得到飞速发展。20世纪50年代,伏花生和狮头企等品种的鉴定和大面积推广,实现了我国花生品种的第一次更替;20世纪70年代末,随着花17、粤油551、天府3号和开农8号等品种的育成推广,实现了第二次品种更替;20世纪80年代,随着海华1号、白沙1016、天府7号和冀油4号等品种的育成推广,实现了第三次品种更替;20世纪90年代,随着粤油223、8130、冀花2号和中花4号等品种的育成推广,实现了第四次品种更替;进入21世纪以来,高产、优质和抗病等专用型花生新品种逐步替代非专用型花生品种,实现了我国花生品种的第五次更替[2]。然而,近年来花生品种改良遗传进度放缓,2011年花生单产为3502.50 kg/hm2,2015年为3561.75 kg/hm2,每年增幅仅为0.34%(http://www.zzys.moa.gov.cn/)。遗传基础狭窄是造成遗传改良进度放缓的可能原因,育种过程中品种的更替通常会引起遗传多样性降低[3-4]。
微卫星标记 SSR(simple sequence repeats)是从分子水平上探讨遗传多样性的实用工具。已被广泛应用于花生的遗传多样性分析、遗传图谱的构建、QTL定位和杂交种鉴定等方面[1,5-7]。由于SSR标记具有多态性丰度高的特点,致使传统银染法检测容易产生实验误差,同一试验中不易准确区分差异小的等位变异;不同试验之间更难判断等位变异是否一致。毛细管电泳检测实现了数据收集和处理的自动化,克服了银染法的不足,有效的降低了人为试验误差[8],提高了试验精度,并使不同试验结果有效整合成为可能。
近年来,国内外学者利用SSR标记对野生花生[9-10]、栽培花生的不同类型间[10-13]、同一栽培花生类型的不同品种间[14-15]和花生核心种质[16-19]进行了遗传多样性分析。然而,由于年代久远,花生品种更替过程中涉及的一些品种分布分散、不易收集,针对历史上5次品种更替前后,主栽品种遗传多样性变化的专门研究尚未见报道。
本研究对中华人民共和国成立以来育成并大面积推广的不同时期花生主栽品种进行系统搜集整理,并应用SSR标记毛细管电泳检测,对我国花生主栽品种遗传多样性变化进行分析,揭示品种更替对花生主栽品种SSR位点遗传多样性演变的影响,为花生遗传育种和品种推广等提供依据和参考。
1 材料和方法
1.1 试验材料
本研究选取我国主栽花生品种共计68份为材料(见表1),品种主要来源于本单位种质资源圃和国家油料作物中期库。其中包含北方花生区(河北、河南、江苏、山东、安徽)38个花生品种,长江流域花生区(四川、湖北、湖南)12个花生品种和南方花生区(福建、广东、广西)18个花生品种。通过参考《中国花生品种及其系谱》[2]和《全国农作物主要品种推广情况统计表》及在地方农科院相关人员的帮助下,将材料分为4个时期进行分析(由于材料数目的限制,本研究中将70年代及以前的材料归为一类进行分析),其中包含20世纪70年代及以前花生品种7个,80年代花生品种15个,90年代和21世纪花生品种均为23个。
1.2 试验方法
剪取来源于15~20个单株的0.1 g新鲜花生叶片用液氮研成粉末,置于2mL离心管中,然后采用生工生物工程(上海)有限公司的基因组DNA快速抽提试剂盒(植物)进行DNA样品制备。DNA提取完成后用分光光度计测量其浓度和纯度,并将部分DNA稀释成20 ng/μL,置于4℃备用。
本研究中共选取多态性高且覆盖花生20对染色体的154对SSR引物对材料进行扫描,引物的序列及所在染色体参考文献[5,20-22]。采用普通引物加M13通用引物的方法检测,即引物合成时前导链5′端添加M13序列[23]。采用FAM、VIC、NED和PET共4种不同颜色的荧光染料直接标记M13互补链的每个碱基。PCR 反应体系(15μL) 为:7.5μL 2×power Taq PCR Master Mix(美国 Microanalysis Inc.),0.2μL 2μmol/L Primers 1(携带 M13),1μL 2μmol/L Primers 2,0.8μL 2μmol/L M13,2μL DNA,3.5μL ddH2O。反应程序为:94℃预变性5min;94℃变性45 s;50℃退火 45 s;72℃延伸 45 s,共 34个循环;94℃变性 45 s;53℃退火 45 s;72℃延伸 45 s,共 8个循环;72℃延伸10min。PCR扩增产物通过ABI3730 DNA分析仪进行毛细管电泳检测,检测结果利用Genemapper 3.2软件(Applied Biosystems)进行数据采集。
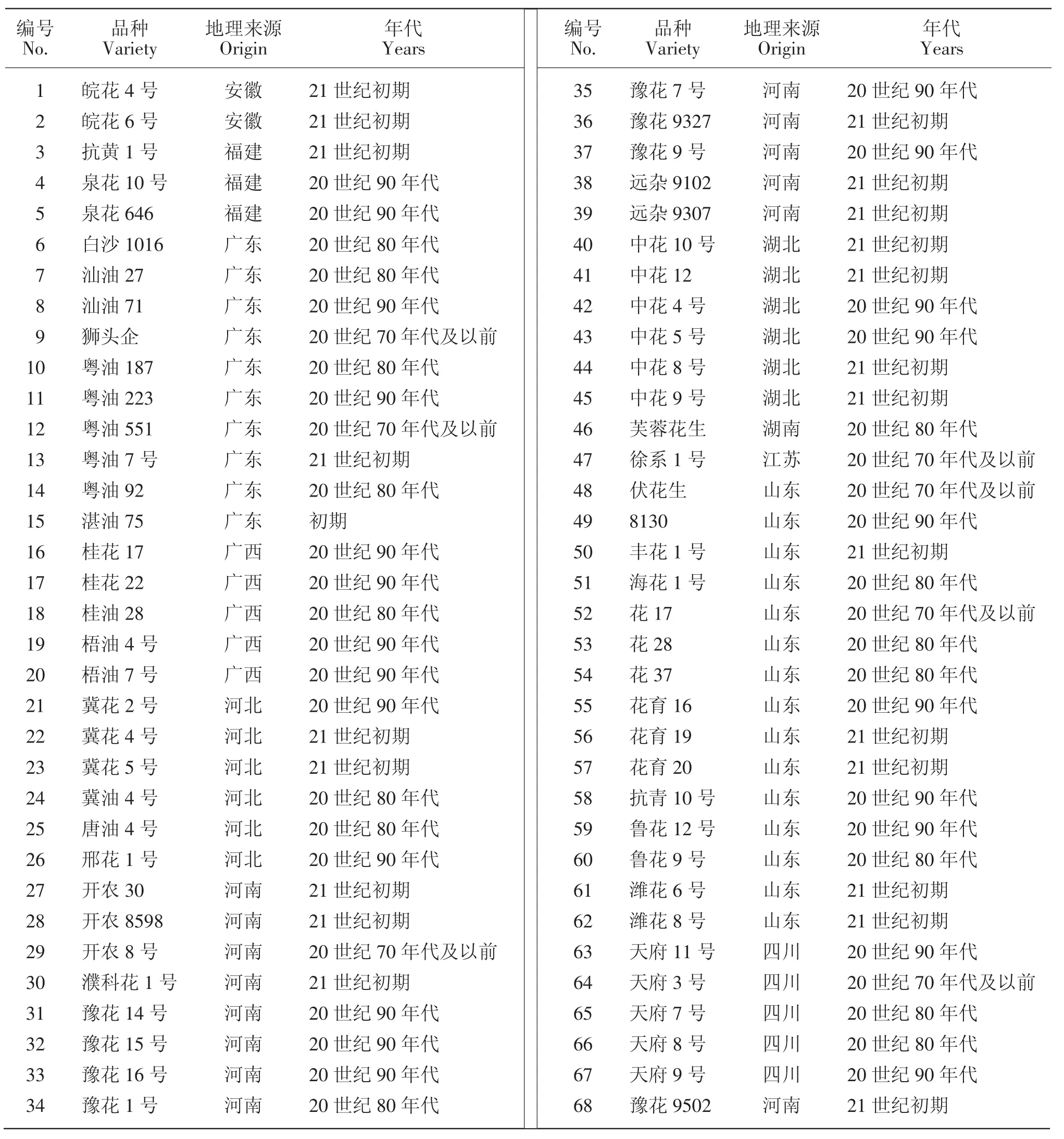
表1 主栽品种及其育成年代Table 1 The origin and the years released of peanut elite cultivars
1.3 数据统计分析
利用PowerMarkerV3.25软件[24]统计每个SSR位点的等位变异丰富度(A)、多态性信息指数(PIC)、品种间的遗传距离(D)等。同时利用此软件进行群体间的遗传距离的估算并以除权配对法(UPGMA)进行聚类分析。用FigtreeV1.4.3(http://tree.bio.ed.ac.uk/software/figtree/)软件对输出图文件进行编辑并绘制聚类图。
多态性信息指数:
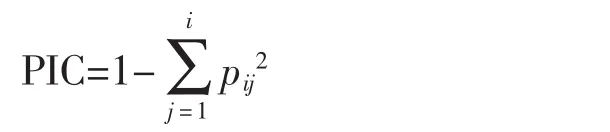
其中,pij表示第i位点第j个等位变异在全部供试材料中出现的频率。
全基因组的遗传丰富度:
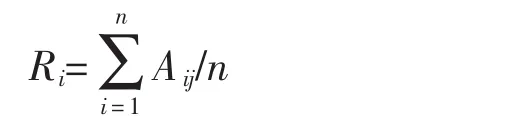
其中,Aij表示基因组第i个基因组第j个位点的等位变异数目;n表示检测位点总数。
全基因组的多样性指数:
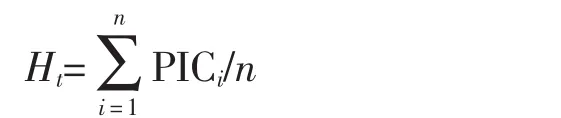
其中,PICi表示第i个位点的多态性信息指数;n表示检测到的位点总数。
年代内品种间遗传距离:
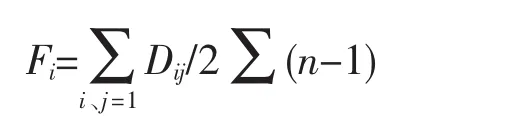
其中,Dij表示基因组第i个位点与第j个位点的遗传距离;n表示检测位点总数。
2 结果与分析
2.1 品种更替前后主栽品种遗传多样性变化
2.1.1 参试品种多样性参数分析
选用154对SSR引物对主栽品种进行检测,共获得173个位点,其中Ah1TC3B05等19对引物扩增得到2个位点。173个SSR位点共检测到872个等位变异,位点的平均等位变异数为5.04个,等位变异最小为2个,分别为seg12E03、GA79和TC3G05等32个位点。等位变异数目最多为15个,为标记seq16F10。等位基因中有200个稀有等位基因(出现频率小于0.05),占总检出等位基因数的22.94%。共检测到1108个杂合位点,占所有多态性位点(11150个)的9.94%。主栽品种遗传多态性指数的变化范围为0.014~0.881,平均0.477,多样性最高位点为TC1A08,最低位点为Ah-408。
对20条染色体的等位基因遗传丰富度和多样性指数比较,不同染色体上的SSR位点等位基因遗传丰富度差别较大,如图1所示,染色体b07最高为7.20个,其次为a10、a01和b04染色体,分别达到了7.00个、6.42个和6.00个,其余染色体都低于6个,a04、a09和b08最低均为4.00个。多样性指数分析结果表明,染色体b07最高为0.65,其次为a07为0.56,染色体b06最低0.28,染色体a04较低为0.37。结合二者进行分析,20条染色体中,b07遗传多样性最高,染色体a04较低。

图1 20条染色体的等位基因丰富度和多样性指数比较Figure 1 The comparisons of genetic richness of allele and gene diversity index in twenty chromosomes
2.1.2 4次品种更替引起的多样性变化
为探讨我国花生主栽品种遗传多样性的演变趋势,在全基因组遗传丰富度、多样性指数和年代内品种间遗传距离等方面对4个群体进行评价分析,结果如表2所示,我国花生全基因组遗传丰富度是逐渐增大的趋势,由20世纪70年代及之前的2.80增加到21世纪初期的4.06。同时其多样性指数也由20世纪70年代及以前的0.42增大到20世纪80年代的0.45,但20世纪90年代和21世纪初期均为0.46与80年代相比,差异不大。以上结果表明,近年来随着育种的发展,我国花生基因组遗传丰富度有所增加,品种遗传基础得到拓宽。但品种的遗传多样性自20世纪80年代以来,并未得到较大提高。
对4个年代主栽品种的年代内品种间遗传距离进行统计计算(见表2),不同年代间遗传距离变异范围为0.5223~0.5407。其中20世纪70年代及以前的7个主栽品种遗传距离最大为0.5407,到20世纪80年代减小为0.5284,但20世纪80年代以后,主栽品种的遗传距离接近。
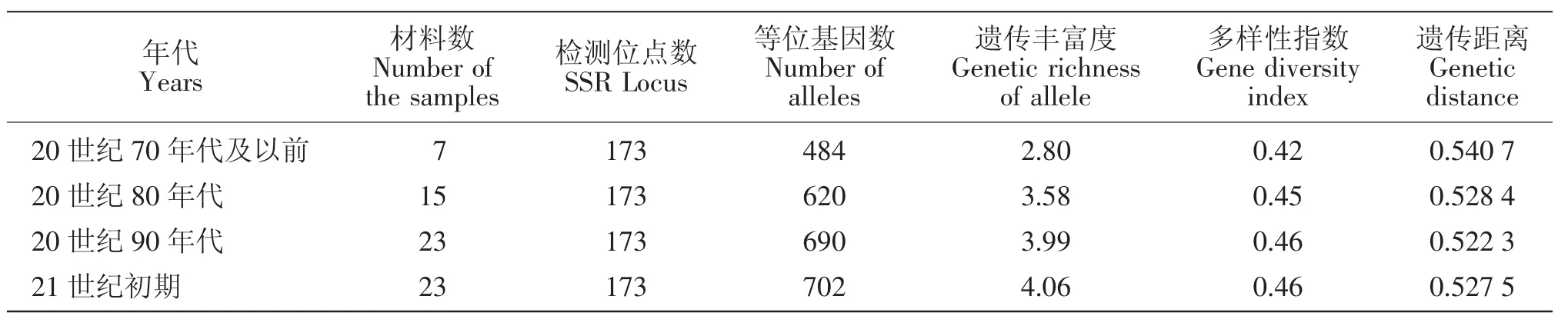
表2 不同阶段花生主栽品种遗传多样性分析Table 2 The genetic diversity of peanut elite cultivars at different period
2.1.3 不同SSR位点对品种更替响应程度分析
对173个SSR位点在不同年代花生主栽品种间等位变异的变化进行分析,结果表明,29个位点在不同年代花生主栽品种间的等位变异未发生改变;69个位点随着年代的增加等位变异逐渐增大;70个位点随着年代的增加等位变异出现先增大后减少的趋势;5个位点的等位变异随着年代的增加而减少。表3中为随着年代的增加等位变异减少的5个标记。
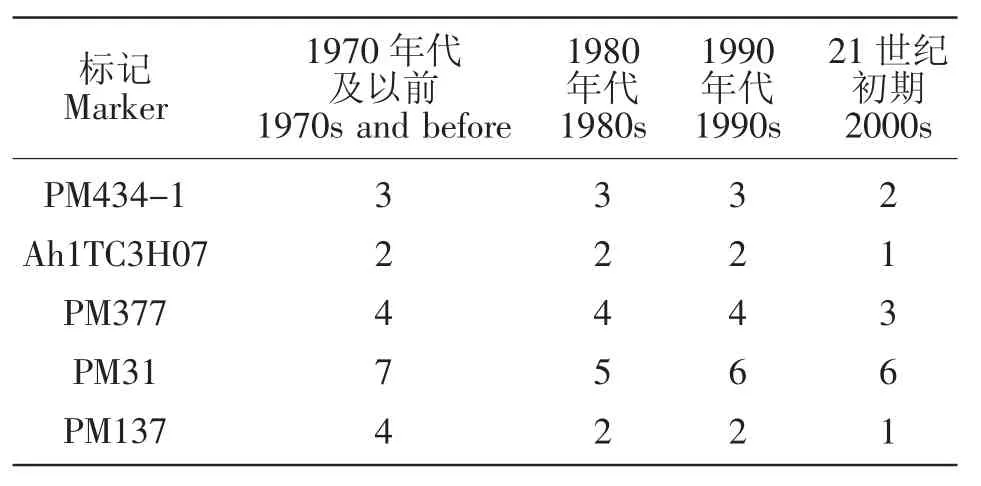
表3 SSR位点在不同年代花生品种间等位变异数目Table 3 The number of the alleles of different periods peanut elite cultivars
对173个位点的等位变异数与多态性信息指数进行相关分析,结果表明二者之间存在极显著正相关(P<0.01),但二者之间线性关系中等(相关系数R<0.8)。在等位变异相同的情况下,多态性信息指数存在较大差异,例如PM31和TC3E05检测到等位变异数目都为8个,多态性信息指数分别为0.65和0.80。图2即为2位点等位变异及其频率分布,从图中可以看出PM31等位变异分布不均匀,其中200 bp的频率远高于其他等位变异,推测此位点可能与某些重要农艺性状基因紧密连锁,由于受到较强的选择压保留下来[3,25]。TC3E05检测结果与PM31相反,等位变异分布相对均匀。

图2 PM31和TC3E05位点等位变异及其频率Figure 2 Alleles and allele frequency of the locus PM31 and TC3E05
2.2 不同年代育成品种聚类分析
68份花生材料基于Nei’s遗传距离的系统聚类分析结果示于图3,所选材料分为4个类群。第Ⅰ类群有29个材料,主要为来源于河南(7个,占24.14%)、山东(7个,占 24.14%)、河北(6个,占20.69%)。来源于20世纪70年代及以前、80年代、90年代和21世纪初期的品种分别为2、7、8和12个;第Ⅱ类群包含22个品种,主要来源于广东(8个,占36.36%)和广西(5个,占22.73%)。来源于20世纪70年代及以前、80年代、90年代和21世纪初期的品种分别为4、5、10和3个;第Ⅲ类群包含11个品种,其中河南和山东材料最多均为4个,其次为四川2个。来源于20世纪70年代及以前、80年代、90年代和21世纪初期的品种分别为1、2、3和5个;第Ⅳ类群有6个品种,其中包含山东和河南各2个。来源于80年代、90年代和21世纪初期的品种分别为1、2和3个。从每个小类群的组成可见,相同省份的材料并未完全聚类于同一小类群,但具有一定的倾向性,如河北省的品种都在第Ⅰ类群中,广东和广西品种大都也在第Ⅱ类群中。第Ⅱ类群主要为狮头企、狮头企与伏花生杂交育成品种及其衍生后代。但每个类群中均包含不同年代的品种。从结果可以看出,类群分布与亲缘关系及地域相关,与品种更替时间无关。
对来源于不同年代花生主栽品种两两间进行遗传距离的计算,表4可以看出20世纪70年代及以前与其他时期间的遗传距离较大,为0.2076~0.2386,并随着年代的增加逐渐增大。20世纪80年代主栽品种与20世纪90年代和21世纪主栽品种之间遗传距离较小在0.1641~0.1651之间。20世纪90年代和21世纪主栽品种遗传距离为0.1754。

图3 68个花生品种的SSR标记遗传聚类图Figure 3 Dendrograph of 68 peanut varieties based on SSR genetic distance

表4 不同阶段花生主栽品种间遗传距离分析Table 4 The genetic distance of peanut elite cultivars between different periods
3 讨论与结论
3.1 不同研究中花生SSR多态性的比较
近年来,SSR标记已广泛用于花生种质资源的遗传多样性检测中。姜慧芳等[16]对我国298份小核心种质进行SSR多样性分析,花生资源平均多态性条带数为4.8,遗传多样性指数为0.22。Wang M.L.等[19]对美国小核心种质112份进行SSR多样性分析,每个位点检测到的等位基因数平均为8.1个,PIC为0.53。近年来,国内外学者利用SSR标记对栽培种进行多态性检测也屡见报道,Wang H.等[26]对中国、印度和美国的79份栽培种和高代品系进行了分析,等位基因丰富度为4.25,PIC为0.43。任小平分析了我国196份花生栽培种,其等位基因丰富度为 3.0,PIC 为 0.38[1]。K.M.Pandey等[27]对 20 个花生亲本进行遗传多样性分析,其等位基因丰富度为3.2,PIC为0.31。本研究对我国68份花生主栽品种进行遗传多样性分析,等位基因丰富度为5.04,PIC为0.48。不同的研究得到的结果不同,主要是由于以下几个方面的原因造成的:一是供试材料的选择,本研究中不同年代主栽品种的等位基因丰富度和遗传多样性指数存在很大差异,花生中也已经被证明地方种的多样性比育成品种丰富[12]。因此,可通过在杂交育种过程中筛选鉴定更多优异地方种作亲本,进而拓宽我国花生品种的遗传基础;二是SSR标记的选择,选择扩增产物稳定、多态性高的标记可大大提高检测效率,节约检测时间和检测成本,因此,可优先选择他人文章中发表的多态性标记进行扩增。本研究中选用的标记主要来源于文献[5,20-22],都是遗传图谱中两亲本间分离的标记;三是检测方法的选择,常规的SSR银染技术在大规模、多批次的数据收集和分析时仍存在相当大的难度,如不同等位变异的准确识别困难及不同批次反应数据难以统一。SSR荧光标记采用了荧光标记技术,利用ABI3700 DNA分析仪进行扩增产物的检测,采用Genemapper3.2软件进行检测数据的采集,实现了数据收集和处理的自动化,明显降低实验结果的判读错误,提高数据分析的可靠性和准确度,并且经分析荧光标记技术较银染方法在每个位点上多检测到3个等位变异,具有更高的灵敏性,更适于进行遗传多样性分析和研究[8]。因此,本研究中利用荧光标记技术对材料进行检测得到较好的多态性结果。
3.2 品种更替对花生主栽品种遗传多样性的影响
我国花生品种改良起步较晚,中华人民共和国成立后生产上利用的大都为农家品种,20世纪50年代鉴定的农家品种伏花生和狮头企大面积推广利用,实现了我国花生品种的第一次更替。后来随着育种工作的蓬勃开展分别于20世纪70年代、80年代、90年代和21世纪又经历了4次花生品种更替[2]。本研究中,我国花生主栽品种的等位基因丰富度随着年代的增加逐渐增加,这主要是由于我国花生主栽品种虽然大都有伏花生和狮头企的血缘,但是随着年代的增加,在杂交过程中开始利用筛选到的优良地方种及国外引进优良品种作亲本,育种方法也开始由地方种的鉴定筛选到单交、复交、诱变等多种途径,因此,检测到的等位基因数目逐渐递增。但是在20世纪80年代以后主栽品种的多样性指数未出现明显差异,遗传距离也是随着年代的增加而降低,虽在2000年以后略有增加,但较20世纪70年代及以前的品种还是降低明显。这是由于我国主栽品种虽然在杂交过程中利用了地方品种和国外引进品种,但大都还是利用育成种作亲本进行修修补补,育成的品种具有的某些特有等位基因多是稀有基因,对群体遗传结构的影响不大,因而通过SSR分析结果计算年代内品种间遗传距离时得到数值并不高。因此,基于以上结果可确认我国花生主栽品种遗传多样性自20世纪80年代以来虽全基因组遗传丰富度得到了提高,但等位基因分布均匀度尚未产生显著性改变。
3.3 SSR多态性与选择牵连效应的关系
张学勇等[25]指出,在基因组中,一些承受强选择作用的基因在群体中的多样性显著降低,同时这些基因附近区域的遗传多样性也明显下降。在遗传学中将这种对个别基因的选择导致其侧翼区域遗传多样性降低的现象称之为选择牵连效应(hitch hiking effect,也称选择搭载效应)。自然选择和人工选择使被选择位点两侧的多样性降低,在育种过程中育种家感兴趣的目标性状如产量性状、品质性状、抗病性等相关基因的选择,可能存在较强的选择牵连作用。对20条染色体进行比较,a04染色体的遗传多样性最低,推测a04染色体在育种过程中承受了较大的选择压,在其中也检测到许多于脂肪酸组成相关的QTL位点[28]。5个等位变异减少的标记中,PM13已被证明与花生油酸含量[29]和黑斑病抗性[30]相关;TC3H07[31]与花生株型有关;PM434和 PM377[32]与花生锈病抗性相关。
3.4 聚类结果与品种更替的关系
利用154对SSR引物扩增结果,将68份材料划分为4个类群,材料类群划分在与地理来源相关的同时,也可以发现与亲缘关系有关,例如:第Ⅱ类群主要为狮头企、狮头企与伏花生杂交育成品种及其衍生后代。除第四类群外,每一类群中也都包含不同年代的主栽品种。因此,聚类结果中类群的划分与地理来源和亲缘关系有关,与育成年代无关。我们要拓宽育成品种的遗传多样性,可以通过挑选不同地理来源及亲缘关系远的材料做亲本,不用考虑其育成年代。
以上结果表明,我国花生自20世纪80年代以来的主栽品种间全基因组遗传丰富度逐渐增大,但遗传多样性指数和年代内品种间的遗传距离变化不明显,可能是影响花生适应性和产量潜力增加的原因。因此,今后花生育种要在进一步引进国外优异资源、鉴定筛选优异的地方资源和挖掘野生近缘植物中的优良基因的同时,结合现在基因克隆、分子标记和转基因等工作,不断拓宽和创新花生育成品种的遗传基础,积极推动我国花生育种及花生产业的蓬勃发展。
[1]REN X P,JIANG H F,YAN Z Y,et al.Genetic diversity and population structure of the major peanut(Arachis hypogaea L.)cultivars grown in China by SSR markers[J].Plos One,2014,9(2):88091.
[2]禹山林.中国花生品种及其系谱[M].上海:上海科学技术出版社,2008:10-11.
[3]郝晨阳,王兰芬,张学勇,等.我国育成小麦品种的遗传多样性演变[J].中国科学,2005,35(5):408-415.
[4]刘传光,张桂权.用SSR标记分析1949-2005年华南地区常规籼稻主栽品种遗传多样性及变化趋势[J].作物学报,2010,36(11):1843-1852.
[5]GAUTAMI B,FONCEKA D,PANDEY M K,et al.An international reference consensus genetic map with 897 marker loci based on 11 mapping populations for tetraploid groundnut(Arachis hypogaea L.)[J].Plos One,2012,7(7):41213.
[6]PANDEY M K,WANG M L,QIAO L X,et al.Identification of QTLs associated with oil content and mapping FAD2 genes and their relative contribution to oil quality in peanut(Arachis hypogaea L.)[J].BMC Genetics,2014,15(1):1-14.
[7]王瑾,李玉荣,张嘉楠,等.花生杂交种F1真伪的SSR标记检测[C].武汉:中国油料作物学报,2013(35):171-175.
[8]郝晨阳,王兰芬,贾继增,等.SSR荧光标记和银染技术的比较分析[J].作物学报,2005,31(2):144-149.
[9]MORETZSOHN M C,HOPKINS M S,MITCHELL S E,et al.Genetic diversity of peanut(Arachis hypogaea L.)and its wild relatives based on the analysis of hypervariable regions of the genome[J].BMC Plant Biology,2004,4(1):11.
[10]BARKLEY N A,DEAN R E,PITTMAN R N,et al.Genetic diversity of cultivated and wild-type peanuts evaluated with M13-tailed SSR markers and sequencing[J].Genetics Research,2007,4(89):93-106.
[11]崔顺立,刘立峰,陈焕英,等.河北省花生地方品种基于SSR标记的遗传多样性[J].中国农业科学,2009,42(9):3346-3353.
[12]姜慧芳,任小平,陈玉宁,等.中国花生地方品种与育成品种的遗传多样性[J].西北植物学报,2011,31(8):1551-1559.
[13]HE G,MENG R,NEWMAN M,et al.Microsatellites as DNA markers in cultivated peanut(Arachis hypogaea L.)[J].BMC Plant Biology,2003,3(1):3.
[14]唐荣华,庄伟建,高国庆,等.珍珠豆型花生的简单序列重复(SSR)多态性[J].中国油料作物学报,2004,26(2):20-26.
[15]张建成,王传堂,杨新道.SSR和STS标记在花生栽培品种鉴定中的应用研究[J].植物遗传资源学报,2006,7(2):215-219.
[16]姜慧芳,任小平,张晓杰,等.中国花生小核心种质SSR遗传多样性[J].中国油料作物学报,2010,32(4):472-478.
[17]JIANG H,HUANG L,REN X,et al.Diversity characterization and association analysis of agronomic traits in a Chinese peanut(Arachis hypogaea L.)mini-core collection[J].Journal of Integrative Plant Biology,2014,56(2):159-169.
[18]任小平,张晓杰,廖伯寿,等.ICRISAT花生微核心种质资源SSR 标记遗传多样性分析[J].中国农业科学,2010,43(14):2848-2858.
[19]WANG M L,SUKUMARAN S,BARKLEY N A,et al.Population structure and marker trait association analysis of the US peanut(Arachis hypogaea L.)mini-core collection[J].Theoretical Applied Genetics,2011,123(8):1307-1317.
[20]SHIRASAWA K,BERTIOLI D J,VARSHNEY R K,et al.Integrated consensus map of cultivated peanut and wild relatives reveals structures of the A and B genomes of Arachis and divergence of the legume genomes[J].DNA Research,2013,20(2):173.
[21]HONG Y,CHEN X,LIANG X,et al.A SSR-based composite genetic linkage map for the cultivated peanut(Arachis hypogaea L.)genome[J].BMC Plant Biology,2010(10):17.
[22]HUANG L,HE H,CHEN W,et al.Quantitative trait locus analysis of agronomic and quality-related traits in cultivated peanut(Arachis hypogaea L.)[J].Theoretical Applied Genetics,2015(128):1103-1115.
[23]OETTINH W,LEE H,FLANDERS D,et al.Linkage analysis with multiplexed short tandem repeat polymorphisms using infrared fluorescence and M13 tailed primers[J].Genomics,1996,30(3):450-458.
[24]LIU K,MUSE S V.PowerMarker:an integrated analysis environment for genetic marker analysis[J].Bioinformatics,2005,21(9):2128-2129.
[25]张学勇,童依平,游光霞,等.选择牵连效应分析:发掘重要基因的新思路[J].中国农业科学,2006,39(8):1526-1535.
[26]WANG H,KHERA P,HUANG B,et al.Analysis of genetic diversity and population structure of peanut cultivars and breeding lines from China,India and the US using SSR markers[J].Journal of Integrative Plant Biology,2016,58(5):452-465.
[27]PANDEY M K,GAUTAMI B,JAYAKUMAR T,et al.Highly informative genic and genomic SSR markers to facilitate molecular breeding in cultivated groundnut(Arachis hypogaea L.)[J].Plant Breeding,2012,131(1):139-147.
[28]WANG M L,KHERA P,PANDEY M K,et al.Genetic mapping of QTLs controlling fatty acids provided insights into the genetic control of fatty acid synthesis pathway in Peanut(Arachis hypogaea L.)[J].Plos One,2015,10(4):0119454.
[29]SARVAMANGALA C,GOWDA M V C,VARSHNEY R K,et al.Identification of quantitative trait loci for protein content,oil content and oil quality for groundnut(Arachis hypogaea L.)[J].Field Crops Research,2011,122(1):49-59.
[30]GAUTAMI B,PANDEY M K,VADEZ V,et al.Quantitative trait locus analysis and construction of consensus genetic map for drought tolerance traits based on three recombinant inbred line populations in cultivated groundnut(Arachis hypogaea L.)[J].Molecular Breeding,2012:DOI 10.1007/s11032-011-9660-0.
[31]FONCEKA D,TOSSIM H A,RIVALLAN R,et al.Fostered and left behind alleles in peanut:interspecific QTL mapping reveals footprints of domestication and useful natural variation for breeding[J].BMC Plant Biology,2012,12(1):26.
[32]KHEDIKAR Y P,GOWDA M V C,SARVAMANGALA C.A QTL study on late leaf spot and rust revealed one major QTL for molecular breeding for rust resistance in groundnut(Arachis hypogaea L.)[J].Theoretical Applied Genetics,2010,121(5):971-984.
Analysis the Genetic Diversity Variation Trends of Peanut Elite Cultivars in China using SSR Markers
WANG Jin,LI YU-rong*,CHENG Zeng-shu,CHEN Si-long,SONG Ya-hui,ZHANG Peng-juan
(Institute of Cereal and Oil Crops,Hebei Academy of Agriculture and Forestry Sciences/Laboratory of Crop Genetics and Breeding of Hebei Province,Shijiazhuang 050035,China)
【Objective】The objective of this study was to detect the genetic diversity in China peanut cultivar replacement since 1950s based on SSR marker in order to provide references for peanut breeding.【Method】The genotype of 68 elite varieties was screened using 154 SSR markers.【Results】As results,173 loci and 872 alleles were detected.The mean number of allele per locus was 5.04.The polymorphism information content(PIC)value ranged from 0.014 to 0.881,with average 0.477.Among 20 chromosomes,b07 had highest PIC value while a04 had lowest one.The peanut cultivar evolution in 1980s had the most significant impact on genetic diversity.The varieties that released in this stage had more allele,higher PIC value and similar genetic distance compared with the varieties that released before 1980s.The varieties that released after 1980s has more allele number than others,but which had no significant impact on PIC value and genetic distance.Furthermore,the allele number of 5 SSR loci showed significant decrease with cultivar evolution.The varieties with related pedigree or geographic origin were assigned into same group,while cluster result had no relationship with released year of varieties.【Conclusion】Our results showed that the allele number had increased while the evenness did not change in the past 50 years.
Arachishypogaea;cultivar evolutions;elite cultivar;SSR marker;genetic diversity
S565.202
A
1000-2650(2017)03-0309-08
10.16036/j.issn.1000-2650.2017.03.004
2017-05-18
国家自然科学基金项目(31301354);河北省科技计划项目(16226301D);国家花生产业技术体系建设项目(CARS-14)。
王瑾,副研究员,博士,主要从事花生遗传育种与栽培研究,E-mail:wangjinnky@163.com。*责任作者:李玉荣,研究员,学士,主要从事花生遗传育种与栽培研究,E-mail:liyrl@163.com。
(本文审稿:任小平;责任编辑:刘诗航;英文编辑:刘诗航)
