蝴蝶兰PhalPI基因的克隆及在花器官突变体中的表达分析
2017-11-10袁秀云许申平王莹博
袁秀云 许申平 王莹博 崔 波*
(1.郑州师范学院生物工程研究所,郑州 450044; 2.河南农业大学生命科学学院,郑州 450002)
蝴蝶兰PhalPI基因的克隆及在花器官突变体中的表达分析
袁秀云1许申平1王莹博2崔 波1*
(1.郑州师范学院生物工程研究所,郑州 450044;2.河南农业大学生命科学学院,郑州 450002)
为深入研究兰科植物花器官发育的调控机理,从蝴蝶兰花瓣中克隆了一个B类MADS-box转录因子PhalPI(GenBank登录号为KY020416)。序列分析表明,该基因的cDNA全长为944 bp,含完整的开放阅读框,可编码210个氨基酸,属于BGLO/PI蛋白家族,与蝴蝶兰属的PhPI10和PeMADS6基因关系最近;表达模式分析表明,PhalPI基因在生殖器官中表达,在营养器官中不表达,在授粉后的子房中,该基因的表达水平降低。在5种花器官突变体中,PhalPI基因在萼片唇瓣化突变体的萼片和蕊柱中表达水平明显升高;在雄蕊花瓣化突变体的萼片和侧瓣中表达水平降低,在其唇瓣和蕊柱中显著升高;在侧瓣合柱化突变体的蕊柱中,PhalPI基因的表达也发生了显著升高;PhalPI基因表达的改变与花器官形态的突变相关;而在侧瓣唇瓣化和侧瓣花药化突变体中,PhalPI基因的表达水平没有变化。推测该基因在决定蝴蝶兰侧瓣和唇瓣的发育中起重要的调控作用。
蝴蝶兰;GLO/PI;MADS-box;花器官突变体
花器官的发育是高等植物发育过程中特别重要的过程,也是人们一直研究的热点。根据对双子叶模式植物如拟南芥、矮牵牛和金鱼草的研究,花器官发育的分子调控从最初的“ABC模型”理论逐步发展到了“ABCDE模型”理论[1~2]。由于调控花器官发育的同源基因随着植物的系统演化,其结构和功能产生了一系列复杂的亚功能和新功能,进而影响了花器官的发育,赋予花器官形态的多样性[3~4],因此花器官发育的调控理论既有普遍性又有特殊性。单子叶植物花的发育不同于双子叶植物,一些单子叶植物花的发育并不能用“ABCDE模型”解释[5]。广泛开展对单子叶植物花器官性状发育的分子调控机制研究,对于揭示高等植物花器官多样性的演化及植物的系统发育具有重要意义。
兰科植物作为单子叶植物中较为进化的类群,其花器官有独有的特征,既有典型的花被轮状排列,花萼和花瓣呈2轮,又有独特的形态和结构:其中萼片的大小和颜色与花瓣相似;1枚花瓣特化为唇瓣,具有多样的形状;子房下位,呈明显的180°的扭转;雌蕊和雄蕊合生为合蕊柱;花药团块状等,这些性状被认为是植物演化过程中进化的表现。通过对兰花MADS-box转录因子的研究,兰科植物也具有A、B、C、D和E类基因[6],这些基因在蝴蝶兰花器官中具有特殊的表达模式[7],其中B类基因在其花器官发育中起着重要作用[8~9]。B类基因有DEFICIENS(DEF)/APETALA3(AP3)-like家族和GLOBOSA(GLO)/PISTILLATA(PI)-like 2个家族,这些基因只在生殖器官中表达,在营养器官中不表达[6,10];兰科植物的DEF/AP3-like有4个分支的同源基因,GLO/PI-like有2个分支的同源基因,这些分支基因的不同组合及差异表达决定了多样的花被形态[12],如PI和AP3B分支决定花萼的发育,PI、AP3A1和AP3B分支决定花瓣侧瓣的发育,PI和AP3A分支决定唇瓣的发育[11]。因此,兰花花器官发育的分子调控有“兰花密码”(Orchid Code)模型及修正的“Orchid Code”模型[12~13],这些理论的提出表明,兰科植物花器官发育的调控具有更加复杂的机制,而这些发现无疑是对传统的“ABC模型”的有益补充和完善。
本研究以蝴蝶兰花瓣为材料,采用RT-PCR和RACE技术克隆了一个B类基因PhalPI,利用实时荧光定量PCR方法分析了该基因在在“萼片唇瓣化”、“侧瓣唇化”、“雄蕊瓣化”、“侧瓣合柱化”、“侧瓣花药化”5类变异花器官和正常花器官中的表达,旨在详细分析B类基因在花器官决定中的调控作用,为丰富和完善高等植物的开花调控理论提供依据。
1 材料与方法
1.1 试验材料
以蝴蝶兰品种‘内山姑娘’及其雄蕊瓣化变异株、‘婚宴’及其侧瓣合柱化变异株、‘S1024’及其萼片唇瓣化变异株、‘火鸟’及其侧瓣唇瓣化变异株、‘富乐夕阳’及其侧瓣花药化变异株均来自郑州师范学院兰花工程中心智能温室,突变株由大量无性繁殖的种苗中获得(图1)。分别取正常株和变异株花器官的萼片、侧瓣、唇瓣、蕊柱,另取‘富乐夕阳’植株营养期、抽葶期、开花期和授粉后(10 d)等不同发育时期的叶片、根和花葶,每个样品取3个重复,取样后迅速用液氮速冻,于-80℃冰箱备用。
1.2 方法
1.2.1 RNA的提取
组织总RNA用RNAprep Pure Plant Kit(天根)提取,具体方法参照说明书进行。提取的RNA分别通过琼脂糖凝胶电泳和核酸蛋白分析仪判断其完整性、浓度和纯度。
1.2.2 蝴蝶兰PhalPI基因全长的克隆及序列分析
以蝴蝶兰品种‘富乐夕阳’花瓣的总RNA为模板,利用M-MLV反转录酶(Takara)将其反转录为cDNA,根据NCBI登录的B类MADS-box基因序列设计简并引物PI-F和PI-R(表1),用于扩增B类MADS基因的中间保守片段,PCR扩增体系为20 μL,含10×buffer 2 μL,4种dNTP各150 μmol·L-1,每条引物0.5 μmol·L-1,cDNA模板1 000~2 000 ng,TaqDNA聚合酶1 U。PCR扩增程序为:95℃变性3 min;95℃ 35 s,58℃ 35 s,72℃ 35 s,35个循环;72℃延伸10 min。回收目的片段,连接到pMD19-T载体上,转入大肠杆菌DH5α菌株,进行克隆和测序。根据得到的中间片段序列,设计1对3′端特异引物GSP3-1和GSP3-2,1对5′端特异引物GPS5-1和GPS5-2(表1),利用SMARTTM RACE Amplification kit(Clontech)进行扩增,回收扩增产物,克隆到pMD19-T载体上,转入大肠杆菌DH5α菌株,鉴定后进行测序。将5′和3′RACE得到的序列与中间片段进行拼接,并根据拼接的序列设计1对引物PI-ORFF和PI-ORRR,进行开放阅读框(ORF)的扩增和测序验证。引物合成和测序由上海立菲生物技术有限公司完成。基因序列的同源性分析在NCBI(http://blast.ncbi.nlm.nih.gov/Blast)上进行,蛋白结构域在NCBI的蛋白保守区数据库(https://www.ncbi.nlm.nih.gov/cdd)中搜索,使用MEGA软件NJ法构建系统发育树。
1.2.3 蝴蝶兰PhalPI基因的表达分析
组织材料总RNA用PrimeScriptRT reagent Kit with gDNA Eraser(TaKaRa)反转录成cDNA。根据基因的全长设计1对特异引物qPI-F和qPI-R,产物长度211 bp,以Actin(JN185655)为内参基因,引物为Act-F和Act-R(表1)。采用SYBR Premix Ex TaqTMⅡ kit(TaKaRa)进行实时荧光定量PCR反应。反应体系为25 μL,反应条件为:95℃ 1 min,接着95℃ 15 s,58℃ 15 s,72℃ 15 s(40个循环)。反应在荧光定量PCR仪(Eppendorf)上进行,3次重复。引物特异性分别通过溶解曲线分析并测序验证,相对表达量按照2-ΔΔct法计算,PhalPI基因在突变中的表达差异通过SPSS 17.0软件进行分析。

表1 引物序列
2 结果与分析
2.1 PhalPI基因全长的克隆
不同组织材料总RNA提取通过凝胶电泳如图28S和18S区分明显,28S亮度大约是18S的2倍(图2)。核酸蛋白分析仪测定显示,这些RNA的OD260/OD280的值在1.87~2.06,浓度在180 ng·μL-1以上,说明所提RNA比较完整,浓度及纯度可以满足后续的实验和分析。
图2 部分材料的总RNA提取Fig.2 Total RNA extraction of several materials
以蝴蝶兰花瓣的cDNA为模板,以简并引物PI-F和PI-R进行保守片段的PCR扩增,得到1个398 bp保守片段(图3A),此片段经Blastn分析,该片段为MADS-box蛋白基因。根据该片段序列,采用RACE技术,利用GSP3-1和GSP3-2引物扩增获得3′端目的片段为339 bp(图3B),利用GSP5-1和GSP5-2引物扩增获得5′端目的片段为425 bp(图3C)。将保守片段、3′和5′端序列拼接,在开放阅读框区域设计引物,进行扩增验证,得到了预期633 bp的片段(图3D),最终拼接得到944 bp的基因cDNA全长。该具有完整的开放阅读框。其中包含95 bp的5′-UTR、633 bp开放阅读框和215 bp的3′-UTR,编码210个氨基酸。将其命名为PhalPI,提交GenBank,登录号为KY020416。
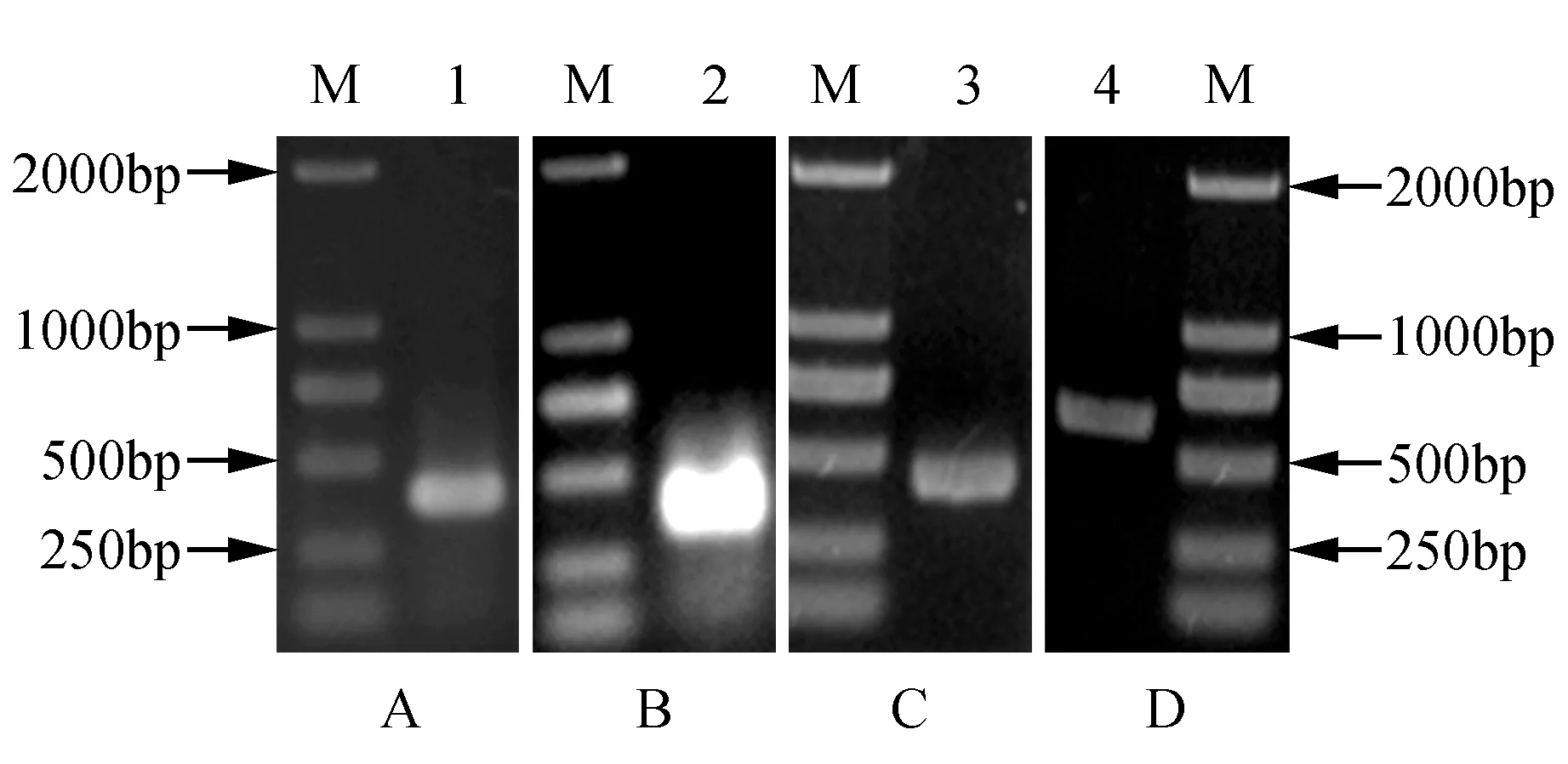
图3 PhalPI基因的克隆 A.保守片段扩增;B. 3′RACE;C. 5′RACE;D. ORF片段扩增 1.保守片段;2. 3′端片段;3. 5′端片段;4. ORF片段;M. DL2000 markerFig.3 The cloning of PhalPI A.Amplification of conservative fragment; B. 3′RACE; C. 5′RACE; D. ORF fragment 1.Conservative fragment; 2. 3′RACE; 3. 5′RACE; 4. ORF fragment; M. DL2000 marker
2.2PhalPI基因编码氨基酸序列的比对及其进化树分析
PhalPI基因编码的氨基酸序列通过Blastp分析,发现与其它植物的PI-like蛋白具有75%以上的一致性,其中与小兰屿蝴蝶兰(Phalaenopsisequestris)的PeMADS6(AAV83997)和蝴蝶兰杂交种(Phalaenopsishybrida)的PhPI10(AAV28490)有98%的一致性。该蛋白包含一个MADS结构域,一个K-box区和一个PI基序(图4),说明该基因是B类转录因子PI基因。在NCBI的蛋白保守区数据库中的搜索显示,该蛋白具有MADS-MEF2-like和K-box结构域,在蛋白的N-端具有DNA结合位点、假定的磷酸化位点和二聚化结合界面等结构,属于MADS和K-box超家族成员(图5)。

图4 PhalPI基因的氨基酸序列与同源序列的比对 方框区域分别代表MADS区、K区和PI结构基序。Fig.4 Aligment of PhalPI amino sequence with other Homologous proteins The boxes are MADS domain,K region and PI motif respectively

图5 PhalPI基因编码蛋白的结构域Fig.5 Domain of the PhalPI protein
为更好地了解蝴蝶兰该PhalPI蛋白与其它MADS-box基因之间的进化关系,选取与PhalPI蛋白一致性高的PI-like蛋白和有代表性的MADS-box蛋白构建进化树。从图6可以看出,所分析的MADS-box蛋白中,有2个大的分支,B类为一个分支,A、C、D和E类为另一个分支,B类中又有GLO/PI和DEF/AP3 2个分支,其中拟南芥(Arabidopsisthaliana)的AP3、金鱼草(Antirrhinummajus)的DEFA、小兰屿蝴蝶兰的PeMADS2、PeMADS3、PeMADS4和PeMADS5为DEF/AP3类,而红门兰(Orchisitalica)的OrcPI2、球茎石槲(Dendrobiumthyrsiflorum)的DthyrPI、文心兰(Oncidiumhybrida)的OncPI、小兰屿蝴蝶兰的PeMADS6、蝴蝶兰品种的PhPI10、PhalPI、番红花(Crocussativus)的CsatPIC1、水稻(Oryzasativa)的MADS4、金鱼草的GLO和拟南芥的PISTILLATA(PI)为GLO/PI类。可以看出在每个分支中单子叶植物和双子叶植物的B类MADS-box蛋白区分明显;本研究中的PhalPI蛋白属于GLO/PI类,与兰科植物的GLO/PI类蛋白在一个分支上,并且与蝴蝶兰品种的PhPI10和小兰屿蝴蝶兰的PeMADS6关系最近(图6)。

图6 PhalPI蛋白的系统进化树分析Fig.6 Phylogenetic tree of PhalPI protein with known B class MADS-box protein
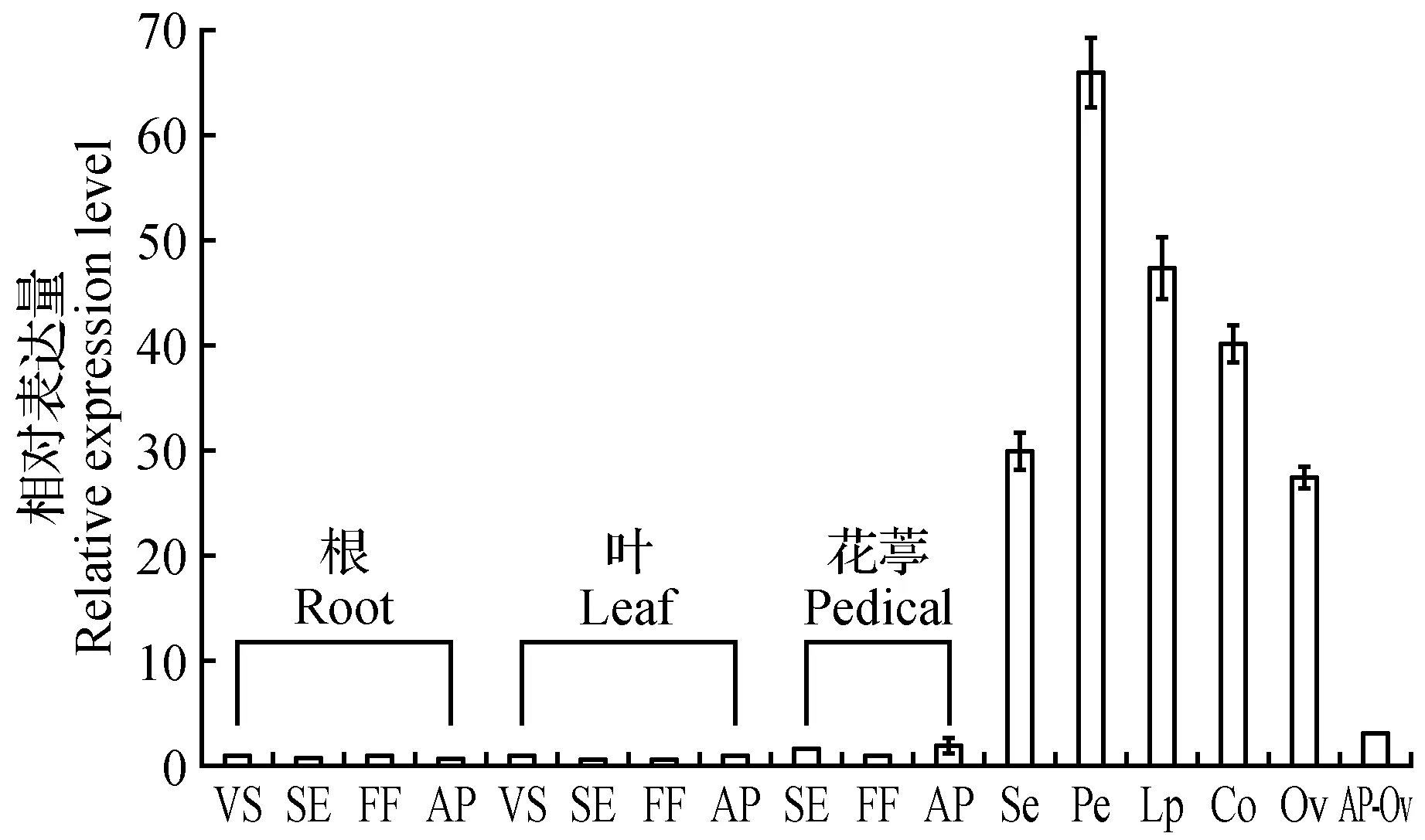
图7 PhalPI基因在蝴蝶兰发育过程中不同组织中的表达 VS.营养期;SE.抽葶期;FF.盛花期;AP.授粉后;Se.萼片;Pe.侧瓣;Lp.唇瓣;Co.蕊柱;Ov.子房;AP-Ov.授粉后子房Fig.7 The expression of PhalPI in different tissues during development VS. Vegetative stage; SE. Spike emergence; FF. Full flowering; AP. After pollination; Se. Sepal; Pe. Petal; Lp. Lip; Co. Column; Ov. Ovary; AP-Ov. Ovary of after pollination
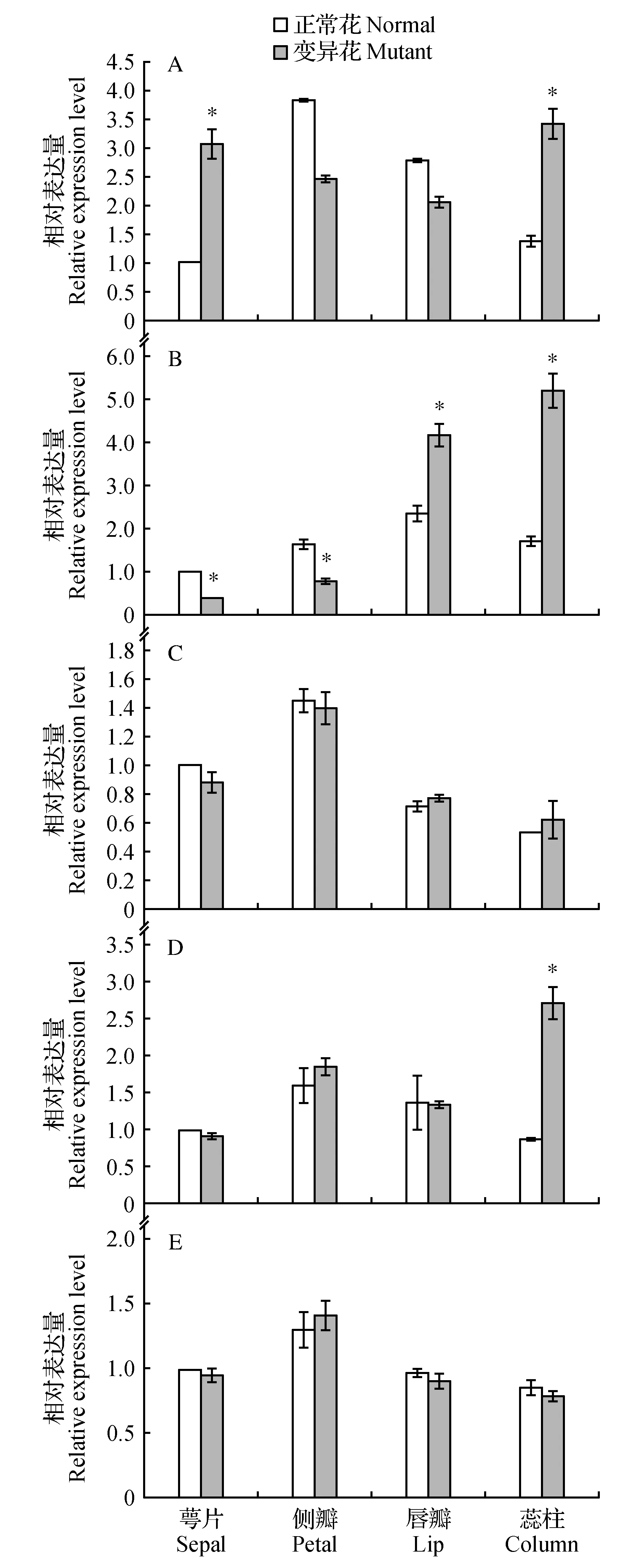
图8 PhPI-like基因在蝴蝶兰花器官突变体中的表达A.‘S1024’;B.‘内山姑娘’;C.‘火鸟’;D.‘婚宴’;E.‘富乐夕阳’ *表示差异显著Fig.8 The expression of PhPI-like in floral organ mutants of PhalaenopsisA. ‘S1024’; B. ‘Nei Shan Gu Niang’; C.‘Huo Niao’; D.‘Hun Yan’; E.‘Fu Le Xi Yang’ * indicate significant difference
2.3 PhalPI基因的表达分析
为了研究PhalPI基因在蝴蝶兰器官建成中的功能,分析了其在发育过程中不同组织及5种变异花器官中的表达特性。结果表明,PhalPI基因主要生殖器官中表达,在营养器官几乎不表达;在花瓣、唇瓣和蕊柱中表达较高,而在花萼和子房中表达较低;授粉后子房中的表达量降低;在花葶中有微量表达(图7)。
5种变异花器官包括:‘S1024’下面两侧的萼片明显变异为唇瓣;‘内山姑娘’雄蕊瓣化的变异,其与正常花主要的区别是蕊柱两侧的雄蕊变异为花瓣状;‘火鸟’的侧瓣完全变异为唇瓣;‘婚宴’花器官的变异除了花明显变小、萼片和花瓣较小外,主要是侧瓣退化并与蕊柱合生;‘富乐夕阳’的花除了两侧萼片颜色有变化,主要的变异为侧瓣变得狭窄,顶端具有花药(图1)。PhalPI基因在5种花器官突变体中的表达分析表明,在萼片唇瓣化突变体中,PhalPI基因在侧瓣和唇瓣中的表达水平与正常花相比无明显改变,而在侧萼和蕊柱的表达水平明显升高(P<0.05)(图8A);在雄蕊瓣化的突变体中,PhalPI基因在萼片和侧瓣中的表达水平明显下降,而在唇瓣和蕊柱中的表达水平显著升高(P<0.01)(图8B);在侧瓣唇瓣化和侧瓣花药化突变体中,PhalPI基因在各轮花器官中的表达量没有显著改变(图8C,E);在侧瓣合柱化突变体中,PhalPI基因在萼片、侧瓣和唇瓣中的表达水平与正常花相比没有显著差异,而在蕊柱中显著升高(P<0.01)(图8D)。这些结果表明,在侧萼唇瓣化、雄蕊花瓣化和侧瓣合柱化突变体中,PhalPI基因的表达发生了显著变化,推测PhalPI基因在决定侧瓣和唇瓣的发育中起重要作用。
3 讨论
B类基因是种子植物中一个古老的MADS-box家族基因[14],在拟南芥、金鱼草等模式植物中,AP3和PI基因具有典型的决定花瓣和雄蕊的发育的功能[15],是典型的“ABC模型”调控模式,其同源基因的表达在基部被子植物中扩展到花被、雄蕊和心皮[16],一些单子叶植物如百合和郁金香的B类基因也在花器官的外面3轮表达,这些表达模式支持“修正的ABC模型”[5],然而随着植物的进化,发生了许多基因复制事件,再经过基因的选择和纯化使其典型的功能得以演化[17~19]。因此,B基因对花器官发育的调控功能发生了微妙的变化。既是在同一种植物的不同花器官中表达特性也不相同。例如,番木瓜(Caricapapaya)的CpPI基因在营养器官中不表达,在花器官中表达,这是该类基因的原有的表达特点,但在两性花和雌花的花萼和花瓣中表达,雄蕊和心皮中不表达,在雄花的花瓣和花药中表达,萼片和心皮中不表达,这是该类基因功能演化的结果[20]。在单子叶植物中,B类基因的表达更为复杂,有的B类基因在花器官第1轮不表达,而有的B类基因的转录调控拓展到第4轮[5]。
在兰科植物,其花型、花色多样,体现了花器官的高度进化,B类基因的复制和转录差异作为一种积极的演化策略在花器官发育调控中扮演着重要角色[21~23],B类基因不同的表达模式,在兰花的形态决定中发挥独特的调控作用[24~25]。蝴蝶兰B类基因(PeMADS2-PeMADS6)及与其它转录因子如AP1/AGL9和AGL6等多样的互作复合体使调控模式更加复杂化[26],而E类基因(PeSEPs)也与B、C和D类基因构成更为复杂的蛋白复合体参与花器官的建成[27],因此提出了“同源性兰花花被”(Homeotic Orchid Tepal)模型,这个模型认为,所有B类MADS-box基因成员在兰花花被早期花组织原基阶段出现,而与其它MADS-box基因组成复合体在晚期的花原基和花芽中具有不同的表达特性,决定了花器官各轮组织的发生[11]。
研究表明蝴蝶兰的PeMADS6是一个GLO/PI基因,在生殖器官中表达,与兰花的超长花期和子房发育有关[28]。蝴蝶兰的PhPI15只在花器官中表达,在营养器官中不表达[29],PhPI10的表达仅限于唇瓣[15]。红门兰中有2个PI基因OrcPI和OrcPI2,在花器官中OrcPI2的表达水平明显高于OrcPI1,二者在未成熟花和成熟花中表达模式不同,但具有相似的表达趋势,即在未成熟花中,二者都在花萼、侧瓣和唇瓣中表达,侧瓣中的表达量最高,在蕊柱中有微量表达;而在成熟花中只在唇瓣中的表达量最高,在萼片和侧瓣中表达水平较低,在蕊柱中几乎不表达;在子房发育过程中,未成熟花的子房中OrcPI和OrcPI2的表达均较低,成熟花未授粉的子房中表达水平较高,而在授粉后的子房中表达水平明显降低,在子房中的表达模式与PeMADS6相似[30]。文心兰一个PI基因OMADS8在营养器官和花器官中都表达[24],OMADS8可与一个AP3基因OMADS3构成异源二聚体参与文心兰萼片、侧瓣和唇瓣的发育[32]。对木石斛的花器官决定基因的研究发现,木石斛和拟南芥的B和C类基因有一定的保守型,其PI基因DcOPI与AP3类基因构成二聚体,导致兰花器官的个体发生[32]。
本研究获得的蝴蝶兰PhalPI基因与PhPI10和PeMADS6关系最近,其在蝴蝶兰发育过程中不同组织中的表达模式与PeMADS6相似[28],即PhalPI主要在生殖器官中表达,在营养器官中几乎不表达;在授粉后的子房中表达量降低,但是又与PhPI10在唇瓣中表达不同[10]。在花器官突变体中的表达分析表明,当萼片演化为唇瓣时,该基因在萼片中和蕊柱中的表达升高,萼片中PhalPI基因表达水平的升高与萼片唇瓣化直接相关,而蕊柱中PhalPI基因表达水平的升高与形态无相关性,说明该基因参与唇瓣的发育调控;当雄蕊演化为花瓣时,唇瓣和蕊柱中的PhalPI表达水平显著升高,说明该基因在决定侧瓣和唇瓣的发育调控中起重要作用;在侧瓣退化与蕊柱合生的突变体花器官中,蕊柱中的表达水平也升高,进一步说明PhalPI基因参与侧瓣的发育调控,因此推测该基因主要参与花器官第2轮(侧瓣和唇瓣)的发育调控;而当侧瓣演化为唇瓣和侧瓣顶端长出花药时,该基因的表达无明显差异。说明这两种突变与PhalPI基因的表达无关,推测可能与其它基因的参与相关。据报道,蝴蝶兰的一个C类(PeMADS1)和D类(PeMADS7)基因在E类基因的作用下构成同源或异源二聚体,调控了侧瓣雄化变异的发育[33]。蝴蝶兰花器官突变体的转录组分析发现,蝴蝶兰的PhAGL6a、PhAGL6b、PhMADS1、PhMADS4、PhMADS5在唇瓣化的侧瓣中表达上调,PhAGL6a、PhAGL6b和PhMADS4基因在唇瓣的发育起决定性作用[34]。这些结果表明,兰花花器官的形态建成具有多个基因复杂的调控机制,深入发掘其调控基因及其分子机制,一方面能进一步补充和完善高等植物花器官的发育调控理论,另一方面为兰花花器官形态多样性的遗传改良提供依据。
1.Litt A,Kramer E M.The ABC model and the diversification of floral organ identity[J].Seminars in Cell & Developmental Biology,2010,21(1):129-137.
2.Kater M M,Dreni L,Colombo L.Functional conservation of MADS-box factors controlling floral organ identity in rice andArabidopsis[J].Journal of Experimental Botany,2006,57(13):3433-3444.
3.Airoldi C A,Davies B.Gene duplication and the evolution of plant MADS-box transcription factors[J].Journal of Genetics and Genomics,2012,39(4):157-165.
4.Soltis P S,Soltis D E.Flower diversity and angiosperm diversification[M].//Riechmann J L,Wellmer F.Flower development.New York:Springer,2014,1110:85-102.
5.Kanno A,Nakada M,Akita Y,et al.Class B gene expression and the modified ABC model in nongrass monocots[J].The Scientific World Journal,2007,7:268-279.
6.Tsai W C,Chen H H.The orchid MADS-box genes controlling floral morphogenesis[J].The Scientific World Journal,2006,6:1933-1944.
7.Su C L,Chen W C,Lee A Y,et al.A modified ABCDE model of flowering in orchids based on gene expression profiling studies of the moth orchidPhalaenopsisaphrodite[J].PLoS One,2013,8(11):e80462.
8.Zahn L M,Leebens-mack J,Depamphilis C W,et al.To B or Not to B a flower:the role ofDEFICIENSandGLOBOSAorthologs in the evolution of the angiosperms[J].Journal of Heredity,2005,96(3):225-240.
9.Mondragón-palomino M,Theiβen G.Why are orchid flowers so diverse? Reduction of evolutionary constraints by paralogues of class B floral homeotic genes[J].Annals of Botany,2009,104(3):583-594.
10.Guo B,Zhang T,Shi J L,et al.Cloning and characterization of a novelPI-likeMADS-box gene inPhalaenopsisorchid[J].DNA Sequence,2008,19(3):332-339.
11.Pan Z J,Cheng C C,Tsai W C,et al.The duplicated B-class MADS-box genes display dualistic characters in orchid floral organ identity and growth[J].Plant and Cell Physiology,2011,52(9):1515-1531.
12.Mondragón-palomino M,Theiβen G.MADS about the evolution of orchid flowers[J].Trends in Plant Science,2008,13(2):51-59.
13.Mondragón-palomino M,Theiβen G.Conserved differential expression of paralogous DEFICIENS- and GLOBOSA-like MADS-box genes in the flowers ofOrchidaceae:refining the ‘orchid code’[J].The Plant Journal,2011,66(6):1008-1019.
14.Becker A,Winter K U,Meyer B,et al.MADS-Box gene diversity in seed plants 300 million years ago[J].Molecular Biology and Evolution,2000,17(10):1425-1434.
15.Vandenbussche M,Zethof J,Royaert S,et al.The duplicated B-class heterodimer model:whorl-specific effects and complex genetic interactions inPetuniahybridaflower development[J].The Plant Cell,2004,16(3):741-754.
16.Soltis D E,Ma H,Frohlich M W,et al.The floral genome:an evolutionary history of gene duplication and shifting patterns of gene expression[J].Trends in Plant Science,2007,12(8):358-367.
17.Irish V F.Evolution of petal identity[J].Journal of Experimental Botany,2009,60(9):2517-2527.
18.Hernández-hernández T,Martínez-castilla L P,Alvarez-buylla E R.Functional diversification of B MADS-box homeotic regulators of flower development:adaptive evolution in protein-protein interaction domains after major gene duplication events[J].Molecular Biology and Evolution,2007,24(2):465-481.
19.Panchy N,Lehti-shiu M,Shiu S H.Evolution of gene duplication in plants[J].Plant Physiology,2016,171(4):2294-2316.
20.Ackerman C M,Yu Q Y,Kim S,et al.B-class MADS-box genes in trioecious papaya:twopaleoAP3 paralogs,CpTM6-1 andCpTM6-2,and aPIorthologCpPI[J].Planta,2008,227(4):741-753.
21.Mondragón-palomino M,Hiese L,Härter A,et al.Positive selection and ancient duplications in the evolution of class B floral homeotic genes of orchids and grasses[J].BMC Evolutionary Biology,2009,9:81.
22.Cantone C,Gaudio L,Aceto S.ThePI/GLO-like locus in orchids:duplication and purifying selection at synonymous sites withinOrchidinae(Orchidaceae)[J].Gene,2011,481(1):48-55.
23.Melzer R,Härter A,Rümpler F,et al.DEF- and GLO-like proteins may have lost most of their interaction partners during angiosperm evolution[J].Annals of Botany,2014,114(7):1431-1443.
24.Chang Y Y,Kao N H,Li J Y,et al.Characterization of the possible roles for B class MADS box genes in regulation of perianth formation in orchid[J].Plant Physiology,2010,152(2):837-853.
25.Tsai W C,Kuoh C S,Chuang M H,et al.FourDEF-likeMADS box genes displayed distinct floral morphogenetic roles inPhalaenopsisorchid[J].Plant and Cell Physiology,2004,45(7):831-844.
26.Tsai W C,Pan Z J,Hsiao Y Y,et al.Interactions of B-class complex proteins involved in tepal development inPhalaenopsisorchid[J].Plant and Cell Physiology,2008,49(5):814-824.
27.Pan Z J,Chen Y Y,Du J S,et al.Flower development ofPhalaenopsisorchid involves functionally divergentSEPALLATA-like genes[J].New Phytologist,2014,202(3):1024-1042.
28.Tsai W C,Lee P F,Chen H I,et al.PeMADS6,aGLOBOSA/PISTILLATA-likegene inPhalaenopsisequestrisinvolved in petaloid formation,and correlated with flower longevity and ovary development[J].Plant and Cell Physiology,2005,46(7):1125-1139.
29.Guo B,Hexige S,Zhang T,et al.Cloning and characterization of aPI-likeMADS-box gene inPhalaenopsisorchid[J].Journal of Biochemistry and Molecular Biology,2007,40(6):845-852.
30.Salemme M,Sica M,Gaudio L,et al.Expression pattern of two paralogs of thePI/GLO-likelocus duringOrchisitalica(Orchidaceae,Orchidinae) flower development[J].Development Genes and Evolution,2011,221(4):241-246.
31.Mao W T,Hsu H F,Hsu W H,et al.The C-terminal sequence and PI motif of the Orchid(OncidiumGower Ramsey)PISTILLATA(PI) ortholog determine its ability to bindAP3 orthologs and enter the nucleus to regulate downstream genes controlling petal and stamen formation[J].Plant and Cell Physiology,2015,56(11):2079-2099.
32.Xu Y F,Teo L L,Zhou J,et al.Floral organ identity genes in the orchidDendrobiumcrumenatum[J].The Plant Journal,2006,46(1):54-68.
33.Chen Y Y,Lee P F,Hsiao Y Y,et al.C- and D-class MADS-box genes fromPhalaenopsisequestris(Orchidaceae) display functions in gynostemium and ovule development[J].Plant and Cell Physiology,2012,53(6):1053-1067.
34.Huang J Z,Lin C P,Cheng T C,et al.Adenovofloral transcriptome reveals clues intoPhalaenopsisorchid flower development[J].PLoS One,2015,10(5):e0123474.
Science and technology research projects of Henan Province(162102110073)
introduction:YUAN Xiu-Yun(1970—),female,Dr.,professor,Mainly engaged in the study of plant biotechnology.
date:2016-11-08
CloningofPhalPIGenefromPhalaenopsisandItsExpressioninFloralOrganMutants
YUAN Xiu-Yun1XU Shen-Ping1WANG Ying-Bo2CUI Bo1*
(1.Institute of Bioengineering,Zhengzhou Normal University,Zhengzhou 450044;2.College of Life Science,Henan Agricultural University,Zhengzhou 450002)
For the regulatory mechanism of floral organ development in orchid,PhalPIgene(GenBank accession number: KY020416) was cloned fromPhalaenopsispetals. By the sequence analysis, the cDNA full-length of this gene was 944 bp encoding 210 amino acid residues. The gene belongs to the GLO/PI group of the B class MADS-box transcription factor, which was highly identified with PhPI10 and PeMADS6 fromPhalaenopsis. ThePhalPIwas expressed in reproductive organs and not in vegetative organs, the expression level was reduced in ovary after pollination. In 5 floral organ mutants, the expression level of thePhalPIgene was higher significantly in sepal and column of lip-like sepal mutants than that of the normal. The expression of this gene was decreased in sepals and petals in petal-like stamens mutant, and it was increased significantly in lip and column in this mutant. In column of Lateral petals pillared mutants, the expression ofPhalPIgene was also increased significantly. The changes of expression level of thePhalPIgene in 3 floral organ mutants were associated with morphological mutations of floral organ. However, the expression levels ofPhalPIgene were not changed in lip-like Lateral petals and anther-like Lateral petals mutants. Therefore, thePhalPIgene plays a crucial role in regulating Lateral petal and lip development inPhalaenopsis.
Phalaenopsis;GLO/PI;MADS-box;floral organ mutants
河南省科技攻关项目(162102110073)
袁秀云(1970—),女,博士,教授,主要从事植物生物技术方面的研究。
* 通信作者:E-mail:laocuibo@163.com
2016-11-08
* Corresponding author:E-mail:laocuibo@163.com
S682.31
A
10.7525/j.issn.1673-5102.2017.03.012
